不同成熟度无花果比较转录组分析
刘姚 李彭生 张金云 阳桂芳 张方秋 杨光

摘 要:【目的】为揭示无花果果实成熟的分子机制及无花果果实品质改良和新品种选育提供参考。【方法】以不同成熟度(S1: 八成熟,S2: 九成熟,S3: 全熟)的‘芭劳奈无花果果实为材料,利用Illumina 高通量测序技术,进行转录组测序和数据de novo 组装,对不同成熟度果实的Unigene 进行比较,筛选差异表达基因,并对差异表达基因进行转录因子分析、GO 富集分析和KEGG 富集分析。【结果】通过测序共获得65 676 条Unigene序列,筛选出差异表达基因15 620 个,其中八成熟与九成熟果实有1 508 个差异表达基因,九成熟与全熟果实有11 385 个差异表达基因。GO 富集分析结果表明:八成熟与九成熟果实的差异表达基因主要富集在核糖体相关的细胞组分以及糖酵解、ADP 和ATP 代谢等过程;九成熟与全熟果实的差异表达基因主要富集在植物细胞壁相关的细胞组分以及果实成熟、发病原和谷氨酸代谢等过程。KEGG 分析结果表明:八成熟与九成熟果实的差异表达基因主要富集在糖酵解、丙酮酸代谢、碳代谢和氨基酸合成代谢等途径;九成熟与全熟果实的差异表达基因主要富集在糖酵解、脂肪酸代谢、淀粉和蔗糖代谢以及氨基酸合成代谢等途径。【结论】筛选出的调控果实成熟的关键基因包括26 个与乙烯合成及信号转导相关基因,44 个与ABA 合成及信号转导相关的基因。
关键词:无花果;转录组;差异表达基因;果实成熟度
中图分类号:S663.3 文献标志码:A 文章编号:1003—8981(2023)03—0214—10
无花果Ficus carica 为桑科Moraceae 无花果属Ficus 多年生灌木或小乔木,原产于地中海沿岸,是人类驯化较早的经济作物之一。其果实具有极高的营养、保健和药用价值,享有“21 世纪人类健康的守护神”“抗癌斗士”等称号[1-2]。无花果是优良的经济树种,为高端果树品种,具有巨大的开发潜力和价值[3]。然而,由于成熟的无花果皮薄多汁,采摘后易腐烂,果实保存期仅为2 ~ 3 d,严重制约了无花果大规模种植和产业发展。
根据成熟生理特性,水果可分为呼吸跃变型和非呼吸跃变型。呼吸跃变型果实成熟时,产生大量乙烯,呼吸速率急剧上升,具有较强后熟作用。例如,苹果一般在成熟前采摘,在储运过程中完成成熟过程。非呼吸跃变型果实在成熟过程中不出现乙烯释放高峰和呼吸速率高峰,其发育和成熟无明显阶段性,无后熟现象。例如,草莓必须在完全成熟时采摘,若在未成熟时采摘,果实不能在储运过程中完成成熟过程[4]。无花果则表现出特殊的双重成熟机制:果实成熟的后期出现乙烯释放高峰和呼吸速率高峰。无花果的果实成熟后迅速衰老,但无明显的后熟现象,只有在树上完熟后才能采摘,未成熟果实采摘后,通过乙烯处理难以诱导果实软化成熟[5]。为了延长保鲜时间,果农往往根据经验在无花果果实八成熟时采摘。
然而,八成熟与全熟的无花果果实间品质差别较大。完熟果实的总糖含量、糖酸比和维生素C 含量显著高于八成熟果实,糖酸比呈现跳跃式增长[6]。市场调查结果也显示,消费者更喜爱全熟的无花果果实[6-7]。无花果的果实品质和保鲜时间的矛盾一直困扰着无花果种植者,限制了无花果的规模生产。近年来,有一些关于无花果果实采收成熟度及采后保鲜的研究报道,主要的采后保鲜方法包括氯化钙浸泡、1- 甲基环丙烯(1-MCP)熏蒸、热激处理、冷激处理、低温保存以及过氧乙酸处理等[8],但无法从根本上解决无花果储存的问题。利用现代生物技术手段筛选调控果实成熟的关键基因,解析无花果果实成熟的分子调控机制,对于果实品质改良和新品种选育具有重要意义。转录组测序技术是探究植物发育分子机制的重要技术方法[9-11],已被广泛应用于文冠果[12]、杜仲[13]和忍冬[14] 等非模式植物果实发育机制的研究中。目前,有关无花果果实成熟机制的研究鲜见报道。为了探明无花果果实不同成熟阶段基因表达情况,为深入开展无花果果实成熟的分子调控机制研究提供参考,本研究中以无花果主栽品种‘芭勞奈为材料,使用转录组测序技术建立不同成熟度无花果果实的转录组数据库,筛选不同成熟度的差异表达基因,并进行分类、富集分析,重点分析激素合成和信号转导途径的差异表达基因。
1 材料方法
1.1 试验材料
供试无花果果实采自广东生态工程职业学院种质资源圃(113°38′E′,23°20′N),无花果树龄为4 a,品种为‘芭劳奈。选择长势一致、无病虫害的植株,采摘八成熟(S1)、九成熟(S2)和全熟(S3)的果实。每个时期设置3 次生物学重复,每次重复选取3 个植株的9 个果实(每植株3 个果实)。切除果实头部和尾部,去除果皮,取中间直径3 cm 左右的果肉部分,将每次重复的样品充分混匀,用锡箔纸包装后置于液氮中速冻,保存于-80 ℃冰箱中备用。
1.2 试验方法
1.2.1 文库构建及转录组测序
使用EASYspin 植物RNA 快速提取试剂盒提取无花果果实的总RNA,使用Nano Photometer 分光光度计和Qubit 2.0 荧光计检测总RNA 的纯度和浓度,使用Agilent 2100 生物分析仪检测RNA完整性。经检测合格的RNA 样品用于cDNA 文库的构建。用带有Oligo(dT)的磁珠富集mRNA,并将mRNA 片段化,以mRNA 短片段为模板,合成cDNA 的2 条链[15]。对双链cDNA 进行加工修饰,末端加poly(A)尾并连接测序接头,最后进行PCR 扩增,完成cDNA 文库的构建[15]。采用HiSeq4000 测序平台进行转录组测序。文库构建及转录组测序由天津诺禾致源科技有限公司完成。
1.2.2 De novo 组装
将经测序获得的原始数据去接头和引物序列,过滤掉低质量序列,即N 含量超过该read 碱基数的10%,或碱基数(Q ≤ 20)超过该条read 碱基数的50%,获得高质量序列[16]。使用Trinity 软件对高质量序列从头组装拼接获得转录本序列[17],然后使用corset 软件去冗余,筛选最长的Cluster序列作为Unigene[18],进行后续分析。
1.2.3 差异表达基因筛选及转录因子预测
采用FPKM(fragments per kilobase of transcriptper million fragments mapped)法衡量基因的表达量,使用DESeq2 软件分析不同成熟度果实样品之间RNA 的差异表达情况[19-20]。设置Rf < 0.05且|log2(Fc)| ≥ 1 为筛选标准(Rf 为错误率,Fc 为变化倍数),筛选差异表达基因(DEGs)。使用iTAK 软件预测转录因子[21]。根据PlnTFDB和PlantTFDB 数据库中定义好的转录因子家族及规则[22-23],通过hmmscan 比对的方式鉴定差异表达基因中的转录因子。
1.2.4 差异基因功能富集分析
使用DIAMOND Blastx 软件将筛选的DEGs序列进行GO 注释和KEGG 注释分析[24],并采用clusterProfiler 软件对差异基因进行GO 功能富集分析和KEGG 通路富集分析[24]。通过差异基因表达分析和KEGG 通路富集分析,筛选无花果果实成熟过程中与乙烯和ABA 信号转导途径相关的差异基因[25],使用Origin 软件绘制差异基因热图。
2 结果与分析
2.1 转录组测序数据分析
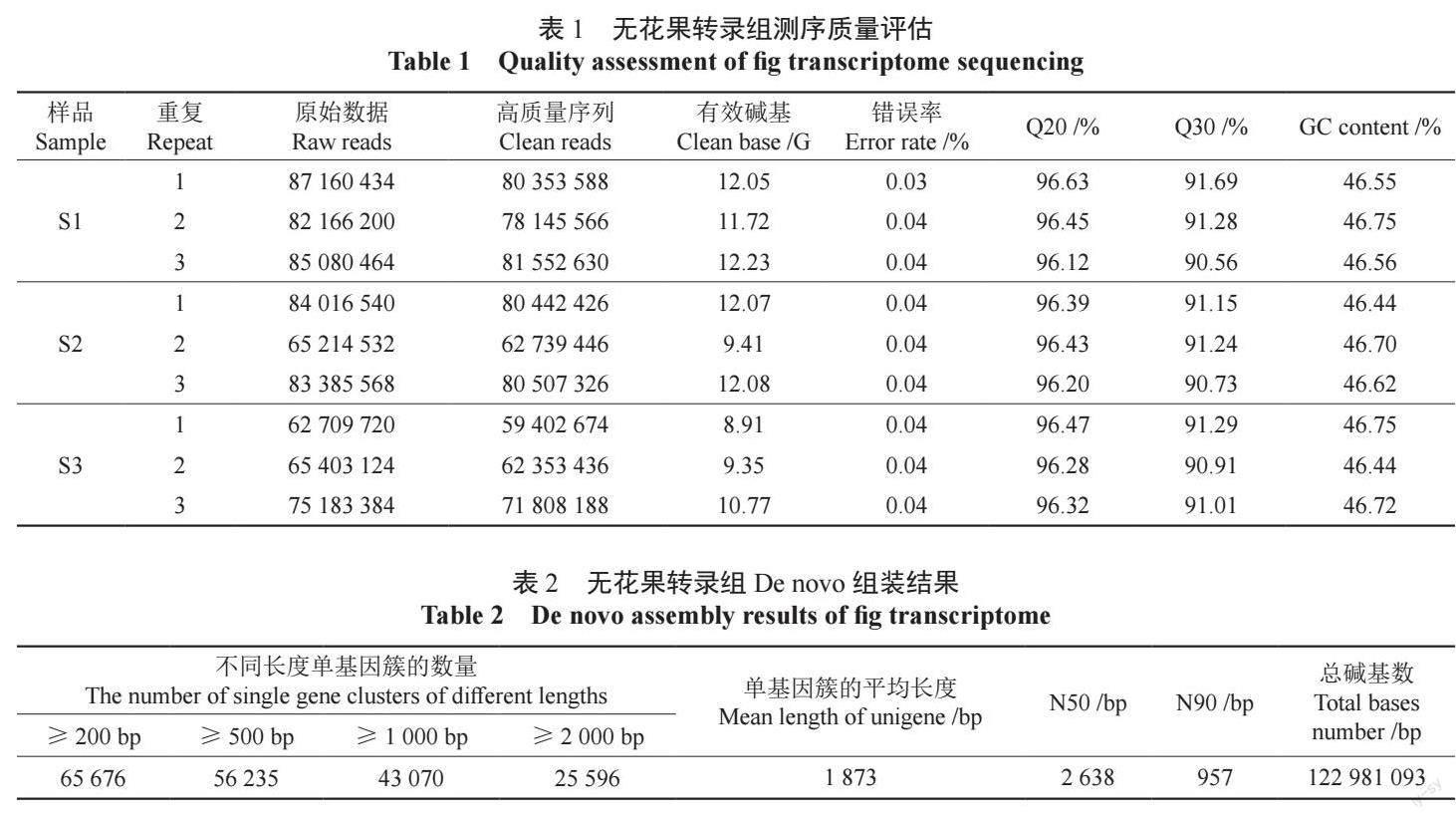
对无花果果实样品进行高通量测序分析,共获得690 319 966 原始读序,各样品的有效碱基平均为10.95 G,测序错误率为0.03% ~ 0.04%,Q20 碱基含量最小值为96.12%,Q30 碱基含量最小值为90.56%,GC 含量最小值为46.44%(表1)。经组装获得65 676 个Unigenes 序列,平均长度为1 873 bp,其中长度不小于1 000 bp 的Unigenes 有43 070 条。一般而言,N50 长度在800 bp 以上被认为组装序列的完整性较好。本研究中组装获得的序列N50 为2 638 bp(表2)。以上各项数据指标说明测序数据质量可满足后续分析的要求。
2.2 差异基因表达分析
通过比较不同成熟度的无花果果实的基因表达,共获得15 620 个差异基因。与九成熟果实相比,八成熟果实有1 508 个差异基因,其中上调1 158 个,下调350 个。与全熟果实相比,九成熟果实有11 385 个差异基因,显著多于与八成熟果实的差异基因,其中上调6 502 个,下调4 883 个。与全熟果实相比,八成熟果实差异基因最多,为13 932 个,其中上调7 986 个,下调5 946 个。经进一步分析发现,在八成熟和九成熟时期特异性差异表达的基因有274 个,在九成熟和全熟时期特异性差异表达的基因有1 219 个,在八成熟和全熟时期特异性差异表达的基因有3 462 个。此外在八成熟、九成熟、全熟时期有540 个共同差异表达的基因。
2.3 差异基因转录因子分析
转录因子是基因表达调控的关键因子。通过对不同成熟度无花果果实的差异表达基因进行转录因子预测,共获得2 936 个转录因子。由图1 可见,在八成熟和九成熟时期果实有113 个显著差异表达的转录因子,在九成熟和全熟时期果实有678 个显著差异表达的转录因子。对转录因子家族排名进行分析后发现:在八成熟和九成熟时期显著差异表达的转录因子中占比最高的是Jumonji 家族(11.5%),包含13 个显著差异表达的转录因子,其次是NAC 和AP2/ERF-ERF 家族,占比均为8.85%;在九成熟和全熟时期显著差异表达的转录因子中占比最高的是SET 家族(5.01%),包含34 个显著差异表达的转录因子,其次是bZIP 和bHLH 家族,占比分别为4.57% 和4.42%。
2.4 差异基因的GO 富集分析
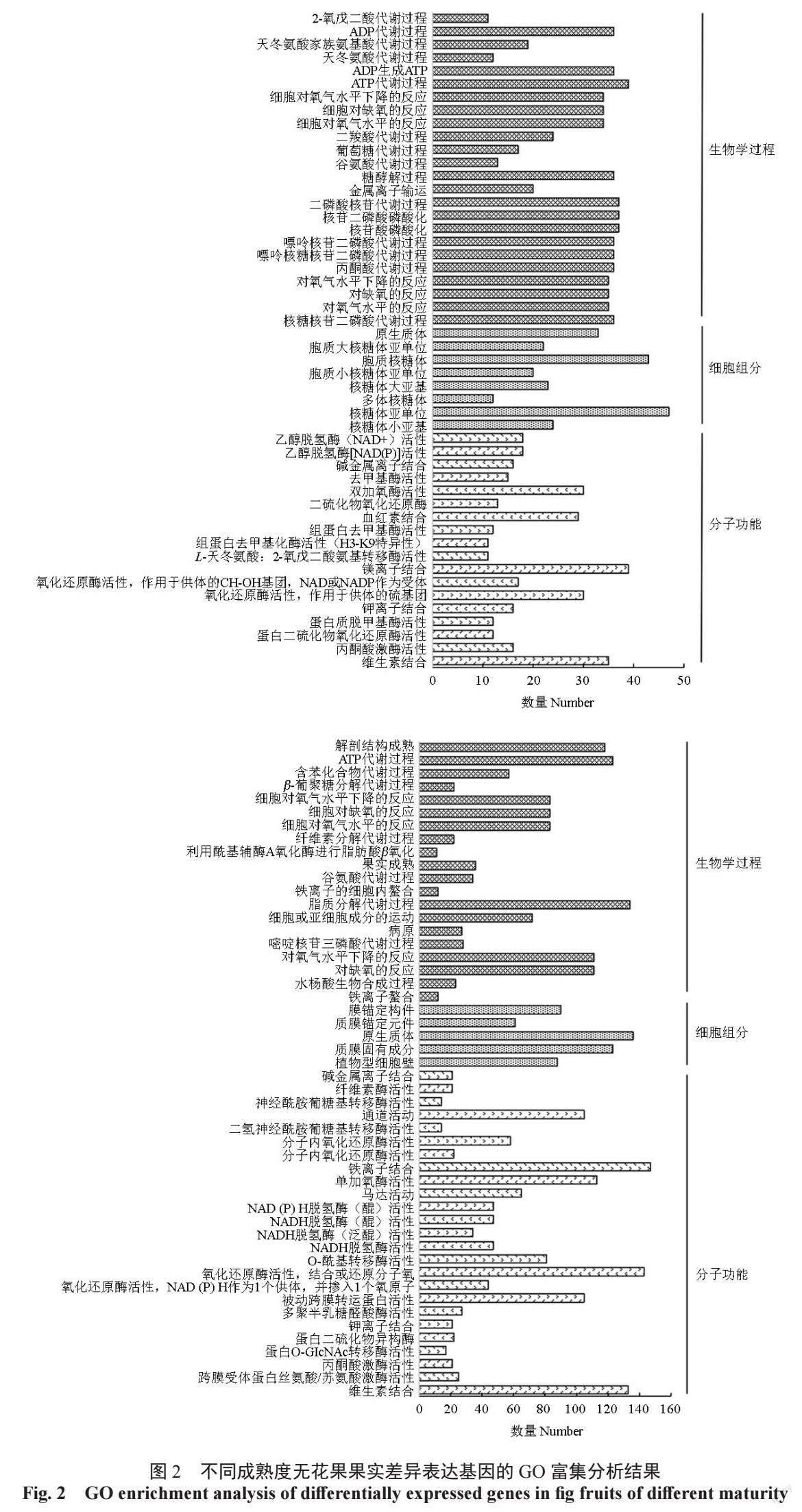
对不同成熟度无花果果实的差异表达基因进行GO 富集分析,结果表明,差异表达基因被富集到生物学过程、细胞组分以及分子功能三大类。排名前50 的GO 组分如图2 所示。在生物学过程方面,八成熟和九成熟时期果实的差异表达基因主要富集在糖酵解、ADP 生成ATP、ADP 代谢、嘌呤核苷二磷酸代谢等过程,而九成熟和全熟时期果实的差异表达基因主要富集在果实成熟、发病原和谷氨酸代谢等过程。在细胞组分方面,八成熟和九成熟时期果实的差异表达基因主要富集在胞质核糖体和核糖核蛋白体亚基,而九成熟和全熟时期果实的差异表达基因主要富集在植物细胞壁、膜(质膜和内膜)及锚定组分。在分子功能方面,八成熟和九成熟时期果实的差异表达基因主要富集在丙酮酸激酶活性、乙醇脱氢酶和NAD+ 活性以及碱金属和钾离子结合;而九成熟和全熟时期果实的差异表达基因主要富集在神经酰胺和二氢神经酰胺葡糖基转移酶活性。
2.5 差异基因的KEGG 富集分析
对不同成熟度无花果果实的差异表达基因进行KEGG 富集分析,富集最顯著的20 条通路如图3 所示。八成熟和九成熟时期果实的差异表达基因主要富集在糖酵解、丙酮酸代谢、碳代谢和氨基酸合成代谢等途径。九成熟和全熟时期果实的差异表达基因主要富集在糖酵解、脂肪酸代谢、淀粉和蔗糖代谢以及氨基酸合成代谢等途径。经进一步分析发现,3 个脂质代谢相关途径(脂肪酸代谢、不饱和脂肪酸的生物合成和甘油脂代谢,以及4 个糖代谢相关途径(淀粉和蔗糖的代谢、戊糖和葡萄糖醛酸转换、其他多糖降解和其他类型的O- 聚糖生物合成)在九成熟和全熟时期果实的差异表达基因中特异性富集。
2.6 乙烯和ABA 信号转导途径关键基因的表达模式分析
在不同成熟度的无花果果实中,检测到参与乙烯合成及信号转导途径相关的差异表达基因共计26 个(图4)。与八成熟果实相比,在九成熟果实中6 个乙烯合成相关基因均上调表达,包括1 个SAMS(Cluster-2772.0),1 个ACS(Cluster-16914.0),3 个ACO(Cluster-20160.0、Cluster-20160.1、Cluster-20160.2) 和1 个信号转导基因(Cluster-14830.0)。与九成熟果实相比,在全熟果实中共检测到12 个乙烯合成基因差异表达,5 个上调表达,包括2 个SAMS(Cluster-1868.0 和Cluster-9519.1), 和3 个ACO(Cluster-20160.0、Cluster-20160.1 和Cluster-20160.2),7 个下调表达, 包括5 个SAMS(Cluster-12282.0、Cluster-12970.0、Cluster-17563.0、Cluster-30389.0 和Cluster-9519.0),2 个ACS(Cluster-16914.0 和Cluster-23294.0)。同时检测到12 个乙烯信号转导基因差异表达,1 个ETR(Cluster-26426.3)下调表达,11个均上调表达,包括8 个CRT1(Cluster-17679.2、Cluster-17679.5、Cluster-21650.0、Cluster-21650.4、Cluster-21650.6、Cluster-26931.4、Cluster-30212.2和Cluster-30212.5),2 个MPK6(Cluster-23275.5和Cluster-23275.4)以及1 个EIN3(Cluster-20645.1)。
在不同成熟度的无花果果实中,检测到参与ABA 合成及信号转导相关的差异基因共计44 个(图4)。与八成熟果实相比,在九成熟果实中检测到6 个ABA 合成基因差异表达,5 个上调表达,包括4 个ZEP(Cluster-24371.5、Cluster-24371.6、Cluster-24371.7 和Cluster-30802.0), 和1 个SDR(Cluster-13733.0),而1 个NCED(Cluster-4218.0)下调表达。同时检测到1 个ABA 信号转导基因PP2C(Cluster-29918.2)下调表达。与九成熟果实相比,在全熟果实中检测到13 个ABA 合成基因,7 个上调表达,包括5 个ZEP(Cluster-24371.0、Cluster-24371.1、Cluster-24371.4、Cluster-24371.5和Cluster-24371.7),2 个NCED(Cluster-31657.4和Cluster-4218.0),6 个下调表达,包括2 个ZEP(Cluster-30802.3 和Cluster-30802.4),2 个NCED(Cluster-10575.0 和Cluster-8141.0),1 个SDR(Cluster-12339.0)以及1 个AAO3(Cluster-7523.12)。
同时检测到PP2C、SnRK2 和ABF 共28 个ABA信号转导基因。其中,20 个上调表达, 包括13 个PP2C(Cluster-9481.1、Cluster-9481.0、Cluster-7565.0、Cluster-29918.9、Cluster-29918.8、Cluster-29918.7、Cluster-29918.4、Cluster-29918.2、Cluster-29918.10、Cluster-29918.1、Cluster-19606.0、Cluster-26571.5 和Cluster-28800.8),5 個ABF(Cluster-18662.1、Cluster-27803.0、Cluster-27803.2、Cluster-27803.4 和Cluster-29950.4)以及2 个SnRK2(Cluster-25974.0 和Cluster-29585.3),8 个下调表达,包括3 个PP2C(Cluster-20288.0、Cluster-26431.7 和Cluster-28161.0)以及5 个SnRK2(Cluster-6322.0、Cluster-29585.8、Cluster-25108.0、Cluster-20892.1 和Cluster-20892.0)。
3 结论与讨论
本研究中利用转录组测序技术,分析了‘芭劳奈无花果果实成熟过程中基因表达的变化,共获得15 620 个差异表达基因。八成熟和九成熟果实的差异表达基因主要富集在核糖体细胞组分、糖酵解、ADP 和ATP 代谢等过程。九成熟和全熟期果实的差异表达基因主要富集在细胞壁相关组分以及果实成熟、发病原代谢等过程。KEGG 分析结果表明,八成熟与九成熟果实及九成熟与全熟期果实的差异表达基因均在糖酵解和氨基酸合成代谢途径富集。在九成熟和全熟期果实的差异表达基因中,脂肪酸代谢、淀粉和蔗糖代谢途径也显著富集。通过进一步筛选,获得26 个与乙烯信号转导相关的差异表达基因,获得44 个与ABA信号转导相关的差异表达基因。基于本研究结果,利用相关的激素或关键基因延长无花果的保鲜时间,将是后续的重点研究工作。
通过对不同成熟度无花果果实进行转录组测序,共获得15 620 个差异表达基因。统计发现,九成熟与全熟期果实的差异表达基因数量(11 385)是八成熟与九成熟果实差异表达基因数量(1 508)的7.5 倍,其中九成熟与全熟期果实的差异转录因子数量(678 个)也显著高于八成熟与九成熟果实差异转录因子数量(113 个),说明从九成熟至全熟转变过程中,无花果基因表达十分活跃,果实内部的代谢活动更加旺盛,这可能与果实处于呼吸跃变时期有关[26]。
GO 分析结果表明,八成熟与九成熟果实的差异表达基因主要富集在核糖体相关的细胞组分。果实成熟初期,核糖体蛋白基因可以在翻译水平上进一步调控成熟相关基因的翻译效率[15]。九成熟和全熟期果实的差异表达基因主要富集在植物细胞壁相关的细胞组分。完全成熟的无花果果实变软,说明细胞壁的细胞成分发生显著变化。KEGG 分析结果表明,八成熟与九成熟果实的差异表达基因主要富集在糖酵解、ADP 和ATP 代谢过程。有研究结果表明,果实开始成熟时,糖酵解途径的关键酶被活化,呼吸活性增强[27],使葡萄糖分解生成ATP,为其他代谢活动提供部分能量。转录组测序结果与果实成熟过程的生理特征变化一致,验证了转录组测序的可靠性。九成熟和全熟期果实的差异表达基因主要富集在果实成熟和发病原等途径。推测由于成熟的无花果散发香气,更易吸引果蝇等病虫从果孔入侵,因此发病原等途径显著富集。部分糖代谢有关途径在九成熟和全熟期果实的差异表达基因中特异性富集,这可能与九成熟至全熟期间,无花果果实中淀粉开始分解,葡萄糖和果糖等糖类快速积累有关[28]。在九成熟和全熟期果实中脂类相关代谢途径也发生显著变化,说明脂类物质的积累也与果实成熟密切相关。
乙烯是调控果实成熟的关键因素,是触发呼吸跃变型果实成熟的重要启动因素[29]。呼吸跃变型果实中乙烯合成有2 个系统:系统Ⅰ负责生产低浓度的乙烯,具有自体抑制的特点,控制组织的基础乙烯含量;系统Ⅱ在呼吸跃变期生产高水平的乙烯,具有自体催化的特点。已有报道验证植物内源乙烯合成的限速酶ACC 合成酶(ACS)和ACC 氧化酶(ACO)调控番茄[30]、香瓜[31]、梨[32]、猕猴桃[33] 和苹果[34] 等多种果实成熟。本研究中,在八成熟与九成熟果实的差异表达基因中6 个乙烯合成基因和1 个信号转导基因均上调表达,说明乙烯合成系统Ⅱ已经开始启动,加速乙烯合成。与九成熟果实相比,在全熟果实中Cluster-23294.0(ACS) 显著下调表达,说明其可能仅参与乙烯合成系统Ⅰ,跃变期果实内乙烯含量升高,负反馈调节ACS 下调表达。Cluster-20160.0,Cluster-20160.1 和Cluster-20160.2(ACO)在果实成熟过程(八成熟至全熟)一直上调表达,说明其同时参与乙烯合成系统Ⅰ和Ⅱ,促进乙烯合成,刺激果实成熟。
乙烯信号转导途径中,乙烯受体ETR[35] 与CTR1[36] 为负调控元件,下游的EIN2[37]、EIN3[38]和ERF[39] 在乙烯信号转导过程中起正调控作用。本研究结果与之相符,与九成熟果实相比,在全熟果实中乙烯负调控元件ETR 显著下调,正调控元件MPK6 和EIN3 均显著上调表达,说明乙烯信号转导通路被激活,促进果实成熟。不同的是,在无花果果实成熟过程中,CTR1 显著上调表达,这与其乙烯信号转导负调控作用相悖,推测乙烯调控无花果果实成熟可能存在新的信号转导模型。
ABA 在水果成熟衰老调控过程中起着重要作用。内源ABA 含量的升高以及外源ABA 处理均会导致水果成熟和果肉软化[40]。张涵等[41] 经研究发现,外源ABA 处理能够促进无花果果实的成熟,ABA 抑制剂能抑制无花果果实的成熟。乔菡[42] 经研究发现,在无花果果实成熟过程中,内源ABA含量持续增加,ABA 与乙烯协同作用诱导果实成熟。目前,尚不清楚ABA 调控果实成熟的作用机制。本研究结果表明,在无花果果实成熟过程中,ABA 合成基因发生显著变化。植物体内ABA是在9- 顺式环氧类胡萝卜素脱氢酶(NCED)的作用下,由类胡萝卜素合成而来,NCED 是调控ABA 生物合成的关键酶[43]。ABA 醛氧化酶(AAO)可将脱落醛转化为ABA,是催化ABA 生物合成的最后一步,拟南芥AtAAO3 沉默后出现明显的ABA 缺失[44]。本研究中,与九成熟果实相比,在全熟果实中检测到4 个NCED 基因(2 个上调表达,2 个下调表达)和1 个AAO3 基因(下调表达)可能参与体内ABA 合成调控。
本研究结果表明,在无花果果实成熟过程中,ABA 信号转导途径的PP2C、SnRK2 和ABF关键基因的表达发生显著变化。PP2C 是ABA 信号途径的负调控元件,SnRK2 是ABA 信号途径的正调控元件。ABA 与受体结合,抑制PP2C 的酶活性,增加SnRK2 的酶活性,磷酸化下游目标蛋白(ABF),ABA 信号通路被激活[45]。本研究中,与九成熟果实相比,在全熟果实中检测到5 个ABF 显著上调表达,表明ABA 的信号通路被激活。同时检测到3 个PP2C(Cluster-20288.0、Cluster-26431.7 和Cluster-28161.0) 下調表达,2 个SnRK2(Cluster-25974.0 和Cluster-29585.3)上调表达,推测ABF 可能参与ABA 信号转导调控无花果果实成熟。
参考文献:
[1] 沈元月. 我国无花果发展现状、问题及对策[J]. 中国园艺文摘,2018,34(2):75-78,122.
SHEN Y Y. Current situation, problems and solutions of figdevelopment in China[J]. Chinese Horticulture Abstracts,2018,34(2):75-78,122.
[2] KISLEV M E, HARTMANN A, BAR-YOSEF O. Earlydomesticated fig in the Jordan Valley[J]. Science,2006,312:1372-1374.
[3] 吴子江, 马翠兰, 郭阳彬, 等. 无花果生产与研究进展[J]. 亚热带农业研究,2013,9(3):151-157.
WU Z J, MA C L, GUO Y B, et al. Overview of fig (Ficus carica)production and research[J]. Subtropical Agriculture Research,2013,9(3):151-157.
[4] KUMAR R, KHURANA A, SHARMA A K. Role of planthormones and their interplay in development and ripening offleshy fruits[J]. Journal of Experimental Botany,2014,65(16):4561-4575.
[5] 李春丽, 沈元月. 无花果果实发育过程中ABA 和乙烯含量与果实成熟的关系[J]. 中国农业大学学报,2016,21(11):51-56.
LI C L, SHEN Y Y. Relationship between ABA and ethylenecontent and fruit ripening during fig fruit development[J]. Journalof China Agricultural University,2016,21(11):51-56.
[6] 孙锐, 孙蕾, 马金辉, 等. 不同成熟度无花果品质指标的变化分析[J]. 经济林研究,2017,35(2):32-37.
SUN R, SUN L, MA J H, et al. Change analysis of figsquality indicators in different maturities[J]. Non-wood ForestResearch,2017,35(2):32-37.
[7] CRISOSTO C H, BREMER V, FERGUSON L, et al.Evaluating quality attributes of four fresh fig (Ficus carica L.)cultivars harvested at two maturity stages[J]. HorticultureScience,2010,45(4):707-710.
[8] 颜道民, 尹金晶, 唐晋文, 等. 鮮食无花果贮藏保鲜技术研究进展[J]. 食品安全质量检测学报,2019,10(9):2462-2467.
YAN D M, YIN J J, TANG J W, et al. Research advances onstorage and fresh-keeping technology of freshFicus carica[J].Journal of Food Safety & Quality,2019,10(9):2462-2467.
[9] MARTIN L B B, FEI Z J, GIOVANNONI J J, et al. Catalyzingplant science research with RNA-seq[J]. Frontiers in Plant Science,2013,4:66.
[10] 王保明, 颜士华, 王胜清, 等.‘ 横冲89 油茶转录组及光合油脂代谢途径基因表达分析[J]. 经济林研究,2022,40(2):31-39.
WANG B M, YAN S H, WANG S Q, et al.Camellia oleifera‘Hengchong 89 transcriptomes and gene expression ofphotosynthesis and lipid pathway[J]. Non-wood Forest Research,2022,40(2):31-39.
[11] 李树战, 王艺儒, 孙鹏, 等.‘ 龙岩野柿1 号 雄花和两性花花芽的转录组SSR 特征分析[J]. 中南林业科技大学学报,2022,42(9):171-177,186.
LI S Z, WANG Y R, SUN P, et al. Analysis of SSR characteristicsofDiospyros kaki ‘Longyanyeshi 1 based on the transcriptome ofmale and hermaphroditic floral buds[J]. Journal of Central SouthUniversity of Forestry & Technology,2022,42(9):171-177,186.
[12] 赵阳阳, 郭雨潇, 张凌云. 文冠果果实转录组测序及分析[J].生物技术通报,2019,35(6):24-31.
ZHAO Y Y, GUO Y X, ZHANG L Y. Transcriptome sequencingand analysis ofXanthoceras sorbifolia Bunge fruit[J].Biotechnology Bulletin,2019,35(6):24-31.
[13] 李铁柱, 杜红岩, 刘慧敏, 等. 杜仲幼果和成熟果实转录组数据组装及基因功能注释[J]. 中南林业科技大学学报,2012,32(10):9-17.
LI T Z, DU H Y, LIU H M, et al. Transcriptome data assemblyand gene function annotation ofEucommia mature fruits andyoung fruits[J]. Journal of Central South University of Forestry &Technology,2012,32(10):9-17.
[14] 张庆田, 李晓艳, 杨义明, 等. 蓝靛果忍冬转录组SSR 信息分析及其分子标记开发[J]. 园艺学报,2016,43(3):557-563.
ZHANG Q T, LI X Y, YANG Y M, et al. Analysis on SSRinformation in transcriptome and development of molecular markersinLonicera caerulea[J]. Acta Horticulturae Sinica,2016,43(3):557-563.
[15] WANG W H, WANG P W, LI X J, et al. The transcriptionfactor SlHY5 regulates the ripening of tomato fruit at both thetranscriptional and translational levels[J]. Horticulture Research,2021,8:83.
[16] GARG R, PATEL R K, TYAGI A K, et al.De novo assembly ofchickpea transcriptome using short reads for gene discovery andmarker identification[J]. DNA Research,2011,18:53-63.
[17] GRABHERR M G, HAAS B J, YASSOUR M, et al. Trinity:reconstructing a full-length transcriptome without a genome fromRNA-Seq data[J]. Nature Biotechnology,2011,29(7):644-652.
[18] DAVIDSON N M, OSHLACK A. Corset: enabling differentialgene expression analysis forde novo assembled transcriptomes[J].Genome Biology,2014,15(7):410.
[19] LOVE M I, HUBER W, ANDERS S. Moderated estimation offold change and dispersion for RNA-seq data with DESeq2[J].Genome Biology,2014,15:550.
[20] VARET H, BRILLET-GU?GUEN L, COPP?E J Y, et al. SARTools: a DESeq2- and EdgeR-based rpipeline forcomprehensive differential analysis of RNA-seq data[J]. PLoS One,2016,11(6):0157022.
[21] ZHENG Y, JIAO C, SUN H H, et al. iTAK: a program forgenome-wide prediction and classification of plant transcriptionfactors, transcriptional regulators, and protein kinases[J].Molecular Plant,2016,9(12):1667-1670.
[22] P?REZ-RODR?GUEZ P, RIA?O-PACH?N D M, CORR?AL G G, et al. PlnTFDB: updated content and new featuresof the plant transcription factor database[J]. Nucleic AcidsResearch,2010,38:D822-D827.
[23] JIN J P, ZHANG H, KONG L, et al. PlantTFDB 3.0: a portalfor the functional and evolutionary study of plant transcriptionfactors[J]. Nucleic Acids Research,2014,42(D1):D1182-D1187.
[24] BUCHFINK B, XIE C, HUSON D H. Fast and sensitive proteinalignment using DIAMOND[J]. Nature Methods,2015,12(1):59-60.
[25] MAO X Z, CAI T, OLYARCHUK J G, et al. Automatedgenome annotation and pathway identification using the KEGGOrthology (KO) as a controlled vocabulary[J]. Bioinformatics,2005,21(19):3787-3793.
[26] ROSIANSKI Y, DORON-FAIGENBOIM A, FREIMAN Z E,et al. Tissue-specific transcriptome and hormonal regulation ofpollinated and parthenocarpic fig (Ficus carica L.) fruit suggestthat fruit ripening is coordinated by the reproductive part of thesyconium[J]. Frontiers in Plant Science,2016,7:1696.
[27] 李昂, 苗玉樂, 孟君仁, 等. 溶质和硬质型桃果实成熟过程果肉多肽组学分析[J]. 中国农业科学,2022,55(11):2202-2213.
LI A, MIAO Y L, MENG J R, et al. Peptidome analysis ofmesocarpin melting flesh and stony hard peach during fruitripening[J]. Scientia Agricultura Sinica,2022,55(11):2202-2213.
[28] LI Y C, ZHU B Z, XU W T, et al.LeERF1 positively modulatedethylene triple response on etiolated seedling, plant developmentand fruit ripening and softening in tomato[J]. Plant Cell Reports,2007,26(11):1999-2008.
[29] GRIERSON D. Ethylene and the control of fruit ripening[M].Hoboken: John Wiley & Sons, Inc.,2013.
[30] LI S, ZHU B Z, PIRRELLO J, et al. Roles of RIN and ethylenein tomato fruit ripening and ripening-associated traits[J]. NewPhytologist,2020,226:460-475.
[31] AYUB R, GUIS M, AMOR M B, et al. Expression of ACCoxidase antisense gene inhibits ripening of cantaloupe melonfruits[J]. Nature Biotechnolgy,1996,14:862-866.
[32] GAO M, MATSUTA N, MURAYAMA H, et al. Gene expressionand ethylene production in transgenic pear (Pyrus communis cv.‘La France) with sense or antisense cDNA encoding ACCoxidase[J]. Plant Science,2007,173:32-42.[33] ATKINSON R G, GUNASEELAN K, WANG M Y, et al.Dissecting the role of climacteric ethylene in kiwifruit (Actinidiachinensis) ripening using a 1-aminocyclopropane-1-carboxylicacid oxidase knockdown line[J]. Journal of ExperimentalBotany,2011,62:3821-3835.
[34] DANDEKAR A M, TEO G, DEFILIPPI B G, et al. Effect ofdown-regulation of ethylene biosynthesis on fruit flavor complexin apple fruit[J]. Transgenic Research,2004,13:373-384.
[35] CHANG C, KWOK S F, BLEECKER A B, et al.Arabidopsisethylene-response geneETR1: similarity of product to twocomponentregulators[J]. Science,1993,262:539-544.
[36] CLARK K L, LARSEN P B, WANG X X, et al. Association oftheArabidopsis CTR1 Raf-like kinase with the ETR1 and ERSethylene receptors[J]. Proceedings of the National Academy ofSciences,1998,95(9):5401-5406.
[37] BISSON M M, BLECKMANN A, ALLEKOTTE S, et al. EIN2,the central regulator of ethylene signalling, is localized at the ERmembrane where it interacts with the ethylene receptorETR1[J].Biochemical Journal,2009,424:1-6.
[38] GAGNE J M, SMALLE J, GINGERICH D J, et al.ArabidopsisEIN3-binding F-box1 and 2 form ubiquitin-protein ligasesthat repress ethylene action and promote growth by directingEIN3 degradation[J]. Proceedings of the National Academy ofSciences,2004,101:6803-6808.
[39] YOO S D, CHO Y H, TENA G, et al. Dual control of nuclearEIN3 by bifurcate MAPK cascades in C2H4 signalling[J]. Nature,2008,451:789-795
[40] 杨方威, 段懿菲, 冯叙桥. 脱落酸的生物合成及对水果成熟的调控研究进展[J]. 食品科学,2016,37(3):266-272.
YANG F W, DUAN Y F, FENG X Q. Advances in biosynthesisof abscisic acid and its roles in regulation of fruit ripening[J].Food Science,2016,37(3):266-272.
[41] 张涵, 乔菡, 廖亚军, 等. ABA 及其抑制剂对无花果果实成熟的影响[J]. 北京农学院学报,2019,34(3):42-45.
ZHANG H, QIAO H, LIAO Y J, et al. Effect of ABA and itsinhibitor on fig fruit ripening[J]. Journal of Beijing University ofAgriculture,2019,34(3):42-45.
[42] 乔菡. 乙烯以ABA 依赖的方式调控无花果果实成熟的分子机理[D]. 北京: 北京农学院,2021.
QIAO H. Molecular mechanism of ethylene regulating FIG fruitripening in an ABA-dependent manner[D]. Beijing: BeijingAgricultural College,2021.
[43] TAYLOR I B, BURBIDGE A, THOMPSON A J. Control ofabscisic acid synthesis[J]. Journal of Experimental Botany,2000,51:1563-1574.
[44] JIANG F, HARTUNG W. Long-distance signaling of abscisicacid (ABA): the factors regulating the intensity of the ABAsignal[J]. Journal of Experimental Botany,2008,59(1):37-43.
[45] LIN Z, LI Y, WANG Y B, et al. Initiation and amplificationof SnRK2 activation in abscisic acid signaling[J]. NatureCommunications,2021,12(1):2456.
[ 本文編校:闻 丽]

