线粒体DNA在鳞翅目昆虫系统学研究中的应用*
房守敏
(西华师范大学生命科学学院,四川南充 637002)
随着分子生物学的发展,DNA序列用于昆虫和其他动物系统发育和分子进化的研究越来越受到人们的重视。除了一些快速进化的核基因如无翅(wingless,Wg)[1]和延伸因子1α(elongation factor 1-alpha,EF-1α)[2]等能用于分子系统学研究外,线粒体DNA(mitochondrial DNA,mtDNA)由于其进化速率快而备受关注。自Nass等[3]于1962年发现mtDNA以来,近年来人们对其基因组结构和进化进行了深入细致的研究,发现线粒体基因组分子量小、拷贝数多、复合母系遗传、不发生重组、突变率高等特点。动物和昆虫的线粒体基因往往比核基因的进化速率分别快5~10倍[3]和2~9倍[4]。不同的线粒体基因有不同的进化速率,通常进化速率最大的是控制区;蛋白编码基因中的细胞色素氧化酶I(cytochrome oxidase I,COI)、COII和NADH脱氢酶亚基5(NADH dehydrogenase subunit 5,ND5)次之;核糖体RNA(ribosomal RNA,rRNA)的大小亚基(12S和16S)较保守;进化速率最慢的是转运RNA(transfer RNA,tRNA)。由于线粒体进化速率快和基因组相对较小等优点,mtDNA已被广泛用于系统发育、分子进化、昆虫分类学和群体遗传学等研究[5]。
鳞翅目(Lepidoptera)有40个以上的总科、约120个以上的科及18万种,是仅次于鞘翅目的第二大目,包括蝶类和蛾类。鳞翅目昆虫是完全变态生物,发育过程分为卵、幼虫、蛹及成虫四个时期,是研究昆虫拟态、与寄主植物关系、翅膀模式发育生物学和昆虫生理等研究的重要模式生物。目前,GenBank(http://www.ncbi.nlm.nih.gov/)中鳞翅目昆虫约有68条线粒体全基因组序列,其中47条来自蚕蛾总科(Bombycoidea)[6-9],这可能是由于家蚕是重要的经济昆虫并且也是鳞翅目昆虫的模式生物的缘故。另外,有成千上万条的鳞翅目线粒体基因序列被测定,这些序列已被广泛应用于群体遗传结果和系统发生分析等研究。本文主要对线粒体DNA在鳞翅目昆虫分子系统学研究的应用,以及不同线粒体DNA区段在鳞翅目昆虫不同分类阶元的适用性进行了综述,为今后鳞翅目昆虫系统发生分析提供参考。
1 鳞翅目昆虫mtDNA结构特征
鳞翅目昆虫mtDNA与其他动物一样,是闭合双连环状DNA分子。目前GenBank中鳞翅目昆虫mt全基因组的大小为14 535 bp(玉米螟Ostrinia nubilalis,Accession no.AF442957)~16 094 bp(玛爱杰凤蝶Papiliomaraho,Accession no.NC_003395)。线粒体全基因组包括13个蛋白编码基因(COI、COII和COIII;细胞色素氧化酶亚基b基因Cytb;ATPase复合物亚基ATP6和ATP8基因;ND1~6和ND4L)、12S rRNA、16S rRNA、22个tRNA及1个A+T富集区。A+T富集区有控制mtDNA的复制和转录的功能,又叫控制区(control region,CR)。线粒体基因组各基因间排列紧密,仅存在较短的基因间隔序列。例如中国野桑蚕mt基因组含有17个基因,间区仅391 bp[10]。而且线粒体基因不含内含子,部分基因还存在重叠现象[10]。因此,线粒体碱基的使用节约、高效。
2 mtDNA应用于鳞翅目昆虫分子系统学的研究
2.1 蛋白编码基因
蛋白编码基因是线粒体的重要组织部分,对于完成线粒体全基因组测序的鳞翅目昆虫而言,常用13个基因的核酸或氨基酸连接序列进行分子进化的研究[10-11]。但更多的是选取线粒体的部分代表性基因进行相关的分子系统学的研究。
2.1.1 COI与COII基因
我们对PubMed(http://www.ncbi.nlm.nih.gov/)中截至2009年的文献进行分析与整理,发现鳞翅目昆虫分子系统学的研究中COI与COII基因的应用最广,至少有51项研究涉及利用了COI和(或)COII基因,而且COI基因的应用更为广泛(表1)。这些研究中还常常结合线粒体的其他基因或区段(16S rDNA、ND5和控制区等)及核基因Wg(wingless)和EF-1α(elongationfactor 1-alpha)共同揭示鳞翅目昆虫的进化关系。表1还显示,利用COI/COII基因分析鳞翅目蛱蝶科Nymphalidae、凤蝶科Papilionidae和灰蝶科Lycaenidae等昆虫系统发生关系的研究较多。
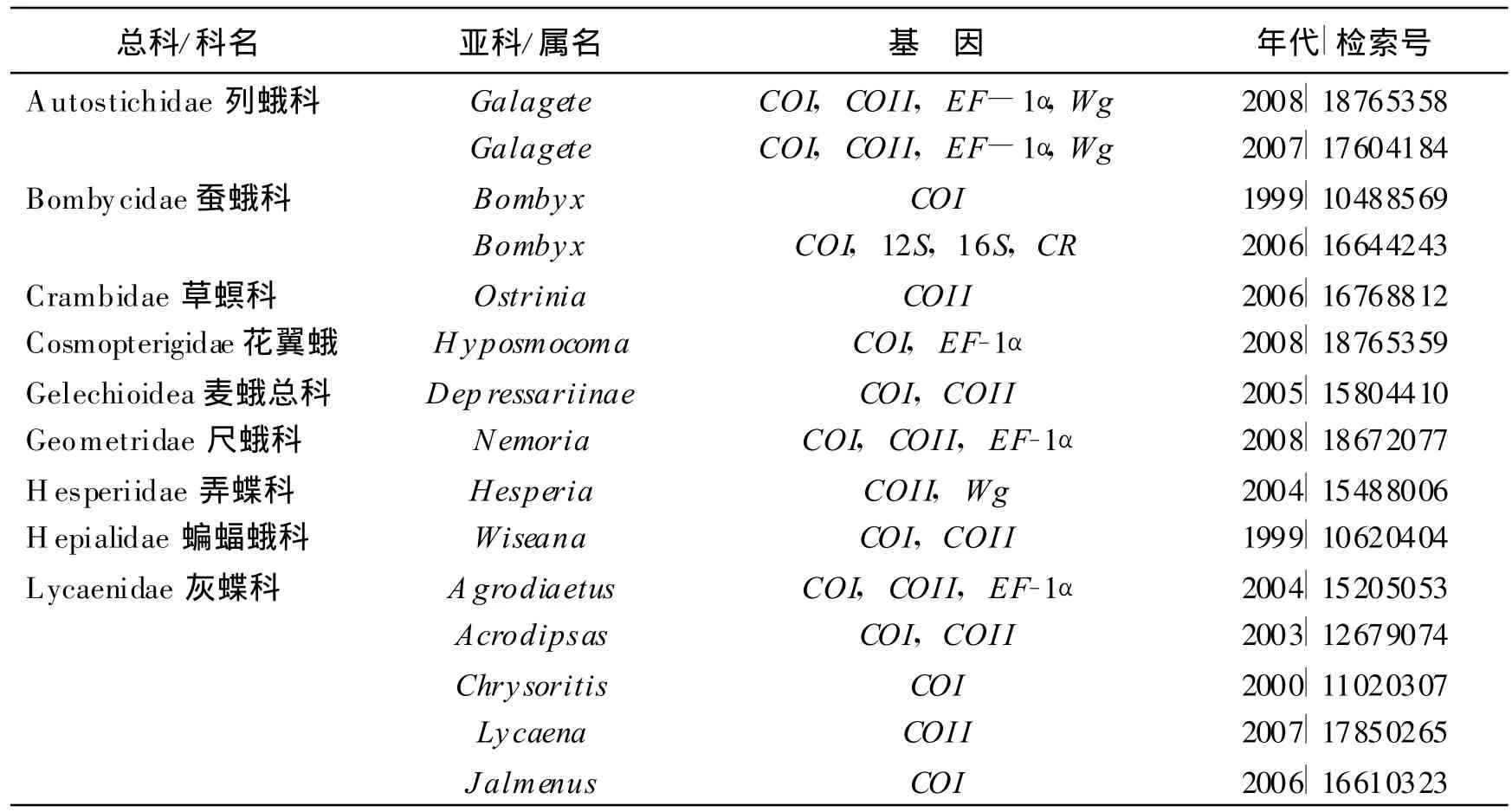
表1 线粒体COI/COI I基因在鳞翅目昆虫分子系统学的研究中的应用
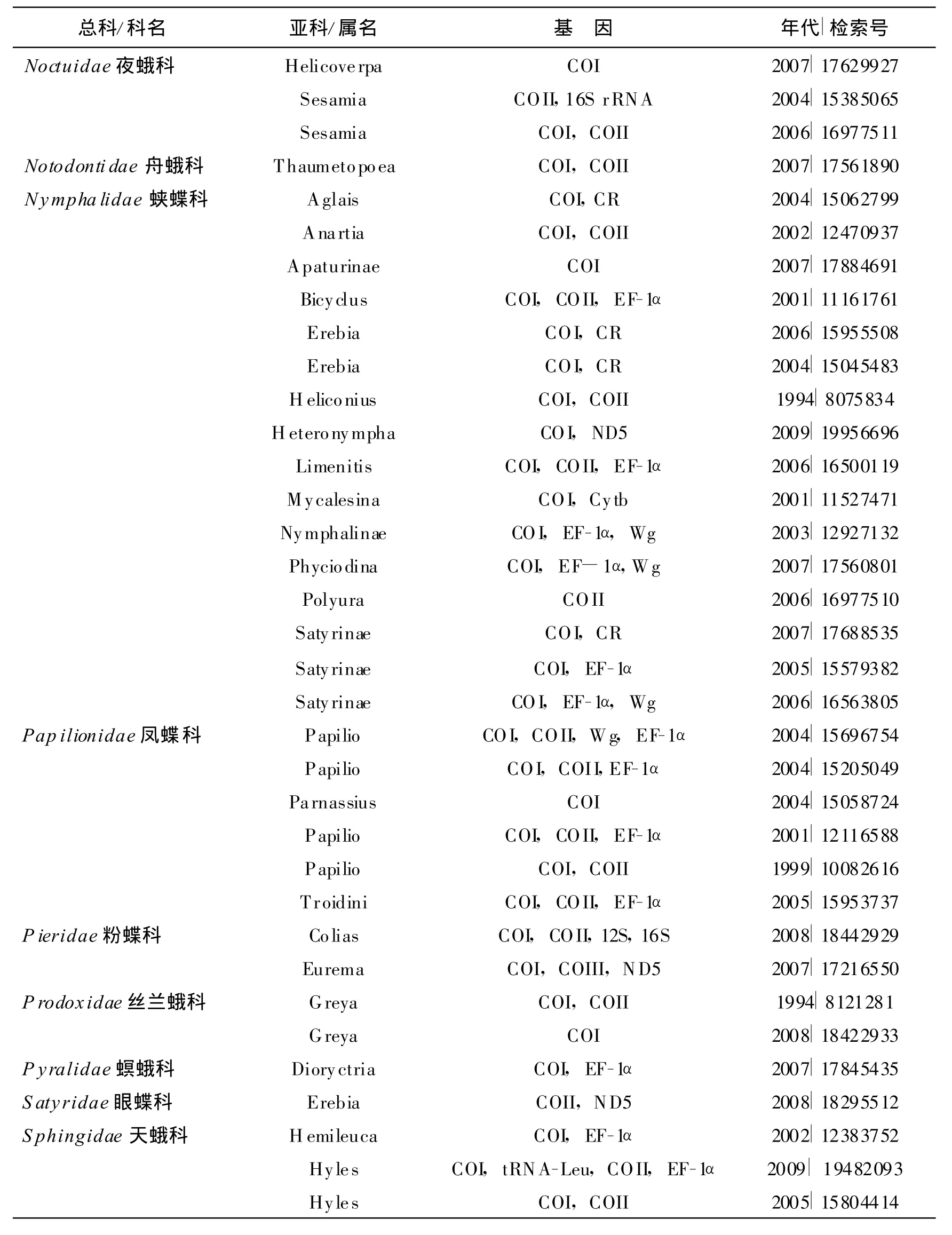
续表1 线粒体COI/COII基因在鳞翅目昆虫分子系统学的研究中的应用
COI基因已被较多地应用于鳞翅目昆虫种内、种间及属间分子系统学研究。DeChaine和Martini[12]对落基山脉20个不同区域帕纳塞斯蝶Parnassius smintheus的385个个体COI基因进行了测序,进化分析发现在落基山脉的北部和南部至少存在两个多样化的中心,并且利用巢式进化支分析(nested clade analysis)揭示出该物种经历了多次的群体扩增和分裂事件。Rand等[13]以COI基因对灰蝶科Chrysoritis、Poecilmitis和Oxychaeta三个属的昆虫进行分子系统发生分析时发现3个属应归属于同一个Chrysoritis属,这与基于形态特征的系统假设一致。Hwang等[14]分析发现家蚕COI基因与野桑蚕、日本柞蚕Antheraea yamamai和中国柞蚕A.pernyi的序列相似性分别为97%、85%和87%,基于COI构建的系统发生树表明大蚕蛾科和蚕蛾科呈单源系起。Behere等[15]以COI基因部分序列对夜娥科Noctuidae的昆虫作了进化分析,表明澳洲棉铃虫Helicoverpa punctigera在进化上先于烟草夜娥H.assulta,而后相继分化形成棉铃虫H.armigera和玉米夜蛾H.zea,并且北美和南美大陆的H.zea从H.armigera祖先分歧年代非常近<1.5百万年前(million years ago,Mya)。
COII基因在鳞翅目昆虫亚种及种间的分子进化研究中有一定的应用。Long等[16]对大二尾蛱蝶Polyura eudamippus 5个亚种的50个样本COII基因的单倍型进行了分析,结果表明在405bp的COII部分序列中共发现了9种单倍型,5个亚种的地理分布与单倍型空间分布基本一致,并且推测在冰河期以后大二尾蛱蝶采取了两条线路的扩张,一条是向北至四川、重庆和湖北,另一条则是向东至中国大陆的东南海和台湾。Ohno等[17]以COII基因对虎杖螟Ostrinia latipennis群体的两个种O.latipennis(Warren)与O.ovalipennis Ohno进行分子进化研究,发现其亲缘关系较近,约在0.3百万年前发生的分歧。王戎疆等[18]利用线粒体COII基因对尾蛱蝶属5个种的12个样品分析,在405bp长的COII片段有11.4%的位点为多态性位点,在进化上尾蛱蝶属蝴蝶分为两大分支,一支包括大二尾蛱蝶、二尾蛱蝶和忘忧尾蛱蝶,另一分支包括窄斑凤尾蛱蝶和黑凤尾蛱蝶。这些分子系统学的结果均与形态学的结果相一致。
相对于COI或COII单基因研究而言,COI和COII基因的连接序列有着更广泛的应用,它可用于属、种及亚种分类阶元的进化分析,而且还常用于分歧年代的估算。1994年,Brown等[19]对丝兰蛾科(Prodoxidae)Greya属16个种的COI+COII基因765 bp序列作了进化分析,其结果与形态分类大致相似。1999年,Caterino和Sperling[20]初次对凤蝶22个种和1个亚种的COI和COII基因测序,研究者分别以COI、COII和COI+COII的核酸序列作进化分析,结果发现仅以COI基因不能支持garamas与scamander世系单系起源,COII则不能解决Papilionini科单系起源问题,而COI+COII则能较好地消除这两个问题,因此非常适宜于凤蝶亚属及种间的分子进化研究。2004年,Zakharov等[21]利用COI+COII对凤蝶的51个种进行了系统发育分析,参考考古学证据,估算出了系统发生树每个节点的分化年代,同时计算出了凤蝶COI+COII的每年每位点的替换率为7.8×10-9~10.2×10-9,由此认为凤蝶COI+COII的进化速率比昆虫的“标准”速率[22]慢2-4倍。2008年,Pan等[10]利用凤蝶COI+COII的进化速率作为分子钟对蚕的进化年代进行了估算,结果表明家蚕与中国野桑蚕的分歧年代为1.08~1.41百万年。
COI/COII与其他一些线粒体基因和核基因结合对鳞翅目昆虫系统发育和分子进化的研究也是非常有用的工具之一。其他的线粒体基因或区域主要包括12S rRNA、16S rRNA、ND5及控制区;核基因则主要是EF-1α和Wg基因(表1)。2001年,Monteiro和Pierce[23]将COI、COII和EF-1α基因应用于Bicyclus属54个种的系统发育分析,他们分别建立了单基因和COI+COII+EF-1α基因连接序列的系统发育树,结果发现COI+COII+EF-1α基因连接序列的系统发育树与形态分类的结果较一致。在此后几年里,有更多的研究人员将COI/COII和EF-1α/Wg等结合起来研究鳞翅目昆虫一些种属间的关系(表1)。因此,mt基因与核基因结合也是研究昆虫系统发育和分子进化一个切实可行的方法。
2.1.2 ND5基因
ND5基因是线粒体中快速进化的基因之一,其基因长度也比较适合分子进化[24]。1999年,Yagi等[25]以ND5基因对日本凤蝶不同科的代表性物种进化关系进行研究时,发现凤蝶科Papilionidae与其他科形成不同的进化类群,而且在Papilionidae中分为两个亚族,这一结论与传统分类一致。2001年,Yagi等[26]又以ND5基因对日本群岛和亚洲大陆东部的凤蝶P.glacialis和P.stubbendorf ii的进化关系作了详细分析,发现这两个种起源于共同的祖先,其大致分歧年代在1.7~2.0 Mya。Omoto等[27]利用ND5基因主要对绢蝶Parnassius的进化关系作了探讨,结果表明该属为单系起源,其内部可以分为多个类群,即显示在进化过程中该物种发生了相对快速的扩散。2002年,Yukuhiro等[7]以甲虫ND5基因的进化速率(遗传距离为0.01,分化时间等于3.6百万年)作为分子钟,估算出了家蚕和日本野桑蚕分歧时间约为7.1百万年前。
除单独用于进化研究外,ND5基因也可结合其他mt基因同时用于鳞翅目昆虫分子系统学研究。2008年,Albre等[28]以ND5和COII基因对Erebia tyndarus种群的种和亚种的亲缘关系进行了研究,结果发现以ND5或COII的进化树拓扑结构基本一致,ND5+COII连接序列构建的进化树表明Erebia tyndarus种群可以分为两个不同的进化枝,且鉴别出了11个新种。因此ND5基因已被用于鳞翅目昆虫的亚种、种、属和科等分类阶元的分子系统学研究。
2.1.3 Cytb基因
2005年,Li等[29]利用Cytb基因探讨了蚕蛾属中的家蚕和野桑蚕的起源与进化关系,结果发现14个家蚕品种与日本野桑蚕Cytb基因核苷酸水平的差异为5.4%~5.8%,而与中国镇江野桑蚕的差异仅为0.8%~1.9%,进化树也显示家蚕与中国野桑蚕的亲缘关系更近,这与公认的家蚕起源于中国野桑蚕一致。陈永久等[30]测定了4种云南白马雪山和1种新疆天山的珍稀绢蝶Cytb基因部分序列,研究表明西猴绢蝶Parnassius simo分化较早,其次是珍珠绢蝶Parnassiusorleans和阿波罗绢蝶Parnassius apollo,巴裔绢蝶Parnassiusbaileyi和爱坷绢蝶Parnassius acco的遗传关系较近,且分化较晚,与形态学结果相吻合。
2.2 核糖体RNA基因
线粒体核糖体RNA分为大小两个亚基,其中16S rRNA应用于分子系统学的研究相对较为广泛。1999年,Hwang等[31]用12S和16S rRNA基因对蚕蛾科与大蚕蛾科的亲缘关系进行研究,分别以12S和16S rRNA构建的系统发生树的拓扑结构相同,进化树显示蚕蛾属与大蚕蛾属为单系起源。2008年,刘彦群等[32]以12S rRNA基因序列对鳞翅目柞蚕属、樗蚕属和桑蚕属等9种绢丝昆虫之间的系统发生关系进行了研究,从分子水平证实了所发现的野生柞蚕仍属于中国柞蚕种,进化分析支持柞蚕属、樗蚕属和桑蚕属的单系起源。Mahendran等[33]利用16S rRNA对大蚕蛾科Satumiidae和蚕蛾科14个不同物种的系统发生分析也佐证了大蚕蛾科和蚕蛾科呈单系起源的观点。2007年,Sobti等[34]以16S rRNA对6个北印度蝴蝶种的遗传关系研究发现,6个种按属聚为2个不同的类群,并指出结合rRNA与蛋白编码基因将能从不同的分类阶元上更好地理解鳞翅目昆虫的系统发生关系。另外,国内也有利用16S rRNA对蛱蝶科、凤蝶科内亚科主要类群进化关系的研究报道[35-36]。
目前,已有部分研究将rRNA与线粒体蛋白编码基因结合起来研究鳞翅目昆虫的进化关系。Aubert等[37]研究了凤蝶属(Papilio)种间的系统发生关系,当分别利用16S rRNA、ND1(仅密码子第一二位碱基)以及16S rRNA+ND1(仅密码子第一二位碱基)连接序列构建了系统发生树,发现16S rRN A+ND1的系统发生树最切合实际。另外,还有少量应用16S rRNA+ND1连接序列在蝴蝶种属分子进化的研究报道[38-39]。Wahlberg和Zimmermann[40]测定了蛱蝶科网蛱蝶族Melitaeini 13属77个物种的16S rRNA和COI基因的部分序列,系统发生树显示大多数前人所定义的属都是并系的,但发现Euphydryas、Phyciodes、Chlosyne和Melitaea为单系群,进化树并暗示melitaeines起源于新北区,然后曾三次扩散到新热带区和两次扩散到古北区。2004年,Lange等[41]利用16S rRNA和COII的部分序列对夜蛾科Noctuidae和螟蛾总科Pyraloidea 10属和6个族的26个种进行分子进化研究,结果发现夜蛾科为单系起源,而螟蛾总科呈并系发生;并且夜蛾科内Sesamia和Bathytricha也是单系发生,而且Busseola是Bathytricha发生的基础。
2.3 控制区
在过去的十年间,线粒体控制区已被广泛应用于动物群体遗传和分子进化等研究。但是昆虫控制区是否能用于分子系统学等研究一直争论不休[42]。2004年,Vila等[43]对控制区是否能用于鳞翅目昆虫分子进化研究进行了新的评价。他们对蛱蝶科的15个种进行系统发生分析时发现,控制区构建的系统发生树比用COI基因更能反映蛱蝶科内真实的起源进化关系。并进一步指出,在解决快速进化的种分类水平的系统发生关系时,CR+COI基因可能是一个非常有用的工具。2005年,Vila等[44]就以CR+COI基因序列对蛱蝶科Erebia triaria蝴蝶世系的初始分化与杂交进行了研究,其结论支持在第四纪期间伊比利亚半岛不是唯一的避难所,而是山脉与不同冰河时期的复杂分布促进了群体的隔离。
3 展望
mtDNA已被广泛应用于鳞翅目昆虫的近缘种、属、科甚至是目等分类阶元的系统发生分析和分子进化等研究。但是不同的mt基因进化速率不同,而且编码基因密码子的第三位碱基和控制区容易发生替换而达到饱和,加之外群选择不当,常会引起系统发生树长枝吸收效应直接影响分析的结果[45]。因此mt基因用于系统发育和分子进化等研究也有其适合的领域,在不同的物种中可能选择mt基因不同,在不同分类阶元也有各自适合的基因。通常亲缘关系较远的物种,一般选取比较保守的基因;亲缘关系较近的物种,则选取选择进化速率较快的序列。纵观鳞翅目的研究,COI和COII基因应用最广,并且结合线粒体其他基因或区段(ND5、12S rRNA、16S rRNA及CR)或核基因(EF-1α、Wg)等进行鳞翅目昆虫的分子系统学研究也有着不可替代的优势。相信mtDNA序列将在昆虫分子系统学研究中有着更为广阔的空间,并随着各物种进化研究的相继开展必将为濒危物种的保护,多样性的保持和种质资源的保存带来新的希望。
[1]Brower AV,DeSalle R.Patterns of mitochondrial versus nuclear DNA sequence divergence among nymphalid butterflies:the utility of wingless as a source of characters for phylogenetic inference[J].Insect Mol.Biol.,1998,7:73-82.
[2]Friedlander TP,Regier JC,Mitter C.Nuclear gene sequences for higher level phylogenetic analysis:fourteen promising candidates[J].Syst.Biol.,1992,41:483-490.
[3]Brown WM,Prager EM,Wang A,et al.Mitochondrial DNA sequences of primates:tempo and mode of evolution[J].J.Mol.Evol.,1982,18:225-239.
[4]DeSalle R,Freedman T,Prager EM,et al.Tempo and mode of sequence evolution in mitochondrial DNA of Hawaiian Drosophila[J].J.M ol.Evol.,1987,26:157-164.
[5]Pakendorf B,Stoneking M.Mitochondrial DNA and human evolution[J].Annu Rev Genomics Hum.Genet.,2005,6:165-183.
[6]鲁成,廖顺尧,刘运强,等.家蚕线粒体基因组全序列测定与分析[J].农业生物技术学报,2002,10:163-170.
[7]Yukuhiro K,Sezutsu H,Itoh M,et al.Significant levels of sequence divergence and gene rearrangements have occurred between the mitochondrial genomes of the wild mulberry silkmoth,Bombyx mandarina,and its close relative,the domesticated silkmoth,Bombyx mori[J].Mol.Biol.Evol.,2002,19:1385-1389.
[8]Hu XL,Cao GL,Xue RY,et al.The complete mitogenome and phylogenetic analysis of Bombyx mandarina strain Qingzhou[J].Mol.Biol.Rep.,2009(In press)
[9]Li D,Guo Y,Shao H,et al.Genetic diversity,molecular phylogeny and selection evidence of the silkworm mitochondria implicated by complete resequencing of 41 genomes[J].BMC Evol.Biol.,2010,10:81.
[10]Pan MH,Yu QY,Xia YL,et al.Characterization of mitochondrial genome of Chinese wild mulberry silkworm,Bomyx mandarina(Lepidoptera:Bombycidae)[J].Sci.China Ser.C-Life Sci.,2008,51:693-701.
[11]Yang L,Wei ZJ,Hong GY,et al.The complete nucleotide sequence of the mitochondrial genome of Phthonandria atrilineata(Lepidoptera:Geometridae)[J].Mol.Biol.Rep.,2008,36(6):1441-9
[12]DeChaine EG,Martini AP.Historic cycles of fragmentation and expansion in Parnassius smintheus(papilionidae)inferred using mitochondrial DNA[J].Evolution Int.J.Org.Evolution,2004,58:113-127.
[13]Rand DB,Heath A,Suderman T,et al.Phylogeny and life history evolution of the genus Chrysoritis within the Aphnaeini(Lepidoptera:Lycaenidae),inferred from mitochondrial cytochrome oxidase I sequences[J].Mol.Phylogenet.Evol.,2000,17:85-96.
[14]Hwang J,Lee JS,Goo TW,et al.The comparative molecular study between Bombycidae and Saturniidae based on mtDNA RFLP and cytochrome oxidase I gene sequences:implication for molecular evolution[J].Z.Naturforsch C,,1999,54:587-594.
[15]Behere GT,Tay WT,Russell DA,et al.Mitochondrial DNA analysis of field populations of Helicoverpa armigera(Lepidoptera:Noctuidae)and of its relationship to H.zea[J].BMC Evol.Biol.,2007,7:117.
[16]Long Y,Wan H,Yan F,et al.Glacial effects on sequence divergence of mitochondrial COII of Polyura eudamippus(Lepidoptera:Nymphalidae)in China[J].Biochem Genet,2006,44:361-77.
[17]Ohno S,Ishikawa Y,Tatsuki S,et al.Variation in mitochondrial COII gene sequences among two species of Japanese knotweed-boring moths,Ostrinia latipennis and O.ovalipennis(Lepidoptera:Crambidae)[J].Bull.Entomol.Res.,2006,96:243-9.
[18]王戎疆,万宏,龙玉,等.利用线粒体CO II基因序列对中国尾蛱蝶属系统分化的研究(鳞翅目:蛱蝶科)[J].昆虫学报,2004,47:243-247.
[19]Brown JM,Pellmyr O,Thompson JN,et al.Phylogeny of Greya(Lepidoptera:Prodoxidae),based on nucleotide sequence variation in mitochondrial cytochrome oxidase I and II:congruence with morphological data[J].Mol.Biol.Evol.,1994,11:128-141.
[20]Caterino MS,Sperling FA.Papilio phylogeny based on mitochondrial cytochrome oxidase I and II genes[J].Mol.Phylogenet.Evol.,1999,11:122-137.
[21]Zakharov EV,Caterino MS,Sperling FA.M olecular phylogeny,historical biogeography,and divergence time estimates for swallowtail butterflies of the genus Papilio(Lepidoptera:Papilionidae)[J].Syst.Biol.,2004,53:193-215.
[22]Beckenbach AT,Wei YW,Liu H.Relationships in the Drosophila obscura species group inferred from mitochondrial cytochrome oxidase II sequences[J].Mol.Biol.Evol,1993,10:619-634.
[23]M onteiro A,Pierce NE.Phylogeny of Bicyclus(Lepidoptera:Nymphalidae)inferred from COI,COII,and EF-1alpha gene sequences[J].Mol.Phylogenet.Evol.,2001,18:264-281.
[24]Clary DO,Wolstenholme DR The mitochondrial DNA molecule of Drosophila yakuba:nucleotide sequence,gene organization,and genetic code J M ol Evol.,1985,22:252-271.
[25]Yagi T,Sasaki G,Takebe H.Phylogeny of Japanese papilionid butterflies inferred from nucleotide sequences of the mitochondrial ND5 gene[J].J.Mol.Evol.,1999,48:42-48.
[26]Yagi T,Katoh T,Chichvarkhin A,et al.M olecular phylogeny of butterflies Parnassius glacialis and P.stubbendorfii at various localities in East Asia[J].Genes Genet.Syst.,2001,76:229-234.
[27]Omoto K,Katoh T,Chichvarkhin A,et al.M olecular systematics and evolution of the"Apollo"butterflies of the genus Parnassius(Lepidoptera:Papilionidae)based on mitochondrial DNA sequence data[J].Gene,2004,326:141-147.
[28]Albre J,Gers C,Legal L.Molecular phylogeny of the Erebia tyndarus(Lepidoptera,Rhopalocera,Nymphalidae,Satyrinae)species group combining CoxII and ND5 mitochondrial genes:a case study of a recent radiation[J].Mol.Phylogenet.Evol.,2008,47:196-210.
[29]Li A,Zhao Q,Tang S,et al.Molecular phylogeny of the domesticated silkworm,Bombyx mori,based on the sequences of mitochondrial cytochrome b genes[J].J.Genet.,2005,84:137-142.
[30]陈永久,张亚平,沈发荣,等.中国5种珍稀绢蝶非损伤性取样mtDNA序列及系统进化[J].遗传学报,1999,26:203-207.
[31]Hwang JS,Lee JS,Goo TW,et al.Molecular genetic relationships between Bombycidae and Saturniidae based on the mitochondria DNA encoding of large and small rRNA[J].Genet.Anal.,1999,15:223-8.
[32]刘彦群,靳向东,秦利,等.九种绢丝昆虫线粒体12S rRNA基因的序列特征和系统发育分析[J].昆虫学报,2008,51:307-314.
[33]Mahendran B,Ghosh SK,Kundu SC.Molecular phylogeny of silk-producing insects based on 16S ribosomal RNA and cytochrome oxidase subunit I genes[J].J.Genet.,2006,85:31-38.
[34]Sobti RC,Sharma VL,Kumari M,et al.Genetic relatedness of six North-Indian butterfly species(Lepidoptera:Pieridae)based on 16S rRNA sequence analysis[J].Mol.Cell.Biochem.,2007,295:145-51.
[35]陈娜,朱国萍,郝家胜,等.基于线粒体rDNA序列探讨蛱蝶科(鳞翅目,蝶亚目)主要分类群的系统发生关系[J].动物学报,2007,53:106-115.
[36]苏成勇,朱国萍,郝家胜,等.凤蝶亚科(凤蝶科,鳞翅目)16S rRNA基因的分子系统发生分析[J].动物分类学报,2007,32:335-342.
[37]Aubert J,Legal L,Descimon H,et al.Molecular phylogeny of swallowtail butterflies of the tribe Papilionini(Papilionidae,Lepidoptera)[J].Mol.Phylogenet.Evol.,1999,12:156-167.
[38]Katoh T,Chichvarkhin A,Yagi T,et al.Phylogeny and evolution of butterflies of the genus Parnassius:inferences from mitochondrial 16S and ND1 sequences[J].Zoolog.Sci.,2005,22:343-51.
[39]M artin J,Gilles A,Descimon H.Molecular phylogeny and evolutionary patterns of the european satyrids(Lepidoptera:Satyridae)as revealed by mitochondrial gene sequences[J].Mol.Phylogenet.Evol.,2000,15:70-82.
[40]Wahlberg N,Zimmermann M.Pattern of Phylogenetic Relationships among Members of the Tribe M elitaeini(Lepidoptera:Nymphalidae)Inferred from Mitochondrial DNA Sequences[J].Cladistics,2000,16:347-363.
[41]Lange CL,Scott KD,Graham GC,et al.Sugarcane moth borers(Lepidoptera:Noctuidae and Pyraloidea):phylogenetics constructedusing COII and 16S mitochondrial partial gene sequences[J].Bull.Entomol.Res.,2004,94:457-64.
[42]Zhang DX,Hewitt GM.Insect mitochondrial control region:A review of its structure,evolution,and usefulness in evolutionary studies[J].Biochem.Syst.Ecol.,1997,25:99-120.
[43]Vila M,Bjorklund M.The utility of the neglected mitochondrial control region for evolutionary studies in lepidoptera(insecta)[J].J.Mol.Evol.,2004,58:280-290.
[44]Vila M,Vidal-Romani JR,Bjorklund M.The importance of time scale and multiple refugia:incipient speciation and admixture of lineages in the butterfly Erebia triaria(Nymphalidae)[J].Mol.Phylogenet.Evol.,2005,36:249-60.
[45]Wheeler WC.Nucleic acid sequence phylogeny and random outgroups[J].Cladistics,1990,6:363-367.

