通用引物多重PCR技术检测3种病原微生物
商 颖,许文涛,,元延芳,梁志宏,石 慧,翟志芳,张雅楠,罗云波,,黄昆仑,,*
(1.中国农业大学食品科学与营养工程学院,北京 100083;2.农业部转基因生物食用安全监督检验测试中心(北京),北京 100083)
通用引物多重PCR技术检测3种病原微生物
商 颖1,许文涛1,2,元延芳2,梁志宏2,石 慧1,翟志芳1,张雅楠2,罗云波1,2,黄昆仑1,2,*
(1.中国农业大学食品科学与营养工程学院,北京 100083;2.农业部转基因生物食用安全监督检验测试中心(北京),北京 100083)
为寻找快速且准确的病原微生物检测方法,在普通多重PCR(polymerase chain reaction)技术的基础上研究一种新的通用引物多重PCR(universal primers-multiplex PCR,UP-M-PCR)技术。利用该技术对食品中主要的3种致病菌——大肠杆菌、单增李斯特菌和沙门氏菌进行同时检测,经过单重PCR验证、二重以及三重UP-PCR反应条件和体系优化。结果表明,该方法的最低检测限为0.5pg目标DNA,复合引物和通用引物的加入量分别为2nmol/L和300nmol/L。此方法检测灵敏度高、病原菌检测限低,可在实践中应用。
大肠杆菌;单增李斯特菌;沙门氏菌;通用引物;多重PCR
食品是人类赖以生存和发展的物质基础,食品安全关系到人体健康和国计民生,食源性致病菌检验更是食品安全检验的重中之重。近些年来,世界范围内大规模的生物性污染引起的食源性疾病爆发事件屡屡发生,如日本的出血性大肠埃希菌O157:H7污染、法国的李斯特菌中毒、美国的沙门氏菌属污染等,食品安全已成为世界性公共卫生问题,而以上3种菌也是存在于食物中并且导致食源性疾病的最主要病原菌[1]。
目前对食源性致病菌的检测仍主要依靠传统的细菌培养、血清学、生化鉴定等方法,这些方法不但检测周期长,而且操作繁琐复杂,特异性、灵敏度均较低,无法对人工难以培养的病原细菌进行检测[2-3],以上缺点都限制了传统检测方法的应用。随着现代免疫学和分子生物学理论和技术的不断发展,聚合酶链式反应(polymerase chain reaction,PCR)技术因其特异性强、灵敏度高、操作简单,而被广泛应用于食品病原细菌的检测中。由于单一PCR检测成本高,食品样品中往往存在的病原细菌种类很多,有可能涉及不同种和型别的细菌,人们进而开发了多重PCR技术[4-6]。多重PCR是在常规PCR基础上改进并发展起来的一种新型PCR扩增技术,是在同一反应体系中加入多对引物同时扩增多条目的DNA片段的方法[7-8]。但是,通常情况下由于引物的性质差异,每个特异性引物的扩增效率不同,扩增产物比例不一致,使得有些目的基因得不到清晰的扩增条带,有时也会出现假阳性[9]。
本研究在普通多重PCR的基础上,建立起一种通用引物——多重PCR技术(universal primer-multiplex PCR,PU-M-PCR),此技术可以克服普通多重PCR扩增效率不一致等的缺点,提高检测灵敏度,降低检测成本和检测限,从而为简便、快速、准确的检测病原微生物提供一种新方法。
1 材料与方法
1.1 菌株以及培养
沙门氏菌Salmonella spp.(strains CGMCC 1.0090和CGMCC 1.1552) 中国科学院微生物所;单增李斯特菌L. monocytogenes(strains CMCC 55004和CMCC 55007) 中国兽医药品监察所;非致病性大肠杆菌E. coli (BL21)和E. coli (JM109) 本实验室分离;E. coli O157:H7 EDL933中国预防医学科学院流行病微生物学研究所景怀奇研究员馈赠。
菌株冻干粉的活化条件:LB培养基(10g/L胰蛋白胨、5g/L酵母浸粉、10g/L NaCl和15g/L琼脂)或胰酶解大豆酪蛋白胨琼脂培养基(胰酶解大豆酪蛋白肉汤、18g/L琼脂)、37℃过夜培养。
活化后细菌的培养条件:选取单菌落于50mL LB液体培养基或500mL胰酶解大豆酪蛋白肉汤培养基,200r/min摇床培养20~22h。
1.2 基因组DNA提取
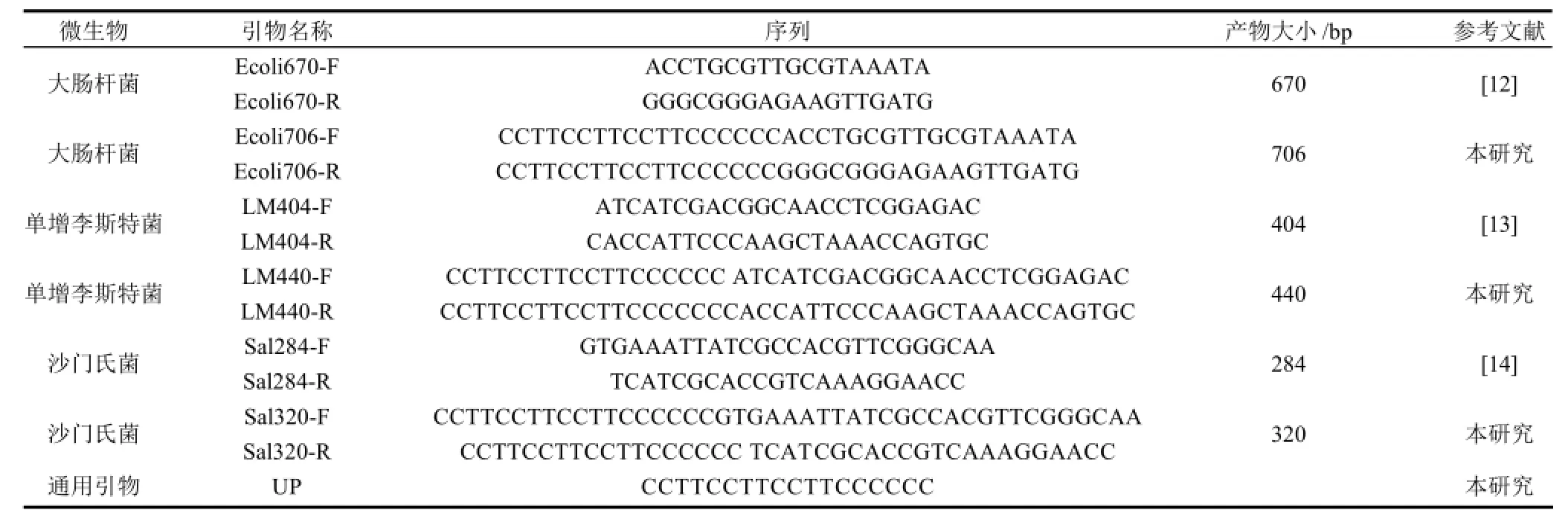
表1 引物序列Table1 Primer sequences used in this study
纯菌培养物基因组提取:2mL菌悬液(OD>1.8) 8000r/min离心5min,1.5mL PBS缓冲液洗涤沉淀并且重悬,反复两次。采用优化的CTAB法提取细菌基因组DNA[10-11]。
将基因组DNA溶于300μL TE缓冲液;采用Wizard DNA Purification Kit(美国普洛麦格公司)对DNA进行纯化;纯化后,用 Pico Green试剂对DNA进行荧光定量。特异性和灵敏性测试时,用双蒸水稀释目的基因组。
1.3 引物序列
大肠杆菌、单增李斯特菌和沙门氏菌的特异引物分别为Ecoli 670-F/R、LM404-F/R、Sal284-R/F[12-14]。UPM-PCR体系中,用于检测大肠杆菌、单增李斯特菌和沙门氏菌的复合引物在以上3种特异性引物5′端接上一段新序列——CCTTCCTTCCTTCCCCCC,此序列即为本研究新PCR体系中的通用引物UP。各引物序列如表1所示。
1.4 复合引物单重PCR特异性验证
复合引物是在普通特异性引物的5′端接上一段序列,其特异性与普通引物特异性是否一致,以及扩增产物片度大小是否相同有待于对比。在此部分的研究中同时进行两组对比实验,其中反应体系中,一组的引物为普通引物,另一组为复合引物,其余试剂均相同。热循环程序为:94℃变性5min;94℃、30s,56℃、30s,72℃、50s,35个循环;最后72℃延伸10min。PCR反应产物用2%(质量浓度)的琼脂糖凝胶电泳分离。
1.5 通用引物多重PCR(UP-M-PCR)反应原理及反应条件
与普通多重PCR体系相比,UP-M-PCR体系中除了含有3对目的基因复合特异性引物之外,还含有一对通用引物UP。在PCR反应的前10个循环,复合引物主要发挥作用,进行扩增,而通用引物UP由于没有目标模板,无法进行扩增。当特异性引物用尽,扩增产物得到一定程度的积累,通用引物开始以得到的产物为模板,进行序列扩增。本研究对此方法的可行性进行验证。
UP-M-PCR体系中,复合引物的特异性根据温度溶解曲线-SYBR GREENⅠPCR方法测试[11],并且采用单重、二重、三重PCR对3种引物的比例进行优化。由于PCR反应中,镁离子的浓度是影响扩增特异性的一个重要因素[15-17],因此,在UP-M-PCR体系中MgCl2含量为1.8mmol/L,比单重PCR含量稍高。优化后的体系为1.8mmol/L MgCl2、200μmmol/L dNTPs和1.5U DNA聚合酶、300nmol/L通用引物、2nmol/L每个复合引物。模板加入量从500、50、5、0.5、0.05pg依次进行测试,反应总体系为30μL,剩余体积用双蒸水补齐。
采用梯度PCR进行退火温度的优化,退火温度范围为56~64℃。最终热循环程序为:94℃变性5min;94℃、30s,60℃、30s,72℃、50s,35个循环;最后72℃延伸10min。
普通多重PCR中不含通用引物,除了特异引物的含量为300nmol/L,其他试剂量与UP-M-PCR体系相同。热循环程序为:94℃变性5min;94℃、30s,56℃、30s,72℃、50s,35个循环;最后72℃延伸10min。PCR反应产物用2%的琼脂糖凝胶电泳分离。
2 结果与分析
2.1 复合引物特异性验证
由于复合引物是在普通引物的5′端加了一段通用引物序列,因此通用引物扩增得到的产物片段较普通引物扩增的产物片段大36bp,所以2、3、4泳道的条带较5、6、7泳道的条带位置略低。此部分研究的结果如图1所示,每一对引物都得到相应特异性的扩增条带,且目的条带的亮度相似。说明复合引物与普通引物对目的片段的特异性一致。

图1 复合引物特异性验证Fig.1 Verification of the specificity of composite specific primers
2.2 通用引物单重PCR可行性验证
此部分的研究中,一组单重PCR反应体系中只加入复合引物,另一组反应体系除了加入复合引物外,还加入了通用引物,结果如图2、3所示。由图可知,当复合引物加入量降低到2nmol/L时,已经得不到扩增条带。而当复合引物量为2nmol/L,同时加入300nmol/L通用引物,得到的条带依然非常清晰,与图2第2泳道得到的扩增产物量相当。因此,为了优化UP-M-PCR反应体系,复合引物和通用引物的加入量即可分别定为2nmol/L和300nmol/L。

图2 复合引物单重PCR结果Fig.2 Composite specific primers in single PCR

图3 通用引物单重PCR可行性验证结果Fig.3 Feasibility of universal primers in single PCR
2.3 通用引物多重PCR可行性验证
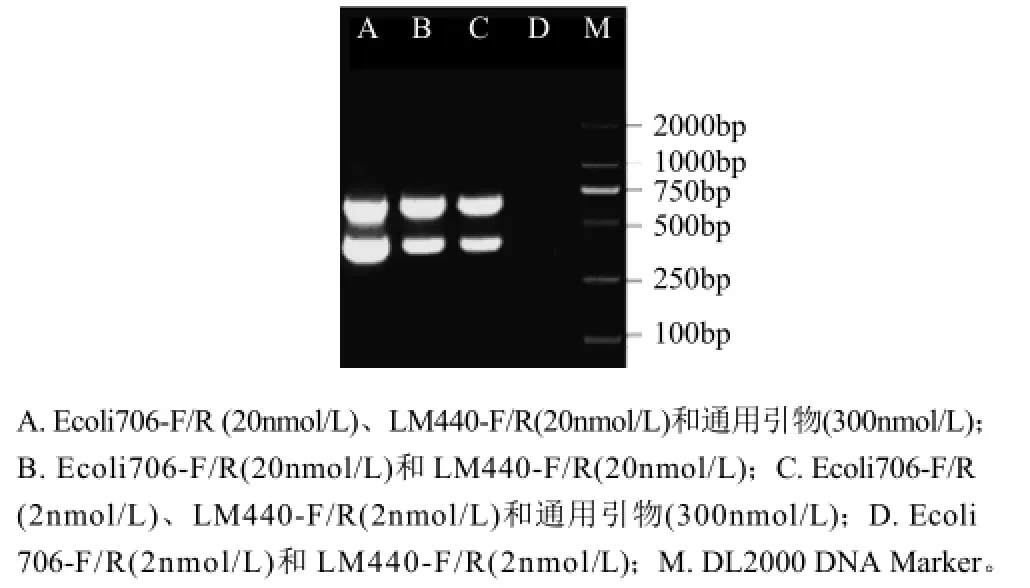
图4 通用引物二重PCR可行性验证结果Fig.4 Feasibility of universal primers in duplex PCR
在反应体系中逐渐减少复合引物的加入量,以优化反应条件。结果如图4所示,其中706bp和440bp为大肠杆菌O157和单增李斯特菌的特异性条带。由图可知,当两对复合引物的加入量都在2nmol/L时,已得不到目的条带;相反,当复合引物的加入量为2nmol/L,同时加入300nmol/L通用引物,依然可以得到非常清晰的目的条带。此结果与2.2节得到的结果一致。
2.4 UP-M-PCR特异性和灵敏度
为研究同时加入3对复合引物是否会产生非特异条带,进行4组多重UP-PCR。每组PCR反应体系包括3对复合引物各2nmol/L、通用引物300nmol/L、模板为3种致病菌基因组DNA的混合物。结果如图5所示,其中706、440bp和320bp分别为大肠杆菌O157、单增李斯特菌和沙门氏菌的特异性条带,每对引物的特异性很高,在整个体系中引物对之间同时存在,没有出现非特异性条带。

图5 UP-M-PCR特异性验证结果Fig.5 Specificity of the developed UP-M-PCR method

图6 UP-M-PCR灵敏度验证Fig.6 Sensitivity of the developed UP-M-PCR method
灵敏度测试结果如图6所示,扩增效率随模板量减少而降低,同时条带的清晰度也随之下降,当模板量减少至0.05pg时,已得不到目的条带。所以,UP-MPCR的检测限为0.5pg目标DNA。
3 讨 论
通用引物三重PCR体系中,反应的前10个循环,复合特异性引物主要发挥作用,进行扩增,而通用引物UP由于没有目标模板,无法进行扩增。当特异性引物用尽,扩增产物得到一定程度的积累,通用引物开始以得到的产物为模板,进行序列扩增,最终产生3个不同长度的目的片段。体系中,复合引物的加入量从200nmol/L减少到2nmol/L时,若不加通用引物,随着引物量的减少,扩增条带的清晰度依次降低,直至最后扩增不到扩增条带。虽然复合引物的加入量仅为2nmol/L,但通过加入通用引物UP,依然可以得到非常清楚的扩增条带。因此,复合引物的最佳反应浓度为2nmol/L,约为普通多重PCR反应体系中浓度的1%,而通用引物的浓度为正常浓度300nmol/L。因此,UP-MPCR体系中,总的引物含量较普通三重PCR体系更少,仅为其1/4,不仅可以节约复合引物消耗,而且通过通用引物消除不用复合引物之间的扩增性质差异。
在对微生物的检测中,提高灵敏度和降低检测限一直是学者们关系的问题,在本研究中,通过对模板加入量的研究得到:当混合模板中,其中有一种目标DNA减少到0.5pg时,依然可以扩增到清晰的条带,而且无非特异性条带出现。此方法技术降低了检测限,提高了检测的灵敏度,为简便、快速、准确的检测病原微生物提供和开辟了一种新方法和手段。对保障食品安全与国民健康具有重要意义。
[1]代娟, 李玉峰, 杨潇. 肠道致病菌多重PCR快速检测体系研究[J].中国公共卫生, 2007, 23(2): 219-220.
[2]张敬平, 吴家林, 肖勇, 等. 沙门菌、志贺茵和副溶血性弧菌的多重PCR快速检测技术的建立与应用[J]. 检验医学, 2008, 23(6): 642-645.
[3]李艳霞, 吴松浩. 食品微生物检测技术的研究进展[J]. 食品工业科技, 2008, 29(7): 270-273.
[4]许一平, 成炜, 陈福生. 多重PCR技术在食源性病原细菌检测中的应用[J]. 食品科学, 2007, 28(2): 355-359.
[5]彭军, 王国芬, 黄俊生, 等. 香蕉两种主要病毒多重PCR检测方法的建立[J]. 园艺学报, 2006, 33(4): 845-848.
[6]ELNIFRO E M, ASHSHI A M, COOPER R J, et al. Multiplex PCR: optimization and application in diagnostic virology[J]. Clin Microbiol Rev, 2000, 13(4): 559-570.
[7]焦豫良, 张兴群, 李振勇, 等. 6种食品致病菌的多重PCR检测[J].临床检验杂志, 2005, 23(4): 256-258.
[8]LUTZ E L, KLAUS P H, THOMAS E, et al. A multiplex real-time PCR assay for rapid detection and differentiation of 25 bacterial and fungal pathogens from whole blood samples[J]. Med Microbiol Immun, 2008, 197(3): 313-324.
[9]EDWARDS M C, GIBBS R A. Multiplex PCR: advantages, development, and applications[J]. Genome Res, 1994, 3(4): 65-75.
[10]RICH C, ALFIDJA A, SIROT J, et al. Identification of human enterovirulent Escherichia coli strains by multiplex PCR[J]. J Clin Lab Anal, 2001, 15(2): 100-103.
[11]MALORNY B, HOORFAR J, BUNGE C, et al. Multicenter validation of the analytical accuracy of Salmonella PCR: towards an international standard[J]. Appl Environ Microbiol, 2003, 69(1): 290-296.
[12]MCDANIELS A E, RICE E W, REYES A L, et al. Confirmational identification of Escherichia coli, a comparison of genotypic and phenotypic assays for glutamate decarboxylase andβ-D-glucuronidase[J]. Appl Environ Microbiol, 1996, 62(9): 3350-3354.
[13]WU S J, CHAN A, KADO C I. Detection of PCR amplicons from bacterial pathogens usingmicrosphere agglutination[J]. J Microbiol Methods, 2004, 56(3): 395-400.
[14]RAHN K S, de GRANDIS S A, CLARKE R C, et al. Amplification of an invA gene sequence of Salmonella enterica by polymerase chain reaction as a specific method of detection of Salmonella[J]. Mol Cell Probes, 1992, 6(4): 271-279.
[15]XU Wentao, HUANG Kunlun, WANG Ying, et al. A cotton-specific gene, stearoyl-ACP desaturase, used as a reference for qualitative and real-time quantitative polymerase chain reaction detection of genetically modified organisms[J]. J Sci Food Agric, 2006, 86(7): 1103-1109.
[16]XU Wentao, BAI Weibin, LUO Yunbo, et al. A novel common single primer multiplex polymerase chain reaction (CSP-M-PCR) method for the identification of animal species in minced meat[J]. J Sci Food Agric, 2008, 88(15): 2631-2637.
[17]MCPHERSON M J, MOLLER S G. Reagents and instrumentation. In PCR[M]. Oxford, UK: BIOS Scientific Publishers Ltd, 2000: 23-66.
Employing Universal Primers-Multiple PCR to Detect Three Pathogenic Microorganisms
SHANG Ying1,XU Wen-tao1,2,YUAN Yan-fang2,LIANG Zhi-hong2,SHI Hui1,ZHAI Zhi-fang1,ZHANG Ya-nan2,LUO Yun-bo1,2,HUANG Kun-lun1,2,*
(1. College of Food Science and Nutritional Engineering, China Agricultural University, Beijing 100083, China;2. Supervision, Inspection & Testing Center of Genetically Modified Food Safety, Ministry of Agriculture, Beijing 100083, China)
A fast, accurate and novel universal primers-multiplex PCR (UP-M-PCR) method was developed to detect three common pathogenic microorganisms in food matrices simultaneously: Escherichia coli, Listeria monocytogenes and Salmonella spp. The specificity of the constructed complex primers was verified by single-PCR, and the duplex and triplex-PCR reaction systems and conditions were optimized. The developed method had a limit of detection of 0.5 pg target DNA. The additions of the complex specific primers and universal primers were 2 nmol/L and 300 nmol/L, respectively. The new UP-M-PCR method has the advantages of high sensitivity and low detection limit and can thus be applied in practice.
Escherichia coli;Listeria monocytogenes;Salmonella spp.;universal primer;multiplex PCR (polymerase chain reaction)
Q789;R446.5
A
1002-6630(2011)10-0103-04
2010-06-22
国家“863”计划项目(2006AA10Z440);国家自然科学基金项目(30800770);科技部转基因生物重大专项(2008ZX08012-001)
商颖(1986—),女,硕士研究生,研究方向为食品安全。E-mail:cauxwt@yahoo.cn
*通信作者:黄昆仑(1968—),男,教授,博士,研究方向为食品安全。E-mail:hkl009@163.com

