转cry1Ac/cpti基因水稻对土壤氨氧化细菌群落组成和丰度的影响
宋亚娜,苏 军,林 艳,王 锋
福建省农业科学院生物技术研究所,福建省农业遗传工程重点实验室,福建福州350003
土壤氮素生物地球化学循环是土壤物质能量循环的重要组成部分,它能够影响土壤质量、氮素的植物吸收利用及农田等生态系统的生产力、可持续性及环境变化。硝化作用是土壤氮素循环的重要环节,对土壤氮素养分的转化具有重要意义。氨氧化细菌是执行硝化作用第一步的关键微生物,即将氨氧化为亚硝酸盐,该步骤也是硝化速率的限制性环节(Phillips et al.,2000)。虽然在水稻淹水时土壤处于厌氧条件而抑制硝化作用,但由于水稻根系能够分泌O2,为水稻根表和根际提供了充足的氧,足以支持非专一性的好氧微生物过程,而使水稻根际具有显著硝化作用的可能(Brune et al.,2000)。近年来的研究发现,稻田土壤中不仅存在丰富的氨氧化微生物基因资源,而且其群落组成和丰度变化活跃。土壤类型、水稻品种、施肥及土壤pH等环境因素均可改变氨氧化细菌的群落组成或丰度(宋亚娜等,2009;钟文辉等,2008;He et al.,2007;Nicol et al.,2008;Shen et al.,2008)。
在转基因水稻栽培中引入外源基因,有可能改变转基因作物的根系分泌物、根系及植株残体的组成及其降解过程,从而影响土壤物质能量的迁移转化过程,改变土壤微生物组成及土壤中矿质营养的转化循环(Masoero et al.,1999;Poerschmann et al.,2005、2008、2009;Saxena & Stotzky,2001)。目前,已有研究表明,转Bt基因水稻秸秆的降解对土壤细菌和真菌群落没有影响(Lu et al.,2010a、2010b;Wu et al.,2004)。但有关转基因水稻对与土壤养分转化密切相关的功能微生物(如氨氧化细菌)群落结构的影响还未见报道。
本研究通过3~4年的田间定位试验研究连续种植转cry1Ac/cpti基因水稻后土壤氨氧化细菌群落组成和丰度的变化,旨在为评价转基因水稻种植的土壤生态风险提供数据支持和理论依据。
1 材料与方法
1.1 材料
供试水稻是由福建省农业科学院农业遗传工程重点实验室研制的转基因杂交稻Ⅱ优科丰8号(GM)和非转基因杂交稻Ⅱ优明恢86(CK)。以三系水稻恢复系明恢86为受体,通过农杆菌介导法将双T-抗虫PCDMARUBAC载体中的cry1Ac(苏云金芽孢杆菌杀虫晶体蛋白基因)和cpti(修饰的豇豆胰蛋白酶抑制剂基因)2个抗虫基因导入水稻,获得能稳定遗传和表达的转基因恢复系科丰8号。以科丰8号的T8代纯合株系为父本、Ⅱ-32A为母本配制成转基因杂交稻Ⅱ优科丰8号。以转基因水稻科丰8号的受体恢复系明恢86为父本、Ⅱ-32A为母本配制获得非转基因杂交稻Ⅱ优明恢86。转基因杂交稻Ⅱ优科丰8号于2006年获得农业部转基因安全委员会批准进行生产性试验。
1.2 定位试验
2008年开始在具有防护措施的联动网室中建立水稻定位试验。地点位于福建省福州市郊的“吴凤水稻试验基地”(26°01' 北纬、119°20'东经)。该试验基地属暖湿亚热带季风气候,年无霜期长达326 d,年均气温19.6℃,平均湿度77%,年均降水量1342.5 mm。供试土壤为砂壤,土壤有机质含量为 23.32 g·kg-1,pH 6.15,全氮 1.65 g·kg-1,全磷(P2O5)0.76 g·kg-1,全钾(K2O)18.28 g·kg-1,速效氮 154.53 mg·kg-1,速效磷 31.34 mg·kg-1,速效钾 70.52 mg·kg-1。
于2008、2009、2010和2011年连续4年种植转cry1Ac/cpti基因抗虫水稻。挑选饱满的水稻种子于30℃催芽2 d,选发芽势强、整齐一致的种子播种,1个月后插秧,株行距为20 cm×20 cm,单本插。4年均作中稻栽培,5月播种,6月插秧,10月收获。前一年收获后留茬10 cm割稻,稻茬留田自然越冬。次年春季将稻茬翻入土中,于6月继续按前一年的小区排列方式种植。每种材料设3次重复,6个小区随机排列,每个小区种植水稻350株。定位试验期间除不使用杀虫剂外,水肥和田间管理措施等均按当地常规。
1.3 样品采集
分别于2010(定位试验第3年)和2011年(定位试验第4年)水稻分蘖期(播种后60 d)、齐穗期(播种后90 d)及成熟期(播种后120 d)采集土壤样品。采用五点取样法,每次重复取5株水稻,将水稻整株挖起后取根系周围分布密集的土层深度为10~20 cm的土壤,将5株水稻根系土壤混合为1个土样,放入4℃冰盒中保存。于实验室内去除土样根系、杂草和石块等杂质后混匀,分装入50 mL塑料离心管中,-20℃保存以用于土壤微生物分析。
1.4 分析方法
1.4.1 土壤微生物总DNA提取 称取0.5 g于-20℃保存的土壤样品,采用FastDNA®SPIN Kit For Soil(Q BIOgene)试剂盒进行土壤微生物总DNA的提取,将DNA样品于-20℃冰箱保存待用。
1.4.2 聚合酶链反应—变性梯度凝胶电泳(PCRDGGE)分析 利用氨氧化细菌16S rRNA基因特异引物(Song et al.,2007)进行 PCR 扩增(表 1),检测氨氧化细菌群落组成。反应体系50 μL,其中,2 μL稀释5倍的DNA模板加反应液48 μL,反应液包括1 μL Taq DNA 聚合酶(2.5 U· L-1,天根生物公司,北京),5 μL dNTPs(2 mmol·L-1各碱基,上海生物工程公司),5 μL 10×PCR-buffer(10倍 PCR缓冲液,天根生物公司,北京),前、后引物各4 μL(5 pmol· L-1,上海英骏生物工程公司)和 29 μL超纯水。PCR产物经琼脂糖电泳EB(溴化乙锭)染色检查后,用40 μL PCR产物进行DGGE分析,采用梯度为35%~50%的8%聚丙烯酰胺凝胶[化学变性剂为100%尿素7 mol·L-1和40%(v/v)去离子甲酰胺]在1倍TAE缓冲液中于150 V、60℃下电泳5 h。电泳后用10 mL SYBR green I(Sigma)(1倍 TAE稀释10000倍)核酸染料染色45 min,然后用Bio-Rad成像系统拍照。

表1 PCR扩增引物及反应条件Table 1 Primers and PCR conditions used for the study
1.4.3 氨氧化细菌的丰度检测 以土壤中氨氧化细菌16S rRNA基因拷贝数表示氨氧化细菌丰度。利用荧光定量PCR技术检测稻田土壤中氨氧化细菌16S rRNA基因数量。如表1所示,氨氧化细菌的荧光定量PCR扩增采用16S rRNA基因特异引物,每个样品进行3次重复。采用SYBR®Premix Ex TaqTM(TakaRa,大连)试剂盒于ABI PRISM7500 Real-Time PCR System扩增仪上进行绝对定量PCR分析。荧光定量 PCR反应体系25 μL,其中,2 μL稀释5倍的DNA模板加反应液23 μL,反应液包括12.5 μL SYBR®Premix Ex TaqTM(× 2),0.5 μL ROX Reference DyeⅡ (×50),前、后引物各 1 μL(5 pmol· L-1)和8 μL 超纯水。
1.4.4 荧光定量PCR标准曲线的建立 以非转基因水稻土样提取的DNA为模板进行氨氧化细菌16S rRNA基因的PCR扩增(表1)。将100 μL PCR产物用1.5%琼脂糖凝胶电泳,切下含有目的基因的片段约 465 bp,用 E.Z.N.A.Gel Extraction Kit(OMEGA,美国)胶回收试剂盒纯化。按照试剂盒方法用pMD18-T载体连接PCR产物,以大肠杆菌DH5α制备的感受态细胞转化连接产物,在氨苄青霉素平板上进行蓝白斑试验筛选阳性克隆。取部分阳性转化菌液送上海英骏生物工程有限公司测序,重组质粒测序结果经GenBank Blast比对,与氨氧化细菌16S rRNA基因(EU127347)同源性达99%。这表明重组质粒可以作为氨氧化细菌进行绝对荧光定量PCR分析的标准DNA。利用E.Z.N.A.Plasmid Mini Kit试剂盒提取重组质粒DNA,然后用 Nanodrop(Gene Co.Ltd.,美国)测定重组质粒DNA 的质量浓度,为99.0 ng·L-1。根据已知重组质粒全序列和阿伏伽德罗常数[6.02×1023(分子数)·moL-1]计算氨氧化细菌16S rRNA基因拷贝数,为5.86 ×1010(cells)·μL-1。以 10 倍梯度稀释氨氧化细菌16S rRNA基因重组质粒以进行荧光定量PCR检测,每个稀释浓度重复3次,获得氨氧化细菌16S rRNA基因标准曲线。
1.5 数据分析
采用Quantity One(Bio-Rad)软件对DGGE图谱中DNA条带的位置和亮度进行数字化;利用CANOCO 4.0(Microcomputer Power,Ithaca,USA)软件,根据不同处理DGGE结果的条带亮度和位置的数字化数值,以不同水稻材料及其生育期为环境因素对微生物群落结构组成进行冗余分析(redundancy discriminate analysis,RDA)(Marschner et al.,2002)。利用SPSS 13.0软件对数据进行方差分析。
2 结果与分析
2.1 转cry1Ac/cpti基因水稻对土壤氨氧化细菌群落组成的影响
利用氨氧化细菌16S rRNA基因特异引物对稻田土壤氨氧化细菌群落组成进行PCR-DGGE分析(图1)。由DGGE图谱可见,无论在定位试验第3年(图1A)还是第4年(图1B),稻田土壤中都存在丰富的氨氧化细菌基因,各个样品均呈现较多的DGGE条带。不同DGGE条带代表土壤中不同种类的氨氧化细菌,条带亮度表示在本研究的PCR试验条件下各类氨氧化细菌的相对丰度。
依据DGGE图谱的条带位置和亮度的数字化数值计算氨氧化细菌群落多样性指数(Shannonwiener index),结果如表2所示。在定位试验第3、4年,水稻各生育期内GM和CK的氨氧化细菌群落多样性指数均没有显著差异。这表明种植转cry1Ac/cpti基因水稻不会改变稻田土壤氨氧化细菌群落多样性。另外,同一种水稻的氨氧化细菌群落多样性指数在水稻不同生育期间的差异也不显著。
由图2可见,在定位试验第3、4年,变异较大的x轴方向上的氨氧化细菌群落结构分布差异主要表现在不同水稻生育期间,而同一生育期内不同水稻材料的氨氧化细菌群落结构分布较为接近。同时,经RDA显著性相关分析也显示,这2年的土壤氨氧化细菌群落组成的变化只与水稻生育期存在显著相关性,显著性水平分别为0.002和0.018。这表明稻田土壤氨氧化细菌群落组成随水稻生长而发生变化,但种植转cry1Ac/cpti基因水稻对土壤氨氧化细菌群落组成没有明显影响。

图1 定位试验第3年(A)和第4年(B)稻田土壤氨氧化细菌群落组成的DGGE图谱Fig.1 DGGE images of community composition of ammoniaoxidizing bacteria in paddy field in the third(A)and fourth(B)year after establishment of the experimental field site
2.2 转cry1Ac/cpti基因水稻对土壤氨氧化细菌群落丰度的影响
利用氨氧化细菌16S rRNA基因的重组质粒作为标准DNA建立了绝对荧光定量PCR的标准曲线(图 3)。其中,标准曲线的 R2为 0.99,斜率为 3.05,线性范围达到5个数量级,为5.86×103~5.86×107(cells)·μL-1。
依据标准曲线,用荧光定量PCR检测到不同处理的土壤氨氧化细菌16S rRNA基因拷贝数为1.66 ×106~1.85 ×107(copies)·g-1(干土)。在定位试验第3、4年,土壤氨氧化细菌的丰度在水稻生长前期均随水稻生长而增大,至水稻齐穗期时达到最大,之后逐渐减小(图4),GM和CK的氨氧化细菌丰度在齐穗期时均显著高于分蘖期(P<0.05);但在水稻生长过程中,氨氧化细菌丰度在GM和CK处理间的差异均未达到显著性水平(P>0.05)。由此可见,稻田土壤氨氧化细菌丰度受水稻生育期的影响较大,而转cry1Ac/cpti基因水稻对土壤氨氧化细菌丰度没有明显影响。

表2 定位试验第3、4年稻田土壤氨氧化细菌群落的多样性指数Table 2 The diversity of the ammonia-oxidizing bacterial community,characterised by the Shannon index of diversity,in the third and fourth year after establishment of the experimental field site

图2 定位试验第3年(A)和第4年(B)稻田土壤氨氧化细菌群落组成的冗余分析Fig.2 RDA of the community composition of ammonia-oxidizing bacteria in paddy field in the third(A)and fourth(B)year after establishment of the experimental field site
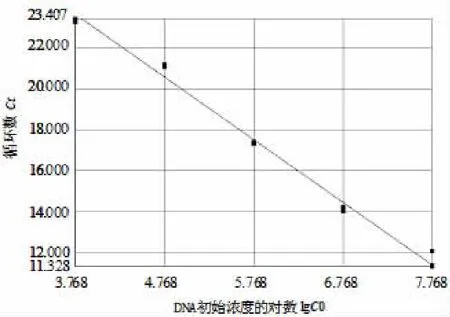
图3 氨氧化细菌16S rRNA基因绝对荧光定量PCR标准曲线Fig.3 Standard curve of real-time PCR of 16S rRNA gene of ammonia-oxidizing bacteria

图4 定位试验第3年(A)和第4年(B)稻田土壤氨氧化细菌的丰度Fig.4 Abundance of ammonia-oxidizing bacteria in paddy field in the third(A)and fourth(B)year after establishment of the experimental field site
3 讨论
目前,关于转基因作物对环境微生物影响的研究结果因作物种类、目标基因、微生物种类等不同而存在差异。在对转抗病基因木瓜的研究中发现,转基因品种的种植能够改变土壤真菌或细菌的群落组成(Wei et al.,2006)。而利用磷脂脂肪酸分析方法研究表明,土壤微生物群落结构在转Bt基因抗虫玉米和非转基因玉米间没有显著差异(Griffiths et al.,2005)。转Bt基因抗虫水稻的秸秆或根系还田对土壤细菌或真菌的群落组成也没有明显影响(Lu et al.,2010a、2010b)。转 Bt基因作物对土壤微生物没有影响可能是由于Bt蛋白在土壤中可快 速 降 解 (Daudu et al.,2009;Wang et al.,2006)。
现有的关于转Bt基因抗虫水稻对环境微生物群落影响的研究多集中于土壤总微生物如细菌或真菌的群落方面,而对一些与土壤养分转化更为密切相关的功能微生物的研究较少。本研究通过田间定位试验分析了转cry1Ac/cpti基因杂交稻Ⅱ优科丰8号连续种植第3、4年,对与土壤氮素转换密切相关的氨氧化细菌群落组成和丰度的影响。结果表明,土壤氨氧化细菌的群落组成和丰度均只与水稻的生长发育期相关,而在转cry1Ac/cpti基因水稻和非转基因水稻间没有显著差异。可见,田间种植转cry1Ac/cpti基因水稻对土壤氨氧化细菌多样性没有明显影响。
同时,本研究采用了PCR-DGGE和荧光定量PCR分子生物学方法。由于土壤中大部分微生物是不可培养的,用常规的培养方法研究土壤微生物群落的局限性很大。而以 DNA为基础的PCRDGGE方法能够较全面地反映土壤微生物组成,荧光定量PCR方法更可准确检测土壤微生物的丰度。另外,本研究通过连续4年的田间定位试验,所得结果能更好地反映种植转基因水稻对土壤微生物的影响。本研究证明,在一定时期内种植转cry1Ac/cpti抗虫基因水稻不会产生影响土壤氨氧化细菌群落组成和丰度的生态风险。
此外,由于土壤中微生物种类繁多、数量庞大等多样性特征有利于保证其群落的相对稳定,使得转基因作物种植对土壤微生物多样性的干扰可能在一定时期内不会表现。因此仍需要通过长期定位试验进一步监测种植转cry1Ac/cpti基因水稻对土壤各类微生物多样性及其生态功能的影响,从而综合评价种植抗虫转基因水稻对稻田土壤生态系统的安全性。
宋亚娜,林智敏,林捷.2009.不同品种水稻土壤氨氧化细菌和氨氧化古菌群落结构组成.中国生态农业学报,17(6):1121-1125.
钟文辉,蔡祖聪,尹力初,张鹤.2008.种植水稻和长期施用无机肥对红壤氨氧化细菌多样性和硝化作用的影响.土壤学报,45(1):105-111.
Brune A,Frenzel P and Cypionka H.2000.Life at the oxicanoxic interface:microbial activities and adaptations.FEMS Microbiology Reviews,24:691 -710.
Daudu C K,Muchaonyerwa P and Mnkeni P N S.2009.Litterbag decomposition of genetically modified maize residues and their constituent Bacillus thuringiensis protein(Cry1Ab)under field conditions in the central region of the Eastern Cape,South Africa.Agriculture,Ecosystems & Environment,134:153-158.
Griffiths B S,Caul S,Thompson J,Birch A N E,Scrimgeour C,Andersen M N,Cortet J,Messean A,Sausse C,Lacroix B and Krogh P H.2005.A comparison of soil microbial community structure,protozoa and nematodes in field plots of conventional and genetically modified maize expressing the Bacillus thuringiens is Cry1Ab toxin.Plant and Soil,275:135-146.
He J Z,Shen J P,Zhang L M,Zhu Y G,Xu M G and Di H.2007.Quantitative analyses of the abundance and composition of ammonia-oxidizing bacteria and ammonia-oxidizing archaea of a Chinese upland red soil under long-term fertilization practices.Environmental Microbiology,9(9):2364 -2374.
Lu H,Wu W,Chen Y,Wang H,Devare M and Thies J E.2010a.Soil microbial community responses to GM transgenic rice residue decomposition in a paddy field.Journal of Soils and Sediments,10:1598 -1605.
Lu H,Wu W,Chen Y,Zhang X,Devare M and Thies J E.2010b.Decomposition of GM transgenic rice residues and response of soil microbial community in rapeseed-rice cropping system.Plant and Soil,336:279-290.
Marschner P,Neumann G,Kania A,Weiskopf L and Lieberei R.2002.Spatial and temporal dynamics of the microbial community structure in the rhizosphere of cluster roots of white lupin(Lupinus albus L.).Plant and Soil,246:167-174.
Masoero F,Moschini M,Rossi F,Prandini A and Pietri A.1999.Nutritive value,mycotoxin contamination and in vitro rumen fermentation of normal and genetically modified corn(cry1Ab)grown in northern Italy.Maydica,44(3):205 -209.
Nicol G W,Leininger S,Schleper C and Prosser J I.2008.The influence of soil pH on the diversity,abundance and transcriptional activity of ammonia oxidizing archaea and bacteria.Environmental Microbiology,10:2966-2978.
Phillips C J,Harris D,Dollhopf S L,Gross K L,Prosser J I and Paul E A.2000.Effects of agronomic treatments on structure and function of ammonia-oxidizing communities.Applied and Environmental Microbiology,66:5410-5418.
Poerschmann J,Gathmann A,Augustin J,Langer U and Górecki T.2005.Molecular composition of leaves and stems of genetically modified GM and near-isogenic non-GM maize—characterization of lignin patterns.Journal of Environmental Quality,34(5):1508-1518.
Poerschmann J,Rauschen S,Langer U,Augustin J and Górecki T.2008.Molecular level lignin patterns of genetically modified GM-maize MON88017 and three conventional varieties using tetramethylammonium hydroxide(TMAH)-induced thermochemolysis.Journal of Agricultural and Food Chemistry,56(24):11906 -11913.
Poerschmann J,Rauschen S,Langer U,Augustin J and Górecki T.2009.Fatty acid patterns of genetically modified Cry3Bb1 expressing GM-maize MON88017 and its near-isogenic line.Journal of Agricultural and Food Chemistry,57(1):127-132.
Saxena D and Stotzky G.2001.GM corn has a higher lignin content than non-GM corn.American Journal of Botany,88:1704-1706.
Shen J P,Zhang L M,Zhu Y G,Zhang J P and He J Z.2008.Abundance and composition of ammonia-oxidizing bacteria and ammonia-oxidizing archaea communities.Environmental Microbiology,10:1601-1611.
Song Y N,Mrschner P,Li L,Bao X G,Sun J H and Zhang F S.2007.Community composition of ammonia-oxidizing bacteria in the rhizosphere of intercropped wheat(Triticum aestivum L.),maize(Zea mays L.)and faba bean(Vicia faba L.).Biology and Fertility of Soils,44(2):307 -314.
Wang H Y,Ye Q F,Wang W,Wu L C and Wu W X.2006.Cry1Ab protein from GM transgenic rice does not residue in rhizosphere soil.Environment Pollution,143:449 -455.
Wei X D,Zou H L,Chu L M,Liao B,Ye C M and Lan C Y.2006.Field release transgenic papaya effect on soil microbial communities and enzyme activities.Journal of Environmental Sciences,18(4):734 -740.
Wu W,Ye Q,Min H,Duan X and Jin W.2004.GM-transgenic rice straw affects the culturable microbiota and dehydrogenase and phosphatase activities in a flooded paddy soil.Soil Biology Biochemistry,36:289-295.

