蛋白质组学在乳酸菌应激反应机制研究中的应用
乌日娜,孙志宏,张文羿,陈永福,刘文俊,钟 智,张和平,*
(1.沈阳农业大学食品学院,辽宁 沈阳 110866;2.内蒙古农业大学 乳品生物技术与工程教育部重点实验室,内蒙古 呼和浩特 010018)
蛋白质组(proteome)是指由一个基因组所表达的全套蛋白质[1]。研究细胞内全部蛋白质的存在及其活动方式的学科即为蛋白质组学(proteomics)。其本质上指的是,在大规模水平上研究蛋白质的特征,包括蛋白质的表达水平、翻译后的修饰、蛋白与蛋白相互作用等,由此获得蛋白质水平上的关于疾病发生、细胞代谢等过程的整体而全面的认识[2]。随着人类基因组测序的完成,后基因组时代随之来临,蛋白质组学作为后基因组研究的主要工具,被列为世界六大研究热点之一。
乳酸菌是一类发酵葡萄糖主要产生乳酸的革兰氏阳性杆菌或球菌的总称[3]。乳酸菌不仅应用于乳制品、蔬菜及肉制品的生产,而且作为益生菌,在工业和医药等重要领域也具有很高的应用价值。研究显示[4],外界环境胁迫可诱导乳酸菌产生应激反应,如在生产和贮藏过程中的酸胁迫、渗透压胁迫和冷胁迫等,可诱导不同种类和数目的蛋白质表达发生变化。通过从蛋白质组水平揭示乳酸菌在环境胁迫下蛋白质表达模式的动态变化,有助于阐明乳酸菌应激反应调节的分子机制,从而为选育和改造优良菌种奠定基础。
1 应激蛋白质
应激蛋白质,通常可以由几种胁迫共同诱导,主要参与DNA或蛋白质的修复。其种类包括热休克蛋白质、冷休克蛋白质和通用应激蛋白质等。
热休克蛋白质(heat shock proteins,HSPs)是较早发现的一类应激蛋白质,属分子伴侣蛋白质,具有修复或解折叠损伤蛋白质的生物学功能,主要包括有HSP70(DnaK)、HSP60(GroEL)、HSP85和HSP100等[5]。蛋白质组研究发现,多种环境胁迫均可诱导乳酸菌热休克蛋白质的表达发生变化。由于胁迫条件不同和乳酸菌种属不同等原因,各种胁迫诱导乳酸菌表达的热休克蛋白质的种类可能有所不同(表1)。其中,DnaK和GroEL是目前报道较多的可能与乳酸菌胁迫应激反应相关的热休克蛋白质。此外,小分子热休克蛋白质sHSP近来也受到了广泛关注。有关益生菌干酪乳杆菌Lactobacillus caseiZhang的研究就报道了小分子HSP20可由胆盐胁迫诱导表达增强,也与其稳定期的耐受性提高有一定关系[6-7]。
据报道[22],当温度急剧降低时,乳酸菌的绝大多数蛋白质的合成会受到抑制,但冷应激蛋白质却急剧增加,其中包括一些小分子质量的酸性蛋白质即冷休克蛋白(cold shock proteins,CSPs)。这些蛋白质对维持细胞活力及功能非常重要。以乳酸乳球菌(Lactococcus lactis)为例,目前发现L.lactis具有冷休克蛋白质Csp A-G[23]。利用双向电泳技术研究发现,Csp A-D及Csp F可以由冷胁迫诱导表达发生变化,而Csp E的变化较小,进而在转录水平的研究也发现了相同的表达趋势[24-25]。嗜热链球菌Streptococcus thermophilus中也具有CSPs蛋白质(Csp A-F),其中研究显示Csp A-E在冷胁迫下表达会有所增强[26]。
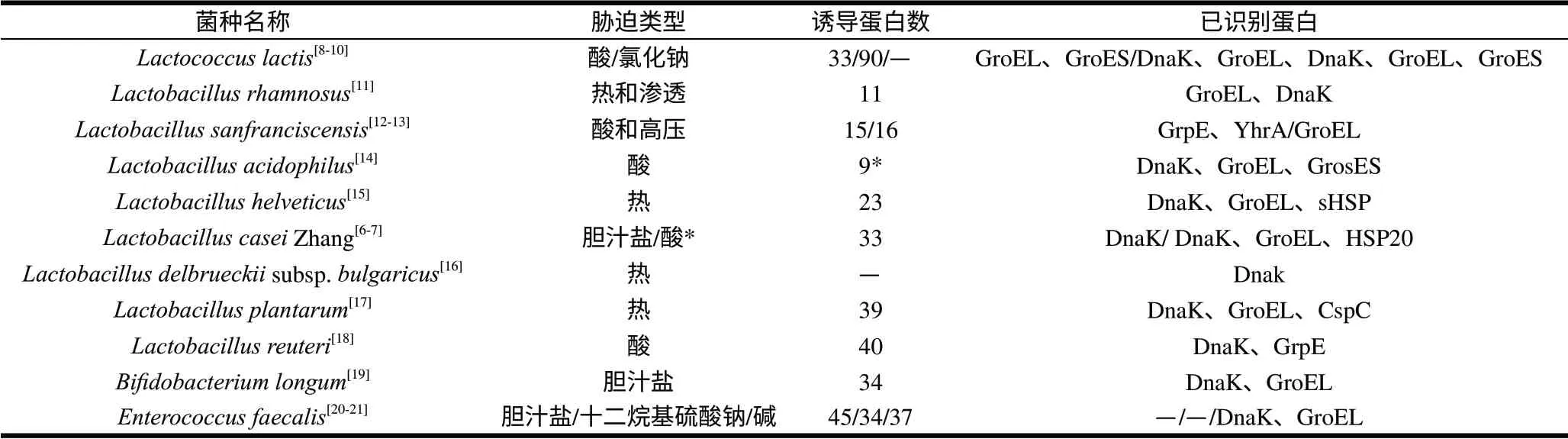
表1 利用蛋白质组学技术发现环境胁迫下乳酸菌HSPs蛋白质表达变化汇总Table 1 Summary of changes of protein expression of HSPs in lactic acid bacteria subjected to environmental stress using proteomics
除以上常见的应激蛋白质外,在L.lactis中还发现了ClpX ATPase(45kD)可能参与该菌株在冷胁迫后修复蛋白损伤[27];2个Clp protease在旧金山乳杆菌(Lactobacillus sanfranciscensis)受到高压胁迫时表达也会增强[13]。而通用应激蛋白质UspA可能与益生菌L.caseiZhang的酸应激胁迫有关[7]。
在2004年Desmond等[28]曾利用乳链菌肽构建了两个质粒pGRO1和pGRO2,且含有副干酪乳杆菌(Lactobacillus paracasei)NFBC338的groESL操纵子,将该质粒分别转入L.lactis(pGRO1)和L.paracasei(pGRO2)中,可使热应激蛋白质GroESL过量表达,结果表明,两株菌的耐渗透压和耐热性都有不同程度的增强。因此,将各种HSPs基因引入不同宿主,可能是提高乳酸菌菌株对一种或多种胁迫耐受性的有效方法之一。
2 代谢途径的调控
从目前报道的乳酸菌参考图谱的分析来看,在所有鉴定的蛋白质中,依据功能分类,糖代谢相关酶类所占比例最高,约25%~30%,说明这类蛋白质在菌体生长中起到重要作用。其次为能量代谢相关蛋白质、翻译和核糖体蛋白质及分子伴侣蛋白质等。
糖代谢是为乳酸菌生长提供能量的主要途径。在某些外界不利环境条件胁迫下,乳酸菌的生长和维持活力所消耗能量会增加,所以糖代谢途径中的某些酶类表达就可能发生变化[29]。Guillot等[30]构建了乳酸乳球菌L.1actisⅡ 1403和L.1actisNCDO763分别在乳糖和葡萄糖培养基中生长的蛋白质组,比较发现不同的碳源可诱导参与乳酸乳球菌糖酵解的相关蛋白酶差异表达,如与含有葡萄糖培养基中生长相比,L.1actis在乳糖培养基中生长时,其塔格糖磷酸激酶等塔格糖途径代谢酶表达会明显上调。在环境胁迫乳酸菌蛋白质组研究中,糖代谢相关酶类的表达变化可能对提高乳酸菌的耐受性起到重要作用。例如,酸胁迫可分别诱导双歧杆菌(Bifidobacterium longum)中糖代谢相关酶类磷酸葡萄糖变位酶(phosphoglucomutase)、丙酮酸激酶(pyruvate kinase)、磷酸尿苷葡糖异构酶(UDP-glucose-4-epimerase)和β-半乳糖苷酶(β-galactosidase)等,以及罗伊氏乳杆菌(Lactobacillus reuteri)中的丙酮酸激酶(pyruvate kinase)、磷酸甘油酸变位酶(phosphoglycerase mutase)、麦芽糖磷酸化酶(maltose phosphorylase)、甘油-3-磷酸脱氢酶(glycerol-3-phosphate dehydrogenase)等参与糖代谢的蛋白质表达发生明显变化[18,31]。
除糖代谢途径,不同的环境胁迫还可以诱导氨基酸代谢途径相关酶类调控变化。例如,胆盐胁迫可以诱导干酪乳杆菌(L.caseiZhang)的半胱氨酸激酶(cysteine kinase,CysK)的蛋白质表达水平增强,且qRT-PCR结果也显示其相应基因CysK在mRNA水平也同样上调[6]。半胱氨酸在细菌中可参与硫氧还原蛋白和谷胱甘肽等蛋白质的合成,而硫氧还原蛋白和谷胱甘肽蛋白具有抗氧化作用。因此,可能说明了L.caseiZhang在受到胆盐压力的同时伴有氧化还原应激反应的产生。而费氏丙酸杆菌Propionibacterium freudenreichii处于胆盐环境压力下时,其CysK酶在蛋白质水平表达也是增强的[32]。
在不同的培养条件下,如将嗜热链球菌S.thermophilus接种于牛乳或化学培养基中生长时,参与氨基酸和核酸代谢的蛋白质的表达会有所不同,尤其是在生长初期,菌体在牛乳中生长时核酸代谢途径相关酶类的表达水平要强于菌体在化学培养基中生长的表达水平[33]。其原因可能是牛乳中缺乏嘌呤碱基,需要S.thermophilus从头合成脱氧核糖核酸。所以,参与核酸代谢的酶类表达量会增强。因此,利用双向电泳发现,PurC、PurL、PurH、PurD、PurB、PurA、PurR及PyrC的表达在S.thermophilus对数生长期的早期和晚期表达有差异。
此外,参与转录和翻译代谢调控的蛋白质,如翻译因子等,也是较为常见的乳酸菌在环境胁迫下诱导的应激蛋白质[7]。
总之,蛋白质组学研究表明,环境胁迫促使乳酸菌产生全面而复杂的应激反应,涉及不同的代谢调控路径,共同对乳酸菌在不同环境条件下的存活和生长能力产生影响。
3 细胞壁和细胞膜的保护作用
细胞壁和细胞膜是细菌应对各种不利环境的第一道屏障,其功能不仅可以维持细胞形态,而且在细胞内外渗透压、pH值、能量代谢和物质运输等调控时起到重要作用。
F0F1-ATPase为细胞膜结合蛋白酶,在细胞膜上催化ATP的合成和水解及跨膜的质子梯度的互相交换[34]。酸胁迫研究发现,当嗜酸乳杆菌(Lactobacillus acidophilus)处于低pH值环境下时,ATPase的表达量上调,可能是因为F0F1-ATPase与能量代谢相关,通过消耗胞体内的ATP,将胞内质子(H+)泵出胞外,以维持胞内一定的pH值[35]。而乳酸菌酸胁迫蛋白质组学研究同样发现,双歧杆菌(Bif idobacterium longum)的ATPase的表达在酸胁迫后有所增强[31]。
N-乙酰氨基葡萄糖-6-磷酸脱醛酶(N-acetylgluco samine-6-phosphate deacetylase,NagA)可催化氨基糖类的代谢,以及催化N-乙酰-D-6-磷酸葡萄糖胺与D-6-磷酸果糖的转化,而N-乙酰-D-6-磷酸葡萄糖胺是N-乙酰-D-葡萄糖胺和UDP-N-乙酰胞壁酸的前体,所以,NagA可能与细菌细胞壁肽聚糖和磷壁酸的合成有密切关系[36]。干酪乳杆菌(L.caseiZhang)的酸胁迫蛋白质组研究发现,NagA的表达量在(L.caseiZhang)受到酸胁迫时所增强,可能有利于维持细胞形态,提高细胞壁的保护作用[37]。
乳酸乳球菌(L.lactis)碱性参考蛋白质组图谱的构建显示,在碱性区间即pH6~12范围内发现了如D-丙氨酸羧肽酶(D-alanyl-D-alanine carboxypeptidase)、甘油-3-磷酸胞苷酰基转移酶(glycerol-3-phosphatecytidiltransferase)、N-乙酰胞壁质酶(N-acetylmuramidase)和肽聚糖合成酶(peptidoglycan synthesis protein,MurG)等与细胞外膜代谢相关的蛋白质[38]。但由于碱性区间的蛋白质组图谱的构建有一定难度,这在一定程度上也阻碍了乳酸菌细胞壁、细胞膜组成和代谢相关蛋白质表达规律的研究。此外,有研究报道,环境胁迫通常会引起细胞膜脂肪酸组成发生改变[39]。
4 适应性反应机制
当细胞先经过温和的环境胁迫后,一般可诱导一种适应性反应,从而对细胞起到保护作用,使细胞在面临更强的环境胁迫时,有良好的抵御能力,有利于细胞生存,这就是所谓的适应性反应[40]。酸适应性反应(acid tolerance response,ATR)是应激保护作用的常见方式之一。例如,德氏乳杆菌(Lactobacillus delbruekii)对数生长期细胞对65℃持续10min敏感,细胞恢复率0.0001%~0.015%,当将对数生长期细胞先在50℃处理30min,再置于致死温度时,菌株的生存力增加了10~1000倍[41]。双向电泳研究显示,当旧金山乳杆菌(L.sanfranciscensis)CB1经过酸耐受性ATR反应后,有63种蛋白质的表达水平发生明显变化,以帮助其在更低酸性胁迫下保持活力[12]。
此外,乳酸菌生长至对数生长期时,所处pH值是非致死的酸性环境且营养比较充分,为乳酸菌进入稳定期提供了一个逐渐适应的过程,以激发其稳定期全面的应激反应,使稳定期的细胞比对数期细胞对酸、热、营养缺陷等环境胁迫更具有抵抗力,即为对数生长期适应性反应(lg-ATR)。植物乳杆菌(Lactobacillus plantarum)不同生长阶段蛋白质组学研究显示,在其生长至稳定期初期,蛋白质合成的数量要远多于对数生长期早期的合成数目[42]。相对于对数生长期,嗜酸乳杆菌(L.acidophilus)CRL 639在稳定期表达量增强的16种蛋白质中,有9种蛋白质随酸度的降低而被诱导表达,Lorca等[14]认为这些蛋白质的表达改变与其ATR反应有密切关系。因此,在乳酸菌的适应过程中,可能通过诱导蛋白质表达发生变化,以提高其对致死环境胁迫的抵抗能力。
5 结 语
利用蛋白质组学相关技术和方法对各种环境胁迫下乳酸菌差异表达蛋白质的深入研究,揭示了乳酸菌响应复杂的应激反应,调控不同的代谢路径相关蛋白质的表达,提高其在环境胁迫下的生存能力和活力。乳酸菌蛋白质组研究是乳酸菌后基因组学系统研究的重要组成部分,也已取得了一定的进展,未来蛋白质组与转录组、代谢组等在乳酸菌研究中的综合应用,将为我们提供更充实的数据和广阔的应用前景。
[1]WILKINS M R, SANCHEZ J C, GOOLEY A A, et al.Progress withproteome projects∶ why all proteins expressed by a genome should be identified and how to do it[J].Biotechnol Genetic Engineering Rev,1996, 13∶ 19-50.
[2]贺福初.蛋白质组学研究∶ 后基因组时代的生力军[J].科学通报,1999, 44(2)∶ 113-122.
[3]金世琳.乳酸菌的科学与技术[J].中国乳品工业, 1998, 26(2)∶ 14-16.
[4]CHAMPOMIET-VERGES M C, MAGUIN E, MISTOU M Y,et al.Lactic acid bacteria and proteomics∶ current knowledge and perspectives[J].J Chromatography, 2002, 771∶ 329-342.
[5]ANG D, LIBEREK K, SKOWYR D, et al.Biological role and regulation of the universally conserved heat shock proteins[J].J Biol Chem, 1991, 266∶ 24233-24236.
[6]WU Rina, SUN Zhihong, WU Junrui, et al.Effect of bile salts stress on protein synthesis ofLactobacillus caseiZhang revealed by twodimensional gel electrophoresis[J].J Dairy Science, 2010, 93∶ 3858-3868.
[7]WU Rina, WANG Weiwei, YU Dongliang, et al.Proteomics analysis ofLactobacillus caseiZhang, a new probiotic bacterium isolated from traditionally home-made koumiss in Inner Mongolia of China[J].MCP, 2009, 8(10)∶ 2321-2338.
[8]HARTKE A, BOUCHE S, GIARD J C, et al.The lactic acid stress response ofLactococcus lactissubsp.lactis[J].Curr Microbiol, 1996, 33∶ 194-199.
[9]BUDIN-VERNEUIL A, VIANNEY P, YANICK A, et al.Proteomic characterization of the acid tolerance response inLactococcus lactisMG1363[J].Proteomics, 2005, 5∶ 4794-4807.
[10]KILSTRUP M, JACOBSEN S, HAMMER K, et al.Induction of heat shock proteins DnaK, GroEL, and GroES by salt stress inLactococcus lactis[J].Appl Environ Microbiol, 1997, 63(5)∶ 1826-1837.
[11]PRASAD J, MCJARROW P, GOPAL P.Heat and osmotic stress responses of probioticLactobacillus rhamnosusHN001 (DR20) in relation to viability after drying[J].Appl Environ Microbiol, 2003,69(2)∶ 917-925.
[12]de ANGELIS M, GOBBETTI M, BINI L, et al.The acid-stress response inLactobacillus sanfranciscensisCB1[J].Microbiol, 2001,147∶ 1863-1873.
[13]HORMANN S, SCHEYHING C, BEHR J, et al.Comparative proteome approach to characterize the high-pressure stress response ofLactobacillus sanfranciscensisDSM 20451T[J].Proteomics, 2006, 6∶ 1878-1885.
[14]LORCA G L, VALDEZ G F.A low-pH inducible, stationary-phase acid-tolerance response inL.acidophilusCRL639[J].Curr Microbial,2001, 42∶ 21-25.
[15]di CAGNO R, de ANGELIS M, LIMITONE A, et al.Response ofLactobacillus helveticusPR4 to heat stress during propagation in Cheese whey with a gradient of decreasing temperatures[J].Appl Environ Microbiol, 2006, 72(7)∶ 4503-4514.
[16]GOUESBET G, JAN G, BOYAVAL P.Two-dimensional electrophoresis study ofLactobacillus delbrueckiisubsp.bulgaricusthermotolerance[J].Appl Environ Microbiol, 2002, 68(3)∶ 1055-1063.
[17]de ANGELIS M, di CAGNO R, HUET C, et al.Heat shock response inLactobacillus plantarum[J].Appl Environ Microbiol, 2004, 70(3)∶ 1336-1346.
[18]LEE K, LEE H G, PI K, et al.The effect of low pH on protein expression by probiotic bacteriumLactobacillus reuteri[J].Proteomics,2008, 8∶ 1624-1630.
[19]SANCHEZ B, CHAMPOMIER-VERGES M, ANGLADE P, et al.Pteomic analysis of global changes in protein expression during bile salt exposure ofBif idobacterium longumNCIMB 8809[J].J Bacteriol,2005, 187∶ 5799-5808.
[20]FLAHAUT S, FRERE J, BOUTIBONNES P, et al.Comparison of the bile salts and sodium dodecyl sulfate stress responses inEnterococcus faecalis[J].Appl Environ Microbiol, 1996, 62(7)∶ 2416-2420.
[21]FLAHAUT S, HARTKE A, GIARD J C, et al.Alkaline stress response inEnterococcus faecalis∶ adaptation, cross-protection, and changes in protein synthesis[J].Appl Environ Microbiol, 1997, 63(2)∶ 812-814.
[22]张英华, 雷雨婷, 霍贵成.细菌中小分子冷休克蛋白的研究现状[J].中国乳品工业, 2008, 36(1)∶ 48-51.
[23]van de GUCHTE M, SERROR P, CHERVAUX C, et al.Stress responses in lactic acid bacteria[J].Antonie van Leeuwenhoek, 2002,82(1/4)∶ 187-216.
[24]WOUTERS J A, SANDERS J W, KOK J, et al.Clustered organization and transcriptional analysis of a family ofcspgenes ofLactococcus lactisMG1363[J].Microbiol, 1998, 144∶ 2885-2893.
[25]WOUTERS J A, JEYNOV B, ROMBOUTS fM, et al.Analysis of the role of 7 kDa cold-shock proteins ofLactococcus lactisMG1363 in cryoprotection[J].Microbiol, 1999, 145∶ 3185-3194.
[26]WOUTERS J A, ROMBOUT fM, de VOS W M, et al.Cold shock proteins and low-temperature response ofStreptococcus thermophilusCNRZ302[J].Appl Environ Microbiol, 1999, 65(10)∶ 4436-4442.
[27]TREMPY S J E.Expression of clpX, an ATPase subunit of the Clp protease, is heat and cold shock inducible inLactococcus lactis[J].J Dairy Sci, 2001, 84(8)∶ 1783-1785.
[28]DESMOND C, FITZGERALD G F, STANTON C, et al.Improved stress tolerance of GroESL-overproducingLactococcus lactisand probioticLactobacillus paracaseiNFBC 338[J].Appl Environ Microbiol, 2004, 70(10)∶ 5929-5936.
[29]KULLEN M J, KLAENHAMMER T R.Identification of the pH inducible, proton-translocating F1F0-ATPase (atpBEFHAGDC) operon ofLactobacillus acidophilusby differential display∶ gene structure,cloning and characterization[J].Mol Microbiol, 1999, 33∶ 1152-1161.
[30]GUILLOT A, GITTON C, ANGLADE P, et al.Proteomic analysis ofLactococcus lactis,a lactic acid bacterium[J].Proteomics, 2003, 3∶ 337-354.
[31]SANCHEZ B, CHAMPOMIER-VERGES M, COLLADO M C, et al.Low-pH adaptation and the acid tolerance response ofBif idobacterium longum[J].Appl Environ Microbiol, 2007, 73(20)∶ 6450-6459.
[32]LEVERRIER P, DIMOVA D, PICHEREAU V, et al.Susceptibility and adaptive response to bile salts inPropionibacterium freudenreichii∶physiological and proteomic analysis[J].Appl Environ Microbiol,2003, 69∶ 3809-3818.
[33]HERVE-JIMENEZ L, GUILLOUARD I, GUEDON E, et al.Postgenomic analysis ofStreptococcus thermophiluscocultivated in milk withLactobacillus delbrueckiisubsp.bulgaricus∶ involvement of nitrogen, purine, and iron metabolism[J].Appl Environ Microbiol,2009, 75∶ 2062-2073.
[34]PAUL C D, COLIN H.Surviving the acid test∶ responses of gram-positive bacteria to low pH[J].Curr Microbiol Mol Bio Rev, 2003, 67∶ 429-453.
[35]乌日娜, 武俊瑞, 孟和, 等.乳酸菌酸胁迫反应机制研究进展[J].微生物学杂志, 2007, 27(2)∶ 62-66.
[36]VINCENT F, YATES D, GARMAN E, et al.The three-dimensional structure of theN-acetylglucosamine-6-phosphate deacetylase, NagA,fromBacillus subtilis∶ a member of the urease superfamily[J].J Biol Chem, 2004, 279∶ 2809-2816.
[37]WU Rina, ZHANG Wenyi, SUN Tiansong, et al.Proteomics analysis of response ofLactobacillus caseiZhang to low acid stress[J].Int J Food Microbiol, 2011, 147∶ 181-187.
[38]DREWS O, REIL G, PARLAR H, et al.Setting up standards and a reference map for the alkaline proteome of the gram-positive bacteriumLactococcus lactis[J].Proteomics 2004, 4∶ 1293-1304.
[39]FOZOL E M, QUIVEY R G, Jr.Shifts in the membrane fatty acid profile ofStreptococcus mutansenhance survival in acidic environments[J].Appl Environ Microbiol, 2004, 70(2)∶ 929-936.
[40]乌日娜, 岳喜庆, 张和平.干酪乳杆菌对数生长期与稳定期蛋白质组比较分析[J].食品科学, 2011, 32(13)∶ 148-151.
[41]de ANGELIS M, GOBBETTI M.Environmental stress responses inLactobacillus∶ a review[J].Proteomics, 2004, 4∶ 106-122.
[42]COHEN D P A, RENES J, BOUWMAN fG, et al.Proteomic analysis of log to stationary growth phaseLactobacillus plantarumcells and a 2-DE database[J].Proteomics, 2006, 6∶ 6485-6493.
- 食品科学的其它文章
- 食盐对咸蛋黄蛋白质特性的影响

