西藏地区传统发酵乳中乳酸菌多样性及微生物数量分析
陈芝兰,杨吉霞,李梦寒,李晓卫,南志强,巩文峰
(1.西藏大学农牧学院,西藏 林芝 860000;2.西南大学食品科学学院,重庆 400715)
西藏自治区是全国五大牧区之一,生活在该区域的藏民族多以饲养牦牛为生,该地区牦牛的数量约占全国总量的30%[1]。相对牛乳而言,牦牛乳含有较高的蛋白质、脂肪、乳糖和矿物质等营养成分,被称为“天然浓缩乳”,是高原地区重要食品和乳品加工原料[2]。藏民族素有食用酸奶的传统习惯,千百年来沿袭古老而传统的方法制作酸奶。一般采用存放发酵奶和鲜奶的相同容器来进行持续发酵[3],这也导致了当地的发酵产品逐渐演变,使产品不仅风味独特、组织细腻、口感纯正,同时还保存了自然环境中的乳酸菌资源。西藏地区海拔高,气温低,昼夜温差大,紫外线辐射强,生态环境特殊,不同牧民家庭制备传统发酵乳制品所采用的原料、工艺等方面的差异,以及不同地域的自然环境和气候,造就了乳酸菌资源丰富的生物多样性。对传统乳制品中乳酸菌的分离、鉴定和生物学特性研究,有利于推动对我国传统乳制品乳酸菌种类认识及其优良菌株在特色乳制品中的应用,具有十分重要的意义。
本实验对从西藏地区采集的传统发酵乳进行乳酸菌的分离、鉴定,旨在发现西藏地区所含乳酸菌类群,以期利用丰富我国的乳酸菌资源。
1 材料与方法
1.1 材料与试剂
1.1.1 样品采集
样品采自西藏自治区7个地区56户牧民家庭。将牧民自然发酵制作的酸奶,在其发酵容器中充分搅拌后,吸取50mL装入无菌容器中,均放入冰盒内,备用。在采样现场用便携式pH计测定酸奶的pH值。样品采集地、样品的代号和样品数见表1。
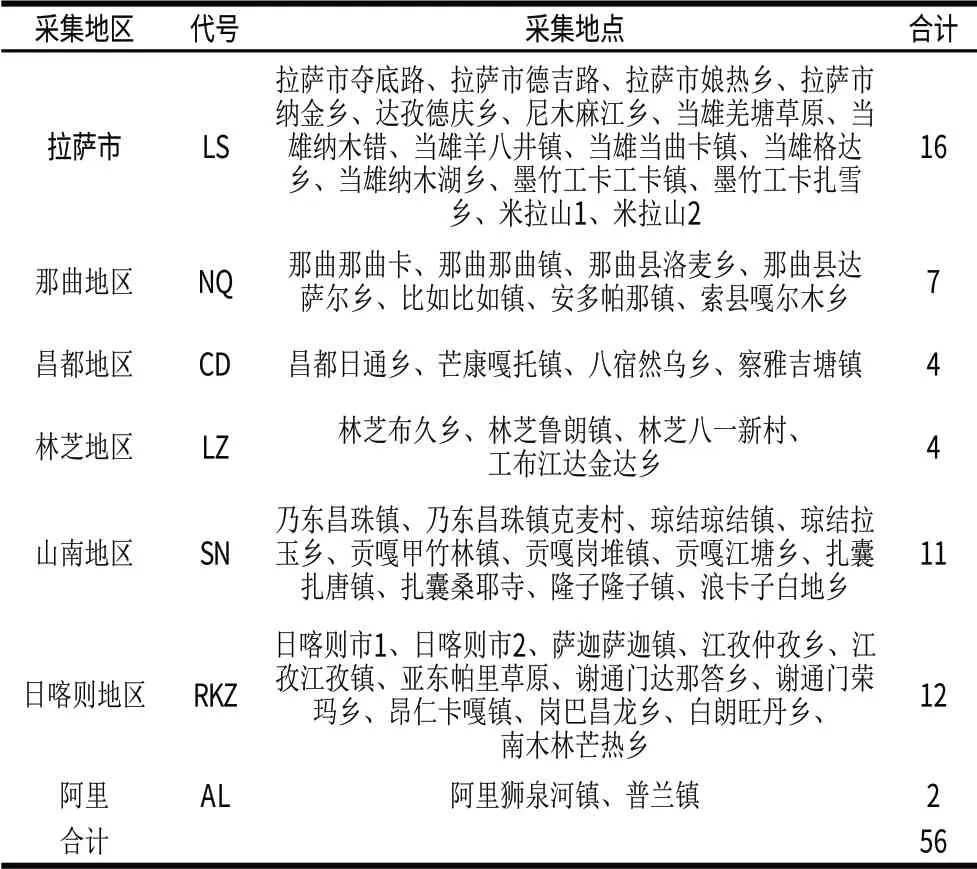
表1 西藏发酵牛乳采集地点及数量Table 1 Fermented milk samples tested in this study
1.1.2 标准菌株
标准菌株Lactobacillus acidophilusATCC 4356T为CICC保藏菌种,其编号为CICC6075T。
1.1.3 试剂
培养基及生理生化实验用试剂参照文献[4-6]中的方法配制;缓冲液与常备试剂参照文献[7]制备。Taq酶、dNTP、DNA Marker 天为时代生物有限公司;GoldView 北京塞百盛基因技术有限公司;溶菌酶、蛋白酶K、十六烷基三甲基溴化铵(CTAB)、十二烷基硫酸钠(SDS) 北京华绿渊生物技术发展公司。
1.2 方法
1.2.1 乳酸菌和酵母菌数量分析
将发酵乳样品用灭菌生理盐水做10倍递增稀释,各取3个稀释度,设3个重复。用MRS(加0.02g/100mL放线菌酮和多黏菌素)、PDA(加0.03g/100mL链霉素)培养基倾注培养,于37℃厌氧条件下培养48h,进行染色、镜检、计数。
1.2.2 乳酸菌的分离与纯化
将发酵乳样品划线接种于含有10μg/g放线菌酮的MRS(加入0.5g/100mL碳酸钙)琼脂培养基上,37℃厌氧培养48h。记录菌落形态,挑取有透明环的菌落,在MRS平板上反复划线分离,至确定为纯菌。将纯化后的菌株进行斜面保存、甘油法低温冻存。
1.2.3 乳酸菌的生理生化鉴定
按照文献[5,8-11]介绍的方法,凡是革兰氏阳性、过氧化氢酶实验阴性的菌株初步确定为乳酸菌,然后按表2的鉴定程序及项目区分不同的乳酸菌菌属,并通过形态特征、生理生化特征、糖发酵实验等对种进行鉴定。
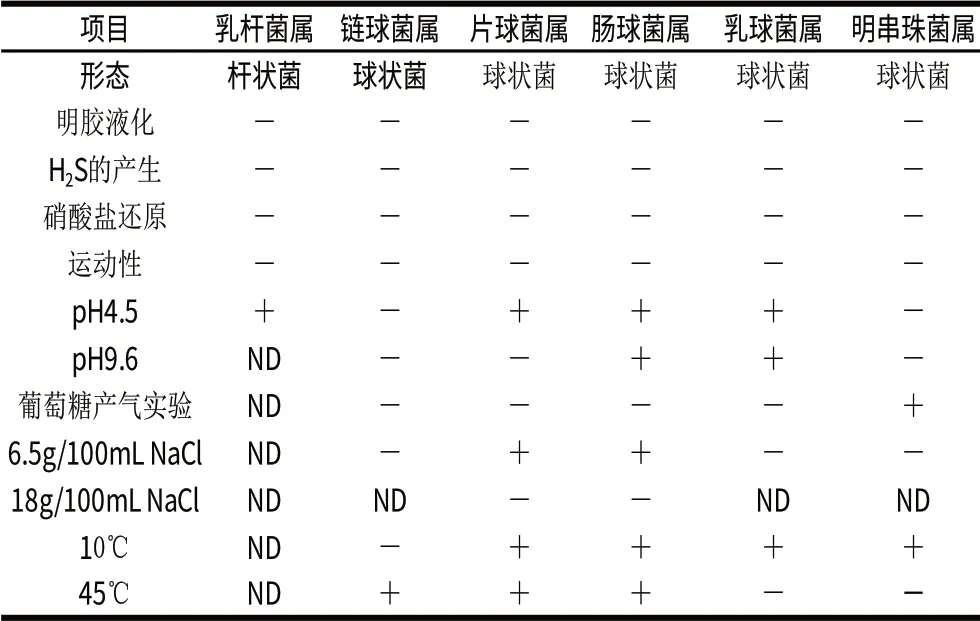
表2 乳酸菌鉴定程序及实验项目Table 2 Identification procedure and indicators of lactic acid bacteria
1.2.4 16S rRNA基因序列同源性分析
将分离的乳酸菌菌株,提取基因组DNA[7,12],采用正向引物27f:5’-AGA GTT TGA TCC TGG CTC AG-3’,反向引物1541r:5’-AAG GAG GTG ATC CACCC-3’,进行PCR扩增[13],PCR纯化产物送生工生物工程(上海)股份有限公司进行测序。
将测得乳酸菌菌株16S rRNA基因序列用BLAST(http://wwwncbi.nlm.nih.gov/blast/)在GenBank中搜索,与待测菌株同源性最高的已知分类地位的菌种初步确定待测菌株的种属。然后从GenBank数据库中下载已知的乳酸菌菌株的16S rRNA序列,以Clustal X[14]进行序列比对后,用MEGA4.0的UPGMA(unweighted pair-groupmethod with arithmetic means)法构建系统发育树[15],并进行1000次Bootstraps检验。利用16S rRNA基因序列同源性分析的方法进行鉴定。
2 结果与分析
2.1 传统发酵牛乳中乳酸菌和酵母菌的分布
采用稀释倾注选择分离法在各样品中均分离到乳酸菌和酵母菌,西藏7个地区56户牧民家庭传统发酵乳样品中微生物数量和种类如表3所示。乳酸菌数量变化幅度为1.01~8.58(lg(CFU/mL)),乳酸菌平均含量为(6.38±1.54)(lg(CFU/mL)),各地区乳酸菌平均含量以山南、那曲>昌都、日喀则>林芝>拉萨>阿里,各个地区间乳酸菌平均含量差异达显著水平(P<0.05),同一地区不同牧民家庭制作发酵乳乳酸菌含量差异以那曲最小,拉萨最大。而酵母菌数量变化幅度为2.09~7.16(lg(CFU/mL)),酵母菌平均含量为(5.27±1.63)(lg(CFU/mL)),各地区酵母菌平均含量以阿里地区显著性(P<0.05)高于其他地区,同一地区不同牧民家庭制作发酵乳酵母菌含量差异以阿里最小,昌都最大。采自阿里的两份发酵乳酵母菌总数比乳酸菌总数大6个对数级,其他54份样品中的乳酸菌总数比酵母菌的总数多,56份酸牛奶样品中乳酸菌平均含量((6.38±1.54)(lg(CFU/mL)))比酵母菌平均含量((5.27±1.63)(lg(CFU/mL)))大1个对数级。结果表明西藏地区传统发酵酸牛乳中富含乳酸菌和酵母菌,以乳酸菌为优势菌群,微生物数量的差异可能和当地的地理位置、气候及制作条件如接种量、接种时间、发酵温度等因素有关。
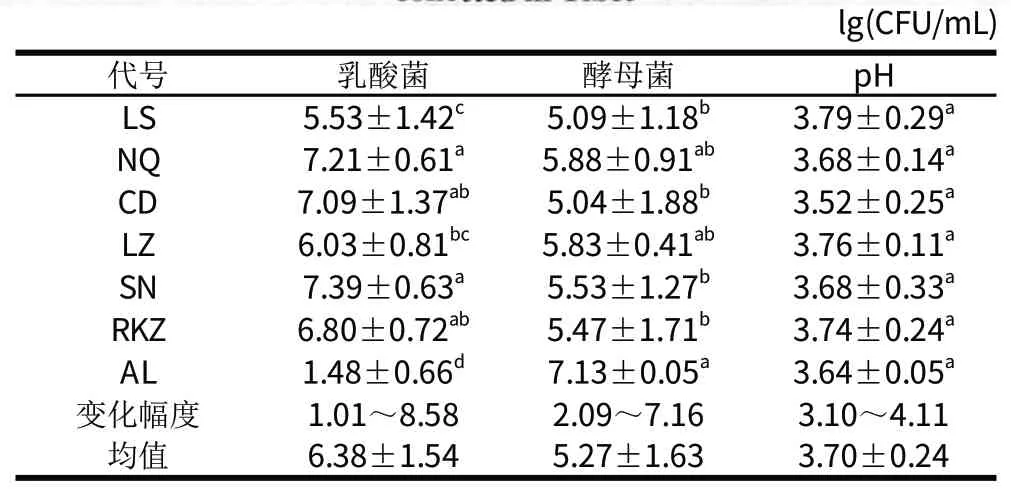
表3 西藏发酵牛乳微生物组成分析Table 3 Microbial composition analysis of fermented milk samples collected in Tibet
2.2 乳酸菌生理生化鉴定结果
将分离的96株杆菌进行属的鉴定,结果表明:均为革兰氏染色阳性,无芽孢,不运动,在H2O2酶实验、硝酸还原实验、H2S实验(与丹毒丝菌属相区分)呈阴性,在pH 4.5的环境中生长(与肉食杆菌属相区分)的杆菌,符合乳杆菌属的生物学特性,被鉴定为乳杆菌属的细菌。进一步以L.acidophilusATCC4356T(CICC保藏菌种,编号为CICC6075)为参考菌株,按照种特性实验方法进行了种的鉴定[5,10]。结果(表4)表明:48个菌株(群1)大部分性状一致,与L.casei的特性相近,18个菌株(群2)大部分性状一致,与L.plantarum的特性相近,7个菌株(群3)大部分性状一致,与L.fermentum的特性相近,4个菌株(群4)大部分性状一致,与L.breris的特性相近,9个菌株(群5)大部分性状一致,与L.parabuchneri的特性相近,5个菌株(群6)大部分性状一致,与L.delbrueckiisubsp.bulgaricus的特性相近,2个菌株(群7)大部分性状一致,与L.acidophilu的特性相近,3个菌株(群8)大部分性状一致,将糖发酵等生理生化实验结果查找文献[5,10]无对应菌种。
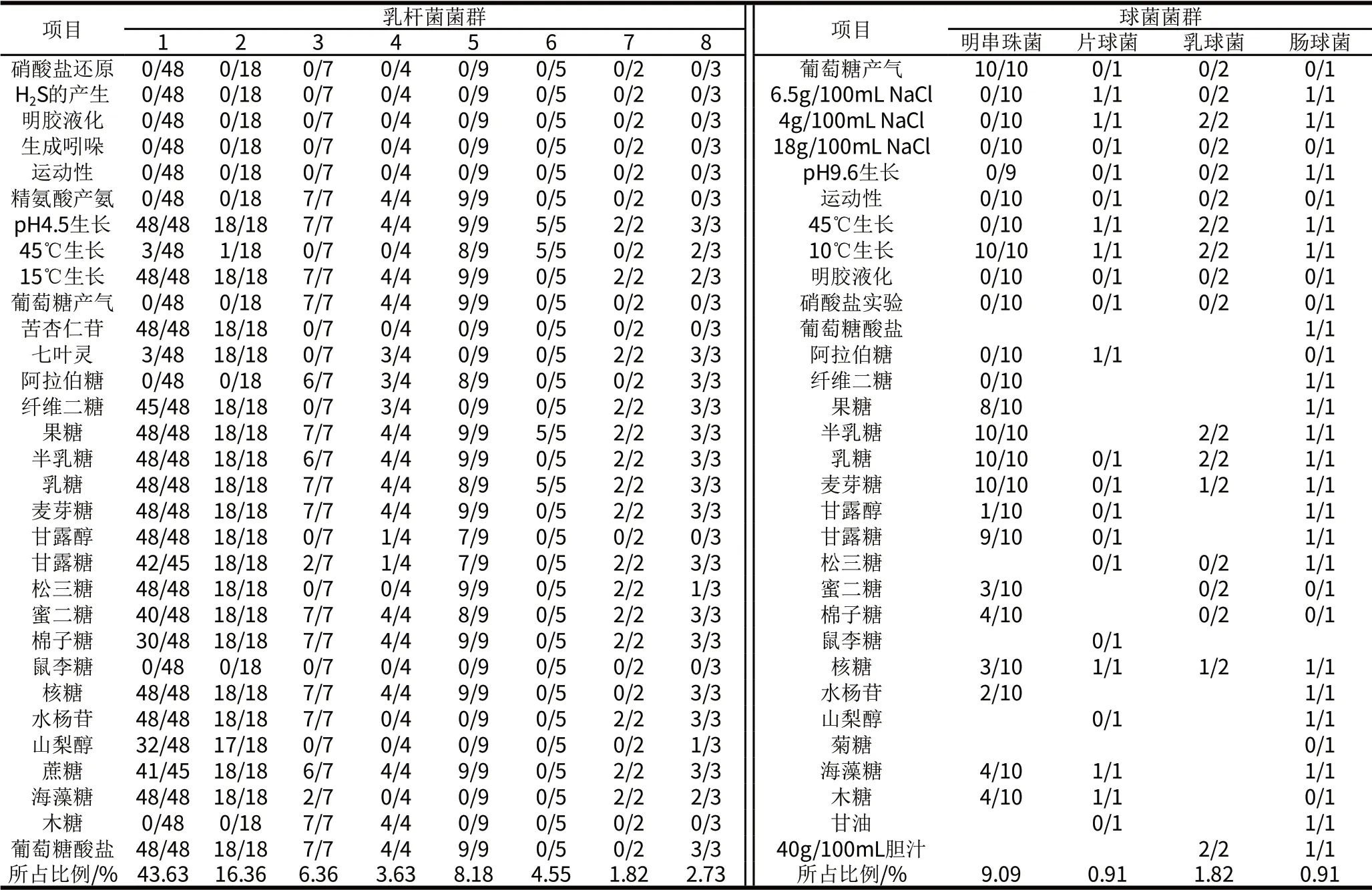
表4 分离自西藏发酵乳中的乳酸菌的生理特性Table 4 Physiological and biochemical characteristics of lactic acid bacteria isolated from Tibetan fermented milk products
由表4可知,于MRS平板划线厌氧分离的14株球菌革兰氏染色阳性、H2O2酶阴性、不运动、硝酸盐实验阴性,符合乳酸球菌的特性。2个菌株葡萄糖产酸产气似明串珠菌属(Leuconostoc)。1个菌株在35℃和45℃均生长,在pH9.6不生长,在4.0g/100mL NaCl、6.5g/100mL NaCl生长,18g/100mL NaCl不生长符合片球菌属(Pediococcus)的特征。2个菌株在10℃生长、45℃不生长,6.5g/100mL NaCl不生长,似乳球菌属(Lactococcus)。1个菌株10℃和45℃、6.5g/100mL NaCl、pH9.6菌生长,葡萄糖产酸实验阴性,似肠球菌属(Enterococcus)。
2.3 16S rRNA基因序列分析
选取Lactobacillus、Leuconostoc等的代表菌株和没有经生理生化鉴定到菌种的共33个菌株,提取其基因组DNA,扩增其16S rRNA基因序列进行测序,将16S rRNA基因序列在GenBank中进行同源序列搜索,各受试菌株与相关菌种的同源性都在99%以上。为显示供试菌株与已知菌种之间的亲缘关系及其系统地位,由待测33株乳酸菌的16S rRNA序列与12株参考菌株的16S rRNA序列用MEGA4.0软件绘制系统发育树,结果见图1,系统发育树以两歧双歧杆菌(B.bifidum)16S rRNA基因序列作为外群,序列一致长度为1400bp。45个序列的系统发育树显示10个分支:LZ3-1、LS11-1、RKZ1-3、SN11-1、RKZ3-1、NQ7-1、RKZ4-1、LS9-3、LS5-1、LS2-1、LS3-1、LS12-2、SN5-1、LS8-2、SN8-1和L.caseiATCC334 D86517处于一个分支;LS14-2、LS6-1、LS7-1、LS7-2、LS6-2和Lactobacillus paracaseiATCC25302 HQ23165、Lactobacillus caseiATCC339 AF469172处于一个分支;LS2-3和Lactobacillus fermentumCIP102980 JN175331处于一个分支;LS14-1、RKZ12-1、RKZ12-2和Lactobacillus diolivoransYIT10368 AB429369处于一个分支;NQ3-1和Lactobacillus parabuchneriDSM5707 AB370877处于一个分支;LS2-2和Lactobacillus brerisNRIC0137 AB362618处于一个分支;SN6-2、CD2-2、LS15-1和Lactobacillus plantarumNBRC15891 AB626055处于一个分支;LS4-1和Lactobacillus delbrueckiiBCRC10696 AY773948处于一个分支;LS3-4和Pediococcus acidilacticiNBRC12218 AB680261处于一个分支;NQ4-1、LS12-1和Leuconostoc mesenteroidesNRIC1541 AB023243处于一个分支。
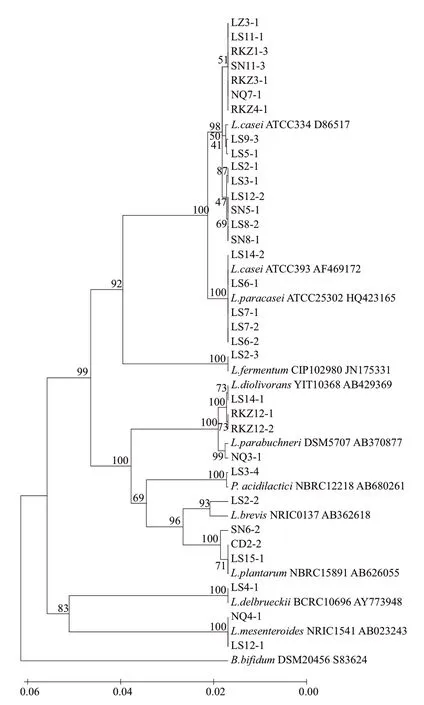
图1 分离乳酸菌菌株和相关菌株16S rRNA 基因序列建立的系统发育树Fig.1 Phylogenetic tree based on the 16S rRNA sequences of tested lactic acid bacteria and relevant strains
2.4 分离菌株的综合鉴定结果
48株生理生化鉴定为L.casei的菌株,经16S rRNA基因序列分析结果为L.casei和L.paracasei,再次证实干酪乳杆菌群内的16S rRNA基因序列相似性达到99%以上,种水平分辨率比较低,采用表型特征鉴定和区分的准确性也会降低[16];19株L.plantarum中,除1株菌可在45℃条件下生长外,其余18株生理生化鉴定结果与文献[5,10]完全相符;分离自日喀则南木林县RKZ12-1与RKZ12-2 和分离墨竹工卡扎雪乡LS14-1的3株菌,糖醇发酵无相似种,16S rRNA基因测序鉴定与L.diolivoransYIT10368基因相似性高达99%;其他菌株生理生化鉴定结果与16S rRNA基因测序鉴定结果完全吻合。
根据表2和表4将乳酸球菌鉴定到4个属,1株生理生化鉴定为片球菌属的菌株与Pediococcus acidilacticiNBRC12218的基因相似性高达99%;明串珠菌属内10个菌株碳水化合物发酵特征十分相似,单纯依据生理生化指标,很难鉴定到种,16S rRNA基因测序鉴定结果,NQ4-14、LS12-1与Ln.mesenteroidesNRIC1541的基因相似性高达99%。总体来说,生理生化结果与16S rRNA基因序列鉴定结果能较好地相吻合。
2.5 发酵乳中乳酸菌种类及分布
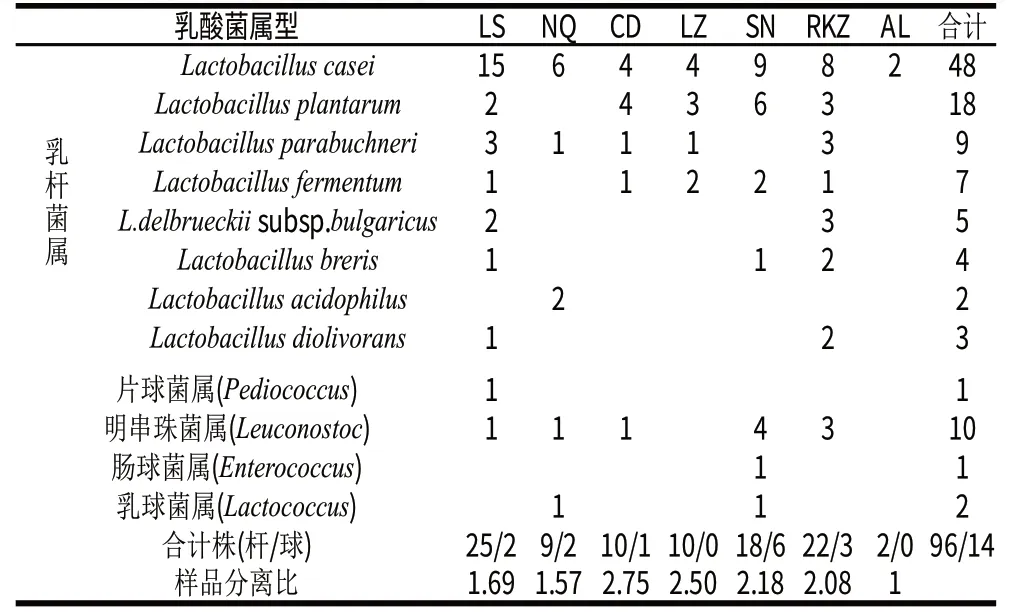
表5 西藏地区乳酸菌属型及乳杆菌分布Table 5 Classification and distribution of the genus and species in fermented milk products from different pasturing areas of Tibet
由表5可知,从西藏7个地区56份发酵乳中均分离到乳酸菌。共分离乳酸菌110株,经鉴定分别归为乳杆菌属(96株)、明串珠菌属(10株)、乳球菌属(2株)、肠球菌属(1株)、片球菌属(1株)5个属。其中,归于乳杆菌属的有8个种,L.casei(占分离株的43.64%)为优势种,分布于整个西藏地区的传统发酵乳制品中,其次为L.plantarum,再次为L.parabuchneri;菌株多样性丰富度以RKZ、LS>SN>NQ>CD>LZ>AL;基物样品分离比在1~2.75间,以CD最高,AL最低;分离到的乳酸杆菌和乳酸球菌的比例相差较大,乳酸杆菌占87.27%,为优势发酵菌群。
3 结 论
3.1 本研究从西藏地区自然发酵的56份酸奶样品中,共分离出110株乳酸菌。其中,乳酸杆菌96株占分离株的87.27%,为优势菌群,分别是L.casei48株、L.plantarum18株、L.parabuchneri9株、L.fermentum7株、L.delbrueckiisubsp.bulgaricus5株、L.breris4株、L.acidophilus2株、L.diolivorans3株,L.casei占分离株的43.64%,是优势菌群;10株乳酸球菌为明串珠菌属(Leuconostoc)10株、乳球菌属(Lactococcus)2株、肠球菌属(Enterococcus)1株、片球菌属(Pediococcus)1株;56份发酵乳中乳酸菌数平均含量为(6.38±1.54)(lg(CFU/mL)),酵母菌平均含量为(5.27±1.63)(lg(CFU/mL)),乳酸菌比酵母菌高一个对数级,为主要发酵菌群。
3.2 传统发酵乳制品常常是混合菌株发酵,不同区域发酵乳中乳酸菌的种类有差异,但菌株与区域之间无必然联系,L.casei分布于整个西藏地区的传统发酵乳制品中,其次为L.plantarum,再次为L.parabuchneri,日喀则和拉萨地区菌株丰富度较好。分菌时根据菌落、菌体形态从同一样品中分出许多不同的菌株,但序列测定发现这些“不同”的菌都归于同种菌,同种菌株间形态和生理生化特性略有差异。
3.3 从西藏牧民家庭自制的发酵乳中分离到的乳杆菌大多是15、37℃生长、45℃弱生长的中温菌株。其原因与牧区牧民家庭制作的发酵乳,往往存放在非恒温状态的环境中,菌株经过长期适应西藏相对低温气候环境,形成了这种在广泛温度范围内生长的特性,说明被分离菌株的适应能力增强。
3.4 分离鉴定的110株乳酸菌,涉及到乳品中常见也是发酵工业上常用的乳酸菌。乳杆菌属中鉴定出8种乳杆菌分别为3种发酵类群,且都能发酵乳糖。分离的乳杆菌普遍具有发酵果糖、棉子糖、蜜二糖的能力,这些差异可能与环境条件有关。分离鉴定的乳球菌菌株无论是种类还是数量都很少,这可能与酸奶较低的pH值(pH值为3.70±0.24)有关[17],致使乳球菌生长受到抑制,活性也不高。14株球菌中,明串珠菌(10株)占优势,可能正是它们赋予了酸奶优良的风味和质地。
3.5 采用传统的培养法在MRS培养基于37℃厌养培养分析传统乳制品中微生物的多样性,但某些乳酸菌由于其活力不够、适应相对低温气候环境或不可在分离培养基中生长,不能被有效地分离,造成对环境微生物多样性的低估[18],须采用更多培养方法和分子生物学的方法相结合考察乳制品中微生物的多样性。
3.6 实验中传统与分子生物学分类结果相符。但传统的生理生化方法鉴定乳酸菌耗时长,而且,其鉴定结果也有不确定性[19],采用16S rRNA序列分析对发酵酸乳中乳酸菌进行分子生物学鉴定,比传统的生化实验鉴定准确度高、结果更可靠,因此,认为只有将分子生物学技术和传统的生理生化实验方法结合起来才能更加准确的鉴定出乳酸菌。
[1]姬秋梅, 普穷, 达娃央拉, 等.西藏牦牛资源现状及生产性能退化分析[J].畜牧兽医学报, 2003, 34(4): 368-371.
[2]于莉, 罗章, 孟云, 等.西藏地区牦牛乳理化和微生物指标的检测分析[J].中国乳品工业, 2006, 34(9): 8-11.
[3]曹凤英.西藏行知书[M].广州: 广东旅游出版社, 2004.
[4]杨洁彬, 郭兴华, 凌代文.乳酸菌: 生物学基础及应用[M].北京: 中国轻工业出版社, 1996: 1-3.
[5]凌代文.乳酸菌分类鉴定及实验方法[M].北京: 中国轻工业出版社,1999: 1-25.
[6]东秀珠, 蔡妙英.常见细菌系统鉴定手册[M].北京: 科学出版社,2001.
[7]萨姆布鲁克J, 拉塞尔D W.分子克隆实验指南[M].2版.黄培堂, 译.北京: 科学出版社, 2002: 1564-1594.
[8]孙天松, 王俊国, 张列兵, 等.中国新疆地区酸马奶中乳酸菌生物多样性研究[J].微生物学通报, 2007, 34(3): 451-454.
[9]倪慧娟, 包秋华, 孙天松, 等.新疆地区酸马奶中酵母菌的鉴定及其生物多样性分析[J].微生物学报, 2007, 47(4): 578-582.
[10]布坎南R E.伯杰细菌鉴定手册[M].8版.北京: 科学出版社, 1984.
[11]FELIS G E, DELLAGLIO F.Taxonomy of lactobacilli and bifidobacteria[J].Curr Issues IntestinalMicrobiol, 2005, 8: 44-61.
[12]WILSON K.Preparation of genomic DNA from bacteria[M]//AUSUBULF M, BENT R.Current protocols in molecular biology.New York: J Wiley and Sons, 1987.
[13]颜世敢, 朱丽萍.16S rRNA PCR鉴定嗜酸乳杆菌鸡源分离株[J].中国微生态学杂志, 2005, 17(2): 115-117.
[14]THOMPSON J D, HIGGINS D G, GIBSO N J, et al.Improving the sensitivity of progressive multiple sequence alignment through sequence weighting, positions-specific gap penalties and weight matrix choice[J].Nucleic Acids Research, 1994, 22: 4673-4680.
[15]SUDHIR K, KOICHIRO T, INGRID B J, et al.Molecular evolutionary genetics analysis software[J].Bioinformatics, 2001, 17: 1244-1245.
[16]刘光全, 刘勇, 李辉, 等.pheS基因序列分析在干酪乳杆菌群种水平鉴定中的应用[J].食品与发酵工业, 2011, 37(9): 94-97.
[17]闫波, 刘丽波, 李艳, 等.国外优质干酪中乳酸菌的分离鉴定与应用[J].中国乳品工业, 2006, 34(12): 21-25.
[18]RANTSIOU K, URSO R, IACUMIN L, et al.Culture-dependent andindependent methods to investigate the microbial ecology of Italian fermented sausages[J].Applied and Environmental Microbiology,2005, 71(4): 1977-1986.
[19]段宇珩, 谈重芳, 王雁萍, 等.乳酸菌鉴定方法在食品工业中的应用及研究进展[J].食品工业科技, 2007(2): 242-244.

