沙门氏菌菌毛研究进展
朱春红,孟 霞,厚华艳,龚建森,张江英,朱国强
(1.江苏省家禽科学研究所,扬州 225125;2.扬州大学兽医学院,扬州 225009)
菌毛是革兰氏阴性菌菌体表面的丝状蛋白附属物,又称柔毛、纤毛等,直径为0.2~1.5 mm。沙门氏菌菌毛多由结构蛋白亚单位组成,具有抗原性,与沙门氏菌的致病性相关。本文以GenBank上已经公布的多株不同血清型沙门氏菌全基因组序列信息为参考,结合相关文献,综述不同血清型沙门氏菌菌毛操纵子编码基因,分子结构特征及其在沙门氏菌进化、宿主嗜性、致病、感染扩散中的可能作用。
1 沙门氏菌菌毛操纵子基因分类
传统的沙门氏菌菌毛分类主要依据其形态和红细胞凝集反应性,但是在与环境以及宿主相互作用过程中,细菌基因组会发生碱基替换、基因转移、染色体倒位,缺失或重组等变异现象,使得细菌遗传信息发生变化,从而产生新的表型性状,因此基于生物大分子(核酸和蛋白质序列)信息的分类方法则显得相对客观[1]。对已登录GenBank的沙门氏菌全基因组序列进行比对分析,表明菌毛编码基因操纵子广泛存在于各种血清型的沙门氏菌基因组中,沙门氏菌基因组通常含有5~20种菌毛编码基因操纵子,依据其可能的表达和组装方式,菌毛主要分为下述三种类型:
1.1 伴侣蛋白-推进蛋白途径类( Chaperone/usher pathway, CUP )菌毛CUP类菌毛即经过伴侣蛋白-推进蛋白途径组装和表达的菌毛,对于这种方式组装与表达机制的菌毛国内外已经有了较为深入的研究。目前研究的大部分菌毛都通过这种机制表达。这类菌毛操纵子基因至少包括三个结构基因,菌毛亚单位蛋白编码基因,伴侣蛋白编码基因,推进蛋白编码基因,有时还有其他亚单位编码基因,如次要亚单位蛋白基因(通常表达于菌毛顶端的粘附素性亚单位),调控基因等。菌毛亚单位蛋白通过在N-末端产生信号肽序列的方式直接进入到细胞周质间隙,N-末端信号肽序列在转运过程中被降解,菌毛结构亚单位蛋白在周质间隙中与伴侣蛋白结合避免非活性形式的聚集或者被相关蛋白酶降解,然后在推进蛋白的辅助作用下穿过外膜蛋白,正确组装并成功表达于细菌表面。
研究者根据GenBank中已经共享的沙门氏菌全基因组信息,对推进蛋白编码序列进行比对分析,进一步将CUP类菌毛分为以下6个亚类:δ型,α型,β型,γ型,κ型和π型(图1)。分析图1中列出的17株沙门氏菌菌株的全基因组序列,发现肠道沙门氏菌肠科亚种除亚利桑那沙门氏菌基因组仅含5个CUP类菌毛编码基因簇外,其他肠科亚种沙门氏菌基因组中均含有11-13种不同CUP类菌毛编码操纵子;邦戈尔沙门氏菌则含有8个CUP类菌毛编码基因簇[2];比对大肠杆菌参考菌株K-12株序列,发现其和肠道沙门氏菌肠科亚种菌株相似,同时分析到12种之多的CUP类菌毛编码基因[3]。
1.2 成核沉淀途径(nucleation-precipitation pathway)类菌毛沙门氏菌中,成核沉淀途径类菌毛主要为细凝 集 性 菌 毛(thin aggregative fimbriae,Tafi), 由操纵子基因agfDEFG、agfBA编码和转录[5,6],这类菌毛形态和其他菌毛不同,直径3~4 nm,其在菌体表面呈现一种异聚体蛋白丝状形态,由一个独特的方式—成核沉淀方式组装表达展呈于菌体表面。

图1 沙门氏菌菌毛操纵子分类[4]Fig.1 Classifi cation of Salmonella fi mbriae
沙门氏菌Tafi菌毛和大肠杆菌卷曲菌毛(Curli菌毛)高度同源,目前对大肠杆菌Curli菌毛的组装机制研究比较深入。大肠杆菌Curli菌毛同样是由操纵子基因操纵子 csg(curli subunit genes)(csgDEFG、csgBA) 编码和转录[7],csgBA编码的亚基CsgB和CsgA通过信号肽的方式分泌到周质中,CsgD调控csgBA的转录,CsgG是一种外膜脂蛋白,与CsgE结合协助CsgA和CsgB完成跨膜运转;在沙门氏菌中,AgfA是Tafi菌毛的主要亚单位蛋白,其含有标准的信号肽序列,在AgfG亚单位的协同作用下通过细胞周质间隙,完成跨膜运转[8],次要亚单位蛋白AgfB,又称为nucleator,与分泌出的AgfA单体成核沉淀成不溶性的β折叠结构[9],在菌体表面形成淀粉纤维样沉淀物[10]。成核沉淀方式与伴侣蛋白-推进蛋白方式最主要不同之处在于,CUP类菌毛是在周质间隙中菌毛亚基蛋白发生聚合,而Curli菌毛和Tafi菌毛组装则发生在细菌外膜上。
1.3 Ⅳ型菌毛Ⅳ型菌毛是主要分布于革兰氏阴性致病菌表面的一种强韧细丝状纤毛,为纤毛蛋白的多聚体,多位于细菌细胞的一端。根据其主要亚单位蛋白的同源性,可以分为两类:ⅣA型和ⅣB型。ⅣA型菌毛含有较短的先导氨基酸序列和成熟的氨基酸序列,其宿主范围较广,可分布于人类、其他哺乳动物、植物、真菌和各种细菌表面;ⅣB型菌毛氨基酸序列较长,只分布于肠道细菌表面。
分析比对已发表的沙门氏菌株全基因组信息发现,仅少数属沙门氏菌含ⅣB型菌毛操纵子基因,如:邦戈尔沙门氏菌的sbeTABCDEFGHIP操纵子,伤寒沙门氏菌的pilM(N',N)OPQRSTUV操纵子,B型副伤寒沙门氏菌和海德堡沙门氏菌基因组也含有类似的同源的操纵子序列[11,12]。
Ⅳ型菌毛的结构研究报道在铜绿假单胞菌和奈瑟琳球菌中较多。他们的组成成分和Ⅱ型分泌系统的成分有同源性,包括菌毛蛋白、前菌毛肽酶、有或无核苷结合基序的可溶性蛋白、膜内蛋白和形成寡聚圆环结构的外膜蛋白。Ⅳ型菌毛蛋白最初是由一个菌毛蛋白亚基开始形成,在这个基础上结构前体蛋白经过前菌毛蛋白肽酶的裂解,前菌毛蛋白肽酶有双重功能(生成菌毛蛋白结构和调节细菌外分泌系统),他能同时使成熟菌毛蛋白的第一个氨基酸(苯丙氨酸或甲硫氨酸)甲基化。Ⅳ型菌毛亚基是在内膜周质中组装形成,在聚集成菌毛蛋白纤维之前,菌毛蛋白分子通过N端高度保守的疏水性区域的氨基酸先附着在内膜上,伤寒沙门氏菌的菌毛亚基pilin通过普遍分泌方式(产生信号肽的分泌方式)转运到周质中,并锚定在内膜上,向外膜拉伸形成完整的菌毛[13]。
2 菌毛基因操纵子的遗传变异
通过对17株测序完成的沙门氏菌株全基因组序列比较发现,肠道沙门氏菌含有多达11~13种之多的菌毛操纵子基因,且每种沙门氏菌基本上都含有一种或多种标准的CUP类菌毛蛋白。虽然菌毛种类极富多样性,但直系同源的沙门氏菌菌毛基因操纵子通常位于基因组序列中相似的位置,据报道,沙门氏菌一旦获得某种菌毛操纵子基因序列,则能通过垂直遗传和水平迁移(包括同源重组,转染,转导)等方式进行遗传[4]。
序列信息分析表明bcf、fim、sth菌毛操纵子基因在沙门氏菌属内高度保守,而lpf、peg、saf、stb、stc、std、ste、stf和sti这9种菌毛基因操纵子基因同源性能大于45%,但是其他菌毛操纵子基因同源性则小于45%[4]。
研究者所在实验室通过PCR扩增方法检测了D群部分沙门氏菌SEF14菌毛基因操纵子的变异情况,研究共检测了18株鸡白痢沙门氏菌、11株肠炎沙门氏菌以及1株都柏林沙门氏菌标准株sefA、sefD和sefR基因序列,结果发现sefA、sefD以及sefR基因在肠炎沙门氏菌和都柏林沙门氏菌中普遍存在,并和已发表的序列100%同源,但sefD和sefR基因在鸡白痢沙门氏菌中或发生碱基缺失,或无相应PCR产物,说明SEF14菌毛基因操纵子在鸡白痢沙门氏菌进化过程中发生了变异,这一变异很可能是鸡白痢沙门氏菌和肠炎沙门氏菌同属D群沙门氏菌,却不能表达SEF14菌毛的原因之一[14]。
3 沙门氏菌菌毛生物学功能
多数致病菌富含多种菌毛结构,如肠道致病菌肠炎沙门氏菌。目前对多种菌毛结构的生物组装及分泌机制研究较多,但对这些菌毛结构在沙门氏菌中的致病作用仍了解甚少。
3.1 CUP类菌毛沙门氏菌基因组中含有丰富的CUP类菌毛编码基因操纵子序列,但对其表达产物—菌毛蛋白在病原菌与易感宿主之间的相互关系仍未能深入研究。目前研究最广泛的I型菌毛,是由fim操纵子编码的,在静置条件下表达于所有的检测的沙门氏菌血清型中。FimA是I型菌毛的主要蛋白亚基,形成菌毛的主干部分,对宿主趋性有影响,但关于FimA的功能还存在争议[15,16];FimH位于菌毛顶端,其氨基酸序列差异决定不同菌种或不同血清型的I型菌毛与不同宿主细胞特异性粘附。研究发现大肠杆菌FimH优先结合到膀胱上皮细胞,而沙门氏菌FimH结合肠细胞[16]。FimH在结合的特异性上的变化不受血清型限制,但是对相同血清型的菌株的FimH进行诱导会产生不同的结合特性。在鼠伤寒沙门氏菌中,存在高粘附性和低粘附性两个FimH变种[15,17],高粘附性变种FimH对人类上皮细胞系有更高的亲和力,低粘附性变种FimH和肠炎沙门氏菌的FimH有同样的结合特性,属于甘露糖敏感型,体外实验中能同时结合人膀胱上皮细胞系和人结肠上皮细胞系[17,18],不同于低粘附性变种FimH,高粘附性变种FimH对不同细胞系的粘附能力差异较大[18]。
研究者所在实验室正致力于限制性表达于肠炎沙门氏菌等D群沙门氏菌的SEF14菌毛的生物学功能探索,部分研究数据显示SEF14菌毛能在优化的体外培养条件下表达。SEF14菌毛优化表达后,可以使活化的小鼠腹腔巨噬细胞对SEF14菌毛主要亚单位突变株吞噬作用增强,借助SEF14菌毛的作用,肠炎沙门氏菌能更好的作用于巨噬细胞并在巨噬细胞中存活[19]。
3.2 细凝集性菌毛细凝集性菌毛除了组装方式和其他菌毛区别外,其还有一些独特的生物学特性。细凝集性菌毛编码基因在多种属细菌中高度保守如:柠檬杆菌属(Citrobacter),肠杆菌属(Enterobacter),埃希氏菌属(Escherichia),克雷伯氏菌属(Klebsiella),沙门氏菌属(Salmonella)和志贺氏杆菌属(Shigella)[20]。细凝集性菌毛在沙门氏菌以及大肠杆菌中是生物膜形成的基质成分之一,细凝集性菌毛促进细菌在基质表面,特别是无生命物质表面,如玻璃、聚丙烯、聚氯乙烯等物质表面形成生物膜,细凝集性菌毛相互缠结,把邻近的细菌联接成小菌群,逐渐发育为生物膜[21]。另外,细凝集性菌毛能作为病原相关分子模式(pathogen associated molecular pattern,PAMP)被TLR2识别,启动先天性免疫保护作用。最新的研究表明,TLR2能启动宿主及病原源性的纤维淀粉样结构物质的先天性免疫反应[22]。鼠伤寒沙门氏菌致牛回肠袢炎的试验中,病菌侵袭后,机体对细凝集性菌毛淀粉样蛋白的识别能启动前炎性因子的生成和中性粒细胞的聚集[23]。
3.3 Ⅳ型菌毛目前关于Ⅳ型菌毛的生物学功能在假单胞菌属和奈瑟菌属研究相对较多,在沙门氏菌属中的研究鲜见报道。革兰氏阴性细菌在半固体表面如黏膜表面可有一种特有的运动形式,在黄黏球菌中这种运动称为滑行运动,而在假单胞菌属和奈瑟菌属中的运动称为蹭行运动。研究者运用镭射显微光钳系统进行体外实验发现奈瑟球菌通过Ⅳ型菌毛的伸缩运动完成在介质表面的运动,而其中PilT蛋白复合体起到了重要的作用[24];相关研究还证明蹭行运动能激活宿主免疫应答反应,介导早期的前炎症因子的释放,如IL-8,IL-6等[25]。Ⅳ型菌毛能通过自聚集在靶组织表面形成微克隆菌群,这种特性和Ⅳ型菌毛介导的介质表面运动共同参与了细菌生物膜的形成。Kube等[25]用实时共聚焦扫描显微镜观察发现铜绿假单胞菌会逐渐游弋形成蘑菇状的生物膜,提示了Ⅳ型菌毛在生物膜形成中的作用。Ⅳ型菌毛还参与一些致病过程比如免疫逃避、DNA吸收、生物膜形成、微菌落形成、粘附等。伤寒沙门氏菌Ⅳ型菌毛通过囊性纤维化跨膜转导调节子(CFTR)来完成附着和吸收,CFTR的水平与伤寒沙门氏菌的吸收效率相联系。prePil结合到CFTR上介导伤寒沙门氏菌与肠上皮细胞的粘附。有趣的是,囊性纤维化的CFTR突变株△F508,小鼠粘膜下层细胞对突变株的吸收严重下降。带有杂合子的突变株对伤寒沙门氏菌感染的敏感性降低[26-28]。上述研究均提示Ⅳ型菌毛在沙门氏菌属相关血清型细菌中的重要生物学致病作用,有较高研究价值。
4 菌毛基因操纵子与宿主嗜性
沙门氏菌菌毛操纵子基因的多样性分布可能与沙门氏菌的宿主嗜性相关[29],爬行动物等冷血动物不具有免疫记忆能力,相应的冷血动物适应性沙门氏菌则含有较少种类的菌毛操纵子基因,如:亚利桑那沙门氏菌含有5种菌毛操纵子基因,邦戈尔沙门氏菌含有8种菌毛操纵子基因(见图2)。
伤寒沙门氏菌以及甲乙丙三型副伤寒等血清型沙门氏菌是高度适应于人类的沙门氏菌,对其他动物不引起自然感染。对伤寒沙门氏菌的测序结果分析表明这一血清型沙门氏菌分化于5万年前[30]。和引起局部肠炎的其他相近血清型沙门氏菌相比,伤寒沙门氏菌能导致感染宿主的全身性败血症反应[31],且长期的胆囊带菌。在此感染机制下,伤寒沙门氏菌表现为多样性菌毛操纵子基因的失活,成为假基因,这一推论表现为伤寒沙门氏菌CT18株11个菌毛操纵子基因中7个演变为假基因,Ty2株则8个菌毛操纵子基因演变为假基因(图2)。研究者所在实验室对SEF14菌毛操纵子基因的遗传变异分析发现,部分鸡白痢沙门氏菌菌株SEF14菌毛亚单位编码基因sefD演变为假基因,表明此遗传变异可能与鸡白痢沙门氏菌的专嗜性相关[14]。
这种宿主偏嗜性沙门氏菌中菌毛操纵子基因的失活为假基因是一种趋势,但是也存在一些例外,如乙型副伤寒沙门氏菌一共11种菌毛操纵子基因,只有1个失活为假基因,其他10个仍是完整的菌毛操纵子基因。这一现象的可能解释是B型副伤寒是出现较晚的宿主偏嗜性沙门氏菌[32]。上述研究表明,假基因的出现以及积累是宿主偏嗜性沙门氏菌利用菌毛操纵子基因失活以趋同方式适应单一宿主的模式[33]。
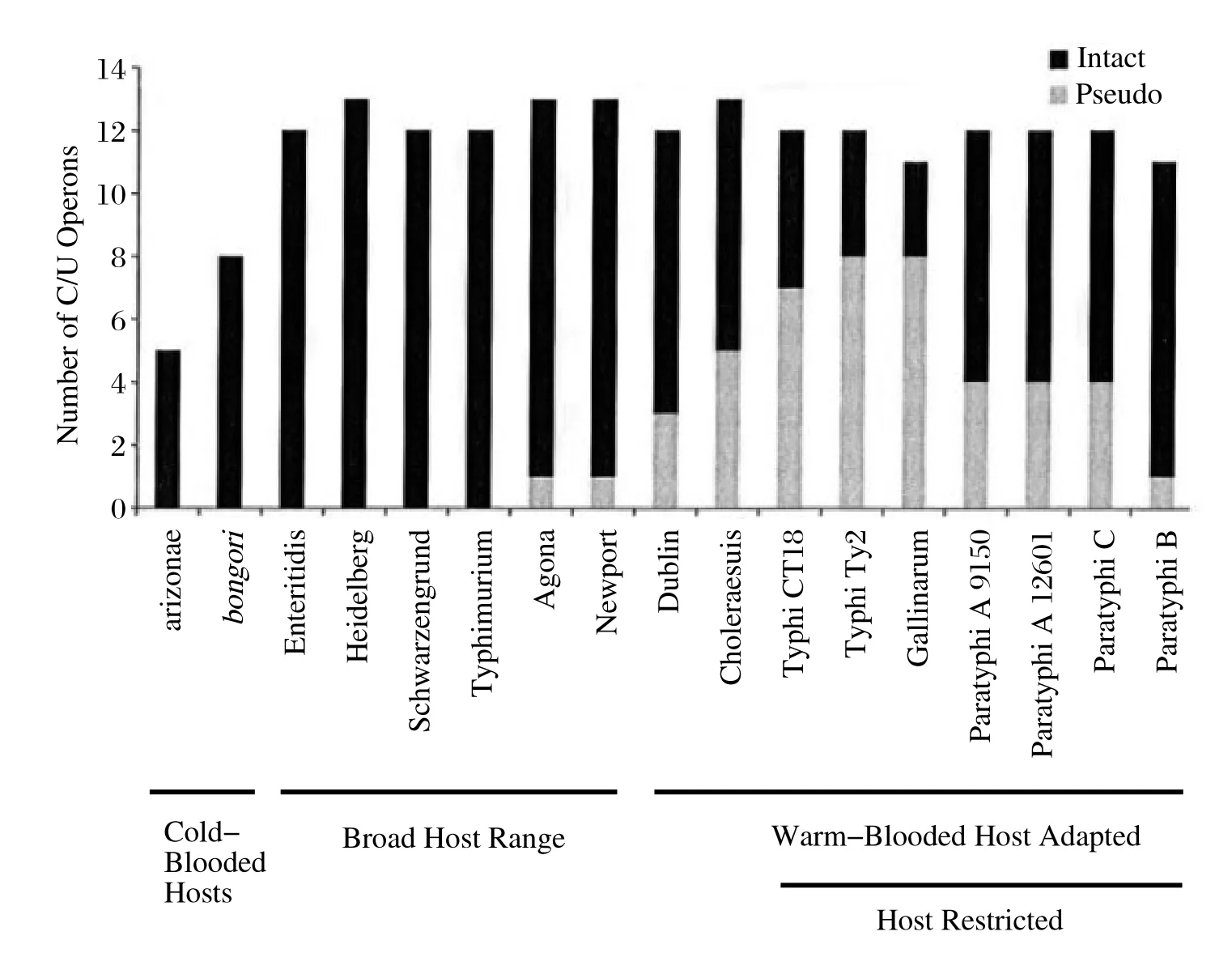
图2 不同血清型沙门氏菌CUP类菌毛操纵子假基因的比例[4]Fig.2 Chaperone/usher fi mbriae repertoire pseudogene content amongst the analysed Salmonella serovars
5 展望
沙门氏菌菌毛操纵子基因的多样性分布可能是不同血清型沙门氏菌适应环境的一种反应,对不同菌毛在沙门氏菌致病及传播过程中的作用,对不同菌毛的鉴定及功能解析等研究还有待加强。相信随着分子生物学研究技术的不断发展,沙门氏菌菌毛的结构和功能研究也会不断深入,菌毛在沙门氏菌特异性检测与预防控制方面将会发挥极其重要的作用。
[1]衣美英, 朱万孚, 刘树林.基因组结构在细菌种系发育研究中的应用[J].中华微生物学和免疫学杂志, 2003,23(12): 995-998.
[2]Nuccio S P, B-umler A J.Evolution of the chaperone/usher assembly pathway: fimbrial classification goes Greek [J].Microbiol Mol Biol Rev, 2007, 71(4): 551-575.
[3]Marchler-Bauer A, Bryant S H.CD-Search: protein domain annotations on the fly [J].Nucleic Acids Res,2004, 32(Web Server issue): W327-331.
[4]Steffen Porwollik.Salmonella: From Genome to Function[M].//Sean-Paul Nuccio, Thomson N R, Fookes M C,et al.Fimbrial signature arrangements in Salmonella.Caister Academic Press; 1 edition (January 1, 2011).
[5]Collinson S K, Em-dy L, Müller K H,et al.Purification and characterization of thin, aggregative fimbriae from Salmonella enteritidis [J].J Bacteriol, 1991, 173(15):4773-4781.
[6]Collinson K S, Doig P C, Doran J L,et al.Thin aggregative fimbriae mediate binding of Salmonella enteritidis to fibronectin[J].J Bacteriol, 1993; 175(1): 12-18.
[7]Olsén A, Jonsson A, Normark S.Fibronectin binding mediated by a novel class of surface organelles on Escherichia coli[J].Nature, 1989, 338(6217): 652-655.
[8]Robinson L S, Ashman E M, Hultgren S J,et al.Secretion of curli fibre subunits is mediated by the outer membrane-localized CsgG protein[J].Mol Microbiol,2006, 59(3): 870-881.
[9]Shewmaker F, McGlinchey R P, Thurber K R,et al.
The functional curli amyloid is not based on in-register parallel beta-sheet structure[J].J Biol Chem, 2009,284(37): 25065-25076.
[10]Chapman M R, Robinson L S, Pinkner J S,et al.Role of Escherichia coli curli operons in directing amyloid fiber formation[J].Science, 2002, 295(5556): 851-855.
[11]Parkhill J, Dougan G, James K D,et al.Complete genome sequence of a multiple drug resistant Salmonella enterica serovar Typhi CT18[J].Nature, 2001, 413(6858): 848-852.
[12]Yoshida T, Kim S R, Komano T.Twelve pil genes are required for biogenesis of the R64 thin pilus[J].J Bacteriol, 1999, 181(7): 2038-2043.
[13]Wolfgang M, van Putten J P, Hayes S F,et al.Components and dynamics of fiber formation define a ubiquitous biogenesis pathway for bacterial pili [J].EMBO J, 2000, 19(23): 6408-6418.
[14]Zhu C H, Wu J, Chen W W,et al.Difference and variation of the sef14 operon gene clusters in S.pullorum[J].J Basic Microbiol, 2010, 50(Suppl 1): S120-123.
[15]Guo A, Cao S, Tu L, Chen P,et al.FimH alleles direct preferential binding of Salmonella to distinct mammalian cells or to avian cells [J].Microbiology, 2009, 155(Pt 5):1623-1633.
[16]Thankavel K, Shah A H, Cohen M S,et al.Molecular basis for the enterocyte tropism exhibited by Salmonella typhimurium type 1 fimbriae [J].Biol Chem, 1999,274(9): 5797-5809.
[17]Boddicker J D, Ledeboer N A, Jagnow J,et al.Differential binding to and biofilm formation on, HEp-2 cells by Salmonella enterica serovar Typhimurium is dependent upon allelic variation in the fimH gene of the fim gene cluster [J].Mol Microbiol, 2002, 45(5):1255-1265.
[18]Kisiela D, Laskowska A, Sapeta A,et al.Functional characterization of the FimH adhesin from Salmonella enterica serovar Enteritidis [J].Microbiology, 2006,152(Pt 5):1337-1346.
[19]Chunhong Zhu, Juan Wu, Hassan Hussein Musa,et al.Construction of S.enteritidis SefA and SefD Mutants and their Role in Invasion and Survival to Host Cell.Microbiological Society of Korea's 50th Anniversary International Symposium on Microbiology 2009 May 28-30, 2009, Jeju Island, Korea.
[20]Zogaj X, Bokranz W, Nimtz M,et al.Production of cellulose and curli fimbriae by members of the family Enterobacteriaceae isolated from the human gastrointestinal tract [J].Infect Immun, 2003, 71(7):4151-4158.
[21]R mling U, Bokranz W, Rabsch W,et al.Occurrence and regulation of the multicellular morphotype in Salmonella serovars important in human disease [J].Int J Med Microbiol, 2003, 293(4): 273-285
[22]Tükel C, Wilson R P, Nishimori J H,et al.Responses to amyloids of microbial and host origin are mediated through toll-like receptor 2[J].Cell Host Microbe, 2009,6(1): 45-53.
[23]Tükel C, Raffatellu M, Humphries A D,et al.CsgA is a pathogen-associated molecular pattern of Salmonella enterica serotype Typhimurium that is recognized by Toll-like receptor 2[J].Mol Microbiol, 2005, 58(1): 289-304.
[24]Maier B.Using laser tweezers to measure twitching motility in Neisseria [J].Cur Opin Microbiol, 2005, 8(3):344-349.
[25]Kube D, Sontich U, Fletcher D,et al.Proinflammatory cytokine responses to P.aeruginosa infection in human airway epithelial cell lines [J].Am J Physiol Lung Cell Mol Physiol, 2001, 280(3): L493-502.
[26]Pier G B, Grout M, Zaidi T,et al.Salmonella typhi uses CFTR to enter intestinal epithelial cells [J].Nature.1998,93(6680): 79-82.
[27]Xu X F, Tan Y W, Lam L,et al.NMR structure of a type IVb pilin from Salmonella typhi and its assembly into pilus[J].J Biol Chem, 2004, 279(30): 31599-31605.
[28]Tsui I S, Yip C M, Hackett J,et al.The type IVB pili of Salmonella enterica serovar Typhi bind to the cystic fibrosis transmembrane conductance regulator [J].Infect Immun, 2003, 71(10): 6049-6050.
[29]Norris T L, B-umler A J.Phase variation of the lpf operon is a mechanism to evade cross-immunity between Salmonella serotypes [J].Proc Natl Acad Sci U S A.1999, 96(23): 13393-13398.
[30]Kidgell C, Reichard U, Wain J,et al.Salmonella typhi,the causative agent of typhoid fever, is approximately 50,000 years old [J].Infect Genet Evol, 2002, 2(1): 39-45.
[31]Kingsley R A, B-umler A J.Host adaptation and the emergence of infectious disease: the Salmonella paradigm[J].Mol Microbiol, 2000, 36(5): 1006-1014.
[32]Selander R K, Beltran P, Smith N H,et al.Evolutionary genetic relationships of clones of Salmonella serovars that cause human typhoid and other enteric fevers [J].Infect Immun, 1990, 58(7): 2262-2275.
[33]Holt K E, Thomson N R, Wain J,et al.Pseudogene accumulation in the evolutionary histories of Salmonella enterica serovars Paratyphi A and Typhi [J].BMC Genomics, 2009, 10: 36.

