基于压缩氨基酸和支持向量机进行膜蛋白类型识别
管翠萍,石 晶,徐惠娟
(宁夏大学生命科学学院,宁夏银川750021)
膜蛋白是一类结构独特的蛋白质,它处于细胞与外界的交界部位,是细胞执行各种功能的物质基础,同时也是很多药物作用的靶点,如最典型的 G蛋白偶联受体家族,它虽然只占人类基因组编码序列的1%,但在药物研发中却有60% ~70% 的目标蛋白是G蛋白偶联受体家族成员[1]。目前随着基因组学和蛋白组学的发展,对膜蛋白结构和功能的研究刻不容缓,而对膜蛋白进行类型预测则是以上工作的一个重要基础。膜蛋白根据其在细胞膜上的不同存在方式,可分为六大类:A.Type I跨膜蛋白,只含有一段α螺旋构成的跨膜区,N末端在细胞外,C末端在细胞内;B.Type II跨膜蛋白,与Type I的方向刚好相反;C.Multipass跨膜蛋白,具有多个跨膜区;D.Lipid Chain锚定膜蛋白,通过脂质锚链与脂双层相结合;E.GPI锚定膜蛋白,通过甘氨酰甘氨酸二肽酶与脂双层相结合;F.外周蛋白,通过与其它膜蛋白之间的非共价键结合,而不是直接与脂双层发生相互作用[2-3]。
目前利用分子生物学方法来验证膜蛋白类型已经不能满足日益增长的膜蛋白序列的需求,而生物信息学则可利用海量的生物数据,进行分类预测。因此,通过膜蛋白的初级序列结合生物信息手段来预测其所属类型,以获取相关的结构和功能信息是目前的一个研究趋势。现已提出了一些预测方法,并取得了较好的预测结果,如Chou等先后提取氨基酸组分、伪氨基酸组成、蛋白质进化等特征进行分类研究[3-8];Feng和Zhang提出了氨基酸指数的自相关函数方法[9];Cai等分别利用部分序列顺序作用和功能结构域方法结合支持向量机(SVM)实现分类预测[10-11];Yang等[12]采用单氨酸和二肽组成方法获取序列顺序信息进行预测;Jiang等融合氨基酸组成和氨基酸位置特征利用支持向量机进行分类预测等[13]。本文将利用压缩的氨基酸对原始膜蛋白序列进行信息压缩,对压缩序列进行氨基酸组成和顺序特征的提取,同时采用SVM构建分类器,实现六种分类模型的构建,利用五叠交叉验证的方法进行验证。
1 材料与方法
1.1 数据集的构建
早期的研究大多数基于 Chou等人[3]构建的CE2059和CE2625两个通用数据集来进行分类模型的构建。这两个数据集中的数据来源于SWISSPROT1997年11月发布的Release 37,建立年限较早,且随着现在数据的不断更新,其中有些信息已经变更。2007年,Chou和 Shen基于 SWISS-PROT Release 51对该数据集做了进一步扩充,其中训练集包含3 249个膜蛋白序列;独立检验集包含4 333个膜蛋白序列[8]。2009年,Zeng又对现有数据集进行改进,收集了5 750条膜蛋白序列[14]。目前,随着数据库中数据的不断增长,膜蛋白序列信息也在不断补充中,采用新的数据集来做分类模型是有必要的,但这样又缺乏了与以往研究的可比较性。所以在本研究中,将采用两个数据集A、B,分别作分类模型构建来对预测结果进行比较。数据集A即采用通用的CE2059和CE2625。数据集B将根据最新的2013年1月发布的uniprotKB/swiss-prot版本进行构建,构建原则参见CE2059和CE2625等通用数据集的建立准则[3,14]:
(1)选择uniprotKB/swiss-prot数据库中清楚明确标示和注释的蛋白质,如出现“fragment”、“probable”、“potential”或“by similarity”的筛除掉;
(2)来自不同物种却同名的蛋白质只入数据集一次;
(3)选择只有唯一类型的蛋白序列入数据集。
经筛选,共选出6 069条膜蛋白序列。其中A.Type I 907 条,B.Type II 273,C.Multipass 4 385 条,D.Lipid Chain 268条,E.GPI 183条,F.Peripheral 53条。以上作为真样本集,相应的假样本集则由除该类型外的其他五组类型数据随机产生,具体分布见表1。
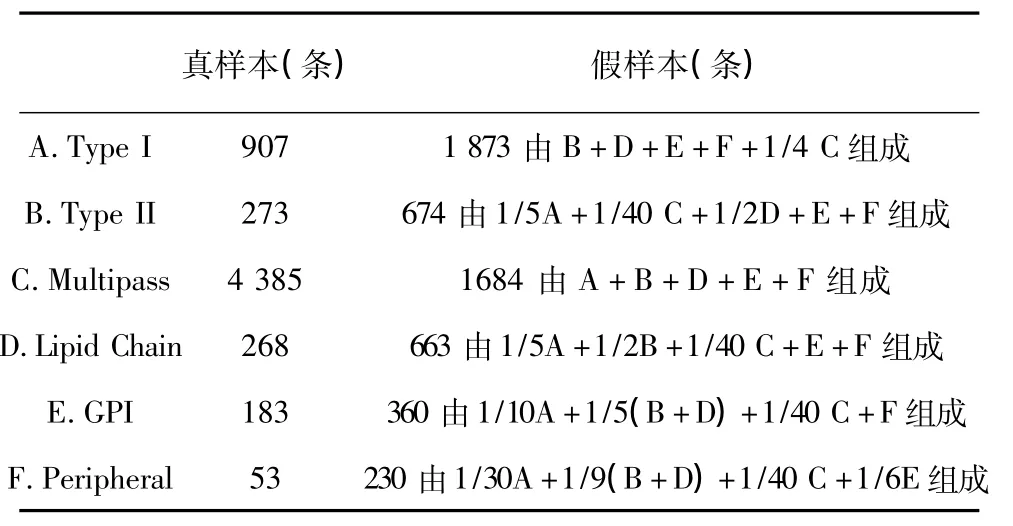
表1 膜蛋白类型数据集Table 1 Database of membrane protein types
1.2 序列特征的提取与转化
1.2.1 由原始序列转换为压缩序列
引入压缩氨基酸的概念,即将原始的20种氨基酸 AA={A,R,N,D,C,Q,E,G,H,I,L,M,K,F,P,S,T,W,Y,V}根据理化性质的不同进行压缩分类,性质相近的归为一类,这样20种氨基酸根据不同的压缩方式[15]形成了不同的压缩种类(见表2)。对表2中所列的11种压缩方式分别进行测试,比较不同的压缩方式对膜蛋白类型识别效果的优劣。

表2 不同的压缩方法对20种氨基酸进行压缩分类Table 2 Compressed alphabets produced by different methods
针对每一种压缩方式,一条原始的由20种氨基酸组成的蛋白质序列,利用压缩的氨基酸转换为压缩序列。
1.2.2 对压缩序列进行氨基酸组分特征提取
蛋白质序列的特征已被普遍用于蛋白质的家族分类、结构预测、信号位点识别等方面,且取得了较好的效果,目前比较常用的序列特征有单氨基酸组成和二肽组成,仅考虑单氨基酸的组成,往往会漏掉许多序列次序信息,二肽的组成分析能很好的补充氨基酸序列之间顺序的特征,考虑了邻近残基之间的耦合作用。通过对压缩序列进行单氨基酸和二肽组成频率的统计,将压缩序列转换为维数固定的特征向量。具体步骤:

其中,Fi表示在压缩序列中氨基酸i的出现频率,Ai表示压缩序列中氨基酸i出现的总次数,n表示压缩序列的长度;Fij表示压缩序列中相邻两个氨基酸ij的出现频率,depij表示压缩序列中相邻两个氨基酸ij出现的总次数,m表示所有两两氨基酸出现的可能组合,N属于表2中所列的11种压缩后的氨基酸种类。最后,根据不同的压缩方式,由公式(1)和公式(2)计算得到的特征向量总维数也是不同的,应为N+N2。
1.3 基于SVM的分类模型构建
支持向量机最大的特点就是泛化能力比较强,即由有限的训练集样本得到的小误差仍能够保证对独立的测试集的小误差,同时也可以防止模型构建过程中问题的产生。以往的研究表明使用支持向量机方法可以很好的对膜蛋白类型进行预测[11-13]。本文采用libsvm3.13软件包[16],选择径向基核函数进行多类分类器的构建,以实现对膜蛋白类型的识别预测。
1.4 五叠交叉验证和评价标准
利用五叠交叉验证的方法随机划分数据集对分类模型进行测试。即将真、假样本数据分别随机分为5个大致相等的子集,依次各取出一个子集合作为测试集,而各自其余4个子集合作为训练集,如此交替反复5次后,将各次的准确度作平均。为了避免随机取样产生的偏好性,将此验证过程重复10次。最后,利用灵敏度(Sensitivity)、特异性(Specificity)和总体准确度(Accuracy)这3个指标来评价模型的性能。具体定义如下:
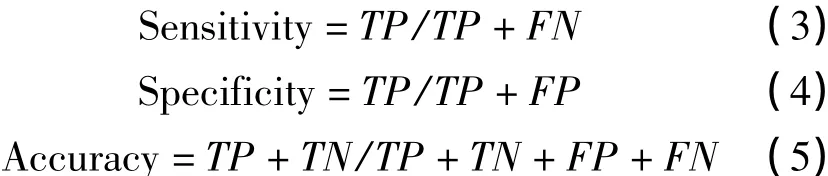
其中,TP为真阳性的数目,TN为真阴性的数目,FP为假阳性的数目,FN为假阴性的数目。
2 结果分析
根据表2所列的不同压缩方法将膜蛋白序列进行压缩,转换为压缩序列;利用单氨基酸和二肽组成的序列信息对序列进行特征提取,根据压缩方式不同最终得到不同维数的特征向量,利用支持向量机(SVM)方法进行分类器构建;采用五叠交叉验证和3个评价指标来衡量不同压缩方法对分类预测结果的影响(见表3)。
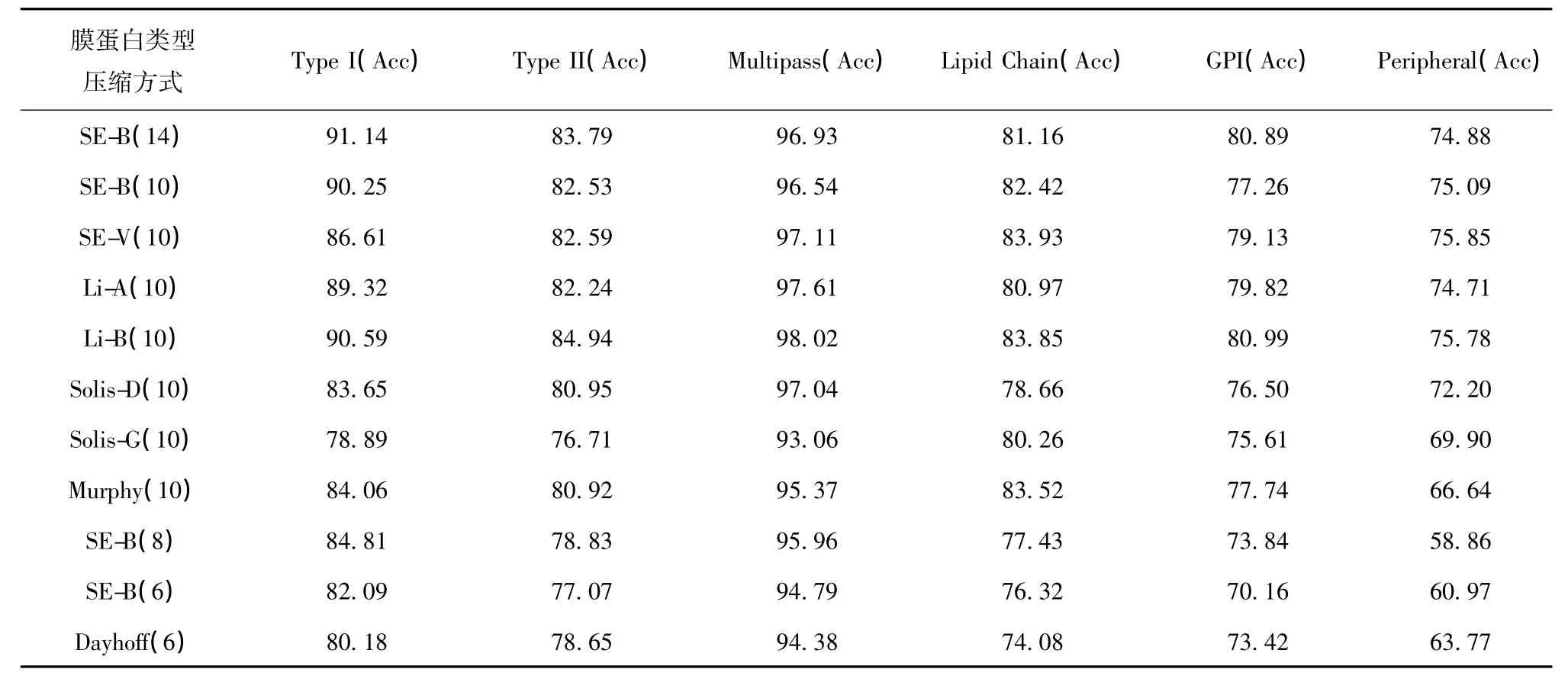
表3 采用不同压缩方法进行分类模型构建的预测结果Table 3 Prediction results of classifiers which construct on different compressed methods
由表3可知,从整体水平来看,采用Li-B(10)的压缩方式可以较好地实现对六种膜蛋白类型的分类。为进一步与以往研究进行比较,我们选取Li-B(10)的压缩方式,再用通用数据集A进行测试(数据集A中只包括5种膜蛋白类型),结果见表4。
由表4结果可知,采用Li-B(10)的压缩方式对通用数据集A进行特征提取同样是有效的,比其他基于数据集A的预测方法效果要好。

表4 采用Li-B(10)的压缩方式对数据集A进行测试Table 4 Test the database A with Li-B(10)compressed method
3 讨论
本研究中采用了与通用数据集CE2059和CE2625同样的构建准则来构建新的膜蛋白类型数据集,与早期通用的数据集 CE2059和CE2625相比,该数据集包含了更为全面的膜蛋白类型(新增的外周蛋白类型)和序列信息,另外在假样本的选取上,我们随机抽取了不同比例的类型数据进行组合,并重复10次随机组成假样本,避免了随机抽样以及假样本过多所引起的结果偏差,有效保证了数据集的全面性与可靠性。其次,有效特征的选取也是成功构建分类器的关键,基于氨基酸组成、氨基酸位置,伪氨基酸以及氨基酸理化性质等特征构建的分类器均取得了较好的分类效果。本研究利用了压缩的氨基酸,将原始序列所包含的信息进行有效压缩,这种方法最早是用在序列比对上,可将序列间的局部相似性最大化,从而发现序列间保守的区域或是鉴定序列的同源性关系等,这里将它应用到分类问题上,再综合氨基酸组成和顺序特征,进行特征提取,由表3和表4结果可知,该方法在膜蛋白类型分类上是有效的。不同的压缩方法得到的结果是有区别的,如对Type I分类预测时,SE-B(14)的压缩方式较好,而SE-V(10)对Lipid Chain和Peripheral的分类效果较好。但从整体上来看,则是Li-B(10)的压缩方式对六种膜蛋白的分类更为合适,平均准确度在85%以上,但对个别类型如Lipid Chain、GPI和Peripheral的分类效果偏低。原因主要有两点:一是这三种类型的数据集所包含的序列数目较少,使如上方法在对该类型进行特征提取时不能很好的体现;二是从类型上分析,Type I、Type II和 Multipass均属于跨膜蛋白,具有跨膜螺旋特征,而Lipid Chain和GPI属于锚定蛋白,还有特殊的一类外周蛋白,这三类与跨膜蛋白差异较大,利用如上方法的特征提取对于跨膜蛋白类型的分类效果较为显著,而对于Lipid Chain、GPI和外周蛋白的区分还需考虑更为有效的特征,如氨基酸的理化性质、序列末端特征等。
4 结论
综上所述,利用压缩的氨基酸结合氨基酸组分和二肽顺序特征来预测膜蛋白类型是一种有效的方法。该方法操作简单,但是仅限于对类型的预测,如要进一步对膜蛋白功能和结构进行分析,还需考虑更多的一些属性特征,挖掘这些特性有待于进一步的研究,为更深入的探讨膜蛋白功能奠定基础。
References)
[1] Oren M.Becker,Yael Marantz,Sharon Shacham,Boaz Inbal,Alexander Heifetz,Ori Kalid,Shay Bar-Haim,Dora Warshaviak,Merav Fichman and Silvia Noiman.G protein coupled receptors:In silico drug discovery in 3D [J].Proceedings of the National Academy of Sciences of the United States of America,2004,101(31):11304-11309.
[2] 张振慧.蛋白质分类问题的特征提取算法研究[D].湖南长沙:国防科学技术大学,2006.
[3] Kuo-Chen Chou,David W.Elrod.Prediction of membrane protein types and subcellular locations[J].Proteins,1999,34(1):137 -153.
[4] Kuo-Chen Chou.Prediction of Protein Cellular Attributes Using Pseudo-amino Acid Com position [J].Proteins,2001,43(3):246-255.
[5] Kuo-Chen Chou,David W.Elrod.Protein Subcellular Locations Prediction[J].Protein Engineering design& selection,1999,12(2):107-118.
[6] Hong-Bin Shen,Kuo-Chen Chou.Using optimized evidence theoretic K-nearest neighbor classifier and pseudo-amino acid composition to predict membrane protein types[J].Biochemical and Biophysical Research Communications,2005,334(1):288 -292.
[7] Hong-Bin Shen,Jie Yang,Kuo-Chen Chou.Fuzzy KNN for predicting membrane protein types from pseudo-amino acid composition [J].Journal of Theoretical Biology,2006,240(1):9 -13.
[8] Kuo-Chen Chou,Hong-Bin Shen.MemType-2L:a web server for predicting membrane proteins and their types by incorporating evolution information through Pse-PSSM[J].Biochemical and Biophysical Research Communications,2007,360(2):339 -345.
[9] Zhi-Ping Feng,Chun-Ting Zhang.Prediction of membrane protein types based on the hydrop-hobic index of amino acids[J].Journal of Protein Chemistry,2000,19(4):269 -275.
[10] Yu-Dong Cai,Xiao-Jun Liu,Xue-Biao Xu and Kuo-Chen Chou.SVM for predicting membrane protein types by incorporating quasisequence-order effect[J].Internet Electronic Journal of Molecular Design,2002,1(4):219 -226.
[11] Yu-Dong Cai,Guo-Ping Zhou and Kuo-Chen Chou.Support vector machines for predicting membrane protein types by using functional domain composition[J].Biophysical Journal,2003,84(5):3257-3263.
[12] Xiao-Guang Yang,Rui-Yan Luo and Zhi-Ping Feng.Using amino acid and peptide composition to predict membrane protein types[J].Biochemical and Biophysical Research Communications,2007,353(1):164-169.
[13]姜彬,王正华,王勇献,贺细平.多特征融合提取算法结合支持向量机预测膜蛋白类型[J].上海交通大学学报,2009,7:1172-1176.
[14]曾聪.蛋白分类的特征提取算法和数据集构建技术研究[D].湖南长沙:国防科学技术大学,2010.
[15] Robert C.Edgar.Local homology recognition and distance measures in linear time using compressed amino acid alphabets[J].Nucleic Acids Research,2004,32(1):380 -385.
[16] Chih-Chung Chang and Chih-Jen Lin.LIBSVM:a library for support vector machines[J].ACM Transactions on Intelligent Systems and Technology,2011,2(3):1 -27.

