我国近海中度嗜盐菌的分离筛选及其产酶多样性分析
产竹华,刘洋,王昭凯,钟添华,曾润颖*
(1.国家海洋局第三海洋研究所海洋生物遗传资源重点实验室,福建厦门361005)
1 引言
中度嗜盐菌(Moderately halophilic bacteria)指一类能够在NaCl的质量分数为3%~15%之间最适生长能力的极端微生物类群[1-2],广泛生活于近海、盐湖、盐场、高盐土壤和盐渍食物等环境[3]。由于中度嗜盐菌独特的生理生化特性,易于在高盐环境下生长及可以利用多种有机物作为碳源和氮源,决定了其在酶制剂生产、食品工业、污水处理等方面的具有可能的应用价值[4-5]。
嗜盐菌由于其独特遗传背景和代谢途径,使其产生的生物酶在食品、化工、环保等领域有重大的应用潜力,因此其逐渐成为研究的热点。Sanchez等[6]从几百株中度嗜盐菌得到多种胞外水解酶并对酶活性进行了详细的研究,同时,他们也分离得到高产蛋白酶的中度嗜盐菌[7]。Mohammad等[8]从中度嗜盐菌Salinivibriosp.AF-2004中得到分泌性的碱性金属蛋白酶,并进行了分离纯化,它具有较宽的pH生长范围和盐耐受性,为它的应用奠定了基础[9]。Hiroko Tokunaga等[10]研究了由中度嗜盐菌中的134个氨基酸组成的二聚体-核酸二磷酸激酶,并对它的结构进行了分析。Moozegar等[11]人对Halobacillus sp.MA-2在高温及高盐条件下分泌的淀粉酶做了详细的研究。这些工作极大地丰富了我们对中度嗜盐菌产酶的认识。
本文从天津滨海、山东东营、江苏连云港、福建厦门、海南三亚等地近海采集样品,富集、分离筛选中度嗜盐菌。利用底物特异性实验分析了嗜盐菌株的产酶多样性。通过16SrRNA基因的系统发育分析产酶嗜盐菌株的分类地位,从而可以了解各菌株的系统进化和产酶多样性,为进一步研究嗜盐微生物资源提供了参考。
2 材料与方法
2.1 试验样品
从天津滨海(39°06′N,117°43′E)、山东东营(37°32′N,118°55′E)、江苏连云港(34°49′N,119°14′E)、福建厦门(24°32′N,118°22′E)、海南三亚(18°12′N,109°28′E)等地近海采集样品。样品放置4℃冰箱保存,然后进行菌株的筛选。
2.2 基础培养基
中度嗜盐菌富集,筛选培养基(g/L):NaCl 100,葡萄糖10,酵母粉10,蛋白胨5,KCl 2.0,MgSO4· 7H2O 1.0,CaCl20.36,NaHCO30.06,FeCl30.001,pH 7.4~7.6。固体培养基加入琼脂15~20。
基本培养基(g/L):NaCl 100,KCl 2.0,MgSO4· 7H2O 1.0,CaCl20.36,FeCl30.001,pH 7.4~7.6。固体培养基加入琼脂15~20。
发酵培养基(g/L):NaCl 100,葡萄糖10,蛋白胨10,酵母粉5。
2.3 实验方法
2.3.1 菌株的富集和分离纯化
称取样品(土样或水样)1g或1mL置于250mL装液量50mL筛选培养基的三角瓶中,37℃、200r/min摇床培养3~5d,富集菌体;然后取富集的菌液逐级稀释从10-2到10-8,取2~3个梯度涂平板;37℃培养箱培养2~3d长出单菌落,挑取单菌落划线、培养,直到得到纯培养菌株。用斜面划线试管和-70℃甘油管两种方法保存。
2.3.2 菌落特征
划线接种于嗜盐培养基,37℃培养2~3d,观察单菌落的形状、大小、透明度、颜色、边缘和表面特征。
2.3.3 菌体特征
采用革兰氏染色法和芽孢染色法[12-13],通过光学显微镜和透射电子显微镜对菌体形态进行观察。
2.3.4 部分生理生化特征
依据《常见细菌系统鉴定手册》[14]和《伯杰氏细菌鉴定手册(第9版)》[15]进行常规生化鉴定。
2.3.5 16SrRNA基因扩增和系统进化分析
使用BioTeke试剂盒提取细菌总DNA。以细菌总DNA为模板,用16SrRNA通用引物27F(5′-AGAGTTTGATCCTGGCTCAG-3′),1492R(5′-GGTTACCTTGTTACGACTT-3′)进行PCR扩增菌株16SrRNA[16]。PCR产物送交到Invitrogen公司测定。测序得到的16SrRNA基因序列通过BLASTN(http://www.ncbi.nlm.nih.gov/BLAST)进行比对。
2.3.6 构建系统发育树
提交各嗜盐菌株的16SrRNA基因序列在EMBL数据库中进行比对,选取与之相似性较高的模式种的16SrRNA基因序列,采用MEGA4.0软件进行系统发育分析,采用Neighbor-Joining法构建系统发育树,Bootstrap1000次进行稳定性验证。
2.4 产酶中度嗜盐菌的筛选
2.4.1 产蛋白酶中度嗜盐菌的筛选
基础培养基中加入1%脱脂奶粉,制成平板。在平板上划线中度嗜盐菌株,37℃培养1周后观察有无透明圈出现[17]。
2.4.2 产淀粉酶中度嗜盐菌的筛选
基础培养基中加入1%可溶性淀粉,制成平板。在平板上点种嗜盐菌株,37℃培养1周后用路戈氏碘液检验,观察有无透明圈出现[18]。
2.4.3 产酯酶中度嗜盐菌的筛选
基础培养基中加入1%Tween80,制成平板。在平板上划线中度嗜盐菌株,37℃培养1周后观察有无白色晕圈[19]。
2.4.4 产纤维素酶中度嗜盐菌的筛选
基础培养基中加入1%羧甲基纤维素钠和0.2%刚果红,制成平板。在平板上点种嗜盐菌株,37℃培养观察有无透明圈出现[20]。
2.5 酶活力测定方法
(1)蛋白酶活力测定方法:参照中华人民共和国专业标准《蛋白酶活力测定方法》SB/T10317-1999进行测定[21]。
(2)淀粉酶活力的测定:采用YoungJ.Y00改良法[22]。
(3)酯酶活力测定法:采用改进过的α-乙酸萘酯比色法[23-25]测定酶活。
(4)纤维素酶活力测定参照文献[26-27],酶活力按照国际单位规定定义为:每分钟催化纤维素水解生成1μmol葡萄糖的酶量为1个酶活力单位U。
3 结果与分析
3.1 部分生理生化特征
通过对天津、三亚、连云港、厦门、东营等地近海和盐田的样品进行富集、分离纯化。得到26株中度嗜盐菌,生长3~4d后,菌落均为干燥或湿润,大多菌株的菌落为白色,其次为黄色。菌株形态和菌落颜色具有多样性。革兰氏染色显示革兰氏阳性菌占绝对优势,各株菌的部分生理生化特征见表1。
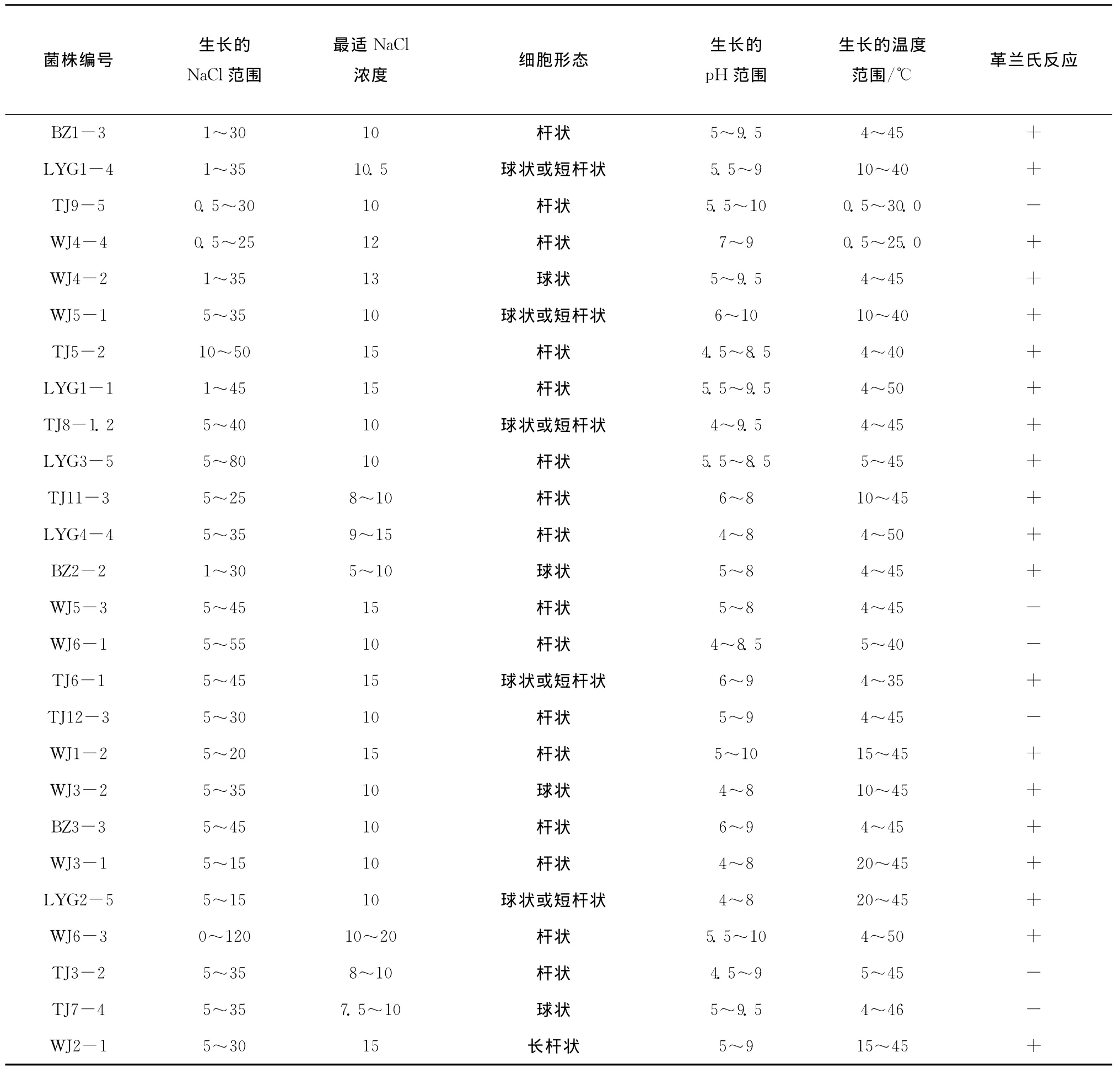
表1 中度嗜盐菌的部分生理生化性质
3.2 中度嗜盐菌产酶多样性
经过筛选得到26株产酶嗜盐菌株,分别对26株菌株进行底物特异性试验,验证了菌株的产酶情况。发现13株菌产蛋白酶,其中LYG1-4、WJ5-1、WJ5-3、TJ5-2、TJ7-4和BZ2-2产蛋白酶能力较强;19株菌产淀粉酶,其中TJ6-1、TJ8-1产酶能力较强;13株菌产酯酶,其中TJ3-2、TJ9-5、TJ12-3、LYG1-1、LYG2-5、LYG3-5产酶能力较强;4株菌产纤维素酶,分别属于Virgibacillus、Halobacillus、 Halomonas等属。从表2中可以看出,没有一株菌可以同时产4种酶,有6株菌同时产3种酶,有11株菌能产2种酶,表明了中度嗜盐菌产酶的多样性。
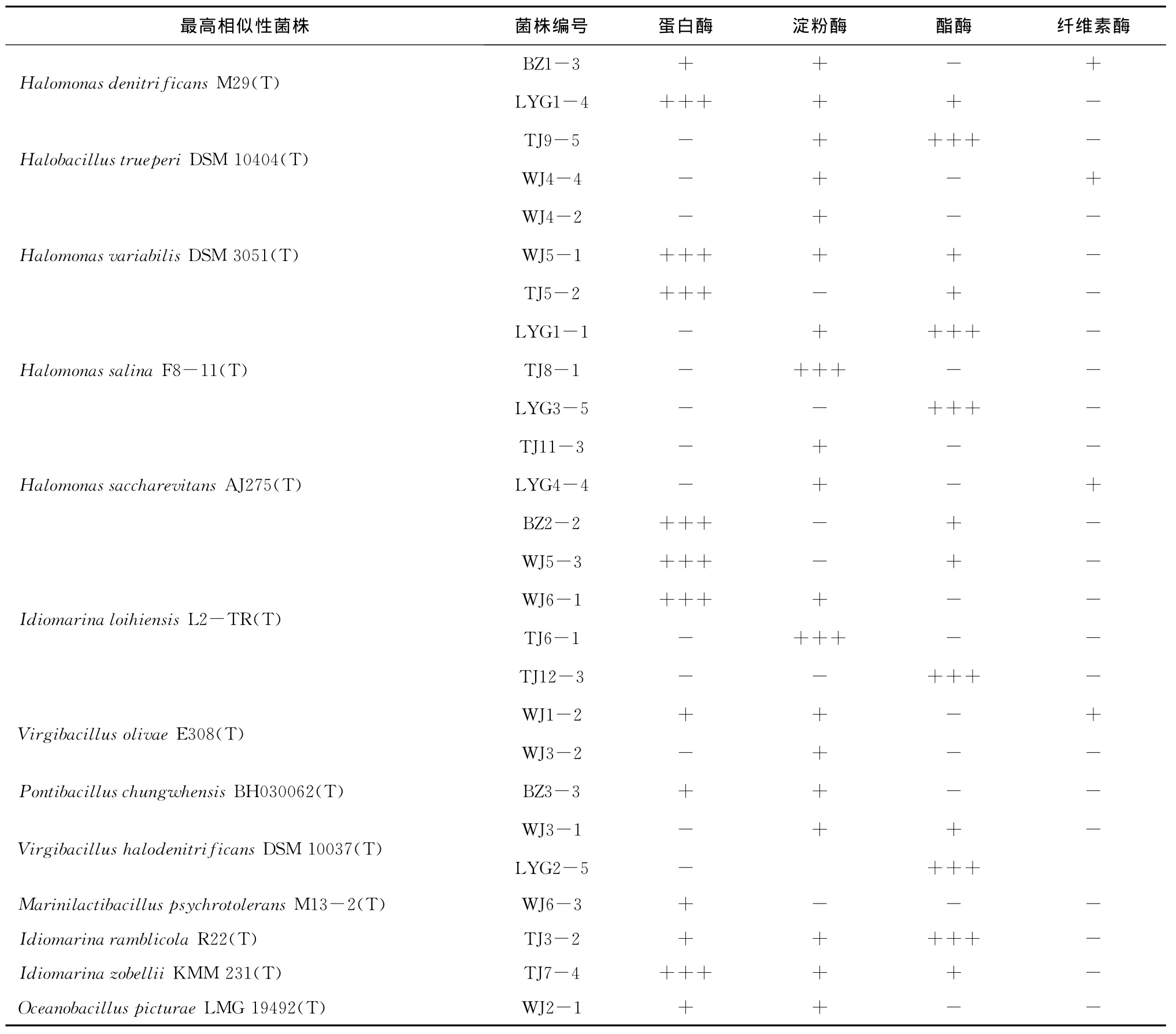
表2 中度嗜盐菌产酶多样性
3.3 嗜盐菌产酶分析
3.3.1 中度嗜盐菌株产蛋白酶
虽然蛋白酶的种类很多,而且有些已在工业生产中应用。但应用环境往往会影响到酶的活力,特别是高盐环境下的活力。因而需要适应于广谱pH、温度、盐度的酶。中度嗜盐菌特殊的生理生化条件决定了其所产蛋白酶的特殊性,耐盐、较宽的pH和温度范围菌保持较高活性。本工作中筛选到产蛋白酶的中度嗜盐菌有13株,占产酶菌株总量的50%。如图1显示,在脱脂奶粉平板上生长的中度嗜盐菌生长1周后,菌落周围出现透明圈,说明这些菌株能产生蛋白酶。通过摇瓶实验,测定产酶菌株的酶活(3组重复取平均),结果表明LYG1-4、WJ5-1、WJ5-3、TJ5-2、TJ7-4和BZ2-2产蛋白酶能力较强,其中BZ2-2菌株酶活力最高,达到765U/g。
3.3.2 中度嗜盐菌株产淀粉酶
图2显示,在淀粉平板上生长的中度嗜盐菌生长1周后,将碘液滴在菌苔上,如果周围无蓝色,而对照仍然为蓝色,说明该菌株能产生淀粉酶(图2)。淀粉酶都需要特定的温度、pH值和盐度范围,故对淀粉酶活较高的中度嗜盐菌进行分离和鉴定,有助于发现新的淀粉酶来适应新的工业需要。通过摇瓶实验,测定产酶菌株的酶活(3组重复取平均),结果表明TJ6-1、TJ8-1产酶能力较强,其中菌株TJ6-1酶活力达到387U/g。
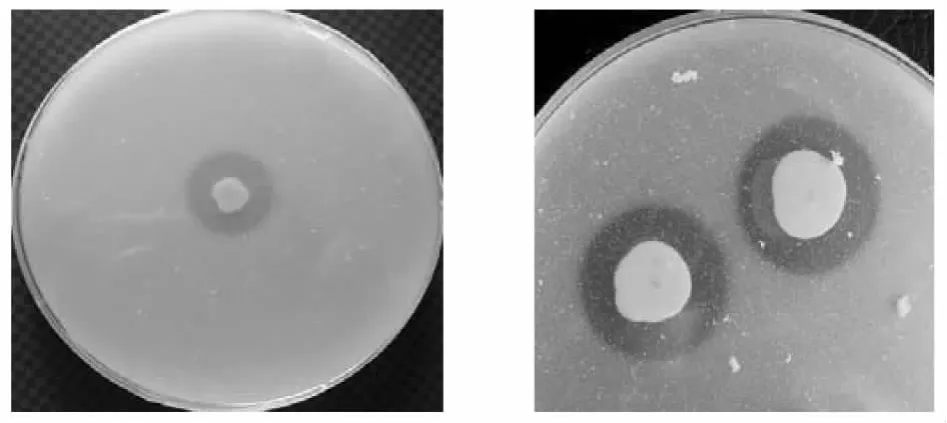
图1 菌株产蛋白酶的照片
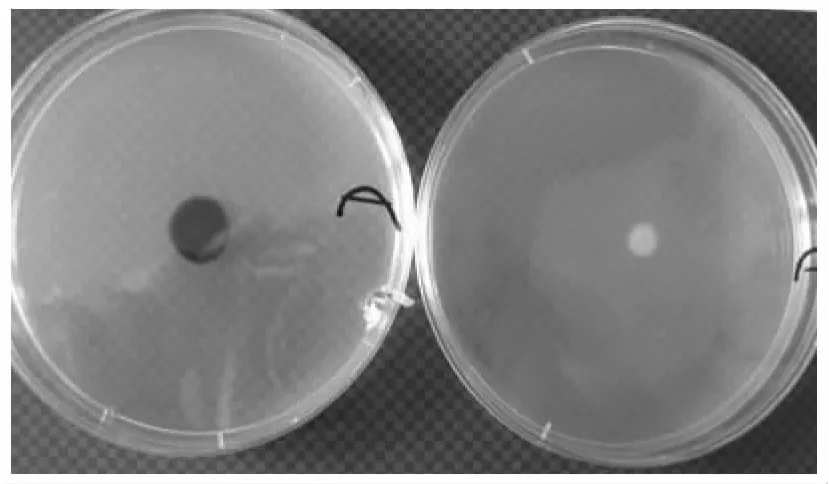
图2 菌株产淀粉酶(左:对照,右:产淀粉酶菌株)
3.3.3 中度嗜盐菌株产酯酶
在酯酶筛选平板上点种嗜盐菌株,培养一定时间后发现有些生长的菌株周围产生白色晕圈,说明该菌株产生酯酶(图3)。酯酶菌株的获得说明中度嗜盐菌的产酶具有多样性,也为更好的利用中度嗜盐菌提供了参考。从图中可以看出,菌株在固体平板上产酯酶的情况较好,明显出现白色晕圈。通过摇瓶实验,测定胞外和胞内酶活(3组重复取平均),该酶大部分属于胞外酶。结果表明TJ3-2、TJ9-5、TJ12-3、LYG1-1、LYG2-5、LYG3-5产酶能力较强,其中菌株LYG3-5的酶活力达到582U/g。
3.3.4 中度嗜盐菌株产纤维素酶
在纤维素的平板上点种嗜盐菌株,结果发现只有4株菌能够生长。进一步通过摇瓶实验,测定纤维素酶活力(3组重复取平均),分别为248U/g、476U/g、207U/g、356U/g,说明这4株菌产纤维素酶,分别属于Virgibacillus,Halobacillus,Halomonas属,产纤维素酶的嗜盐菌株占总菌数不到20%。
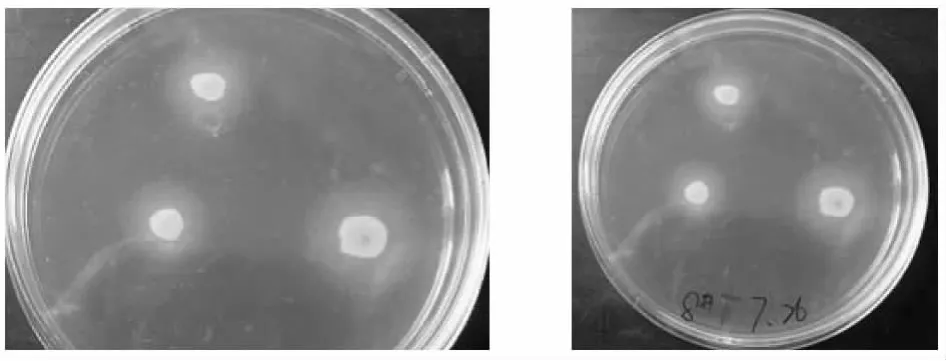
图3 菌株产酯酶的情况
3.4 系统进化分析
经过筛选,得到了至少产一种胞外酶的26株嗜盐菌,其中有11株与盐单胞菌属的细菌相近,占分离细菌总数的42.31%,其次有6株与Idiomarina属的细菌相近,是占优势的类群。16SrRNA基因的系统发育分析表明大部分菌株的进化地位相当明确(见图4),26株菌分别属于7个属(Halomonas,Oceanobacillus,Idiomarina,Virgibacillus,Pontibacillus,Halobacillus和Marinilactibacillus)的13个不同的种,系统进化地位不等,表明中度嗜盐菌分布的多样性和广泛性,以及资源的丰富性,为嗜盐微生物以及嗜盐酶的研究提供了参考。
4 讨论
4.1 中度嗜盐菌系统发育多样性
通过对天津、三亚、连云港、厦门、东营等地近海或盐田的样品进行分离筛选和鉴定,得到26株产酶菌株,分别属于13个种,7个属。其中WJ5-1,TJ3-2分别属于Halomonas,Idiomarina属,与各属中的模式菌株Halomonas variabilis DSM 3051T,Idiomarina zobellii KMM 231T的16SrRNA基因同源性分别为97.62%、97.83%,可能代表该属内的新种,要确定该菌是否为新种需通过GC含量、DNA分子杂交等实验进一步分析和鉴定[28]。表明我国近海中度嗜盐菌分布多样性以及系统进化多样性。
4.2 中度嗜盐菌产酶多样性
对嗜盐菌株进行底物特异性试验表明,筛选到的菌株没有一株可以同时产4种酶,有6株菌同时产3种酶,有11株菌能产2种酶。通过嗜盐产酶情况分析,表明我国近海中度嗜盐菌产酶的多样性。经过性质研究发现,大多数嗜盐酶活力最适盐度为10%,且在NaCl浓度为20%、pH 10和温度20~55℃条件下均有活性,且活性都保持在50%以上。嗜盐酶独特的生理特性将为嗜盐菌在工业应用方面提供重要参考。

图4 中度嗜盐菌菌株基于16SrRNA基因序列相似性构建的系统发育树
4.3 中度嗜盐菌酶的应用分析
随着工业的发展,工业废水的污染越来越严重,电子废水、造纸废水、高盐废水的治理需要更高的成本。中度嗜盐菌酶具有能在高盐、较高pH以及较宽温度均有活性,为工业废水的治理提供了一个新的思路。因此,开发出高酶活的嗜盐菌酶和能较好治理工业废水的嗜盐菌酶是下一步的研究重点。
[1] Wohlfarth A,Erwin J S,Galinki E A.The spectrum of compatible solutes in heterotrophic halophilic eubacteria of the family Halomonadaceae[J].Gen Microbiol,1990,136(44):705—712.
[2] Poli A,Esposito E,Orlando P,et al.Halomonas alkaliantarcticasp.nov.,isolated from saline lake Cape Russell in Antarctica,an alkalophilic moderately halophilic,exopolysaccharide-producing bacterium[J].Syst Appl Microbiol,2007,30(1):31—38.
[3] 任培根,周培瑾.中度嗜盐菌的研究进展[J].微生物学报,2003,43(3):427—431.
[4] 赵百锁,杨礼富,宋蕾,等.中度嗜盐菌在生物技术中的应用[J].微生物学通报,2007,32(2):359—362.
[5] 刘铁汉,周培瑾.嗜盐微生物[J].微生物学通报,1999,26(3):232—235.
[6] Sanchez P C,Martin S,Mellado E,et al.Diversity of moderately halophilic bacteria producing extracellular hydrolytic enzymes[J].Journal of Applied Microbiology,2003,94,295—300.
[7] Sanchez P C,Mellado E,Bertoldo C,et al.Screening and characterization of the proteinase CP1produced by the moderately halophilic bacterium Pseudoalterpmonas sp.CP76[J].Extremophiles,2003,7(3):221—228.
[8] Mohammad A A,Azadeh Z F,Hamid R K,et al.Production of an extracellular alkaline metalloprotease from a newly isolated,moderately halophile,Salinivibriosp.AF-2004[J].Microbiological Research,2007,162:369—377.
[9] Hamid R K,Abed A Z,Johann S,et al.Purification and characterization of an extracellular haloalkaline protease produced by the moderately halophilic bacterium,Salinivibriosp.AF-2004[J].Enzyme and Microbial Technology,2007,40:266—272.
[10] Hiroko T,Matsujiro I,Fumio A,et al.Residue 134determines the dimer-tetramer assembly of nucleoside diphosphate kinase from moderately halophilic bacteria[J].FEBS Letters,2008,582:1049—1054.
[11] Amoozegara M A,Malekzadeha F,Khursheed A.Production of amylase by newly isolated moderate halophile,Halobacillus sp.strain MA-2[J].Journal of Microbiological Methods,2003,52:353—359.
[12] Smibert R M,Krieg N R.Phenotypic characterization In Methods for General and Molecular Bacteriology[J].American Society for Microbiology,1994:607—654.
[13] Ventosa A,Quesada E,Francisco R V,et al.Numerical taxonomy of moderately halophilic Gram-negative rods[J].J Gen Microbiol,1982,128:1959—1968.
[14] 东秀珠,蔡妙英.常见细菌系统鉴定手册[M].北京:科学出版社,2001.
[15] 布坎南,吉本斯.伯杰氏细菌鉴定手册(第8版)[M].北京:科学出版社,1984.
[16] Wilson K H,Blitchington R B,Greene R C.Amplification of bacterial 16Sribosomal DNA with polymerase chain reaction[J].Journal of Clinical Microbiology,1990,28:1942—1946.
[17] Tang X Y,Pan Y,Li S,et al.Screening and isolation of an organic solventtolerant bacterium for high-yield production of organic solvent-stable protease[J].Bioresource Technol,2008,99(15):7388—7392.
[18] Rajendra K S,Kakoli D,Lata A,et al.A highly thermostable and alkaline amylase from a Bacillus sp.PN5[J].Original Research Article Bioresource Technology,2007,98(2):260—265.
[19] Geun J K,Gi S C,Ji Y K,et al.Screening,production and properties of a stereospecific esterase fromPseudomonas sp.S34with high selectivity to(S)-ketoprofen ethyl ester[J].Original Research Article Journal of Molecular Catalysis B:Enzymatic,2002,17(1):29—38.
[20] Kasana R C,Salwan R,Dhar H,et al.A rapid and easy method for the detection of microbial cellulase on agar plate using gram's iodine[J].Current Microbiology,2008,57:503—507.
[21] SB/T10317-1999,蛋白酶活力测定方法[S].
[22] Guinee P A,Jansen W H,Maas H M,et al.An unusual Hantigen(z66)in strains of Salmonella typhi[J].Ann Microbiol(Paris),1981,132:331—334.
[23] 禹邦超,邓辉胜,王旭,等.多种形式酯酶总活力的混合底物分光光度测定法[J].华中师范大学学报(自然科学版),1996,30(1):87—91.
[24] Lanz W W,Williams P P.Characterization of esterases produced by a ruminal bacterium identified as Butyrivibrio fibrisolvens[J].J Bacteriol,1973,113(3):1170—1176.
[25] Hespell R B.Esterase activities in Butyrivibrio fibrisolvens strains[J].Appl EnvironMicrobiol,1988,54(8):1917—1922.
[26] Hardin M T,Mitchell D A,Howes T.Approach to designing rotating drum bioreactors for solid state fermentation on the basis of dimensionless design factors[J].Biotechnology and Bioengineering,2000,67(3):274—282.
[27] Ghose T K.Measurement of cellulase activities[J].Pure and App Chem,1987,59(2):257—268.
[28] Ripka K,Denner E,Michaelsen A,et al.Molecular characterisation of Halobacillus strains isolated from different medieval wall paintings and building materials in Austria[J].Int Biodeterior Biodegrad,2006,58:124—132.
