川西北牧区传统发酵牦牛酸奶中发酵毕赤酵母菌的分离鉴定
王远微,张诚民,索化夷,岳 华,李 键,汤 承,*(.西南民族大学生命科学与技术学院,四川成都6004;.西南大学食品科学学院,重庆40075)
川西北牧区传统发酵牦牛酸奶中发酵毕赤酵母菌的分离鉴定
王远微1,张诚民1,索化夷2,岳 华1,李 键1,汤 承1,*
(1.西南民族大学生命科学与技术学院,四川成都610041;
2.西南大学食品科学学院,重庆400715)
对川西北部分牧区的10份传统发酵牦牛酸奶样品进行酵母菌的分离,通过常规形态特征和26S rRNA基因测序分析鉴定出6株发酵毕赤酵母菌(Pichia fermentans)。同源性分析显示6株分离菌与已知发酵毕赤酵母菌的同源性高达99.7%~100%。实验结果为该地区传统发酵牦牛酸奶中微生物组成多样性研究以及发酵毕赤酵母菌遗传多样性研究提供参考。
传统发酵牦牛酸奶,发酵毕赤酵母菌,分离鉴定
传统发酵牦牛酸奶是青藏高原地区牧民采用传统方法制作而成,营养价值很高,蛋白质和脂肪含量分别达到4.91%和6.89%[1-4],且含有大量的挥发性脂肪酸[5],并富有良好的风味。其在维持肠道菌群生态平衡、促进肠道蠕动、促进消化、抗衰老、抗癌、提高人体免疫力等方面有很好的效果[6]。它是牧区非常传统的奶制食品,也是牧民重要的经济收入来源。
牦牛酸奶发酵过程受海拔地理环境、气候环境、发酵温度、发酵时间、制作方法、文化及奶源的影响,因此微生物菌群非常复杂。张和平等[3]报道青海西北部的高原牦牛酸奶和青海东部的环湖牦牛酸奶中乳酸菌是优势菌,而青海南部的环湖牦牛酸奶中酵母菌是优势菌。Xiao-He Wu等[4]发现3种乳酸菌以及5种酵母菌是西藏不同海拔地区牦牛酸奶中主要的发酵菌群,其中发酵乳杆菌是优势菌。Koichi[7]等发现蒙古牦牛酸奶中乳酸菌量明显高于酵母菌的量。牦牛酸奶中的微生物菌群尽管比较复杂,但其主要组成是乳酸菌和酵母菌[8],只是不同地区优势菌的种类不同。随着对传统发酵乳制品中酵母菌研究的深入,越来越多的酵母菌种属在各种乳制品中被检测出来。发酵毕赤酵母也是其中之一,在开菲尔酸奶、中国酸马奶、西藏的曲拉、台北的酸奶中都相继发现了该菌[9-12]。张和平和阚建全、Mei Bai等[13]也分别从青海地区和西藏地区传统发酵牦牛酸奶中发现该菌,其中张和平等发现青海地区牦牛酸奶中发酵毕赤酵母的分离率达到43.2%,是青海地区酸牦牛奶中的优势菌。但对于川西北高原牧区传统发酵牦牛酸奶中的发酵毕赤酵母的分离报道比较少。
本研究对川西北地区的红原、若尔盖、康定等地的10份传统发酵牦牛酸奶样品进行酵母菌的分离鉴定,并对分离菌进行了系统进化分析,以期对川西北高原地区传统发酵牦牛酸奶中蕴含的大量原始微生物菌种资源的保护及研究提供菌株,为该地区传统发酵牦牛酸奶中微生物组成多样性研究和发酵毕赤酵母的遗传多样性研究提供一定参考。
1 材料与方法
1.1 材料与仪器
样品采集及储存运输 样本采自川西北地区的四川省阿坝洲红原县(3份)、若尔盖县(3份)和甘孜州康定县(4份),样本置于4℃样品箱运输到实验室,立即进行分离培养;YPD培养基、革兰氏染液 杭州微生物制剂有限公司;Taq DNA聚合酶、PCR试剂、DNA Markers、dNTPs 购自TaKaRa公司;琼脂糖 Oxoid公司;细菌基因组DNA提取试剂盒 TIANGEN公司;AXY prepTM DNA Gel extraction kitAXYGEN公司;
恒温培养箱 上海齐欣公司;centrifuge 5804高速离心机 德国Eppendorf公司;my cyclerTMPCR仪、核酸电泳仪powerpac universalTM、VerSa Doc2000凝胶成像系统 美国Bio-Rad公司;Milli-Q超纯水仪 法国Millipore公司;移液器 德国Eppendorf公司;LD2X-30KA立式电热压力蒸汽灭菌锅 上海申安医疗器械厂;HR40-ⅡA2生物安全柜 青岛海尔特种电器有限公司;AR2130/C精密电子天平 梅特勒-托利多仪器(上海)有限公司。
1.2 实验方法
1.2.1 酵母菌的分离与纯化 分别吸取1mL牦牛酸奶置于装有9mL无菌生理盐水的试管中,充分振荡使其混匀。将混合后牦牛酸奶进行倍比稀释,稀释度为10-1、10-2、10-3、10-4、10-5、10-6、10-7,选取10-3、10-4、10-5、10-6、10-75个稀释度,用移液器吸取1mL样液,采用涂布法,将样液均匀涂布于YPD培养基上,将制备好平板置于28℃恒温培养箱中培养24~48h,观察并记录菌落特征,挑取菌落较大,且呈白色或乳白色的单个菌落进行革兰氏染色,观察细胞形态,呈圆形或椭圆形、腊肠状且细胞较大者,将其再接种于YPD培养基,于28℃恒温培养箱中培养24~48h,重复几次纯化酵母菌,直至镜检结果为同一细胞形态后,将其接种于YPD液体培养基中,于28℃恒温培养24~48h后,加入30%的灭菌甘油,混匀后置于-80℃保存备用。
1.2.2 形态结构观察 将上述纯培养菌,接种于YPD培养基斜面,置28℃恒温培养18h后涂片,经革兰氏染色后镜检细菌形态。
1.2.3 酵母菌的26S rRNA序列测定与系统发育学分析
1.2.3.1 PCR模板DNA的制备 将上述纯化的酵母菌接种于YPD液体培养基中置于28℃振荡培养24h,取培养液按照细菌基因组DNA提取试剂盒的操作说明提取细菌基因组DNA,作为本实验的PCR模板。
1.2.3.2 PCR引物 PCR扩增目的片段在酵母菌的大亚基26S rRNA的D1/D2可变区域内,引物序列为NL-1:5’-GCATATCAATAAGCGGAGGAAAAG-3’、NL-4:5’-GGTCCGTGTTTCAAGACGG-3’[14],扩增的目的片段大小在500~600bp之间,引物由上海生工合成。
1.2.3.3 PCR方法 PCR反应采用25μL反应体系,其中含DNA模板1μL,上下游引物各1μL(10pmol),4× dNTPs(10mmol/L)2μL,Taq酶(5U/μL)0.125μL,Mg2+3μL(10mol/mL),10×PCR buffer 2.5μL,用超纯水补足至25μL。PCR程序如下:95°C预变性5min,94℃1min,52℃1min,72℃1.5min,36个循环,再72℃温浴10min,降至室温后使用2%琼脂糖凝胶电泳检测。
1.2.3.4 目的片段的纯化回收及测序 按照1.2.3.3的反应体系和反应条件扩增100μL PCR产物,2%琼脂糖凝胶电泳后按照AXYGEN胶回收试剂盒操作说明进行目的片段的纯化回收,回收后再2%琼脂糖凝胶电泳检测。将电泳检测后的回收产物送上海生工进行测序。
1.2.3.5 序列分析与系统发生树构建 将上述测序得到的26S rRNA基因序列通过NCBI的Blast检索系统(http://www.ncbi.nlm.nih.gov/Blast/)进行序列同源性分析,选择同源性大于99%的10个酵母序列的片段,同时调取GenBank数据库中收录的克鲁维菌属的18个种的32株酵母的相同区段基因序列,使用ClustalX1.83软件进行多序列匹配比对(Multiple Alignments),通过Mega4.1软件采用neighbor-joining法构建分离酵母的同源序列系统进化树和克鲁维酵母属内系统进化树,采用自举分析(Boot strap)进行置信度检测,自举数据集为3000次。
2 结果与分析
2.1 酵母菌分离菌株的菌落形态及细胞形态特征
从四川红原、若尔盖、康定等地的10份传统发酵牦牛酸奶样品中分离到的6个酵母分离株,菌落形态呈圆形,表面光滑湿润,颜色呈乳白色,表面凸起,边缘整齐,不透明。显微镜下细胞染色为蓝紫色,形态为圆、椭圆、卵圆等,无性繁殖为芽殖,一端出芽,细胞形态符合酵母菌特征,见图1。

图1 分离菌的细胞形态(1000×)Fig.1 Cell morphology of isolates(1000×)
2.2 酵母菌26S rRNA序列PCR扩增结果
按照PCR反应条件进行扩增,经琼脂糖电泳检测结果显示酵母26S rRNA基因PCR扩增产物在约587bp左右处出现一条明亮的条带,与预期片段大小相符,证明PCR扩增正确,见图2。
2.3 酵母菌26S rRNA基因测序同源比对及系统发育分析结果
经26S rRNA基因序列测定,扩增序列长度为586~ 588bp,与Genbank中同源性较高的菌株进行同源性比对。结果显示分离的6株酵母菌与Genbank中发酵毕赤酵母的同源性最高,相似性为99.7%~100%,见图3。
系统发育分析显示,6株分离酵母菌与Genbank中的发酵毕赤酵母菌序列独立于毕赤酵母属的其他种酵母形成一个大的分支,发酵毕赤酵母菌大分支中形成3个小的分支,6株分离酵母菌分列其中两个小的分支中,见图4。
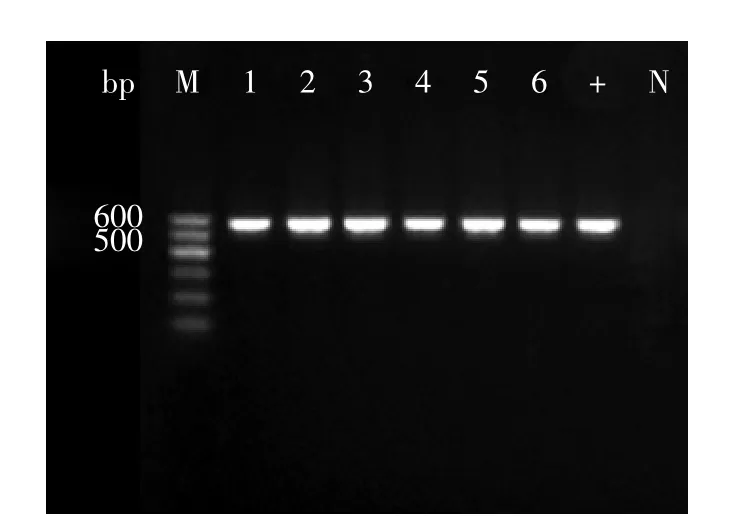
图2 分离菌的26S rRNA基因的PCR扩增结果Fig.2 PCR amplification of 26S rRNA gene of isolates

图3 分离株与发酵毕赤酵母的26S rRNA基因序列的同源性比较结果Fig.3 Sequence homology comparison of 26S rRNA gene sequences of Pichia fermentans and isolates
3 讨论
Kuttzman&Robnett测定了大约500种酵母的大亚基26S rRNA基因的D1/D2可变区的序列,包括假丝酵母和其他有性型及无性型子囊菌酵母几乎所有已知种的模式菌株,发现用这段序列(500~600bp)可以将绝大部分种区分开,同一种内不同菌株间D2区的核苷酸差异不超过1%[14]。这些序列均已公布在GenBank=等国际核酸序列数据库中,给酵母菌的分子鉴定提供了很大方便。本研究扩增了6个分离菌的26S rRNA基因的D1/D2可变区进行测序分析,对分离菌进行分子鉴定。结果发现6株酵母菌与发酵毕赤酵母的同源性最高,相似性为99.7%~100%,按照Kuttzman的同一种内不同菌株间D2区的核苷酸差异不超过1%的结论,所以将6株分离菌鉴定为发酵毕赤酵母菌。本研究为川西北高原地区传统发酵牦牛酸奶中原始微生物菌种资源的保护及研究提供了菌株。
26SrRNA基因是核糖体RNA基因中的一员,具有进化速率慢,序列保守性高的特点,但同时由于各种原因其基因序列中又具有一定的突变,而这些突变的序列具有种属的差异,使其作为一种良好的生物分类鉴定材料而受到广泛的重视,被作为种属的鉴定以及分子分型的工具[15-17]。本研究中6株分离酵母菌与Genbank中的发酵毕赤酵母菌序列独立于毕赤酵母属的其他种酵母形成一个大的分支,发酵毕赤酵母菌大分支中形成3个小的分支,6株分离酵母菌分列其中两个小的分支中,说明发酵毕赤酵母菌在遗传进化过程中具有一定多样性,为发酵毕赤酵母菌的遗传多样性研究提供一定参考。

图4 分离菌与毕赤酵母属的26S rRNA基因序列系统发生树Fig.4 Phylogenic tree of 26S rRNA gene sequences of Pichia and isolates
从川西北地区采集的10份传统发酵牦牛酸奶样品中的6份中分离到了发酵毕赤酵母,分离率为60%,可见发酵毕赤酵母在该地区牦牛酸奶中是一种广泛存在的菌株,这与张和平以及Mei Bai等[13]的研究结果基本相符。虽然在很多食品中,Pichia属的酵母被认为是污染酵母,但是在传统发酵乳制品中,由于它的某些菌种具有很强的利用乙醇和乳酸的能力,能在过度的酒精和乳酸发酵过程中平衡酒精和乳酸的含量,因此在很多情况下都有Pichia属酵母菌的检出,可以认为它是传统发酵乳制品中正常酵母微生态的一部分。本研究为该地区传统发酵牦牛酸奶的微生物组成多样性研究提供参考。
4 结论
本研究从川西北地区的红原、若尔盖、康定等地的10份传统发酵牦牛酸奶样品中分离鉴定出6株发酵毕赤酵母。为川西北高原地区传统发酵牦牛酸奶中蕴含的大量原始微生物菌种资源的保护及研究提供菌株,为该地区传统发酵牦牛酸奶中微生物组成多样性研究以及发酵毕赤酵母菌的遗传多样性研究提供参考。
[1]吴春生,李键,骞宇,等.牦牛乳及牦牛酸乳营养价值的研究现状[J].乳业科学与技术,2012,35(3):43-46.
[2]ZHANG Guo-qiang,MA Chang-zhong,LI Ya-hui,et al. Chemical and microbiological composition analyses of yak milk in tibet[J].Food Research and Development,2011,30(2):4-7.
[3]Heping Zhang,Jie Xua,Junguo Wang,et al.A survey on chemicaland microbiologicalcomposition ofkurut,naturally fermented yak milk from Qinghai in China[J].Food Control,2008,19(6):578-586.
[4]Xiao-He Wu,Zhang Luo,Li Yu,et al.A survey on composition and micro biota of freshand fermented yak milk at different Tibetan altitudes[J].Dairy Sci Technol,2009,89(2):201-209.
[5]苟钰姣,丁路明,王玉鹏.牦牛乳及乳制品、犏牛和黑白花奶牛乳的脂肪酸组成分析[J].草业科学,2013,30(2):274-280.
[6]梁春年,阎萍,郭宪.牦牛乳的特性及利用现状[C].第四届中国牛业发展大会论文集,2010.
[7]Koichi Watanabe.Diversity of lactic bacteria and yeasts in Airag and Tarag,traditional fermented milk products of Mongolia [J].Microbiol Biotechnol,2008,24(8):1313-1325.
[8]吴春生,舒畅,李键,等.牦牛酸乳中乳酸菌的研究进展及前景展望[J].食品工业,2012,33(9):129-133.
[9]Mu Z,Yang X,Yuan H.Detection and identification of wild yeast in Koumiss[J].Food Microbiol,2012,31(2):301-308.
[10]Qing M,Bai M,Zhang Y,et al.Identification and biodiversity of yeasts from Qula in Tibet and milk cake in Yunnan of China [J].Wei Sheng Wu Xue Bao,2010,50(9):1141-1146.
[11]Chen TH,Wang SY,Chen KN,et al.Microbiological and chemicalproperties ofkefir manufactured by entrapped microorganisms isolated from kefir grains[J].J Dairy Sci,2009,92(7):3002-3013.
[12]Wang SY,Chen HC,Liu JR,et al.Identification of yeasts and evaluation of their distribution in Taiwanese Kefir and Viili starters[J].J Dairy Sci,2008,91(10):3798-3805.
[13]Mei Bai,Manjun,Qing Zhuang Guo,et al.Occurrence and dominance of yeast species in naturally fermented milk from the Tibetan Plateau of China[J].Canadian Journal of Microbiology,2010,56(9):707-714.
[14]CletusP.Kurtzman,Christie JRobnett.Identification and phylogeny of ascomycetous yeasts fromanalysis of nuclear large subunit(26S)ribosomal DNA partial sequences[J].Antonie Van Leeuwenhoek,1998,73(4):331-371.
[15]白逢彦,贾建华,梁慧燕.假丝酵母属疑难菌株大亚基rDNA D1/D2区域序列分析及其分类学意义[J].菌物系统,2002,21(1):27-32.
[16]Fell JW,Boekhout T,Fonseca A,et al.Biodiversity and systematics of basisiomycetous yeast asdetermined by largesubunit rDNA D1/D2domain sequenceanalysis[J].Int J Syst Evol Microbiol,2000,50:1351-1371.
[17]Cletus P.Kurtzman,Christie J.Robnett.Phylogenetic relationships among yeasts of the‘Saccharomyces Complex’determined from multigene sequence analyses[J].FEMS Yeast Research,2003(3):417-432.
Isolation and identification of Pichia fermentans strains from traditional fermented yak yoghurt in the northwest of Sichuan province
WANG Yuan-wei1,ZHANG Cheng-min1,SUO Hua-yi2,YUE Hua1,LI Jian1,TANG Cheng1,*
(1.College of Life Science and Technology,Southwest University for Nationalities,Chengdu 610041,China;2.College of Food Science,Southwest University,Chongqing 400715,China)
Based on the conventional morphological and 26S rRNA gene sequencing analysis,the 6 stains of Pichia fermentans were isolated and identified from the 10 samples from traditional fermented yak yogurt in the northwest of Sichuan province.The 6 isolates had 99.7%~100%nucleotide sequence homology with Pichia fermentans which were available from GenBank.Therefore,the results of this study provided information for research of the diversity of microbial compositions of traditional fermented yak yogurt and genetic diversity of Pichia fermentans.
traditional fermented yak yoghurt;Pichia fermentans;isolation and identification
TS201.3
A
1002-0306(2014)12-0184-04
10.13386/j.issn1002-0306.2014.12.031
2013-09-26 *通讯联系人
王远微(1982-),男,硕士研究生,研究方向:分子生物学。
公益性行业(农业)科研专项(201203009);重庆市基础与前沿研究计划项目(cstc2013jcyjA80006)。

