酶连接探针杂交芯片特异性检测转基因水稻品系
张明哲,陈吴健,张晓峰,陈笑梅,陈 曦,吴 蓉
酶连接探针杂交芯片特异性检测转基因水稻品系
张明哲1,陈吴健1,张晓峰2,陈笑梅2,陈 曦1,吴 蓉3
(1.浙江省检验检疫科学技术研究院,浙江 杭州 310016;2.浙江出入境检验检疫局,浙江 杭州 310016;3.杭州出入境检验检疫局,浙江 杭州 310012)
酶连接探针杂交芯片是一种基于连接酶催化的探针杂交芯片技术,其原理是采用硫代引物保护法,利用Lambda DNA外切酶将多重聚合酶链式反应产物切割成单链。在氨基修饰的芯片上固定5’端探针,并与制备的产物单链进行杂交,加入荧光修饰的第2个探针与之前的探针进行酶连接反应,从而实现高特异性、高通量检测。结果表明:酶连接探针杂交芯 片技术检测4 种转基因水稻品系特异性好,灵敏度可达0.1%(m/m),可以应用于进出口检验检疫、食品安全监测等领域。
酶连接探针杂交芯片;转基因水稻;品系检测
近年来,随着各个国家对农业转基因生物实行检验检疫和标识制度管理的加强,转基因米制品一直是欧盟和其他一些国家关注的重点。2006年9月以来,已有法国、德国、意大利、奥地利和日本等7个国家因为转基因大米污染对我国出口米制品采取了拒绝入境或撤架、召回等措施。欧盟在2008年4月15日起对中国出口的大米及其米制品采取保障性措施,要求由中方认可的实验室对出口产品实施检测,并出具统一的卫生证书才能出口[1]。另一方面,我国对转基因作物培育的投入也逐年增加,近年来具有良好生长性能的转基因新品种不断涌现,2009年农业部审放了转基因抗虫水稻“Bt汕优63”等3个转基因农作物的安全证书[2],因此加强进出口领域转基因水稻品系检测成为新的研究热点。
转基因品系特异性检测[1]是通过分析外源插入载体与植物基因组的连接区序列实现的,由于每一个转基因植物品系都具有特异的外源插入载体与植物基因组的连接区序列,并且连接区序列是低拷贝的,品系特异性检测方法具有高度特异性和准确性,因此品系特异性检测已经成为目前转基因检测研究的重点。
以聚合酶 链式反应(polymerase chain reaction,PCR)为代表的传统检测技术在转基因检测中已有了很大发展[3-5],如转基因水稻Bt63[6]、LLRICE62[7]、LLRICE601[7]、科丰6号[8],转基因大豆GTS 40-3-2[9-11],转基因玉米MON531[12]、MON1445[12]、MON810[13-14]、Bt11[15-16]、GA21[17]、MON863[18-19]、T25[20]、NK603[21]、CBH351[22]和转基因油菜GT73[23]等,但是这些方法大部分都是以单品系检测为主。随着转基因品系的不断增多,PCR检测技术已经无法满足转基因高通量检测的需求[24-25]。
传统的芯片检测技术一般只做1次杂交,而且由于杂交的局限性,如探针设计不合理、退火温度过低、洗涤条件不严格等情况,可能会出现非特异性杂交,导致芯片的背景信号高、出现大量假阳性现象。针对这种情况,本研究建立了一种高特异性的酶连接探针杂交芯片技术并应用于转基因水稻品系检测。酶连接探针杂交芯片是一种基于连接酶催化的探针杂交芯片技术,其原理是采用硫代引物保护法,利用Lambda DNA外切酶将多重PCR产物切割成单链。在氨基修饰的芯片上固定5’端探针,并与制备的产物单链进行杂交,加入荧光修饰的第2个探针与之前的探针进行酶连接反应,从而实现高特异性、高通量检测。
1 材料与方法
1.1 材料与试剂
转基因水稻品系Bt汕优63、科丰6号为中国检验检疫科学研究院馈赠;转基因水稻品系LLRICE62、LLRICE601,转基因玉米品系GA21、Bt176,转基因大豆品系GTS-40-3-2,转基因油菜籽品系GT73、T45 欧洲标准局;非转基因水稻、大豆和油菜籽购自国内市场。
DNeasy Plant Mini Kit DNA提取试剂盒凯杰生物技术有限公司;引物和探针由宝生物工程(大连)有限公司合成;dNTP、Buffer、TaqDNA聚合酶、内切酶宝生物工程(大连)有限公司;无水乙醇、异丙醇、NaOH(均为分析纯) 美国Sigma公司。
1.2 仪器与设备
Sorvall micro21R台式冷冻离心机、Nano Drop 3300核酸测定仪、MAXQ6000摇床 美国Thermo Scientific公司;S-1000 PCR仪 美国BioRad公司;Imager HP核酸电泳及凝胶成像系统 美国Alpha公司;HS-800D数显恒温水浴锅 常州国华电器有限公司;XS104电子天平 瑞士Mettler Toledo公司;Pixsys5500点样仪 美国Cartician Tech公司;LS300扫描仪 瑞士Tecan集团公司;Milli-Q超纯水系统 美国Millipore公司;真空泵Auto Science公司;HR2860研磨机 荷兰Philips公司。
1.3 方法
1.3.1 材料制备
将转基因水稻Bt汕优63、科丰6号、LLRICE62、LLRICE601研磨至粉末状(0.2 mm左右),并按表1混合转基因和非转基因的材料,分别获得含量为5%、1%、0.1%、0.01%(m/m)的转基因样品。

表1 样品制备Table 1 Sample preparation
1.3.2 DNA提取
取100 mg上述制备样品,置于1.5 mL离心管中;加入裂解液及RNA酶后65 ℃水浴30 min;冰上极冷后20 000×g离心5 min取上清液;加入滤柱1(QIAshredder maxi spin column)20 000×g离心2 min后取下清液;与1.5 倍体积洗脱缓冲液1(Buffer AP3/E)混匀后,加入滤柱2(DNeasy maxi spin column)中,6 000×g离心1 min弃下清液;加入洗脱液2(Buffer AW),20 000×g离心2 min;将滤柱2置于1.5 mL离心管中,加入溶解液,室温孵育5 min后6 000×g离心1 min;得到纯化DNA,测定浓度。
1.3.3 引物和探针
实验所用的引物来自于文献[26],探针由本实验室设计合成。探针分为固定探针和杂交探针,其中固定探针进行氨基修饰并固定在芯片上,杂交探针则进行磷酸-Cy3修饰。由于涉及到专利和知识产权保护,引物和探针序列在本实验中不公开。
1.3.4 多重PCR扩增
以转基因样品中提取的DNA为模板,同时加入多对引物进行复合PCR扩增,对复合PCR各引物添加量、Mg2+浓度、TaqDNA聚合酶用量进行优化,建立最优反应模式。扩增程序为:94 ℃预变性5 min;94 ℃变性30 s,55 ℃退火30 s,72 ℃延伸30 s,40个循环;72 ℃延伸10 min。
1.3.5 DNA芯片制备及DNA杂交
点样固定模板:在醛基片上点样固定 阳性探针(15 ømol/L)及氨基修饰的探针(30 ømol/L)。水化8 h,70 ℃干燥2 h,分别用2×柠檬酸缓冲液、0.5%十二烷基硫酸钠洗液洗2 次,每次5 min,水洗2 次,每次5 min,吹干芯片于4 ℃保存,备用。
产物酶切:取约8 øg多重PCR产物进行酶切以制备单链产物。酶切体系:8 øg PCR产物、1 øL Lambda E酶(DNA外切酶)、5 øL 10×buffer、水补足至50 øL。同时设对照组,37 ℃酶切。72 ℃灭活0.5 h,加入40 øL异丙醇后离心纯化,取上清液。
单链产物杂交:将单链产物与杂交液混合后与探针阵列杂交。48 ℃孵育30 min,室温静置10 min。用洗液洗5 min,水洗5 min,吹干,待用。
杂交探针:将终浓度为2.5 ømol/L的探针与杂交液混合后,取1 øL与探针阵列杂交。48℃孵育30 min,室温静置10 min。用洗液洗5 min,水洗5 min,吹干,待用。
酶连及NaOH处理:采用T4连接酶反应体系,室温孵育60 min;0.3 mol/L NaOH室温洗涤芯片5 min,用洗液洗5 min,水洗5 min,吹干,扫描杂交结果。
2 结果与分析
2.1 特异性分析
利用醛基片PCR芯片检测不同的转基因水稻品系Bt汕优63、科丰6号、LLRICE62、LLRICE601,并应用转基因玉米品系GA21、Bt176、转基因大豆品系GTS-40-3-2、转基因油菜籽品系GT73、T45进行特异性分析,结果如图1所示。图1A中1、2、12号位点出 现阳性信号,分别对应为大米內源基因GOS、Bt汕优63品系和阳性对照,其余位点均为阴性。其他水稻品系(图1B~D)只在其1、12号位点和对应的品系位点出现阳性杂交信号。因此,酶连接探针杂交芯片技术检测4 种转基因水稻品系的特异性较好,同时背景信号值和特异信号值均比较稳定,检测结果准确可靠。
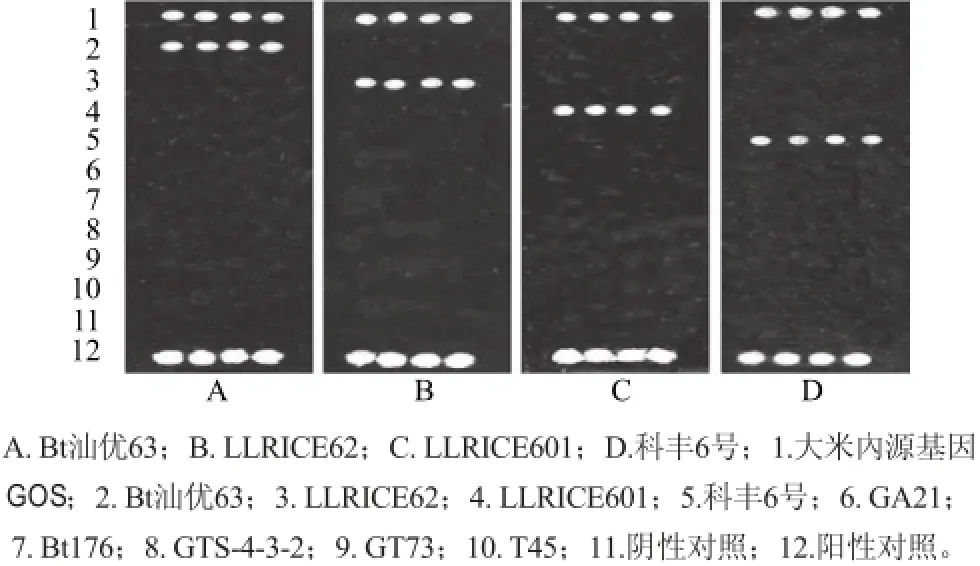
图1 酶连接探针杂交芯片检测转基因水稻品系的特异性分析Fig.1 Specificity of the enzyme-linked probe hybridization chip for different genetically modified plants
2.2 灵敏度

图2 酶连接探针杂交芯片检测转基因水稻品系的灵敏度分析Fig.2 Sensitivity of the enzyme-linked probe hybridization chip for different genetically modified contents
在本实验中,利用酶连接探针杂交芯片进行灵敏度分析,检测结果如图2所示,GOS基因的信号值基本稳定,能有效满足芯片灵敏度检测的需要。而对Bt汕优63品系的基因检测发现,转基因含量为5%、1%、0.1%的样品均有阳性信号出现,含量为0.01%的样品无阳性信号出现。3 次重复检测显示,0.1%的检测结果基本稳定,与其他3个品系基因芯片检测结果相似,因此认为本实验中酶连接探针杂交芯片的灵敏度为0.1%。
3 讨 论
本研究通过2 次杂交,使得只有与2 条探针都匹配的序列才能杂交,然后通过T4 DNA连接酶形成磷酸二酯键,再通过NaOH变性洗涤,就能够把所有未形成磷酸二酯键以及非特异性杂交的片段都洗去,从而保证了检测的特异性。本研究建立的酶连接探针检测转基因水稻品系的检测方法特异性好,灵敏度达到0.1%,可以在进出口检验检疫、食品安全监测等领域进行应用。
[1] 吴志毅, 张明哲, 陈曦. 大米和米制品Bt63转基因检测PCR方法的灵敏度研究[J]. 浙江农业学报, 2009, 21(6): 549-554.
[2] 农业部农业转基因生物安全管理办公室. 2009年农业转基因生物安全证书批准清单[EB/OL]. (2014-03-28) [2012-02-03]. http://www. moa.gov.cn/ztzl/zjyqwgz/spxx/201202/t20120203_2474158.htm.
[3] GACHET E, MARTIN G G, VIGEAU F, et a1. Detection of genetically modified organisms (GMOs) by PCR: a brief review of methodologies available[J]. Trends in Food Science & Technology, 1998, 11(9): 380-388.
[4] 邱良焱, 肖有玉, 刘佳, 等. 多重PCR法检测转Bar、Bt基因双抗稻米[J].食品科学, 2013, 34(6): 139-142.
[5] 王媛, 葛毅强, 陈颖, 等. 大豆加工食品中转基因成分多重PCR检测体系的建立[J]. 食品与发酵工业, 2004, 30(10): 117-121.
[6] BERDAL K G. Roundup ready soybean event-specific real-time quantitative PCR assay and estimation of the practical detectionand quantification limits in GMO analysis[J]. European Food Research and Technology, 2001, 213(6): 432-438.
[7]DING Jiayu, JIA Junwei, YANG Litao, et al. Validation of a rice specific gene, sucrose phosphate synthase, used as the endogenous reference gene for qualitative and real-time quantitative PCR detection of transgenes[J]. Journal of Agricultural and Food Chemistry, 2004, 52(11): 3372-3377.
[8] RALF R, LUTZ G, DIETRICH M. A testing cascade for the detection of genetically modified rice by real-time PCR in food and its application for detection of an unauthorized rice line similar to Ke Feng 6[J]. Journal für Verbraucherschutz und Lebensmittelsicherheit, 2010, 5(2): 185-188.
[9]BERDAL K G, HOLST-JENSEN A. Roundup Ready®soybean event specific real-time quantitative PCR assay and estimation of the practical detection and quantification limits in GMO analysis[J]. European Food Research and Technology, 2001, 213(6): 432-438.
[10] TERRY C F, HARRIS N. Event-specific detection of Roundup Ready® soya using two different real time PCR detection chemistries[J]. European Food Research and Technology, 2001, 213(6): 425-431.
[11] HUANG C C, PAN T Z. Event-specific real-time detection and quantification of genetically modified Roundup Ready®soybean[J]. Journal of Agricultural and Food Chemistry, 2005, 53(10): 3833-3839.
[12] YANG Litao, PAN Aihua, ZHANG Kewei, et al. Qualitative and quantitative PCR methods for event-specific detection of genetically modified cotton Mon 1445 and Mon 531[J]. Transgenic Research, 2005, 14(6): 817-831.
[13] HOLCK A, VA˛TILINGOM, DIDIERJEAN L, et al. 5’-Nuclease PCR for quantitative event-specific detection of the genetically modified Mon810 MaisGard maize[J]. European Food Research and Technology, 2002, 214(5): 449-454.
[14] HERNANDEZ M, PlA M, ESTEVE T, et al. A specific real-time quantitative PCR detection system for event MON810 in maize YieldGard R based on the 3’-transgene integration sequence[J]. Transgenic Research, 2003, 12(2): 179-189.
[15] RONNING S B, VA˛TILINGOM M, BERDAL K G, et al. A. Event specific real-time PCR for genetically modified Bt11 maize (Zea mays)[J]. European Food Research and Technology, 2003, 216(4): 347-354.
[16] ZIMMERMANN A, LÜTHY J, PAULI U. Event specific transgene detection in Bt11 corn by quantitative PCR at the integration site[J]. Lebensmittel-Wissenschaft & Technologie, 2001, 33(3): 210-216.
[17] HERNANDEZ M, ESTEVE T, PRAT S, et al. Development of realtime PCR systems based on SYBR Green I, AmplifluorTMand TaqMan technologies for specific quantitative detection of the transgenic maize event GA21[J]. Journal of Cereal Science, 2004, 39(1): 99-107.
[18] YANG Litao, XU Songci, PAN Aihua, et al. Event-specific qualitative and quantitative PCR detection of genetically modified MON863 maize based on the 5’-transgene integration sequence[J]. Journal of Agricultural and Food Chemistry, 2005, 53(24): 9312-9318.
[19] PAN Aihua, YANG Litao, XU Songci, et al. Event-specific qualitative and quantitative PCR detection of MON863 maize based upon the 3’-transgene integration sequence[J]. Journal of Cereal Science, 2006, 43(2): 250-257.
[20] COLLONNIER C, SCHATTNER A, BERTHIER G, et al. Characterization and event specific-detection by quantitative real-time PCR of T25 maize insert[J]. Journal of AOAC International, 2005, 88(2): 536-546.
[21] NIELSEN C R, BERDAL K G, HOLST-JENSEN A. Characterization of the 5’-integration site and development of an event-specific realtime PCR assay for NK603 maize from a low starting copy number[J]. European Food Research and Technology, 2004, 219(4): 421-427.
[22] WINDELS P, BERTRAND S, DEPICKER A, et al. Qualitative and event-specific PCR real time detection methods for Star LinkTMmaize[J]. European Food Research and Technology, 2003, 216(3): 259-263.
[23] TAVERNIERS I, WINDELS P, VAITILINGOM M, et al. Eventspecific plasmid standards and real-time PCR methods for transgenic Bt11, Bt176, and GA21 maize and transgenic GT73 canola[J]. Journal of Agricultural and Food Chemistry, 2005, 53(8): 3041-3052.
[24] 许文涛, 杨蓉, 陆姣, 等. 转基因玉米59122品系的特异性检测[J]. 食品科学, 2011, 32(4): 139-142.
[25] 宋君, 雷绍荣, 刘勇, 等. 转基因玉米MON863品系特异定量PCR方法的建立[J]. 湖北农业科学, 2011, 50(24): 5250-5253.
[26] 厦门出入境检验检疫局. SN/T 2584—2010 水稻及其产品中转基因成分实时荧光PCR检测方法[S]. 北京: 中国标准出版社, 2010.
Event-specific Detection of Genetically Modified Rice Using Enzyme-Linked Probe Hybridization Chip
ZHANG Ming-zhe1, CHEN Wu-jian1, ZHANG Xiao-feng2, CHEN Xiao-mei2, CHEN Xi1, WU Rong3
(1. Zhejiang Academy of Science and Technology for Inspection and Quarantine, Hangzhou 310016, China; 2. Zhejiang Entry-Exit Inspection and Quarantine Bureau, Hangzhou 310016, China; 3. Hangzhou Entry-Exit Inspection and Quarantine Bureau, Hangzhou 310012, China)
The enzyme-linked probehybridization chip is a method based on ligase-hybridizing probe chip technology with the principle of using thio-primers for protection, and using Lambda DNA exonuclease to cut multiple PCR amplicons into single products. 5’-End probe chain is fi xed in the amino-modified chip, and hybridized with the single-chain product by adding the fl uorescent-modif i ed probe for the enzyme-linked reactions with the anterior probe in order to achieve highly specif i c, parallel and high-throughput detection. In this study, the enzyme-linked probe hybridization chip could effectively detect genetically modif i ed rice with high specif i city and the sensitivity limit was 0.1%. This chip can be used by the entryexit inspection and quarantine authorities and be applied for food safety testing.
enzyme-linked probe hybridization chip; genetically modified rice; event-specific detection
Q783
A
1002-6630(2014)08-0222-04
10.7506/spkx1002-6630-201408044
2013-07-31
浙江省科技厅重大科技专项重点农业项目(2011C12023);国家质检总局科技计划项目(2011IK249);
浙江省科技厅公益性项目(2011C22065);浙江出入境检验检疫局科技计划项目(ZK200958)
张明哲(1982—),男,高级农艺师,博士,研究方向为分子生物学和植物检疫。E-mail:mzzhang429@163.com

