肺炎链球菌血清型鉴定的分子生物学检测方法
窦珍珍(综述),刘 钢(审校)
(首都医科大学附属北京儿童医院感染科,北京 100045)
肺炎链球菌(streptococcus pneumoniae)能够引起肺炎、胸膜炎、中耳炎、脑膜炎、败血症等疾病。世界卫生组织(world health organization,WHO)2005年的统计数据表明,全世界每年至少有160万人死于侵袭性肺炎链球菌感染,其中70万~100万是<5岁的儿童,且发病人群主要集中在发展中国家[1]。我国的统计数据表明,肺炎链球菌是儿童肺炎最常见的病原体,也是细菌性脑膜炎主要的病原体之一,1970~2005年肺炎链球菌引起的细菌性脑膜炎在占全部确诊病例的10%~30%[2]。
目前,接种肺炎链球菌疫苗是最有效的预防肺炎链球菌疾病的方法。以分子生物学为基础的检测方法由于试剂相对便宜、结果客观、能直接检测临床标本的可能性[3]而日益受到关注。不同的科研团队设计出了不同的检测方法,以下对各种实验方法予以综述。
1 肺炎链球菌疫苗的重要意义
美国2000~2008年推广使用肺炎链球菌7价蛋白多糖结合疫苗(7-valent polysaccharide-protein conjugate vaccine,PCV7)(PCV7覆盖的血清型为4、6B、9V、14、18C、19F和23F)期间,人群中侵袭性肺炎链球菌疾病的发病率在<5岁的儿童中下降了77%,因肺炎入院治疗的<2岁儿童下降了39%[4]。欧洲有文献报道称,应用PCV7的国家其侵袭性肺炎链球菌疾病的发病率下降[5]。基于PCV7在降低侵袭性肺炎链球菌疾病发病率的显著成绩,WHO已经推荐PCV7作为国家免疫计划中的优先项目[1]。肺炎链球菌疫苗中除了PCV7外,目前还有13价蛋白多糖结合疫苗、23价荚膜多糖疫苗。这些疫苗均为血清型特异性疫苗,它们对人群的保护作用与疫苗对当地致病性肺炎链球菌血清型的覆盖率有关。虽然目前PCV7在预防肺炎链球菌疾病方面已有效果,但肺炎链球菌血清型监测仍然十分重要。对于已将PCV7纳入国家免疫计划项目的国家,PCV7对侵袭性肺炎链球菌血清型分布的长期影响尚不十分明确。有地区报道称,应用PCV7后导致了血清型替换现象,即非疫苗血清型定植、致病增多的现象[6-8]。如果肺炎链球菌疫苗对侵袭性肺炎链球菌血清型覆盖率降低,对人群的保护效益可能降低。对未纳入PCV7作为国家免疫计划项目的国家(主要是发展中国家和地区),监测肺炎链球菌的重要性主要体现以下三个方面:①目前肺炎链球菌感染死亡病例主要是在发展中国家;②这些国家和地区缺乏完善的肺炎链球菌血清型监测信息;③目前应用的PCV7覆盖的血清型种类的设计主要是基于欧洲和北美发达国家侵袭性肺炎链球菌血清型的监测,而已有文献报道称PCV7的覆盖率具有显著的地区差异[1]。
目前检测肺炎链球菌的金标准是荚膜肿胀实验。荚膜特异性抗体和荚膜结合后在显微镜下可观察到荚膜肿胀显著,荚膜肿胀实验就是利用这种原理用血清型已知的抗体检测菌株的血清型。这种方法的缺点主要是试剂昂贵、结果判读主观,且检测过程需要培养细菌。血清型检测试剂的昂贵限制了经济欠发达地区肺炎链球菌血清型的检测,主观的判读使部分菌株的结果有误,且检测结果还依赖于细菌培养,而使得血清型检测结果也受培养阳性率的限制。
2 肺炎链球菌荚膜基因结构
肺炎链球菌荚膜的生成主要受荚膜基因(capsular polysaccharide synthesis locus,CPS)控制,除血清型3和37外,其余血清型的荚膜均由Wzy/Wzx依赖的途径生成并由cpsA-D调节,这些基因均存在于dexB和aliA之间。cpsA-D是靠近dexB端的前4个相对保守的基因,cpsD和aliA之间的基因是血清型特异性的基因区域[9](图1)。血清型37的肺炎链球菌通过cps外的tts基因指导,由合酶途径合成荚膜[10]。但在dexB和aliA之间,血清型37有与33F非常相似的结构,只是在cap37B,cap37E,cap37N和cap37O存在沉默子,这些沉默子导致该区域的基因结构不能编码荚膜(图2)[10]。血清型3亦依赖合酶途径合成荚膜[11]。血清型3无cpsA-D基因,但是其orf1和dexB之间的一段序列和血清型19F的cpsA、cpsB相似,orf1、orf2分别与cpsC、cpsD相似,orf2下游和cap3A之间是一段长度为1096 bp 19F缺失的序列(图3)[12]。cap3A、cap3B、cap3C是血清型3特异性序列[13]。虽然cpsA-D在所有的血清型之间相对保守,但cpsA和cpsB仍存在与血清型相关的多态性[14-17]。荚膜基因是以分子生物学为基础的血清型检测方法的基础。目前,93种血清型的荚膜基因序列都已测出[9](http://www.sanger.ac.uk/Projects/S_pneumoniae/CPS/),这将助于以分子生物学方法研究肺炎链球菌的血清型。
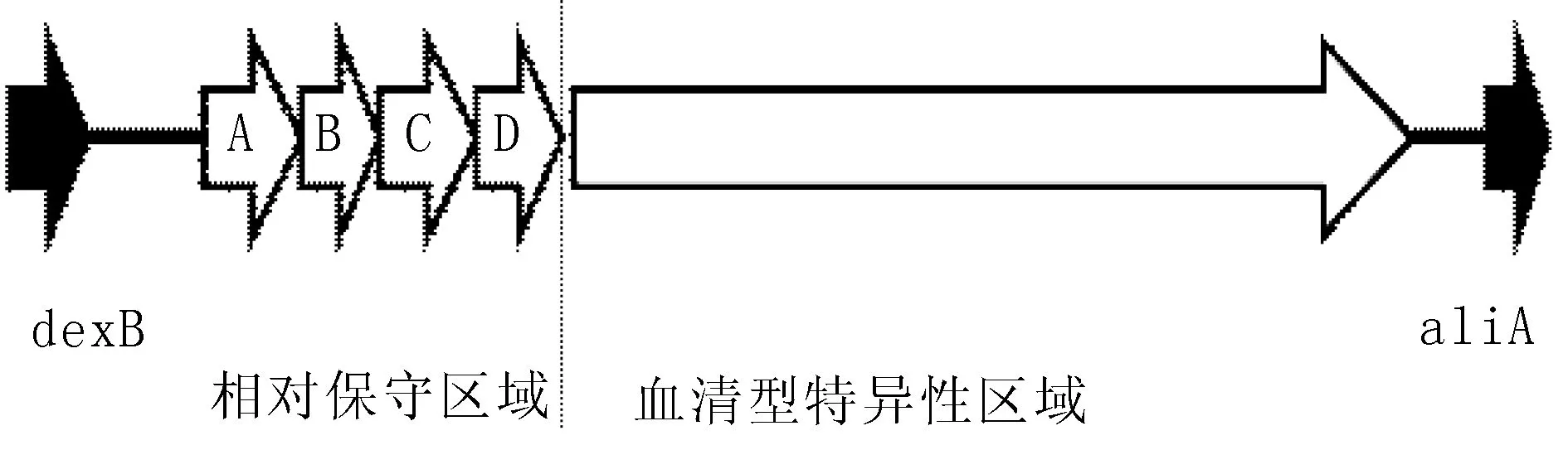
图1 血清型cps结构图
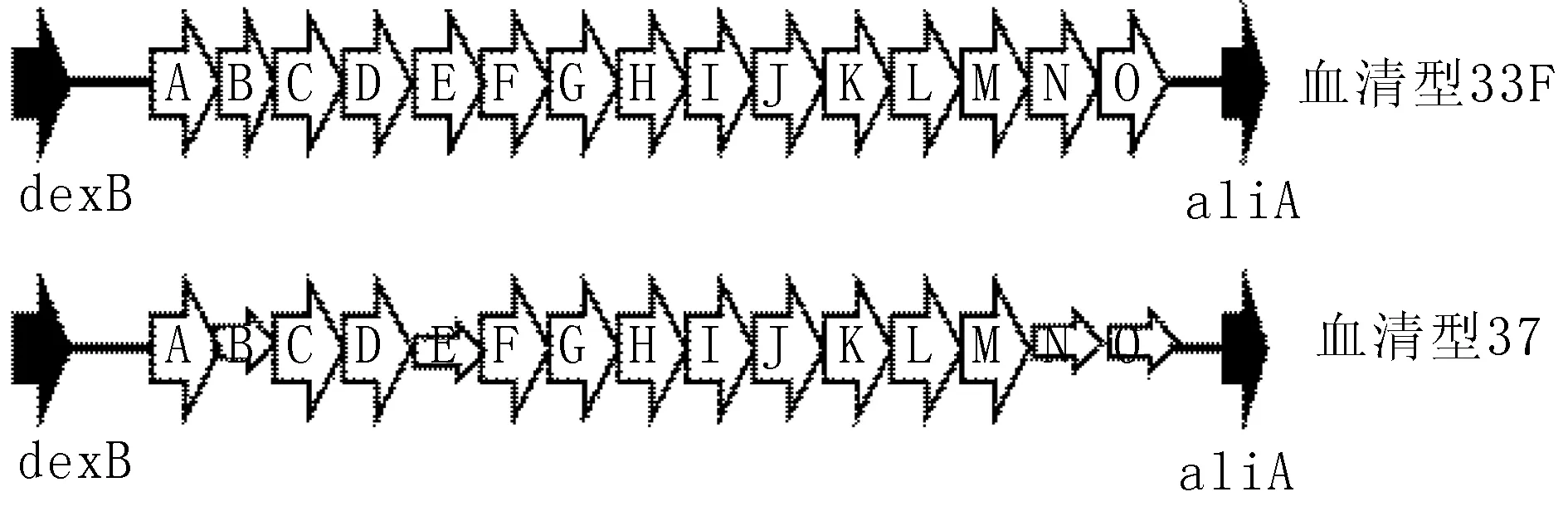
图2 血清型33F和血清型37 cps结构图
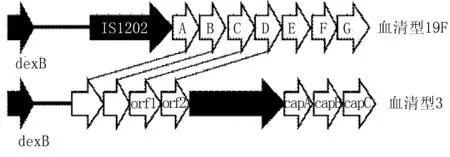
图3 血清型3和血清型19Fcps结构图
3 已有的分子生物学检测方法
已有的以分子生物学为基础的血清型检测方法可分为两类,一类是以检测cps基因血清型特异性区域为基础的方法,如多重聚合酶链反应(polymerase chain reaction,PCR)、多重PCR反向线性杂交、基因芯片;另一类是以检测与血清型相关的cps基因多态性为基础的方法,如限制性片段多态性、以测序为基础的血清型检测方法。
3.1多重PCR和连续多重PCR 多重PCR检测血清型是利用多种血清型特异性引物的混合物与待测标本提取的DNA进行PCR扩增,通过PCR扩增产物的大小判断待测标本的血清型。2005年,O′Halloran等[18]设计了用1个反应体系检测PCV7覆盖的7种肺炎链球菌血清型,结果显示这种方法能够准确预测所覆盖的7种血清型的所有菌株。但是,PCV7推广应用后出现的血清型替换现象可使PCV7覆盖率下降的同时,也削弱该方法检测肺炎链球菌血清型的能力[6-8]。
增加血清型检测范围可削弱血清型替换对血清型检测方法的影响,但是多重PCR体系需要考虑引物的兼容性以及扩增产物的大小[19-20],因此在一个PCR反应体系中能够检测的血清型的种类是有限的。连续多重PCR通过多次多重PCR(每次多重PCR引物组成不同)增加了血清型检测范围。2006年,Pai等[19]设计的连续多重PCR能够检测28种常见致病性肺炎链球菌。此方法共包含有7步PCR反应,每一步PCR反应能够检测4种血清型,每一步PCR均扩增cpsA基因作为内对照,常见的致病血清型用第一步PCR和第二步PCR检测,以便使用最少的PCR反应步骤检测血清型的分布。检测过程中,血清型特异性PCR产物对应的血清型为标本的血清型,PCR反应后若cpsA阳性但血清型特异性PCR阴性则进入下一步PCR,7步PCR完成后若仍只有cpsA阳性,则该标本的血清型未包括在连续多重PCR检测范围内,cpsA阴性的标本为未分型菌株[19]。
连续多重PCR的检测效率高,检测结果准确,且检测成本远低于荚膜肿胀实验。用连续多重PCR的方法检测了随机从2002~2003年收集的菌株中挑选的421株菌株,前3步多重PCR能检测62.5%菌株的血清型,7步PCR完成后401(95.2%)株菌株能确定血清型/群,20株菌株不能确定血清型/群的菌株为未分型菌株或检测范围外的血清型[19]。应用连续多重PCR预测的血清型结果与荚膜肿胀实验预测结果均相符[19]。Pai等[19]统计,荚膜肿胀实验检测肺炎链球血清型的平均成本是28美元,而应用连续多重PCR,3步PCR完成血清型检测的成本是2.28美元,7步PCR完成血清型检测的成本是5.32美元。此外,还可根据当地肺炎链球菌血清型流行情况调整血清型的检测步骤、增加血清型的检测范围。在血清型分布不同的地区连续多重PCR的应用有很强的灵活性,已有巴西[21]、西班牙[22]、比利时[23]、孟加拉国[24]、芬兰[25]、韩国[26]、中国[27]、撒哈拉以南的非洲地区[28]的研究人员根据当地肺炎链球菌血清型流行病学资料在原始连续多重PCR的基础上设计并验证了新的血清型检测方案。以连续多重PCR为基础的血清型检测方法可直接检测生物标本中的肺炎链球菌血清型,孟加拉国的研究人员已经利用这种方法检测了培养阴性的脑脊液中肺炎链球菌的血清型[29]。连续多重PCR还可以检测存在多种血清型标本的血清型。检测中耳炎患者鼻咽部分泌物中肺炎链球菌血清型的研究发现,鼻咽部分泌物分离培养的菌株用荚膜肿胀实验检测结果为19F,而用连续多重PCR直接从鼻咽部分泌物提取的DNA,可同时检测出血清型19F和23F[30]。
多重PCR产生错误结果的主要原因是读胶时产生错误[20],PCR扩增产物大小接近,同一个PCR反应体系检测的血清型种类过多则增加发生这种错误发生的概率,因此单个PCR反应体系检测的血清型不宜过多。
3.2多重PCR反向线性杂交和基因芯片技术 多重PCR反向线性杂交和基因芯片技术检测肺炎链球菌血清型的技术基础为特异性探针与目标DNA杂交,它们共同的特点是在扩大血清型检测范围的同时最大限度地减少PCR反应次数。
2006年,Kong等[31]利用多重PCR反向线性杂交技术,设计了能够检测23组共40种血清型的血清型检测方法。2007年,Zhou等[32]又设计了20组共50种少见血清型的检测方法(不包括6C、6D和11E)。多重PCR反向线性杂交技术检测肺炎链球菌的血清型,首先对标本提取的DNA进行多重PCR扩增(血清型特异性PCR),然后利用尼龙膜上结合的血清型特异性的单核苷酸探针和生物素标记的多重PCR反应产物杂交,再利用过氧化物酶标记的抗生蛋白链菌素和生物素结合并利用化学发光物质检测过氧化物酶,最后利用探针位置及种类与尼龙膜对应的胶片检测荧光。胶片上产生荧光的位置对应的探针种类就是被检测标本的血清型[31]。Kong等[31]用设计的多重PCR反向线性杂交技术检测266株菌株,Zhou等[32]用设计的多重PCR反向线性杂交技术检测了152株菌株,除血清型未覆盖菌株和未分型菌株杂交结果阴性外,其余菌株的血清型预测结果和荚膜肿胀实验一致或包含了荚膜肿胀实验结果(血清型组包含2种或以上的血清型)。
基因芯片是不同血清型的DNA探针按照一定顺序排列的微小基片。
2007年,Wang等[33]设计了通过检测肺炎链球菌wzy基因和cap基因(血清型3)来检测24组共43种血清型的基因芯片(多重PCR发生交叉反应的血清型归为同一组)。基因芯片上,每组血清型有两种探针,同时有两种16SrDNA探针作为阳性对照,40个T组成的探针作为阴性对照。用这种基因芯片检测血清型过程如下:DNA样本经一次多重PCR扩增后,以扩增产物为模板,将双向引物换成单向引物并加入Cy3-dUTP后,在相同的实验条件下再次扩增,再次扩增的产物与芯片杂交,基因芯片平扫仪检测杂交后芯片的信号,所收集的数据通过相关软件分析后可确定血清型种类。
Wang等[33]用设计的基因芯片检测了169株标本(147株属于检测范围的43种血清型,11株为检测范围外的血清型,11株为其他病原体),结果与荚膜肿胀实验的结果相符。此外,这种基因芯片检测血清型的灵敏度较高。研究发现,DNA提取浓度为1010 cfu/L时,用基因芯片能确定血清型种类,而用多重PCR则无法检测血清型[32]。因此,在检测DNA含量较少的临床标本时,基因芯片检测血清型有显著的优势。
2011年Tomita等[34]设计了一种新的检测血清型的基因芯片。芯片通过与肺炎链球菌GT基因(糖基转移酶基因)杂交而能检测23组共37种血清型(多重PCR发生交叉反应的血清型归为同一组)。基因芯片上,每组血清型检测1~6个GT基因,每一个GT基因有3种不同的探针。此外,基因芯片上包含了16SrDNA以及与肺炎链球菌的7个看家基因(aroE/ddl/gdhA/glcK/spi/tktA/xpt)互补的探针作为阳性对照,与肺炎克雷伯菌、金黄色葡萄球菌、嗜肺军团菌、肺炎衣原体、肺炎支原体、绿脓杆菌、化脓性链球菌的看家基因互补的探针作为阴性对照。血清型检测过程中,基因芯片直接与提取的菌株基因组DNA杂交,分析数据判断血清型。该方法仅检测了检测范围内的36种血清型的36株肺炎链球菌,其中1株结果和荚膜肿胀实验结果不符。目前,基因芯片血清型检测的准确性还需要更进一步的提高。
与连续多重PCR相比,多重PCR反向线性杂交及基因芯片技术最大的特点是检测的血清型种类多,且不需要多次进行PCR反应,但是这种方法涉及的引物及探针种类多(多重PCR反向线性杂交检测90种血清型需200种引物或探针),多种血清型之间可发生交叉反应(多重PCR反应线性杂交检测的90种血清型中有58种血清型能发生交叉反应),能发生交叉反应的血清型需与其他血清型检测方法结合方能进一步区分。目前发现的肺炎链球菌血清型种类虽已达93种,但常见的血清型种类有限,连续多重PCR前三步PCR反应就能够检测60%~90%的肺炎链球菌菌株的血清型[19,21-23,25-26,28-30]。因此在检测常见血清型时,上述两种血清型检测方法在时间消耗、操作以及血清型检测成本上并无明显优势。利用基因芯片检测血清型的缺点是该方法的设备有特殊要求(需要基因芯片扫描仪),对于经济欠发达地区的应用可能受到限制。
3.3限制性片段长度多态性 限制性片段长度多态性是一种可用于检测基因多态性的分子生物学分析方法,同源DNA,因为存在基因多态性,应用限制性内切酶后可产生不同的DNA片段图谱。Lawrence等[16]发现,肺炎链球菌cpsA和cpsB基因限制性片段长度多态性与血清型相关,根据这一特点,设计了通过检测限制性片段长度的多态性而预测肺炎链球菌11种血清型的检测方法。每种血清型的cpsA-cpsB PCR扩增产物分别用AluI、HinfI和RsaI3种酶进行酶切,同种酶得到的条带分型用谱图分析软件(GelCompar,version 4.1)进行对比分析并命名,每一种血清型可获得3种酶切后的条带分型。通过研究11种血清型条带分型,建立条带分型和血清型关系数据库。血清型未知的菌株的条带分型和数据库中条带分型的相似度≥90%时,认为是相同的条带分型,可以通过对比血清型已知的条带分型数据库预测未知肺炎链球菌的血清型。
Lawrence等[16]用建立的数据库检测了93株菌株,87株检测结果与荚膜肿胀实验相符,1株血清型为1的菌株被预测为血清型1或19F,4株血清型为6A的菌株被预测为6B,2株血清型分别为6B和9V的菌株出现在数据库中,未有条带分型被错误判断为非疫苗菌株。提示某些菌株只能给出血清型可能范围,这种方法不能正确区分血清型6A和6B,新的条带分型可导致血清型判断错误。
Batt等[35]扩增dexB和ailA之间的所有序列(大小为14~23 kpb),并单独应用Hinfl酶切获得条带分型。通过46种血清型已知的菌株建立了46种血清型条带分型的数据库。用该数据库检测了73株菌株,所有菌株的结果均和荚膜肿胀实验相符。与Lawrence等[16]设计的方法相比,Batt等[35]设计的方法在构建条带、分型数据库以及条带对比时更简单。Lawrence设计的方法,每种血清型对应3种条带分型。Batt等设计的方法是每种血清型对应1种条带分型。但是,Batt设计的方法PCR扩增产物高达14~23 kpb,这在扩增产物方面增加了难度。
限制性长度片段多态性的缺点是每次进行检测都必须有标准株建立比对的数据库,而且,血清型检测范围内的菌株如出现新的条带分型,会导致结果判读错误。
3.4测序为基础的血清型检测方法 肺炎链球菌cpsA 3′端到cpsB 5′端之间的序列多态性与血清型相关[15-16]。Kong等[17,36]通过对90种血清型的这段基因进行测序,建立了部分cpsA-cpsB血清型预测系统。血清型未知的菌株在cpsA 3′端和cpsB 5′端之间的序列和部分cpsA-cpsB血清型预测系统比对可预测血清型未知菌株的血清型。
2011年,Leung等[14]设计了一种新的以测序为基础的血清型检测方法。他们扩增的区域位于cpsA下游1351位点到cpsC下游224位点之间,扩增产物达1064 kpb,但是两侧的测序结果可能不确切,因此采用中间的732 kpb与基因库比对,比对后分数最高的对应的血清型为血清型检测结果。这种新的以测序为基础的血清型检测方法通过比对PPV23覆盖的血清型的基因序列设计引物,除 PPV23覆盖的血清型外,还可以扩增另外61种血清型。Leung等用该方法检测了138株菌株,仅1株菌株血清型预测错误。
以这两种测序为基础的血清型检测方法的引物结合区都是相对保守区域,能与引物结合的血清型种类多,这使血清型检测过程中涉及的引物种类少(Kong等[17,36]设计的检测方法需要三对引物;Leung等[14]设计的检测方法需要一对引物)。这一特点简化了血清型检测过程。但是,由于引物不是血清型特异性引物,混杂其他血清型的DNA对血清型检测的影响还需要进行进一步试验来判断。
4 小 结
与荚膜肿胀实验相比,以分子生物学为基础的血清型检测方法虽然有试剂相对便宜、结果客观以及有直接检测培养阴性临床标本的可能性的优点,但也有不足。首先,目前设计的以分子生物学为基础的血清型检测方法均不能完全区分所有的血清型。一些血清型相近的菌株荚膜基因差异很小而在PCR过程中会发生交叉反应,如血清型6A和6B在wciP基因上存在单个核苷酸多态性[37]。一些血清型虽并不相似,但可能因为PCR扩增的目的基因相似而发生交叉反应,如35F和47F,33F、33A和37[19,31]。其次,肺炎链球菌的血清型属于荚膜抗原特异性,以分子生物学为基础的血清型检测方法只是一种间接的血清型检测方法,它的检测结果可以与荚膜肿胀实验的结果不一致。有文献报道称,用荚膜肿胀实验检测血清型为19F的2株基因背景不一样的菌株血清型特异性PCR扩增产物为19A,将该菌株wzy基因测序比对后发现,这2株菌株wzy基因与血清型19Fwzy基因的相似度为92%,而与血清型19Awzy基因的相似程度为80%[25]。因此,以分子生物学为基础的血清型检测方法不能完全替代荚膜肿胀实验。尽管如此,其还是可以与荚膜肿胀实验联合应用以降低血清型检测成本。目前,连续多重PCR经验证是使用最多的血清型检测方法,仅极少量菌株的检测结果出现错误,极少量的菌株因引物区的基因多态性而PCR扩增失败[23]。芬兰已将连续多重PCR作为血清型检测的常规方法,用连续多重PCR不能检测出血清型的菌株或因交叉反应血清型不能确定的菌株再用荚膜肿胀实验确定血清型。联合应用连续多重PCR和荚膜肿胀实验,降低了血清型检测成本[25]。
[1] Pneumococcal conjugate vaccine for childhood immunization--WHO position paper[J].Wkly Epidemiol Rec,2007,82(12):93-104.
[2] Yao KH,Wang LB,Zhao GM,etal.Pneumococcal serotype distribution and antimicrobial resistance in Chinese children hospitalized for pneumonia[J].Vaccine,2011,29(12):2296-2301.
[3] Azzari C,Moriondo M,Indolfi G,etal.Molecular detection methods and serotyping performed directly on clinical samples improve diagnostic sensitivity and reveal increased incidence of invasive disease by Streptococcus pneumoniae in Italian children[J].J Med Microbiol,2008,57(Pt 10):1205-1212.
[4] Centers for Disease Control and Prevention (CDC).Progress in introduction of pneumococcal conjugate vaccine—worldwide,2000-2008[J].MMWR Morb Mortal Wkly Rep,2008,57(42):1148-1151.
[5] Isaacman DJ,Mcintosh ED,Reinert RR.Burden of invasive pneumococcal disease and serotype distribution among Streptococcus pneumoniae isolates in young children in Europe:impact of the 7-valent pneumococcal conjugate vaccine and considerations for future conjugate vaccines[J].Int J Infect Dis,2010,14(3):e197-e209.
[6] Weinberger DM,Malley R,Lipsitch M.Serotype replacement in disease after pneumococcal vaccination[J].Lancet,2011,378(9807):1962-1973.
[7] Klugman KP.The significance of serotype replacement for pneumococcal disease and antibiotic resistance[J].Adv Exp Med Biol,2009,634:121-128.
[8] Hanage WP.Serotype replacement in invasive pneumococcal disease:where do we go from here?[J].J Infect Dis,2007,196(9):1282-1284.
[9] Bentley SD,Aanensen DM,Mavroidi A,etal.Genetic analysis of the capsular biosynthetic locus from all 90 pneumococcal serotypes[J].PLoS Genet,2006,2(3):e31.
[10] Llull D,Muoz R,López R,etal.A single gene (tts) located outside the cap locus directs the formation of Streptococcus pneumoniae type 37 capsular polysaccharide.Type 37 pneumococci are natural,genetically binary strains[J].J Exp Med,1999,190(2):241-251.
[11] Caimano MJ,Hardy GG,Yother J.Capsule genetics in Streptococcus pneumoniae and a possible role for transposition in the generation of the type 3 locus[J].Microb Drug Resist,1998,4(1):11-23.
[12] Guidolin A,Morona JK,Morona R,etal.Nucleotide sequence analysis of genes essential for capsular polysaccharide biosynthesis in Streptococcus pneumoniae type 19F[J].Infect Immun,1994,62(12):5384-5396.
[13] Arrecubieta C,García E,López R.Sequence and transcriptional analysis of a DNA region involved in the production of capsular polysaccharide in Streptococcus pneumoniae type 3[J].Gene,1995,167(1/2):1-7.
[14] Leung MH,Bryson K,Freystatter K,etal.Sequetyping:Serotyping Streptococcus pneumoniae by a Single PCR Sequencing Strategy[J].J Clin Microbiol,2012,50(7):2419-2427.
[15] Jiang SM,Wang L,Reeves PR.Molecular characterization of Streptococcus pneumoniae type 4,6B,8,and 18C capsular polysaccharide gene clusters[J].Infect Immun,2001,69(3):1244-1255.
[16] Lawrence ER,Arias CA,Duke B,etal.Evaluation of serotype prediction by cpsA-cpsB gene polymorphism in Streptococcus pneumoniae[J].J Clin Microbiol,2000,38(4):1319-1323.
[17] Kong F,Wang W,Tao J,etal.A molecular-capsular-type prediction system for 90 Streptococcus pneumoniae serotypes using partial cpsA-cpsB sequencing and wzy- or wzx-specific PCR[J].J Med Microbiol,2005,54(Pt 4):351-356.
[18] O′Halloran DM,Cafferkey MT.Multiplex PCR for identification of seven Streptococcus pneumoniae serotypes targeted by a 7-valent conjugate vaccine[J].J Clin Microbiol,2005,43(7):3487-3490.
[19] Pai R,Gertz RE,Beall B.Sequential multiplex PCR approach for determining capsular serotypes of Streptococcus pneumoniae isolates[J].J Clin Microbiol,2006,44(1):124-131.
[20] Miernyk K,Debyle C,Harker-Jones M,etal.Serotyping of Streptococcus pneumoniae isolates from nasopharyngeal samples:use of an algorithm combining microbiologic,serologic,and sequential multiplex PCR techniques[J].J Clin Microbiol,2011,49(9):3209-3214.
[21] Dias CA,Teixeira LM,Carvalho Mda G,etal.Sequential multiplex PCR for determining capsular serotypes of pneumococci recovered from Brazilian children[J].J Clin Microbiol,2007,56(Pt 9):1185-1188.
[22] Iraurgui P,Torres MJ,Gandia A,etal.Modified sequential multiplex PCR for determining capsular serotypes of invasive pneumococci recovered from Seville[J].Clin Microbiol Infect,2010,16(9):1504-1507.
[23] Jourdain S,Dreze PA,Drèze PA,etal.Sequential multiplex PCR assay for determining capsular serotypes of colonizing S.pneumoniae[J].BMC Infect Dis,2011,11(11):100.
[24] Saha SK,Al Emran HM,Hossain B,etal.Streptococcus pneumoniae serotype-2 childhood meningitis in Bangladesh:a newly recognized pneumococcal infection threat[J].PLoS One,2012,7(3):e32134.
[25] Siira L,Kaijalainen T,Lambertsen L,etal.From quellung to multiplex PCR,and back when needed,in pneumococcal serotyping[J].J Med Microbiol,2012,50(8):2727-2731.
[26] Yun KW,Cho EY,Hong KB,etal.Streptococcus pneumoniae type determination by multiplex polymerase chain reaction[J].J Korean Med Sci,2011,26(8):971-978.
[27] Zhang YJ,Chen YS,Wang ZW,etal.Serological and molecular capsular typing,antibiotic susceptibility and multilocus sequence typing of Streptococcus pneumoniae isolates from invasive and non-invasive infections[J].Chin Med J (Engl),2013,126(12):2296-2303.
[28] Morais L,Carvalho Mda G,Roca A,etal.Sequential multiplex PCR for identifying pneumococcal capsular serotypes from South-Saharan African clinical isolates[J].J Med Microbiol,2007,56(Pt 9):1181-1184.
[29] Saha SK,Darmstadt GL,Baqui AH,etal.Identification of serotype in culture negative pneumococcal meningitis using sequential multiplex PCR:implication for surveillance and vaccine design[J].PLoS One,2008,3(10):e3576.
[30] Billal DS,Hotomi M,Suzumoto M,etal.Determination of pneumococcal serotypes/genotypes in nasopharyngeal secretions of otitis media children by multiplex PCR[J].Eur J Pediatr,2008,167(4):401-407.
[31] Kong F,Brown M,Sabananthan A,etal.Multiplex PCR-based reverse line blot hybridization assay to identify 23 Streptococcus pneumoniae polysaccharide vaccine serotypes[J].J Clin Microbiol,2006,44(5):1887-1891.
[32] Zhou F,Kong F,Tong Z,etal.Identification of less-common Streptococcus pneumoniae serotypes by a multiplex PCR-based reverse line blot hybridization assay[J].J Clin Microbiol,2007,45(10):3411-3415.
[33] Wang Q,Wang M,Kong F,etal.Development of a DNA microarray to identify the Streptococcus pneumoniae serotypes contained in the 23-valent pneumococcal polysaccharide vaccine and closely related serotypes[J].J Microbiol Methods,2007,68(1):128-136.
[34] Tomita Y,Okamoto A,Yamada K,etal.A new microarray system to detect Streptococcus pneumoniae serotypes[J].J Biomed Biotechnol,2011,2011:352736.
[35] Batt SL,Charalambous BM,Mchugh TD,etal.Novel PCR-restriction fragment length polymorphism method for determining serotypes or serogroups of Streptococcus pneumoniae isolates[J].J Clin Microbiol,2005,43(6):2656-2661.
[36] Kong F,Gilbert GL.Using cpsA-cpsB sequence polymorphisms and serotype-/group-specific PCR to predict 51 Streptococcus pneumoniae capsular serotypes[J].J Med Microbiol,2003,52(Pt 12):1047-1058.
[37] Mavroidi A,Godoy D,Aanensen DM,etal.Evolutionary genetics of the capsular locus of serogroup 6 pneumococci[J].J Bacteriol,2004,186(24):8181-8192.

