泥东风螺4个群体遗传多样性的AFLP分析
秦溱,王晓清*,曾志南,*,朱丹丽,熊钢,2,巫旗生,宁岳
(1.湖南农业大学动物科学技术学院,湖南 长沙 410128;2.湖南生物机电技术学院,湖南 长沙 410127;3.福建水产科学研究所,福建 厦门 361000)
泥东风螺4个群体遗传多样性的AFLP分析
秦溱1,王晓清1*,曾志南1,3*,朱丹丽1,熊钢1,2,巫旗生3,宁岳3
(1.湖南农业大学动物科学技术学院,湖南 长沙 410128;2.湖南生物机电技术学院,湖南 长沙 410127;3.福建水产科学研究所,福建 厦门 361000)
利用AFLP分子标记方法对4个群体(福建连江野生、连江养殖、长乐养殖、广西野生)的泥东风螺进行遗传多样性分析。结果:用11对引物共扩增出908条有效片段,其中684条(75.33%)为多态性片段,224条片段(24.67%)为4个群体所共有;遗传多样性指数分析显示,4个群体的有效等位基因数、平均等位基因数、Shannon’s多样性指数和平均杂合度依次为1.500 6、1.974 0、0.464 7、0.303 4,Nei’s遗传距离为0.128 4~0.180 6,遗传相似系数为0.834 8~0.879 5,表明4个泥东风螺群体具有较为丰富的遗传多样性,且群体间具有较高的遗传相似性;分子方差分析(AMOVA)结果显示,4个群体中 83.49%的变异来源于群体内,14.65%的变异来源于地区间,而群体间的遗传变异仅为1.86%,且4个群体间的遗传分化(GST)为0.195 4,基因流(NM)为2.058 6,说明群体间的基因交流水平较低;UPGMA聚类分析和主坐标(PCA)分析结果表明,长乐养殖群体和广西野生群体遗传距离最近,而连江野生群体与其他3个群体的遗传距离最远。
泥东风螺;遗传多样性;遗传距离;扩增片段长度多态性(AFLP)
泥东风螺(BabyLonia lutosa (Lamer))[1],俗称“黄螺”,属海洋底栖腹足纲(Gastropoda)、新腹足目(Neogastropoda)、蛾螺科(Buccinidae)、东风螺属(BabyLonia)成员,是热带、亚热带贝类品种。泥东风螺在中国主要分布于福建、海南、广东、广西等地,是中国沿海重要的经济软体动物[2–3]。泥东风螺不仅生长速度快、肉质鲜美、营养成分含量高,还含有人体所必须的氨基酸和稀有元素,易被人体消化吸收,属于健康食品,具有较高的经济价值。
扩增片段长度多态性(AFLP)主要是通过分析限制性内切酶不同的片段长度,进而检测基因组DNA多态性的一种分子标记技术。目前,AFLP标记由于具有多态性丰富、信息量多等优点,已被广泛用于黄姑鱼、棘头梅童、华贵栉孔扇贝、文蛤[4–7]等海洋动物群体遗传多样性及各类遗传变异的研究。近年来,对泥东风螺捕捞强度的增大导致其自然资源严重降低。本研究首次利用AFLP技术对中国东南沿海泥东风螺 4个群体进行遗传多样性分析,旨在为泥东风螺种质资源的开发、保护及合理增殖放流提供参考依据。
1 材料与方法
1.1 材 料
1.1.1 供试螺
2013年3—7月,从福建的长乐、连江和广西随机采集的4个泥东风螺群体,包括2个野生群体(连江、广西)和2个养殖群体(连江、长乐),泥东风螺共计120个。
1.1.2 试 剂
Mse I酶、EcoRI酶、T4连接酶及Taq DNA聚合酶均购自NEB公司。
1.1.3 引 物
AFLP分析所用引物序列见表 1。引物均由上海英骏公司合成。
1.2 方 法
1.2.1 基因组DNA的提取
采用盐析法[8]分别提取120个泥东风螺的总DNA。用DNA检测仪检测DNA的浓度和纯度。将吸光比值(A260 nm/A280 nm)在1.8~2.0的样品DNA分装,保存于–20 ℃冰箱,备用。

表1 试验所用接头和引物组合Table 1 AFLP primers and adapters used in the experiment
1.2.2 AFLP分析
1) 酶切。酶切反应体系(20 μL):10×(EcoRI) Buffer 2.0 μL,100×BSA 0.2 μL,20U/μL EcoR I 0.2 μL,10 U/μL Mse I 0.2 μL,20 pmol/μL DNA模板1 μL,ddH2O补齐至20 μL。37 ℃酶切5 h,65 ℃灭活20 min,置于室温使DNA复性。
2) 连接。反应体系为:10×Buffer 1.0 µL,5 pmol/μL EcoR I接头引物(Ead5和Ead3) 0.25 μL,50 pmol/μL Mse I引物(Mad5和Mad3) 0.5 μL,400 U/μL T4连接酶0.1 μL,酶切产物5 μL,ddH2O补足至10 μL,16 ℃连接16 h后,65 ℃灭活20 min。
3) 限制性片段的扩增。预扩增PCR反应体系为:5 U/µL Taq酶 0.2 µL,10×PCR Buffer(含MgCl2) 2.0 µL,10 mmol/L dNTP 0.4 µL,10 µmol/L 选择性扩增引物(E1、E2、E3、E4、E5、E6、E7或E8) 0.6 µL,10 µmol/L选择性扩增引物(M1、M2、M3、M4、M5、M6、M7或M8) 0.6 µL,ddH2O补齐至20 μL。PCR预扩增反应程序为:72 ℃预变性5 min;95 ℃变性30 s,56 ℃退火30 s,72 ℃延伸2 min,20个循环;最后72 ℃延伸5 min。预扩增PCR产物稀释20倍后,用于选择性扩增。
4) 选择性扩增PCR。反应体系为:5 U/µL Taq 酶 0.2 µL,10×PCR Buffer(含 MgCl2)2.0 µL,10 mmol/L dNTP 0.4 µL,10 µmol/L选择性扩增引物(E1、E2、E3、E4、E5、E6、E7或E8)0.4 µL,10 µmol/L选择性扩增引物(M1、M2、M3、M4、M5、M6、M7或M8) 0.8 µL,ddH2O补齐至20 μL。PCR选扩增反应程序为95 ℃变性2 min,95 ℃退火30 s,65℃延伸30 s;72 ℃变性1 min,共13个循环(每个循环降0.7 )℃;95 ℃变性30 s,56 ℃退火30 s,72℃延伸1 min,30个循环;最后在72 ℃延伸5 min。
5) 变性聚丙烯酰胺凝胶电泳。通过对AFLP各条件的优化,筛选出多条带清晰的选择性扩增引物组合进行PCR。PCR反应体系及程序同上。扩增产物用6%变性聚丙烯酰胺凝胶100 W恒定功率电泳2 h,进行银染检测。
1.3 数据统计与分析
对银染后得到的指纹图谱进行读取分析,有条带的记为“1”,无条带的记为“0”。将AFLP指纹图谱的统计结果用由“1”和“0”构成的矩阵数列表示。采用PopGene 1.32软件计算扩增片段总数、多态性位点比例、Shannon指数、期望杂合度、Nei’s遗传距离及遗传相似系数、遗传分化与基因流等,并根据群体间的遗传距离用UPGMA方法进行聚类分析,采用 Winamova1.55软件进行群体间以及群体内的分子变异分析,运用Genalex 6[9]软件进行主坐标分析。
2 结 果
2.1 AFLP扩增谱带统计
本研究共进行了64条随机引物的扩增,其中11对有效引物可获得扩增片段条带清晰、重复性好的条带,11对引物组合从4个群体中共扩增出908条有效扩增片段,每对引物组合可扩增出 56~108条片段,平均82.55条片段,其中224条片段(24.67%) 为4个群体所共有,684条(75.33%)为多态性片段,表现出群体多样性。
E6/M7扩增片段总数最多(108条),E8/M3扩增片段总数最少(56条);E8/M4扩增多态性片段比例最高(78.75%),E8/M5扩增多态性片段比例最低(69.86%),表现出群体多样性(表2)。

表2 11对AFLP引物组合的扩增结果Table 2 Amplification results of 11 different AFLP primer combinations
引物 E2/M3对部分泥东风螺样品的AFLP扩增结果见图1。
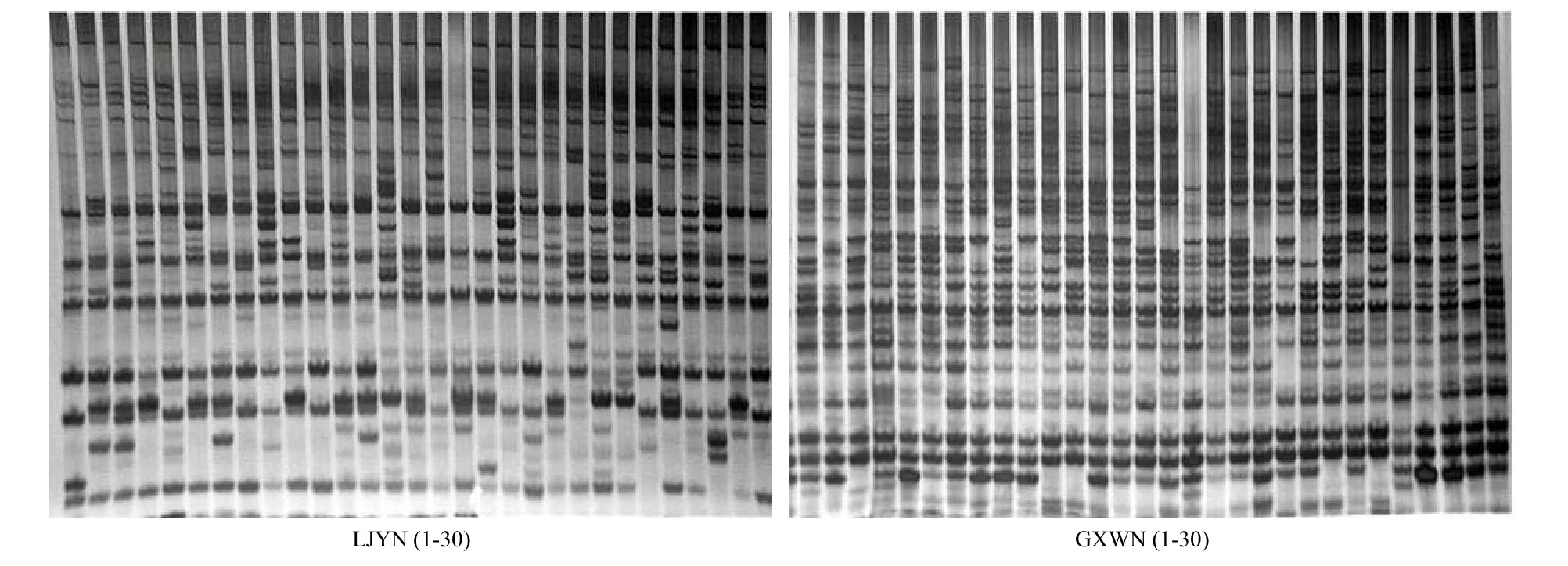
图1 E2/M3引物组合扩增LJYN和GXWN群体得到的AFLP指纹图谱Fig.1 AFLP patterns of LJYN and GXWN populations using primer E2/M3
连江野生泥东风螺(LJWN)、广西野生泥东风螺(GXWN)、连江养殖泥东风螺(LJYN)、长乐养殖泥东风螺(CLYN)4个群体的扩增结果见表3。连江野生群体和长乐养殖群体的多态性比例高于其他2个群体,分别为72.90%和72.86%。

表3 4个泥东风螺群体的扩增结果Table 3 Amplification results of four populations of Babylonia lutosa
2.2 遗传多样性分析
对4个群体的遗传多样性指数进行分析,发现4个群体平均的等位基因数、有效等位基因数、Shannon’s多样性指数和平均杂合度依次为1.974 0、1.500 6、0.464 7和0.303 4(表4)。从等位基因数和有效等位基因数的分析结果来看,长乐养殖群体和广西野生群体的等位基因数及有效等位基因数高于连江野生群体和连江养殖群体,且长乐养殖群体的有效等位基因数最多,连江养殖群体的最少。Shannon’s多样性指数分析结果表明,长乐养殖群体和广西野生群体的遗传变异大于连江野生群体和连江养殖群体,且长乐养殖群体的遗传变异最大,连江养殖群体的最小。平均杂合度分析结果也显示出相同趋势,长乐养殖群体和广西野生群体高于连江野生群体和连江养殖群体,且长乐养殖群体杂合度最高,连江养殖群体杂合度最低。4种遗传多样性指数均表明,长乐养殖群体和广西养殖群体的遗传多样性高于连江野生群体和连江养殖群体,其中长乐养殖群体的遗传多样性最高,连江养殖群体的遗传多样性最低;4个群体的遗传多样性大小依次为长乐养殖群体、广西野生群体、连江野生群体、连江养殖群体。

表4 4个群体的遗传多样性指数Table 4 Parameters of genetic diversity of four populations of Babylonia lutosa
2.3 遗传距离及遗传相似系数分析
依据AFLP指纹图谱,用Nei’s(1979)[10]公式,计算4个群体的遗传距离和遗传相似系数(表5)。4个群体间遗传距离为0.128 4~0.180 6,平均遗传距离为0.152 0;遗传相似系数为0.834 8~0.879 5,平均遗传相似系数为0.859 2,其中,长乐养殖群体和广西野生群体的遗传距离最小,遗传相似系数最高;连江野生群体和连江养殖群体的遗传距离最大,遗传相似系数最低。分析表明,长乐养殖群体和广西野生群体可能是由一个群体遗传变异而得,而同一地区不同养殖模式的群体间存在着较为明显的遗传变异。

表5 4个群体的遗传距离和遗传相似系数Table 5 Genetic distance and genetic similarity of four populations of Babylonia lutosa
2.4 4个群体间遗传多样性来源的分子方差分析(AMOVA)
利用分子方差分析方法得出群体间、群体内和地区间 3种变异来源对总的遗传变异的贡献率(表6),表明泥东风螺的遗传变异主要来源于群体内(83.49%),其次来源于群体间(14.65%),而发生在地区间的遗传变异最少,为1.86%。群体间的PhiPT遗传分化系数为0.201,表明4个群体间处于中等偏低的遗传分化水平,且群体内的遗传变异为最主要的变异源。

表6 泥东风螺4个群体的AMOVA分析Table 6 Data derived from AMOVA of four populations of Babylonia lutosa
2.5 群体间的遗传分化与基因流
用PopGene软件分析得出4个群体的总遗传多样性值(HT)为0.377 0,群体内遗传多样性(Hs)高达0.303 3,遗传分化值(GST)=19.54%,即19.54%的遗传变异存在于群体间,而高达80.46%的遗传变异存在于群体内。4个群体间的基因流(NM)仅为2.058 6,说明由于4个群体间的地理隔离,造成了群体间的基因交流水平较低。
2.6 群体间的遗传距离和聚类分析
根据 Nei’s遗传距离对 4个泥东风螺群体的AFLP指纹图谱进行聚类分析,采用UPGMA法构建系统进化树(图3)。从UPGMA聚类分析结果来看,4个泥东风螺群体中长乐养殖群体和广西野生群体遗传距离最近,其次是连江养殖群体,与广西野生群体遗传距离最远的是连江野生群体。主坐标分析(PCA)得出第一主坐标(coord.1,横坐标)占全部变异的30.22%,而第二主坐标(coord.2,纵坐标)占25.82%,第一主坐标和第二主坐标总共占全部变异的56.04%。表明长乐养殖群体明显与广西野生群体在主坐标上分布距离最近,而连江野生群体与其他3个群体的分布距离最远(图4),说明主坐标分析结果与上述Upgma法聚类分析结果一致。

图3 4个泥东风螺群体构建的UPGMA系统进化树Fig.3 The phylogenetic tree of four populations of Babylonia lutosa with UPGMA

图4 4个泥东风螺群体的主坐标分析(PCA)Fig.4 PCA for four populations of Babylonia lutosa
3 讨 论
a. 泥东风螺4个群体的遗传多样性。AFLP在螺类上的运用较少。潘英等[11]首次利用AFLP技术对5个管角螺群体的遗传多样性进行分析研究。本试验用11对不同的选择性扩增引物对4个泥东风螺群体进行AFLP分析,多态性片段比例、等位基因数和有效等位基因数Shannon’s多样性指数、期望杂合度等指标均表明4个泥东风螺群体具有较为丰富的遗传多样性。遗传多样性高低依次为长乐养殖群体、广西野生群体、连江野生群体、连江养殖群体,且长乐养殖群体和广西野生群体的遗传多样性高于连江野生群体和连江养殖群体。其原因可能是长乐养殖的群体引种于广西野生泥东风螺,经过福建长乐地区长期养殖后,使其遗传多样性在原有的基础上更为丰富。影响遗传多样性的自然因素主要来自区域差异,而人为因素如环境污染、捕捞过度或者人工放流等则都有可能造成遗传多样性的丢失[12–13]。尽管本研究中的泥东风螺群体目前均有较高的遗传多样性,但捕捞方式的不合理及环境污染都有可能致使遗传多样性降低。
b. 泥东风螺4个群体间的遗传相似性分析。有研究[14–18]表明,同一区域的人工养殖群体经过长期的人工选育,由于种间杂交、近亲繁殖、遗传漂变等原因,其遗传多样性低于野生群体,在一定程度上显示出种质资源退化的现象。笔者对4个泥东风螺群体的Nei’s遗传距离和遗传相似系数分析的结果表明,长乐养殖群体和广西野生群体的遗传距离最小,遗传相似系数最高,连江野生群体和连江养殖群体的遗传距离最大,遗传相似系数最低,这说明长乐养殖群体和广西野生群体可能具有一定的遗传同源性,而同一区域内不同的养殖模式会使群体的遗传多样性发生变化,且人工养殖群体的遗传多样性低于野生群体。分子方差分析(AMOVA)结果显示,4个泥东风螺群体的遗传变异主要来源于群体内(83.49%),其次来源于地区间(14.65%),而发生在群体间的遗传变异最少(1.86%),这与群体间遗传分化及基因流结果相一致。基因流是指生物个体由于交配或迁移而导致的基因从一个居群向另一个居群扩散,使得繁殖群体中等位基因频率发生变化的现象。本试验中的基因流(NM)仅为2.058 6,说明本试验中的4个群体间由于地理隔离而引起的群体间基因交流的水平较低,因此导致群体间的遗传变异主要来源于群体内的遗传变异。从UPGMA聚类分析结果来看,4个泥东风螺群体中长乐养殖群
体与广西野生群体遗传距离最近,其次是连江养殖群体,与广西野生群体遗传距离最远的是连江野生群体,这与4个泥东风螺群体在主坐标轴上的分布结果一致,进一步证明长乐养殖群体和广西野生群体在遗传上存在亲缘性,且与连江养殖群体和野生群体存在着一定的遗传分化。
[1] 董津茂,蔡如星.浙江动物志(软体动物)[M].杭州:浙江科学技术出版社,1991.
[2] 王如才.中国水生贝类原色图鉴[M].杭州:浙江科学技术出版社,1988.
[3] 陈利雄,吴进锋.东风螺的养殖技术及产业化前景[J].齐鲁渔业,2004,21(10):9–11.
[4] 韩志强,高天翔,王志勇,等.黄姑鱼群体遗传多样性的AFLP分析[J].水产科学,2006,30(5):640–646.[5] 郑德锋,赵金良,周文玉,等.我国沿海棘头梅童鱼(Collichthys lucidus)群体遗传结构的AFLP分析[J].海洋与湖藻,2011,42(3):443–447.
[6] 欧燕燕.华贵栉孔扇贝体色多态性研究及AFLP分析[D].湛江:广东海洋大学水产学院,2012.
[7] 赫崇波,丛林林,葛陇利,等.文蛤养殖群体和野生群体遗传多样性的AFLP分析[J].中国水产科学,2008,15(2):215–221.
[8] 谭建明,谢桐,徐琴君,等.快速盐析法提取DNA在HLA基因分型中的应用[J].中华器官移植杂志,1996,17(1):9–11.
[9] Peakall R,Smouse P.GENALEX 6:Genetic anaLysis in Excel.Population genetic software for teaching and research[J].Molecular Ecology Notes,2006,6(1): 288–295.
[10] Nei M,Li W.Mathematical model for studying genetic variation in terms of restriction endonucleases[J]. National Acad Sciences,1979,76(10):139–147.
[11] 潘英,慕翠敏,李琪,等.管角螺遗传多样性的AFLP分析[J].中国海洋大学学报,2013,43(4):58–62.[12] 王爱民,阎冰,叶力,等.三个野生种群马氏珠母贝遗传多样性的RAPD分析[J].农业生物技术学报,2003,11(2):163–168.
[13] 高悦勉,孙洋,才慧梅.3种扇贝遗传结构的RAPD分析[J].海洋水产研究,2007,28(6):25–31.
[14] 刘芳,刘卫东,王强,等.微卫星标记及其在贝类遗传选育研究中的应用[J].水产科学,2006,25(5):268–269.
[15] 夏德全,王文君.动物线粒体DNA研究及在鱼类种群遗传结构研究中的应用[J].水产学报,1998,22(4):364–369.
[16] 张全启,徐晓斐,齐洁,等.三种鲆鲽鱼养殖群体的遗传多样性分析及特异性AFLP标记研究[J].中国海洋大学学报,2001,32(5):512–518.
[17] 高祥刚,葛陇利,刘卫东,等.皱纹盘鲍野生与养殖群体遗传多样性的 AFLP分析[J].生物技术通报,2008(增刊):324–330.
[18] 许宝红,肖调义,苏建明,等.南洞庭湖15种野生蚌类遗传多样性分析[J].湖南农业大学学报:自然科学版,2009,35(1):88–103.
责任编辑:罗 维
英文编辑:罗 维
Genetic distance in four populations of Babylonia lutosa (Lamer) assessed by AFLP makers
QIN Qin1, WANG Xiao-qing1*, ZENG Zhi-nan1,3*, ZHU Dan-li1, XIONG Gang1,2, WU Qi-sheng3, NING Yue3
(1.College of Animal Science and Technology, Hunan Agricultural University, Changsha 410128, China; 2.Hunan Biological Electromechanical Vocational Technical College, Changsha 410127, China; 3.Fisheries Science Research Institute of Fujian, Xiamen, Fujian 361000, China)
Genetic diversity of Lianjiang wild population (LJWN), Lianjiang cultured population (LJYN), Changle cultured population (CLYN), Guangxi wild population (GXWN) of Babylonia lutosa (Lamer) was analyzed by amplified fragment length polymorphisms (AFLP). For the assessment, total 908 bands were generated from four populations using 11 different primer combinations, 684 (75.33%) were polymorphic fragments of which 224 (24.67%) are shared by the four populations. Genetic diversity index analysis showed, the effective number of alleles, the average number of alleles, Shannon’s diversity index and average heterozygosity were 1.500 6, 1.974 0, 0.464 7, 0.303 4, respectively; Nei’s genetic distance and genetic similarity coefficient were ranged from 0.128 4 to 0.180 6 and 0.834 8 to 0.879 5, indicating relatively rich genetic diversity and high genetic similarity among the four populations of Babylonia lutosa (Lamer).Results of analysis of molecular variance (AMOVA) and assignment test revealed 83.49% variation within population, 14.65% variation from region difference and 1.86% variation among the four populations. Genetic variance GSTand NMwere 0.195 4 and 2.058 6 among four population groups indicating that the gene exchange level is very low. The analysis results of UPGMA and PCA showed that the nearest genetic distance exists between CLYN population and GXWN population. Genetic distance between LJWN population and other 3 populations was the farthest.
Babylonia lutosa (Lamer); genetic distance; genetic diversity; amplified fragment length polymorphisms (AFLP)
Q959.212;Q347
A
1007−1032(2014)03−0299−06
10.13331/j.cnki.jhau.2014.03.015
投稿网址:http://www.hunau.net/qks
2014–04–11
国家海洋公益性行业科研专项(201205021)
秦溱(1988—),女,湖南长沙人,硕士研究生,主要从事水生生物学研究,51953385@qq.com;*通信作者,wangxiao8258@ 126.com;xmzzn@sina.com

