大豆球蛋白的红外和Raman光谱分析
龙国徽,纪 媛,潘洪斌,孙泽威,王锦欣,秦贵信
(1.吉林农业大学生命科学学院,长春 130118;2.吉林大学物理学院,长春 130012;3.云南农业大学云南省动物营养与饲料重点实验室,昆明 650201;4.吉林农业大学动物科学技术学院,长春 130118)
大豆球蛋白的红外和Raman光谱分析
龙国徽1,纪 媛2,潘洪斌3,4,孙泽威4,王锦欣1,秦贵信4
(1.吉林农业大学生命科学学院,长春 130118;2.吉林大学物理学院,长春 130012;3.云南农业大学云南省动物营养与饲料重点实验室,昆明 650201;4.吉林农业大学动物科学技术学院,长春 130118)
利用Fourier变换红外光谱(FTIR)和激光Raman光谱研究大豆球蛋白的结构,并对大豆球蛋白的红外光谱和Raman光谱的特征峰进行指认,计算Raman费米共振I850/830的比值.结果表明:红外光谱和Raman光谱的酰胺Ⅰ带去卷积和曲线拟合获得二级结构,其中β-折叠结果差异较小,α-螺旋、β-转角和无规卷曲结果差异较大(p<0.05);红外光谱在1 618,1 682cm-1处的吸收峰归属于大豆球蛋白的分子间和分子内聚集,拟合峰面积百分数分别为11.1%和9.5%;包埋和外露的酪氨酸残基占酪氨酸残基总量的14%和86%.
红外光谱;Raman光谱;大豆球蛋白;分子结构
Fourier变换红外(FTIR)光谱和Raman光谱同属分子振动光谱.目前利用FTIR光谱研究大豆球蛋白结构的报道较多[8-10],而利用Raman光谱对大豆球蛋白结构的研究较少[11-12].FTIR光谱由偶极距变化引起,Raman光谱由分子振动的极化率变化引起.FTIR光谱对C—O,N—H和O—H等极性基团伸缩振动检测灵敏,Raman光谱对C—C,C—C和S—S等非极性基团检测灵敏,因此二者可以相互补充提供分子结构线索[13].利用计算机辅助解析方法(如去卷积、二阶导数和曲线拟合)可定量分析蛋白质的二级结构[14-15].本文利用FTIR光谱和Raman光谱研究大豆球蛋白的结构.
1 实 验
1.1 试剂与仪器
大豆品种为丰交7607(吉林农业大学春雨种业有限公司);正己烷、硫酸铵、β-巯基乙醇、三羟甲基氨基甲烷、丙烯酰胺、甲叉双丙烯酰胺和BCA蛋白检测试剂盒(北京鼎国试剂公司);琼脂糖凝胶Sephrose CL-6B(英国Amersham Pharmacia Biotech AB公司);其他试剂均为国产分析纯.
低温高速离心机(美国Sigma公司);110cm×4.5cm层析柱(上海精科实业有限公司);1-4LD型冷冻干燥机(德国Alpha公司);Nicolet 360Fourier变换红外光谱仪(美国Nicolet公司);JY-HR800型显微共聚焦激光Raman光谱仪(法国Jobin Yvon公司);JEOL-6480LV扫描电镜(日本电子株式会社).
1.2 实验过程
1.2.1 大豆球蛋白的制备 参照文献[16]方法:大豆粉用正己烷常温脱脂,脱脂豆粉用pH=8.0的Tris-HCl缓冲液(含10mmol/L 2-ME)抽提,m(料)∶V(液)=1∶15,搅拌2h后于4℃以9 000r/min离心30min,所得上清液用1mol/L HCl调至pH=6.4,以9 000r/min离心30min.所得沉淀物溶解于pH=7.6的磷酸盐缓冲溶液中,静止过夜,6 500r/min离心20min.上清液加入固体硫酸铵调至质量分数为51%的饱和状态,离心后弃去沉淀,上清液继续调至质量分数为66%的硫酸铵饱和状态,离心所得沉淀溶于磷酸盐缓冲液(pH=7.6)中.琼脂糖凝胶CL-6B装柱(110cm× 4.5cm),充分平衡后上样,先用磷酸盐缓冲液(pH=7.6)洗脱,再用紫外-可见分光光度计在280nm处检测,分别收集洗脱液组分.洗脱液于4℃蒸馏水中透析,冻干后得到纯化的大豆球蛋白,用BCA蛋白检测试剂盒检测蛋白质含量,样品于-20℃冷藏备用.
1.2.2 SDS-PAGE电泳和Native-PAGE电泳 参照文献[17]方法:在不连续的缓冲体系中进行电泳,采用质量分数为12%的分离胶和质量分数为4%的浓缩胶,将蛋白样品溶于含有SDS-PAGE的Tris-HCl缓冲液(pH=8.0),含质量分数为1%的SDS、体积分数为2%的巯基乙醇、体积分数为5%的甘油和质量分数为0.025%的溴酚蓝中,电泳前煮沸5min.上样量为10μL,凝胶电泳于恒流模式下进行,在浓缩胶中电流为40mA,进入分离胶后增至80mA.凝胶染色液采用质量分数为0.25%的考马斯亮蓝(R-250)溶液,用高甲醇的醋酸溶液脱色,V(甲醇)∶V(冰乙酸)∶V(去离子水)=1∶1∶8.Native-PAGE的条件与SDS-PAGE类似,但Tris-HCl中不含SDS和巯基乙醇,且样品不需加热处理.
1.2.3 FTIR光谱的测定 参照文献[18]方法:将冻干大豆球蛋白样品置于干燥器内用P2O5充分干燥,称取2mg干燥的样品,加入200mg的KBr,研磨混合均匀后压片,恒温箱中平衡温度5min后进行红外光谱扫描,扫描波段4 000~1 000cm-1,扫描次数32,分辨率4cm-1.图谱处理采用OMNIC 8.0数据处理软件,原谱进行基线校正,所有蛋白样品的红外光谱与不含蛋白质样品的KBr图谱做差谱,去卷积参数控制半峰宽为30和增强因子为4.0得到去卷积谱.结合去卷积图谱,利用Origin 7.5软件对酰胺Ⅰ带进行Gauss曲线拟合,多次拟合使残差最小,使重叠在一起的不同谱带可完全分辨开.
1.2.4 激光Raman光谱的测定 采用JY-HR800型显微共聚焦激光Raman光谱仪,激发光源为氩离子激光器,波长为514.5nm,功率为40mW.狭缝宽度50μm,光栅为1 800刻线,分辨率1cm-1.显微镜头为50倍常规镜头,通过5倍物镜背向散射,积分时间为30s.先将干燥处理的大豆球蛋白置于凹面载玻片上,再将凹面载玻片置于Raman显微镜下,Raman位移范围400~2 000cm-1,重复扫描10次,计算机自动累加平均.Raman图谱基线校正,归一化处理以苯丙氨酸的1 004cm-1为内标,Raman谱图归属采用Labspec 5.0,由OMNIC 8.0处理得到酰胺Ⅰ带去卷积谱,利用Origin 7.5软件进行曲线拟合.
1.2.5 数据分析 采用SPSS 12.0软件包进行数据统计分析,实验数据为3次测定取平均值,蛋白质二级结构含量采用LSD方差分析,比较平均值之间的差异显著性.
2 结果与讨论
2.1 SDS-PAGE和Native-PAGE电泳分析
制备的大豆球蛋白SDS-PAGE电泳图谱具有典型的酸性和碱性亚基,如图1(A)所示.其对应分子量分别为35 000~40 000和18 000.提纯的大豆蛋白各亚基分子量与理论值相符,经电泳鉴定的大豆球蛋白质量分数大于90%,与文献[19]结果相符.Native-PAGE电泳图谱(图1(B))中的3个泳道显示出大豆球蛋白单体、三聚体和六聚体3种形态,不同类型的聚集体表明大豆球蛋白在分离纯化过程中发生部分解聚或聚合.
2.2 大豆球蛋白的FTIR图谱分析
图2为大豆球蛋白的FTIR谱,表1列出了其红外谱特征吸收峰对应的峰位和峰强及归属.由图2和表1可见,3 303cm-1处的峰位归属于O—H伸缩振动和N—H伸缩振动,反映了大豆球蛋白中形成分子内的C—O…H—N氢键和分子间的H—O…N—H氢键水平,氢键强弱反映了水分子的 O—H 伸缩振动或吸附水量的大小[20].2 888cm-1处的峰位归属于大豆球蛋白分子饱和结构中CH3和CH2基团的C—H伸缩振动,反映了大豆球蛋白中脂肪族氨基酸侧链中C—H对称与非对称伸缩振动,该峰位变化可评定脂肪族氨基酸侧链结构在大豆球蛋白中的变化[21].
大豆球蛋白的红外光谱1 800~1 000cm-1是指纹图谱区域:1 652cm-1峰位的酰胺Ⅰ带归属于80%的C O伸缩振动和20%的C—N伸缩振动;1 540cm-1峰位的酰胺Ⅱ带归属于60%的N—H变形振动、30%的C—N伸缩振动和10%的C—C伸缩振动;1 280,1 240cm-1峰位的酰胺Ⅲ带归属于C—N伸缩振动和N—H面外弯曲振动[22];位于1 342cm-1处的吸收峰由CH3基团对称变形振动产生,位于1 467cm-1处的吸收峰由CH3基团不对称变形振动和CH2基团变形振动产生,位于1 342,1 467cm-1处的吸收峰多数来自大豆球蛋白中脂肪族氨基酸的侧链结构[22];1 403cm-1峰位归属于COOH中的C—OH伸缩振动[23];位于1 149cm-1处的吸收峰归属于大豆球蛋白中环状结构C—C振动或C—O—O糖苷键振动[24].

图1 大豆球蛋白的SDS-聚丙烯酰胺(A)和非还原聚丙烯酰胺(B)凝胶电泳图谱Fig.1 SDS-PAGE profiles(A)and native-PAGE profiles(B)of glycinin
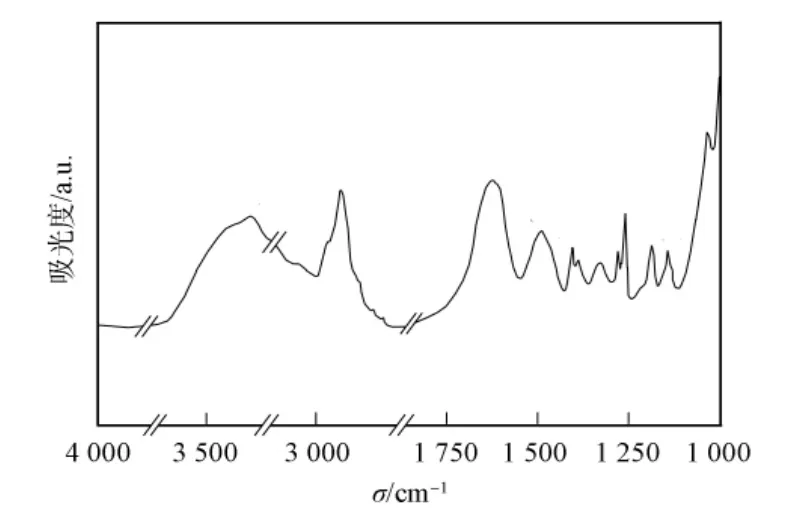
图2 大豆球蛋白的红外光谱Fig.2 FTIR spectrum of glycinin

表1 大豆球蛋白的红外光谱峰值及归属Table 1 Characteristic absorption peaks in FTIR spectrum of glycinin
大豆球蛋白的红外光谱酰胺Ⅰ带1 700~1 600cm-1是二级结构的敏感区域,是计算蛋白二级结构的主要谱带.酰胺Ⅰ带1 600~1 700cm-1进行去卷积获得10个单一峰,如图3(A)所示,FTIR酰胺Ⅰ带特征峰1 618,1 627,1 634,1 674cm-1归属为β-折叠;1 658cm-1归属于为α-螺旋;1 664,1 682,1 692cm-1归属为β-转角;1 646cm-1归属为无规卷曲[11],见表2.1 606cm-1峰位归属于酪氨酸残基的振动或与精氨酸残基相关[24].图3(B)为结合去卷积峰位对红外光谱的酰胺Ⅰ带进行曲线拟合,大豆球蛋白二级结构β-折叠、α-螺旋、β-转角和无规卷曲的百分数分别为(40.67±1.40)%,(15.72±0.44)%,(28.33±1.38)%和(15.27±0.40)%(表2).FTIR酰胺Ⅰ带中1 618,1 682cm-1峰位可定量反映大豆球蛋白分子内聚集和分子间聚集[20],其曲线拟合积累面积的百分数分别为11.1%和9.5%(表2).
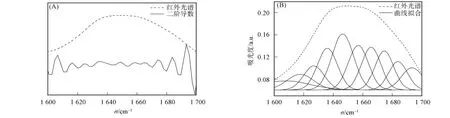
图3 大豆球蛋白的红外光谱酰胺Ⅰ带(1 600~1 700cm-1)去卷积谱(A)和曲线拟合谱(B)Fig.3 Deconvoluted spectrum(1 600~1 700cm-1)region(A)and the curve-fitted individual component bands(B)of amideⅠin glycinin FTIR spectrum
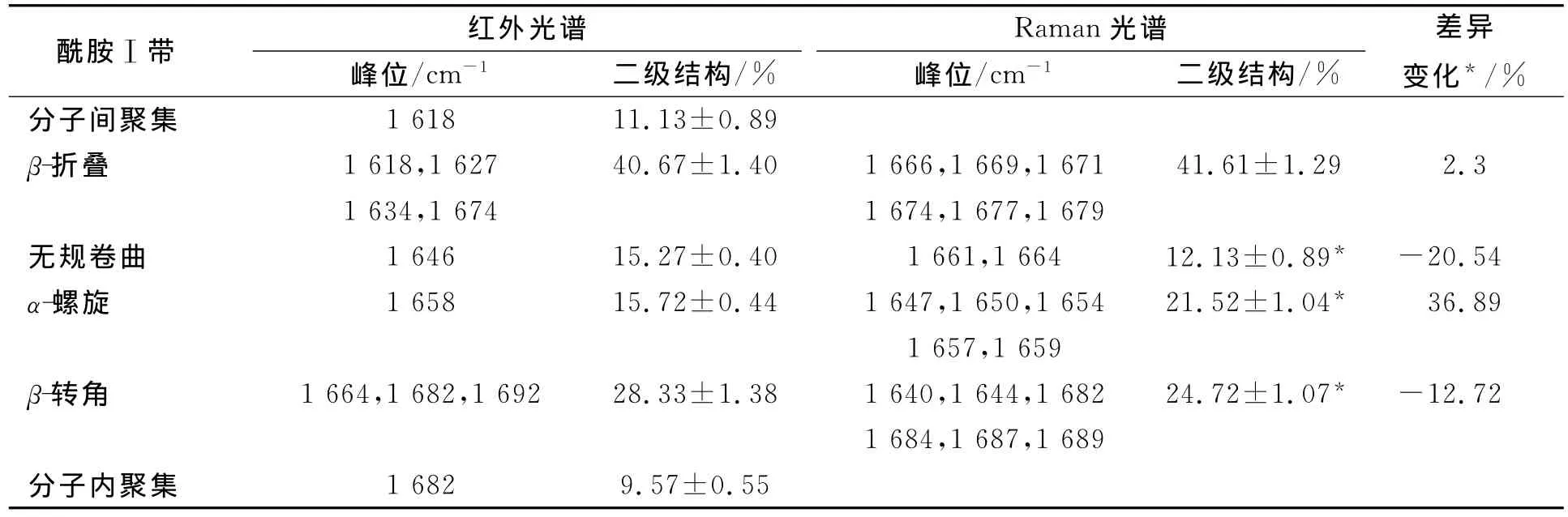
表2 大豆球蛋白的红外光谱和Raman光谱酰胺Ⅰ带曲线拟合结果Table 2 Results of amideⅠcurve-fitting obtained in FTIR spectrum and Raman spectrum for glycinin
2.3 大豆球蛋白的Raman谱分析
图4为大豆球蛋白Raman光谱400~2 000cm-1的特征吸收峰,大豆球蛋白的Raman光谱峰值及归属列于表3.由图4和表3可见,1 667cm-1峰位为酰胺Ⅰ带,1 242cm-1峰位为酰胺Ⅲ带.酰胺Ⅰ带和酰胺Ⅲ带常用于蛋白质主链骨架分析和定量计算二级结构.酰胺Ⅰ带中80%为C=O键伸缩振动,20%为C—N伸缩振动和N—H弯曲振动[25].酰胺Ⅲ谱带中40%为肽键的C—N伸缩振动,30%为N—H弯曲振动,由于酰胺Ⅲ谱带常混杂其他振动能带,因此很少单独用于判断蛋白质的二级结构[21].酰胺Ⅰ带1 640~1 690cm-1去卷积产生19个单 一 峰, 如 图 5(A)所 示, 其 特 征 峰 1 665~1 680cm-1归属为β-折叠,1 645~1 660cm-1归属为α-螺旋,1 640~1 645cm-1和1 680~1 690cm-1归属为β-转角,1 660~1 670cm-1归属为无规卷曲[13](表2).图5(B)结合去卷积峰位对酰胺Ⅰ带进行曲线拟合,获得的二级结构结果中β-折叠的百分数为(41.61±1.29)%,比红外光谱得到的结果高0.31%,差异不显著[12];而α-螺旋、β-转角和无规卷曲的百分数分别为(21.52±1.04)%,(24.72±1.07)%和(12.13±0.89)%,与红外光谱得到的结果相比,分别产生36.89%,-12.72%和-20.54%的显著差异性变化(p<0.05)(表2).Raman光谱不易得到适合做曲线拟合的理想蛋白质二级结构,去卷积提高光谱分辨率时也会增加噪声.由于噪音峰的随机性和信号峰稳定不变,因此通过多次扫描累加平均可消减噪声信号峰的影响.
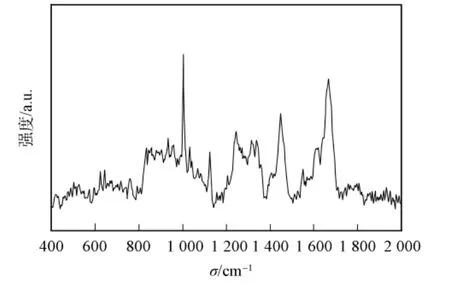
图4 大豆球蛋白Raman光谱Fig.4 Raman spectrum of glycinin
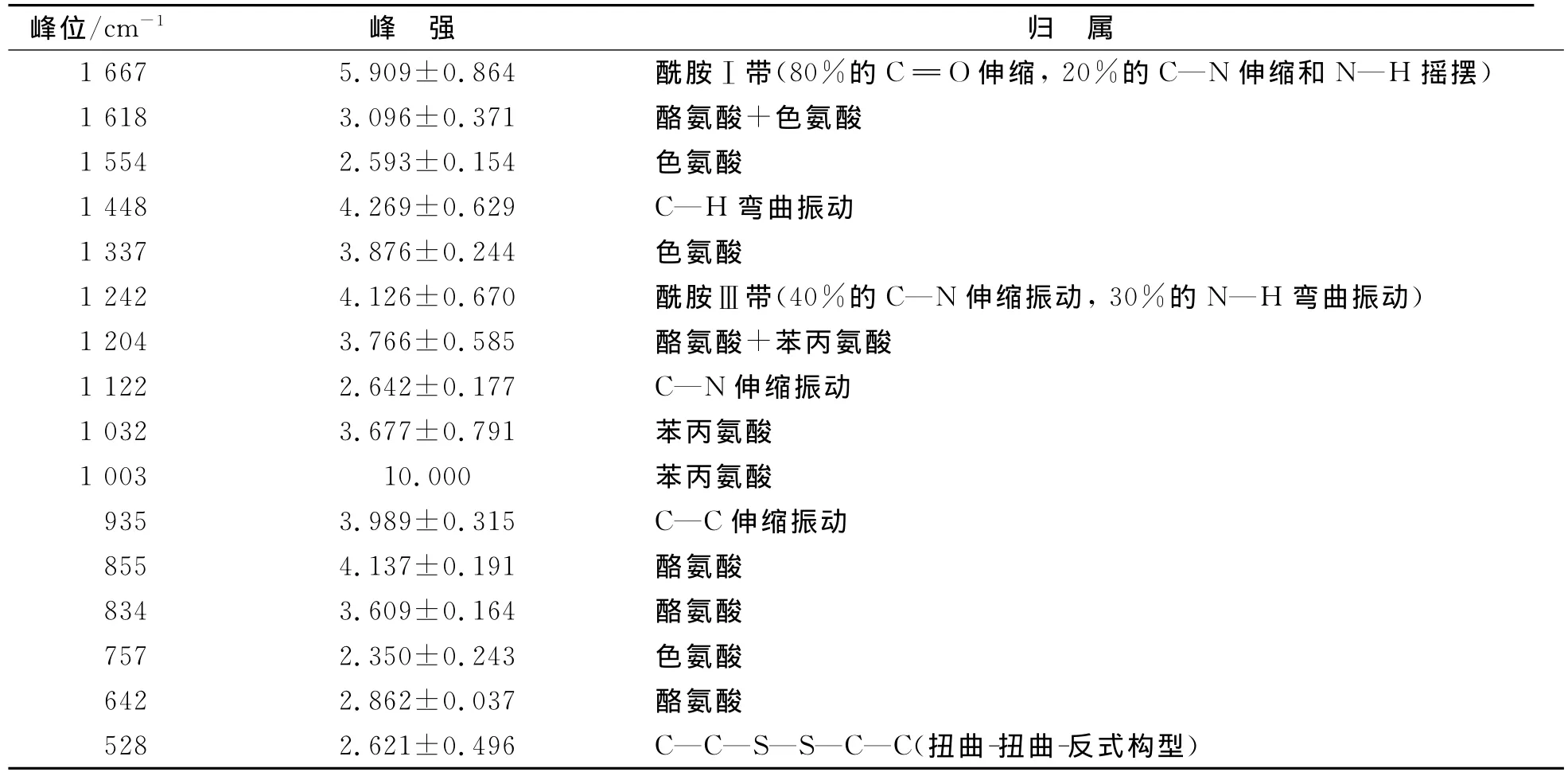
表3 大豆球蛋白的Raman光谱峰值及归属Table 3 Assigmnenst of Raman shift of glycinin
2.4 大豆球蛋白的侧链构象
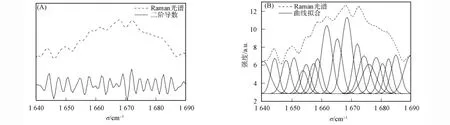
图5 Raman光谱酰胺Ⅰ带(1 640~1 690cm-1)去卷积谱(A)和曲线拟合谱(B)Fig.5 Deconvoluted spectrum(1 640~1 690cm-1)region(A)and the curve-fitted individual component bands(B)of amideⅠin glycinin Raman spectrum
855,834cm-1为酪氨酸残基的苯环呼吸振动和面外弯曲振动产生的费米共振双峰,可反映蛋白质中酪氨酸所处的环境.当I850/I830=1.25~1.40时,表明酪氨酸残基完全暴露于分子表面;当I850/I830=0.3~0.5时,酪氨酸残基完全埋藏于分子内部;当I850/I830=0.7时,酪氨酸残基为电离状态[26].本文中大豆球蛋白的I850/I830=1.146,“包埋”和“外露”的酪氨酸残基占酪氨酸残基总量的14%和86%.通过500~550cm-1处C—C—S—S—C—C键的特征谱带可判断二硫键的构型.500~510cm-1谱带属于扭曲-扭曲-扭曲式构型(g-g-g),515~525cm-1谱带属于扭曲-扭曲-反式构型(g-g-t),535~545cm-1谱带属于反式-扭曲-反式构型(t-g-t)[26].500~550cm-1多峰拟合结果表明,大豆球蛋白的二硫键构型中g-g-g,g-g-t和t-g-t构型的百分数分别为34.9%,32.0%和33.1%.大豆球蛋白中芳香族氨基酸苯丙氨酸、色氨酸和酪氨酸能提供重要的侧链信息.苯丙氨酸的特征谱峰主要位于622,1 003,1 032,1 204cm-1处,其中1 003cm-1处尖锐的振动峰为苯环的呼吸振动峰,其强度不受蛋白构象变化的影响,通常用作归一化因子;1 032cm-1峰位归属于苯丙氨酸的C—H面内弯曲振动.大豆球蛋白中色氨酸的吲哚环Raman谱峰位于757,1 337,1 554cm-1处,其中757cm-1峰位对色氨酸吲哚环的水化环境敏感[27],1 337cm-1处的谱线较强,表明大豆球蛋白中的色氨酸趋向于埋藏.642,834,855,1 204cm-1峰位归属于酪氨酸侧链的谱峰[28],I850/I830表明酪氨酸侧链主要趋向于暴露.大豆球蛋白的侧链构象分析能够反映大豆球蛋白的三级结构和四级结构.
综上所述,本文利用FTIR光谱对大豆球蛋白进行了分子内聚集和分子间聚集的定量分析,与FTIR光谱比较,Raman光谱指纹区数据显示了更高的分辨率,能提供更多的结构信息.
[1] Hojilla-Evangelista M P,Sessa D J,Mohamed A,et al.Functional Properties of Soybean Lupin Protein Concentrates Produced by Ultrafiltration-Diafiltration[J].J Am Oil Chem Soc,2004,81(12):1153-1157.
[2] Preeti L,Anil N N.Thermal and Mechanical Properties of Environment-Friendly‘Green’Plastics from Stearic Acid Modified-Soy ProteinIsolate[J].Industrial Crops and Products,2005,21(1):49-64.
[3] Staswick P E,Hermodson M A,Nielson N C,et al.Identification of the Acidic and Basic Subunit Complexes of Glycinin[J].J Biol Chem,1981,256(16):8752-8755.
[4] Adachi M,Kanamori J,Masuda T,et al.Crystal Structure of Soybean 11SGlobulin:Glycinin A3B4 Homohexamer[J].Proc Natl Acad Sci USA,2003,100(12):7395-7400.
[5] Thygesen L G,Lokke M M,Micklander E,et al.Vibrational Microspectroscopy of Food Raman vs FT-IR[J].Trends in Food Science &Technology,2003,14(1):50-57.
[6] Mills E N,Huang L,Noel T R,et al.Formation of Thermally Induced Aggregates of the Soya Globulin β-Conglycinin[J].Biochem Bioph Acta,2001,1547(2):339-350.
[7] Huson M G,Strourina E V,Kealley C S,et al.Effects of Thermal Denaturation on the Solid-State Structure and Molecular Mobility of Glycinin[J].Biomacromolecules,2011,12(6):2092-2102.
[8] Abbott T P,Nabetani H,Sessa D J,et al.Effects of Bound Water on FTIR Spectra of Glycinin[J].J Agric Food Chem,1996,44(8):2220-2224.
[9] Subirade M,Kelly I,Gueguen J,et al.Molecular Basis of Film Formation from a Soybean Protein:Comparison between the Conformation of Glycinin in Aqueous Solution and in Films[J].Int J Biol Macromol,1998,23(4):241-249.
[10] L’Hocine L,Boye J I,Jouve S.Ionic Strength and pH-Induced Changes in the Immunoreactivity of Purified Soybean Glycinin and Its Relation to Protein Molecular Structure[J].J Agric Food Chem,2007, 55(14):5819-5826.
[11] ZHAO Xiaoyan,CHEN Fusheng,XUE Wentong,et al.FTIR Spectra Studies on the Secondary Structures of Soybean 7Sand 11SGlobulins Using AOT Reverse Micellar Extraction[J].Food Hydrocolloids,2008,22(4):568-575.
[12] ZHAO Xiaoyan,QIANG Ao,CHEN Fusheng,et al.Effect of Reverse Micelle on Conformation of Soy Globulins:A Raman Study[J].Food Chemistry,2009,116(1):176-182.
[13] Lakemond C M,Jongh H H,de,Hessing M,et al.Soy Glycinin:Influence of pH and Ionic Strength on Solubility and Molecular Structure at Ambient Temperatures[J].J Agric Food Chem,2000,48(6):1985-1990.
[14] Byler D M,Brouillette J N,Susi H,et al.Quantitative Studies of Protein Structure by FT-IR Spectral Deconvolution and Curve Fitting[J].Spectroscopy,1986,1:29-32.
[15] 王斌,王靖,余江,等.FT-Raman光谱对蛋白质二级结构的定量分析[J].光谱学与光谱分析,1999,19(5):674-676.(WANG Bin,WANG Jing,YU Jiang,et al.A Quantitative Study on Secondary Structure of Proteins by FT-Raman Spectroscopy[J].Spectroscopy and Spectral Analysis,1999,19(5):674-676.)
[16] Nagano T,Hirotsuka M,Mori H,et al.Dynamic Viscoelastic Study on the Gelation of 7SGlobulin from Soybeans[J].J Agric Food Chem,1992,40(6):941-944.
[17] Laemmli U K.Cleavage of Structural Proteins during the Assembly of the Head of Bacteriophage T4[J].Nature,1970,227:680-685.
[18] Souillac P O,Middaugh C R,Rytting J H.Investigation of Protein/Carbohydrate Interactions in the Dried State Diffuse Reflectance FTIR Studies[J].Inter J Pharmaceut,2002,235(1/2):207-218.
[19] Tsutomu S,Mitsutaka K,Kazunobu T.Novel Method Using Phytase for Separating Soybeanβ-Conglycinin and Glycinin[J].Biosci Biotechnol Biochem,2001,65(4):884-887.
[20] Mahler H C,Friess W,Grauschopf U,et al.Protein Aggregation:Pathways,Induction Factors and Analysis[J].Journal of Pharmaceutical Sciences,2009,98(1):2909-2934.
[21] Ramirez F J,Luque P,Heredia A,et al.Fourier Transform IR Study of Enzymatically Isolated Tomato Fruit Cuticular Membrane[J].Biopolymers,1992,32(11):1425-1429.
[22] Sivakumar S,Sivasubramanian J,Raja B.Aluminium Induced Structural,Metabolic Alterations and Protective Effects of Desferrioxamine in the Brain Tissue of Mice:An FTIR Study[J].Spectrochimica Acta Part A:Molecular and Biomolecular Spectroscopy,2012,99:252-258.
[23] Kaddour A A T,Mondet M,Cuq B,et al.Description of Chemical Changes Implied during Bread Dough Mixing by FTTR Mid-infrared Spectroscopy[J].Cereal Chemistry,2008,85(5):673-678.
[24] Robertson G H,Gregorski K S,Cao T K,et al.Changes in Secondary Protein Structures during Mixing Development of High Absorption(90%)Flour and Water Mixtures[J].Cereal Chem,2006,83:136-142.
[25] Catalina D,Cristiano D,Eloïse L,et al.Raman and IR Spectroscopy of Manganese Superoxide Dismutase,a Pathology Biomarker[J].Vibrational Spectroscopy,2012,62:50-58.
[26] Hartwig S,Malgorzata B.Identification and Quantification of Valuable Plant Substances by IR and Raman Spectroscopy[J].Vib Spectrosc,2007,43(1):13-25.
[27] Susi H,Byler D M.Fourier Transform Infrared Spectroscopy in Protein Conformation Studies[M].Champaign:American Oil Chemists’Society,1988:235-250.
[28] Ellepola S W,Choi S M,Phillips D L,et al.Raman Spectroscopic Study of Rice Globulin[J].Journal of Cereal Science,2006,43(1):85-93.
(责任编辑:单 凝)
Analysis of Soy Glycinin by FTIR and Raman Spectroscopy
LONG Guohui1,JI Yuan2,PAN Hongbin3,4,SUN Zewei4,WANG Jinxin1,QIN Guixin4
(1.College of Life Science,Jilin Agricultural University,Changchun130118,China;2.College of Physics,Jilin University,Changchun130012,China;3.Key Laboratory of Animal Nutrition and Feed of Yunnan Province,Yunan Agricultural University,Kunming650201,China;4.College of Animal Science and Technology,Jilin Agricultural University,Changchun130118,China)
The molecular structure of glycinin was investigated by FTIR and Raman spectroscopy.The frequency and signal intensity of IR and Raman bands were assigned.The data suggest that FTIR and Raman spectra reflect a large amount of structural information.The secondary structure of glycinin was assessed by the deconvolution amideⅠband and curve-fitting.Quantitative analysis of secondary structure reveals that the results of the folded glycininβ-sheet had no significant difference,while the glycininα-helix,turning angle,random coil had significant difference(p<0.05).The IR bands at 1 618cm-1and 1 682cm-1were considered to reflect the formation of intermolecular aggregates(11.1%)and intramolecular aggregates(9.5%).The I850/830intensity ratio of Raman tyrosine doublet suggests that the contents of the buried tyrosine residue and exposed tyrosine residue were 14%and 86%in glycinin.
FTIR;Raman spectroscopy;glycinin;molecular structure
Q512
Q
1671-5489(2014)04-0840-07
蛋白质是生命体中最基本的物质,各种蛋白质的生物学功能与其特有的各级结构密切相关[1-2].大豆球蛋白约占大豆蛋白总量的40%,相对分子质量为300 000~380 000,由2个三聚体堆叠成为六聚体,6种酸性多肽(A1a,A1b,A2,A3,A4,A5)和5种碱性多肽(B1a,B1b,B2,B3,B4)组成5个单体A1bB2,A2B1a,A1aB1b,A5A4B3和A3B4,每个单体均由1个酸性亚基(约35 000)和1个碱性亚基(约20 000)构成,并通过1个二硫键连接[3-4].大豆球蛋白的分子结构紧密,亚基之间高度聚集和压缩,大量酶切位点包藏于蛋白质分子内部,具有较强的抗蛋白酶消化能力[5].大豆球蛋白聚集方式对食物的流变和质构等功能性质影响较大[6].天然大豆球蛋白具有典型的二级结构、三级结构及四级结构,同时其亚基有规律的空间排布形成了部分晶态结构,是研究蛋白质生物聚合物较好的模式蛋白[7].
10.13413/j.cnki.jdxblxb.2014.04.41
2013-10-15.
龙国徽(1971—),男,汉族,硕士,讲师,从事生物化学与分子生物学的研究,E-mail:longguohui@jlau.edu.cn.通信作者:秦贵信(1956—),男,汉族,博士,教授,从事大豆抗营养因子的研究,E-mail:qgx2013@sohu.com.
国家自然科学基金(批准号:11274138).

