北太平洋柔鱼微卫星标记的筛选及遗传多样性
刘连为,陈新军,2,3,*,许强华,2,3,李伟文
(1. 上海海洋大学海洋科学学院, 上海 201306; 2. 国家远洋渔业工程技术研究中心,上海 201306;3. 上海海洋大学大洋渔业资源可持续开发省部共建教育部重点实验室,上海 201306)
柔鱼(Ommastrephesbartramii)广泛分布于全球温带、副热带海域,目前被规模性开发利用的海域主要在北太平洋[1- 2]。根据不同的产卵季节与地理位置,北太平洋柔鱼被划分为冬春生西部群体、冬春生中东部群体、秋生中部群体与秋生东部群体[3]。其中,冬春生西部群体为我国大陆鱿钓船的传统捕捞对象,而秋生群体主要为原流刺网作业海域捕捞对象。柔鱼资源量丰富,且在海洋生态系统中占据重要的地位,被认为是最重要的大洋性柔鱼类之一[4]。国内外学者对柔鱼渔业生物学[5- 6]、资源评估[7- 8]等方面进行深入的研究,有关其群体遗传学的研究体现在不同的研究方法上。Katugin[9]运用蛋白质电泳技术检测出北太平洋柔鱼东西部群体由于等位基因频率不连续分布产生的遗传差异。而刘连为等[10]基于线粒体DNA标记研究了北太平洋柔鱼种群遗传结构,认为柔鱼冬春生群体与秋生群体以及不同的地理群体之间不存在显著的遗传分化。作为渔业管理的基本单元,柔鱼各群体间是否存在显著的遗传变异有待进一步研究。
微卫星DNA是一种广泛分布于真核生物基因组中的串状重复序列,又称简单序列重复(simple sequence repeat,SSR)。SSR标记具有高度多态性、共显性、实验操作相对简单等优点,使得它在群体遗传学分析、基因克隆、育种改良以及连锁图谱构建等领域得到广泛的应用[11]。目前,头足类很多种类的微卫星标记已被分离、筛选,并应用到群体遗传多样性与种群遗传结构研究中[12- 13]。而柔鱼微卫星DNA的分离在国内外尚未见报道,本研究采用磁珠富集法筛选柔鱼微卫星标记,并应用于群体遗传多样性检测,以期为柔鱼资源的可持续利用和科学管理提供理论基础。
1 材料与方法
1.1 实验材料
柔鱼样本按照地理位置划分为西北群体和东北群体,存放于船舱冷库中并运回至实验室。取套膜肌肉组织,置于95%乙醇中,保存于-20 ℃冰箱内。西北群体和东北群体分别由3个部分组成,命名为西北群体1(northwest population 1,NW1)、西北群体2(northwest population 2,NW2)、西北群体3(northwest population 3,NW3)与东北群体1(northeast population 1,NE1)、东北群体2(northeast population 2,NE2)、东北群体3(northeast population 3,NE3)。利用耳石微结构获得的日龄数据并结合捕捞日期推算孵化时间,划分出冬春生群体(NW1、NW2、NE1)和秋生群体(NW3、NE2、NE3)(表1)。限制性内切酶Sau3AI、T4 DNA连接酶、pMD19-T载体、大肠杆菌DH5α购自宝生物工程(大连)有限公司。Biospin Gel Extraction Kit购自杭州博日科技有限公司,Dynabeads M- 280 Streptavidin为Invitrogen Dynal AS产品。探针(AG)12、(AC)12、接头引物及微卫星引物由上海捷瑞生物工程有限公司合成。

表1 柔鱼样本采集信息
1.2 基因组DNA的提取
采用组织/细胞基因组DNA快速提取试剂盒(北京艾德莱生物科技有限公司)提取DNA,洗脱缓冲液EB溶解,-20 ℃保存备用。用1%琼脂糖凝胶电泳检测DNA质量,紫外分光光度计检测DNA浓度。
1.3 微卫星富集文库的构建及微卫星标记的筛选
微卫星富集文库的构建主要参考孙效文等[14]方法。限制性内切酶Sau3AI对基因组DNA进行酶切,Biospin Gel Extraction Kit回收250—1000 bp大小片段。基因组DNA片段与Brown接头连接:
A:5′-GATCGTCGACGGTACCGAATTCT
B:5′-GTCAAGAATTCGGTACCGTCGAC
最后将富集的微卫星DNA片段经pMD19-T载体连接并转化到大肠杆菌DH5α中,从而建立柔鱼部分基因组文库。挑选含有微卫星片段的阳性克隆进行测序,根据其核心序列两端保守的侧翼序列,用Primer Premier 5.0软件进行引物设计。PCR扩增筛选具有较高特异性与多态性的微卫星标记。
1.4 微卫星PCR扩增
PCR反应总体积为25 μL,其中10×PCR Buffer 2.5 μL、TaqDNA polymerase(5 U/μL)0.2 μL、dNTP(各2.5 mmol/L)2 μL、上下游引物(10 μmol/L)各0.6 μL、DNA模板20 ng、ddH2O补足体积。PCR扩增反应程序为:94 ℃预变性2 min;94 ℃变性30 s,退火30 s,72 ℃延伸45 s,35个循环;72 ℃最后延伸2 min。PCR产物经过1.2%琼脂糖凝胶电泳分离、Biospin Gel Extraction Kit纯化后,送至上海迈浦生物科技有限公司,通过ABI3730XL全自动DNA测序仪分析,以ROX- 500为分子量内标,通过Genemapper Version 3.5软件读取微卫星扩增产物的分子量数据。
1.5 数据统计与分析
根据分子量数据确定个体各位点基因型,利用POPGEN 3.2[15]进行群体遗传学分析,计算等位基因数(Na),有效等位基因数(Ne),观测杂合度(Ho),期望杂合度(He)与Shannon多样性指数(Shannon′s information index,I)。多态信息含量(PIC)由CERVUS 3.0[16]软件计算,并采用马尔科夫链(Markov Chain)方法进行Hardy-Weinberg平衡检验。利用ARLEQUIN 3.5[17]计算群体遗传分化的F-统计量(F-statistics,Fst)及分子方差分析(AMOVA)。利用POPGEN 3.2计算群体间的Nei′s遗传距离,并基于该遗传距离用MEGA 5.0[18]构建UPGMA系统发生树。根据遗传距离及地理距离数据在Excel中作线性相关图,其中两地理坐标间直线距离计算方法参照韩忠民[19]的方法。利用STRUCTURE 2.3[20]进行群体遗传结构分析,分析最佳K值,即群体遗传结构的理论群体数。
2 结果
2.1 微卫星序列特征
随机挑取800个菌落,经菌落PCR检测后,共有68个阳性克隆,阳性克隆率为8.5%。对所有的阳性克隆进行测序,有60个(88.24%)克隆含有微卫星序列(部分微卫星序列已提交到Genbank,Genbank登录号为:KC855227—KC855255)。重复次数在11—20次间的34个(44.74%),重复20次以上的微卫星32个(42.10%),最高重复次数为33次。其中,完美型微卫星46个(60.53%),非完美型微卫星28个(36.84%),混合型微卫星2个(2.63%)(表2)。除探针使用的AC/TG、AG/TC重复外,还得到ACAG、AGAC重复序列。

表2 柔鱼微卫星不同类型重复序列所占比例
2.2 微卫星位点的多态性及群体的遗传多样性
选择合成的20对微卫星引物对柔鱼30个个体进行PCR扩增,结果有12对能扩增出特异条带,其中8对具有多态性,并应用于6个不同地理位置的柔鱼群体遗传学分析,引物的核心序列、产物大小及退火温度等情况如表3所示。各位点在所有群体中的扩增结果见表4,等位基因数(Na)为44—52个,有效等位基因数(Ne)介于4.11—30.15之间;观测杂合度(Ho)介于0.640—0.840之间,期望杂合度(He)介于0.747—0.969之间;多态信息含量(PIC)介于0.787—0.987之间,均为高度多态性位点(PIC>0.5),Shannon多样性指数(I)介于1.772—3.638之间。位点Bo103与位点Bo105极显著偏离Hardy-Weinberg平衡(P<0.01)。
6个不同地理位置的柔鱼群体遗传多样性如表5所示。NW1群体的平均等位基因数最多(Na=25.13),NW3群体最少(Na=15.00);NW2群体的平均有效等位基因数最多(Ne=9.91),NW3群体最少(Ne=7.96);NE2群体的平均观测杂合度最高(Ho=0.761),NW3群体最低(Ho=0.672);NW1群体的平均期望杂合度最高(He=0.851),NE1群体最低(He=0.808);NW1群体的多态性息含量最高(PIC=0.918),NE3群体最低(PIC=0.779)。NW1群体的Shannon多样性指数最高(I=2.571),NW3群体最少(I=2.215)。总体上,6个不同地理位置的柔鱼群体均具有较高的遗传多样性。

表3 柔鱼8对微卫星引物特征

表4 柔鱼8个微卫星位点多态性

表5 6个不同地理位置的柔鱼群体遗传多样性
2.3 群体遗传分化与遗传结构分析
AMOVA分析显示群体间遗传差异主要来自于个体间,仅有0.56%的遗传差异来自于群体间,遗传变异不显著(表6)。NW1群体与NE2群体间遗传分化系数Fst为负值,说明两群体间无遗传分化。其余群体间遗传分化系数介于0.0016—0.0244之间,属于轻微程度的分化(Fst<0.05),且统计检验不显著,进一步表明群体间不存在显著的遗传差异。基于Nei′s遗传距离(表7)的UPGMA聚类树显示,NE1群体与NE3群体聚为一族群,其余4个群体聚为一族群,且NW1群体与NE2群体首先聚在一起(图1)。群体间的遗传距离与地理距离相关性分析结果如图2所示,二者没有呈现出正相关性(R=0.175,P>0.05)。STRUCTURE 2.3软件执行2—6的假设K值,重复次数为10次,根据K值对应参数的趋势分析,推断本研究所有参试个体最佳分组为1个理论群。

表6 柔鱼群体分子方差分析
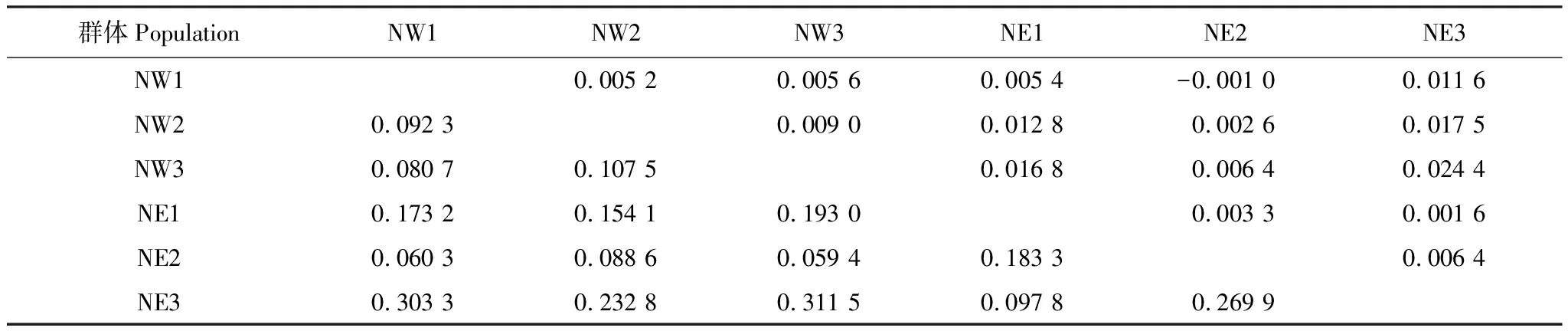
表7 柔鱼群体间Nei′s遗传距离(DA,对角线以下)及F统计值(Fst,对角线以上)

图1 基于Nei′s遗传距离的UPGMA聚类树

图2 群体间Nei′s遗传距离和地理距离的散点图
3 讨论与分析
本文以北太平洋传统作业渔场柔鱼为样本对分离的微卫星位点进行多态性检测,筛选出8个多态性位点(Ho=0.754),所有样本均扩增出条带。应用这些微卫星位点对北太平洋6个不同地理位置的柔鱼群体进行遗传多样性分析,平均每个微卫星位点获得50.5个等位基因与17.08个有效等位基因,多态性信息含量均较高(PIC>0.5),适用于柔鱼群体遗传学分析。位点Bo103与位点Bo105对NE1群体及NE3群体扩增时,部分样本未扩增出条带,个别样本仅显现小片段等位基因的条带,造成该位点纯合体个体过剩。Hardy-Weinberg平衡检验结果显示这2个位点极显著偏离Hardy-Weinberg平衡,因此,2个位点很可能存在无效等位基因。无效等位基因产生的原因主要为微卫星侧翼序列变异,从而导致该位点无法正常扩增[21]。而大片段等位基因丢失也会造成无效等位基因,这些在实验结果中均体现出[22]。无效等位基因会对遗传学相关研究结果造成显著影响,在以后的实验中可通过重新设计微卫星引物或者估算无效等位基因频率进行群体遗传学研究。
6个不同地理位置的柔鱼群体平均观察杂合度介于0.672—0.761,平均期望杂合度介于0.808—0.851,与太平洋褶柔鱼(Todarodespacificus)和阿根廷滑柔鱼(Illexargentinus)的杂合度相近,分别为Ho:0.200—0.950,He:0.644—0.950;Ho:0.59—0.93,He:0.62—0.96[23- 24]。因此,各群体显示出较高的杂合性。Katugin[9]利用18个蛋白酶位点检测北太平洋柔鱼东西部2个群体的遗传变异水平,只有1个蛋白酶位点显示出明显的遗传差异及较高的杂合度(Ho=0.294,He=0.322)。东西部群体平均观察杂合度分别为:0.060 ± 0.021与0.043 ± 0.022,平均期望杂合度分别为0.056 ± 0.020与0.043 ± 0.022,远远低于微卫星位点检测的杂合度水平。蛋白质标记为基因表达的产物,所检测到的遗传差异水平很容易受到周围环境的影响。刘连为等[10]利用线粒体标记检测出北太平洋柔鱼2个产卵季节群体具有较低的核苷酸多样性水平。由此可见,微卫星标记应用于物种群体遗传变异水平研究较其它分子遗传标记有一定的优越性。
本研究所取样本为东西部2个不同产卵群体,其中NW1、NW2、NE1群体为冬春生群体,NW3、NE2、NE3群体为秋生群体。两两群体间的Fst值与AMOVA分析结果显示,群体间的遗传分化程度较低,未达到显著性水平,遗传差异主要存在于个体间。6个群体UPGMA聚类分析显示,NE1群体和NE3群体亲缘关系最近,聚为一类,而地理位置相距较远的NW1群体与NE2群体亲缘关系最近,另聚为一类,而且它们属于不同的产卵季节群体。群体间的遗传距离与地理距离相关性结果进一步说明了不同地理群体间的亲缘关系远近与地理距离不成正相关性。北太平洋海域缺乏显著的地理障碍,而且柔鱼个体具有较强的游泳能力,在海流的作用下,群体之间存在较强的基因交流[25]。Chen等[26]基于胴长与耳石轮纹半径的线性关系在东北太平洋和西北太平洋柔鱼群体间的差异,推测两部分大型雌性群体(胴长>350 mm)可能来自于同一群体,这与利用STRUCTURE 2.3软件推断的北太平洋柔鱼存在1个理论群相一致。
柔鱼资源量丰富,已有研究认为传统作业渔场最大可持续产量为8×104—10×104t[7],而原流刺网作业海域柔鱼资源量则超过30×104t[8]。但作为短生命周期种类,柔鱼种群资源量的大小很容易受到环境变化的影响而发生波动[27]。2009年8—10月旺汛期间由于水温变动,传统作业渔场柔鱼产量出现大幅度下降,其日产量仅为正常年份的一半[28]。2010—2012年本课题组对原流刺网作业海域柔鱼资源量进行探捕调查,取得了较高的产量。因此,在对北太平洋柔鱼资源进行评估与管理时,为了更加合理地开发、利用与保护该渔业资源,建议将不同的产卵季节—地理群体看作1个管理单元。
:
[1] Murata M. Oceanic resources of squids. Marine and Freshwater Behaviour and Physiology, 1990, 18(1): 19- 71.
[2] Araya H. Fishery, biology and stock assessment ofOmmastrephesbartramiiin the North Pacific Ocean. Memoirs of the National Museum Victoria, 1983, 44: 269- 283.
[3] Yatsu A, Midorikawa S, Shimada T, Uozumi Y. Age and growth of the neon flying squid,Ommastrephesbartramii, in the North Pacific Ocean. Fisheries Research, 1997, 29(3): 257- 270.
[4] Coll M, Navarro J, Olson R J, Christensen V. Assessing the trophic position and ecological role of squids in marine ecosystems by means of food-web models. Deep-Sea Research II, Topical Studies in Oceanography, (2012-08- 23) [2013-03-01]. http://dx.doi.org/10.1016/j.dsr2.2012.08.020.
[5] Ma J, Chen X J, Liu B L, Tian S Q, Cao J. Review of fisheries biology of neon flying squidOmmastrephesbartramiiin the North Pacific Ocean. Journal of Shanghai Ocean University, 2011, 20(4): 563- 567.
[6] Chen X J, Tian S Q, Chen Y. Fisheries Biology of the Neon Flying Squid,OmmastrephesBartramiiin the North Pacific Ocean. Beijing: Science Press, 2011: 1- 3.
[7] Chen X J, Chen Y, Tian S Q, Liu B L, Qian W G. An assessment of the west winter-spring cohort of neon flying squid (Ommastrephesbartramii) in the Northwest Pacific Ocean. Fisheries Research, 2008, 92(2/3): 221- 230.
[8] Ichii T, Mahapatra K, Okamura H. Stock assessment of the autumn cohort of neon flying squid (Ommastrephesbartramii) in the North Pacific based on past large-scale high seas driftnet fishery data. Fisheries Research, 2006, 78(2/3): 286- 297.
[9] Katugin O N. Patterns of genetic variability and population structure in the North Pacific squidsOmmastrephesbartramii,Todarodespacificus, andBerryteuthismagister. Bulletin of Marine Science, 2002, 71(1): 383- 420.
[10] Liu L W, Xu Q H, Chen X J. Population genetic structure ofOmmastrephesbartramiiin the North Pacific Ocean based on theCOI andCytb gene sequences analysis. Journal of Fisheries of China, 2012, 36(11): 1675- 1684.
[11] Liu Y G. DNA Molecular Marker Technology in Aquatic Organisms. Beijing: Science Press, 2009: 59- 62.
[12] Dillane E, Galvin P, Coughlan J, Lipinsiki M, Cross T F. Genetic variation in the lesser flying squidTodaropsiseblanae(Cephalopoda, Ommastrephidae) in east Atlantic and Mediterranean waters. Marine Ecology Progress Series, 2005, 292: 225- 232.
[13] Doubleday Z A, Semmens J M, Smolenski A J, Shaw P W. Microsatellite DNA markers and morphometrics reveal a complex population structure in a merobenthic octopus species (Octopusmaorum) in south-east Australia and New Zealand. Marine Biology, 2009, 156(6): 1183- 1192.
[14] Wen X X, Sun X W. Isolation and screening of microsatellite markers from Argus snakehead fishOphiocephalusargus. Journal of Dalian Ocean University, 2010, 25(3): 260- 264.
[15] Nei M. Estimation of average heterozygosity and genetic distance from a small number of individuals. Genetics, 1978, 89(3): 583- 590.
[16] Kalinowski T, Taper M L, Marshall T C. Revising how the computer program cervus accommodates genotyping error increases success in paternity assignment. Molecular Ecology, 2007, 16(5): 1099- 1106.
[17] Excoffier L, Lischer H E. Arlequin suite ver3.5: a new series of programs to perform population genetics analyses under Linux and Windows. Molecular Ecology Resources, 2010, 10(3): 564- 567.
[18] Tamura K, Peterson D, Peterson N, Stecher G, Nei M, Kumar S. MEGA5: molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods. Molecular Ecology and Evolution, 2011, 28(10): 2731- 2739.
[19] Han Z M. Exact calculation of the distance between two points according to longitude and latitude. Applied Technology, 2011(11): 196, 174- 174.
[20] Pritchard J K, Stephens M, Donnelly P. Inference of population structure using multilocus genotype data. Genetics, 2000, 155(2): 945- 959.
[21] Lehmann T, Hawley W A, Collins F H. An evaluation of evolutionary constraints on microsatellite loci using null alleles. Genetics, 1996, 144(3): 1155- 1163.
[22] Wattier R, Engel C R, Saumitou-Laprade P, Valero M. Short allele dominance as a source of heterozygote deficiency at microsatellite loci: experimental evidence at the dinucleotide locus Gv1CT in Gracilaria gracilis (Rhodo-phyta). Molecular Ecology, 1998, 7(11): 1569- 1573.
[23] Iwata Y, Lian C L, Sakuri Y. Development of microsatellite markers in the Japanese common squidTodarodespacificus(Ommastrephidae). Molecular Ecology Resources, 2008, 8(2): 466- 468.
[24] Adcock G J, Shaw P W, Rodhouse P G, Carvalho G R. Microsatellite analysis of genetic diversity in the squidIllexargentinusduring a period of intensive fishing. Marine Ecology Progress Series, 1999, 187: 171- 178.
[25] Tanaka H. Tracking the neon flying squid by the biotelemetry system in the central North Pacific Ocean. Aquabiology, 2001, 23(6): 533- 539.
[26] Chen C S, Chiu T S. Variations of life history parameters in two geographical groups of the neon flying squid,Ommastrephesbartramii, from the North Pacific. Fisheries Research, 2003, 63(3): 349- 366.
[27] O′Dor R K, Dawe E G.Illexillecebrosus// Rodhouse P G, Dawe E G, O′Dor R K, eds. Squid Recruitment
Dynamics: The Genus Illex as a Model, the Commercial Illex Species and Influences on Variability. Rome: FAO, 1998: 77- 104.
[28] Chen F, Chen X J, Qian W G, Liu B L, Tian S Q. Influence of variability of temperature on decline of catch forOmmastrephesbartramiiin the Northwestern Pacific Ocean in 2009. Journal of Guangdong Ocean University, 2010, 30(1): 65- 71.
参考文献:
[5] 马金, 陈新军, 刘必林, 田思泉, 李思亮, 曹杰. 北太平洋柔鱼渔业生物学研究进展. 上海海洋大学学报, 2011, 20(4): 563- 567.
[6] 陈新军, 田思泉, 陈勇. 北太平洋柔鱼渔业生物学. 北京: 科学出版社, 2011: 1- 3.
[10] 刘连为, 许强华, 陈新军. 基于线粒体COI和Cytb基因序列的北太平洋柔鱼种群遗传结构研究. 水产学报, 2012, 36(11): 1675- 1684.
[11] 刘云国. 水产生物DNA分子标记技术. 北京: 科学出版社, 2009: 59- 62.
[14] 温晓曦, 孙效文. 乌鳢微卫星DNA分子标记的分离及筛选. 大连海洋大学学报, 2010, 25(3): 260- 264.
[19] 韩忠民. 知经纬度计算两点精确距离. 科技传播, 2011(11): 196, 174- 174.
[28] 陈锋, 陈新军, 钱卫国, 刘必林, 田思泉. 水温变动对2009年西北太平洋柔鱼产量下降的影响. 广东海洋大学学报, 2010, 30(1): 65- 71.

