大肠杆菌苏氨酸合成途径动力学模型的构建与分析
杨雪,张彦飞,郑阳阳,马红武
中国科学院天津工业生物技术研究所 中国科学院系统微生物工程重点实验室,天津 300308
伴随着组学数据的大量获得,各种组学尺度的代谢网络模型不断发展和完善。利用通量平衡分析等算法可以由代谢网络模型求得某一产物生成的最优途径。获得的结果在代谢途径设计、敲除靶点预测等方面都具有重要指导作用[1]。与基因组规模代谢网络模型相对应,动力学模型由于需要较多的动力学参数数据一般只针对包含较少反应的代谢途径。但动力学模型具有更强的预测能力,可以分析在各种扰动下途径速率的变化,通过模型模拟和控制分析更准确地预测敲除和扩增靶点,从而为高效细胞工厂的理性设计提供指导[2]。目前人们进行动力学模型构建主要是基于已有的生化知识来选择在模型中包括哪些反应,而由基因组尺度代谢网络模型求得的最优途径也可以作为动力学模型构建的出发点。与传统方法相比,这种方法得到的模型自身就是满足能量和还原力平衡的,因此不需要人为设定一些代谢物如ATP、ADP、NADH等的浓度。本文中即采用这种新的方法确定了大肠杆菌中由葡萄糖出发合成苏氨酸的代谢途径的动力学模型的初始反应集,进而结合以前发表的相关模型中的动力学数据确定各反应的动力学方程和参数,最终得到了一个完整的能量和还原力平衡的苏氨酸合成途径动力学模型。
作为鸟类和哺乳类动物的必需氨基酸之一,苏氨酸对维持人类与动物的营养和健康具有重要意义,被广泛应用于食品、饲料、药品及化工领域,全球需求量逐年增长[3]。目前,苏氨酸主要通过大肠杆菌等细菌的生物发酵制备[4]。为选育苏氨酸高产菌株提供指导是人们构建苏氨酸合成途径动力学模型的主要目的。Chassagnole等曾提出一个基于实验数据确立的苏氨酸合成动力学模型,通过模型对苏氨酸合成途径中各步反应对苏氨酸合成通量的影响进行了深入分析。但该模型中仅包括从天门冬氨酸到苏氨酸的5步反应,过于简单[5-6]。2009年,该实验室进一步将该模型发展为包括了从葡萄糖出发到苏氨酸的完整合成途径,并结合代谢途径的通量分布的测量结果预测和验证了丙酮酸激酶敲除对苏氨酸合成的促进作用[7]。苏氨酸合成是一个高还原力和能量需求的过程,实际苏氨酸菌种改造过程中常常涉及到包括中心代谢内的其他代谢途径以满足还原力和能量平衡的需求,但在已发表的模型中均未考虑能量和还原力的供给而是直接固定ATP、NADH等的浓度,这样就会使得计算结果的准确度受到影响。本文针对这一问题,以基因组规模代谢网络分析求得的最优途径为出发点来构建动力学模型,不但考虑苏氨酸合成的碳源需求,还考虑了其合成过程中消耗的ATP、还原力等对碳源的需求,可以使计算结果更为可靠。
1 动力学模型构建
1.1 动力学模型结构的确定
我们用大肠杆菌的基因组规模代谢网络模型iJO1366[8]计算了从葡萄糖出发合成苏氨酸的最优代谢途径。发现最优途径中糖酵解主要通过磷酸戊糖途径进行,但考虑到实验测得的通量分布中以EMP途径为主[7],我们构建的动力学模型中将EMP途径和磷酸戊糖途径都包括了进来,整体代谢途径如图 1所示。为了构建该完整代谢途径的动力学模型,我们主要参考了两个已发表的模型来确定动力学方程和参数,分别为从葡萄糖到丙酮酸的中心代谢途径动力学模型 (图 1中细实线部分)[9]和从天冬氨酸到苏氨酸合成途径的模型(图 1中虚线框内的反应)[5-6]。以上两个模型已被BioModels Database收录[10],模型编号分别为051和066,下文简称为模型a和模型b。其中模型a模拟的是细胞生长过程,其中包括了大量用于生长的稀释反应及用于生物质生成的合并反应。因为我们建模的目的是模拟能量和还原力平衡的苏氨酸合成过程,所以去掉了这些与生长相关的反应。同样的,模型 b中包含的两个与生长过程相关的NADPH和ATP的内源消耗反应也去掉了。由于赖氨酸和苏氨酸均由天门冬氨酸合成得到且第一步反应相同 (同工酶),我们在模型中亦考虑了赖氨酸合成这一分支途径,以及赖氨酸对天冬氨酸激酶III的抑制影响,以考察酶量和调控特性的改变对两个分支途径间通量分配比的影响。
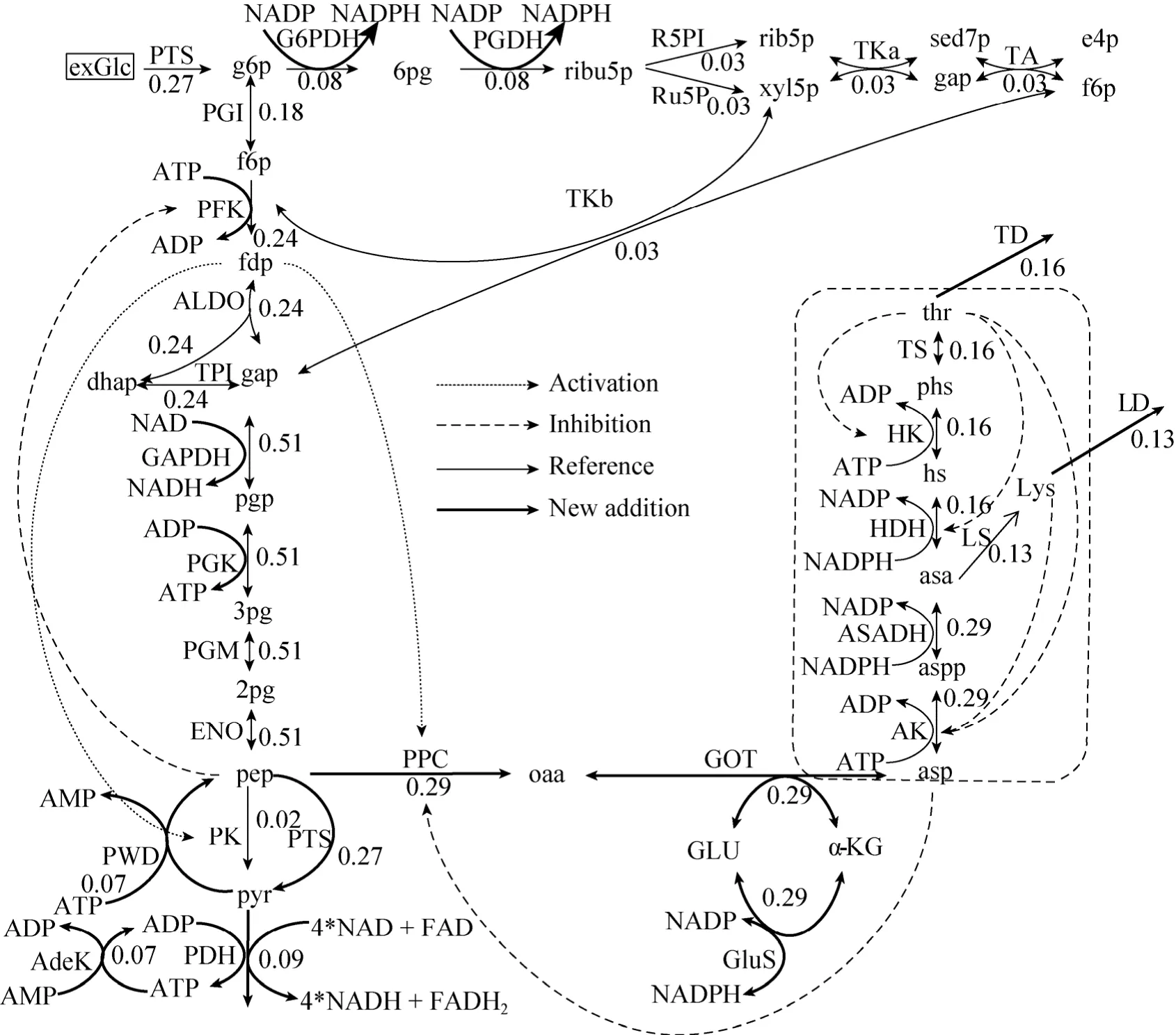
图1 大肠杆菌产苏氨酸模型的生物合成途径及稳态下的通量分布 (酶和代谢物的全名见表2、表3)Fig. 1 Metabolic pathways in the E. coli threonine biosynthesis model and the flux distribution in the metabolic pathways at steady state.
模型 a和b之间存在缺口,为了整合两个模型,我们通过BRENDA数据库的动力学数据和其他文献发掘工作为13个新反应添加了相应的动力学方程和参数值等相关信息,模型中新增反应及其方程详见表1。下面对这些新增反应的根据进行详细阐述。
PPC和GOT两步反应使模型a中的磷酸烯醇式丙酮酸可以形成草酰乙酸继而生成天冬氨酸,与模型b实现连通。由于谷草转氨酶反应 (GOT)同时将谷氨酸转化为α-酮戊二酸,因此需要新反应 GluS由酮戊二酸生成谷氨酸以实现两个代谢物的平衡。以上两个反应的动力学方程均遵守Ping-Pong机制[11-12],动力学参数的取值参考自BRENDA数据库,各反应的rmax取值见表3。
模型a中葡萄糖的转运是通过PTS实现的,在这个过程中pep转化为丙酮酸,并且pep还通过回补途径生成草酰乙酸 (反应 PPC)用于苏氨酸合成,因此在代谢网络分析求得的最优途径中还包括反应PWD以将pyr转化回pep。同时由于该过程生成了 AMP,还需要 AdeK反应以保证AMP的产生与消耗之间的平衡。以上两个反应均为不可逆反应,动力学方程采用双底物不可逆顺序反应机制[15-16]。

表1 模型中新增反应及其动力学方程Table 1 New reactions and kinetic rate equations in the integrated model

续表1
Rodríguez-Prados等的实验分析表明苏氨酸合成时三羧酸循环各反应亦有较高活性, 主要是为苏氨酸合成提供能量和还原力[7]。在我们的模型中类似于模型a采用了一个合并反应 (PDH)来表示pyr经过三羧酸循环后分解产生 ATP、NADH和FADH2的过程。由于大部分的ATP都是通过氧化磷酸化途径产生的,我们在模型中添加了 RCR1(Respiratory chain reaction)和RCR2两个反应用以体现NADH和FADH2经呼吸链电子传递后生成ATP的过程。其中 ATP/NADH的转化计量系数1.85和ATP/FADH2的转化计量系数1都是基于文献报道的呼吸链效率确定,均低于其最大理论值[20]。由于PDH、RCR1和RCR2这3个反应均为多个反应的合并反应,无法通过酶动力学机理确定合适的动力学方程,因此我们补全PDH反应式中的计量关系后,采用了模型a中原有的PDH反应动力学方程及参数。RCR1反应的动力学方程以呼吸链第一步反应的 Ping-Pong机制及其动力学参数值代表[18]。由于RCR2中的FADH2的消耗速率与PDH中的FADH2生成速率必须保持相同,RCR2的动力学方程及其参数值设置与PDH中基本相同,不同之处在于其反应速率随FADH2的浓度变化而变化[9]。NDPK反应实现了 ATP和GTP(三羧酸循环途径中产生)之间的转换,反应动力学机制为Ping-Pong机制[19]。三羧酸循环过程产生的主要是 NADH,而苏氨酸合成需要的是NADPH。PP途径产生的NADPH不足以满足需要,需要由NADH转化得到。因此我们添加了NT反应以实现NADH和NADPH之间的转化,反应动力学方程采用可逆的双底物顺序反应机制[17]。赖氨酸合成途径也用一个合并反应LS表示, 其受产物赖氨酸的抑制,反应动力学方程参考 Contador等构建的模型[14]。苏氨酸和赖氨酸的排出反应TD和LD均采用一级反应动力学方程,其反应速率与浓度成正比 (文中所涉及的全部反应与代谢物名称简写的全名参见表2、表3)。
1.2 模型修正及初始浓度设置
为保障模型构建的准确性,首先检查两个模型中的计量关系和反应可逆性信息。两个模型由于反应物的代谢途径关系不完整,往往需要固定大量的反应物浓度才可使模型达到稳态。整合后的模型由于添加了13个新反应和完善了能量及还原力的计量关系,在极大程度上提高了模型的自洽能力。新模型中,ATP、NADH、NADPH和FADH2等物质从模型 a中固定浓度的全局变量,调整为随系统运行发生浓度变化并最终可以达到动态平衡的反应物。因此,在固定外部葡萄糖浓度的前提下,只需固定磷酸根离子和二氧化碳的浓度为较高的数值 (视作充足供应),对其余包括能量和还原力在内的反应物的初始浓度赋值不再严苛要求。除葡萄糖外的各反应物的初始浓度只影响模型最初运行阶段的反应速率,并不会影响系统最终的稳定状态和控制关系,所以模型中的初始反应物浓度多沿用模型a和b中的数据。新增反应物的初始浓度参考细胞组成数据范围进行赋值[21-22],最终所有反应物初始浓度值设置详见表2。
1.3 模型调试及最终参数设置
由于两个模型出于不同的构建目的、创建于不同的反应体系,各自的通量水平存在很大的差异。添加新反应以及能量和还原力的计量关系后,模型初步运行后无法达到稳定状态。通过观察稳态运行的结果,发现无法得到稳定解的原因是中心代谢部分的通量远高于苏氨酸合成部分的通量,导致 asp不断积累。这种现象在整合不同模型的过程中非常普遍,也是模型整合要解决的首要问题。从pep形成oaa开始,至最后生成thr的反应过程为线性途径,且pep生成oaa的反应决定了糖酵解途径向苏氨酸合成子途径的通量分配,因此asp的积累意味着模型a向模型b的通量分配高于模型b所能承受的通量,需将合成asp的PPC反应速率降低,或者提高消耗asp的AK反应的速率。调整asp的相关反应速率后,asp的积累得到控制,但随之会带来其他代谢物的积累,但可采用类似思路进行调整以最终得到稳定解。需要说明的是由于代谢网络中存在的复杂调控关系及非线性动力学方程,这个过程常常需要不断的试差才能得到一个可以稳定运行的模型。目前阶段还没有一套标准化的系统的调试方法,但可根据经验把主要的调试方法分为3种情况:1) 对于存在于线性途径中的反应物,可以先将途径外的其他反应的速率设置为零,然后根据途径中反应物的积累情况,增加其消耗反应的最大反应速率rmax或者降低其作为生成物的最大反应速率即可。2) 针对类似 ATP(ADP和 AMP)、NAD(NADH)以及NADP(NADPH)等这样在很多反应中均涉及的、但存在固定转化关系的反应物组,可以先将其他组反应物的浓度固定,如固定 ATP(ADP和AMP)和NAD (NADH)两组代谢物的浓度,单独调整NADP和NADPH之间的平衡,就很容易发现导致崩溃的原因。同理再逐步将其他两组反应物改为可变的,一一调整即可。3)通过COPASI[23–24]软件自带的参数扫描功能对各反应rmax进行鲁棒性分析,着重调整鲁棒性较差的反应的rmax。经过上述过程的反复调试,我们最终使模型得到稳态解,此时模型中的部分参数设置如表3所示。
2 模型模拟分析
2.1 稳态通量分布
我们利用COPASI软件对上述构建好的模型进行稳态运算,存在赖氨酸分支时,得到 PTS反应的通量为 0.268 mmol/(L·s),苏氨酸合成的通量为 0.155 mmol/(L·s),六碳的葡萄糖转化为四碳的苏氨酸的碳摩尔得率为 38.56%,敲除赖氨酸分支,苏氨酸得率提高为91.17%,接近FBA计算的途径最大得率,分支途径的敲除对苏氨酸得率的提高非常重要[3]。途径中各反应的通量分布和主要调控关系如图1所示。
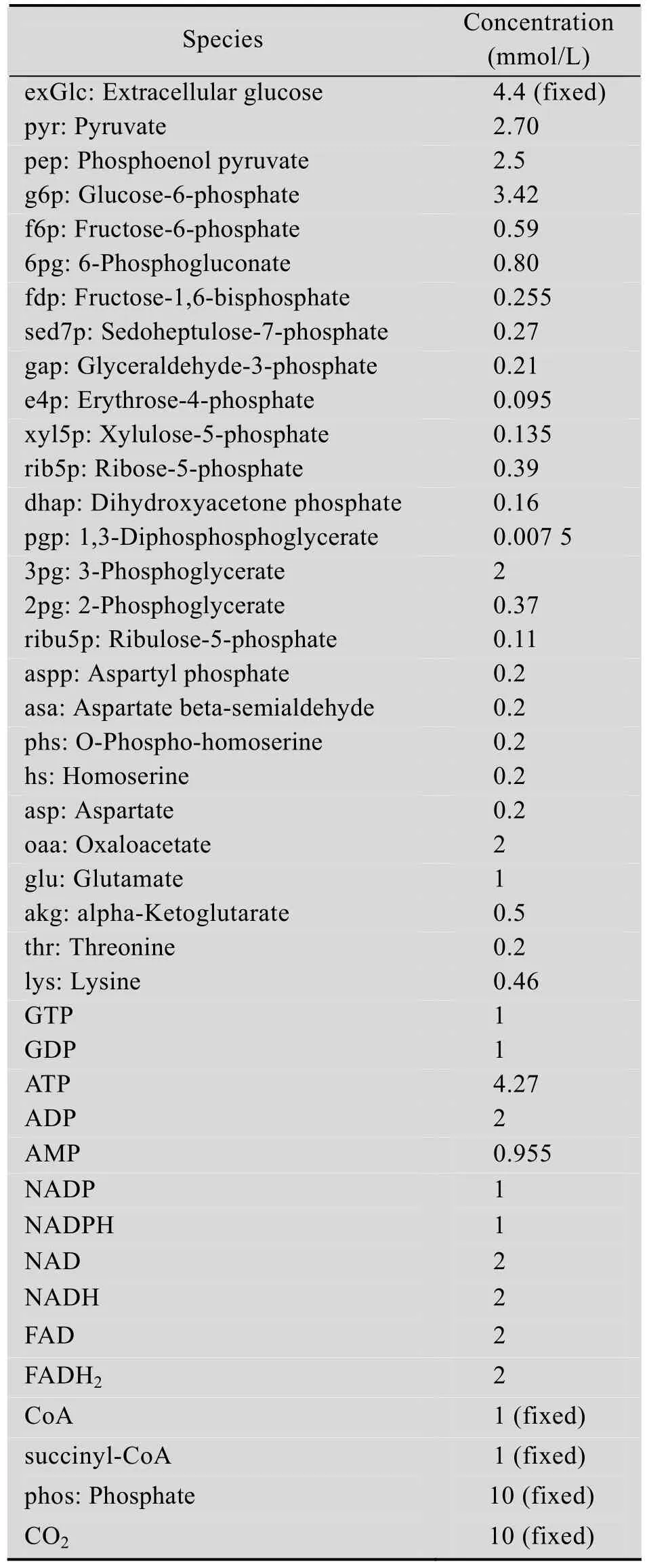
表2 模型中设定的代谢物的初始浓度Table 2 Initial concentrations of metabolites given in the integrated model
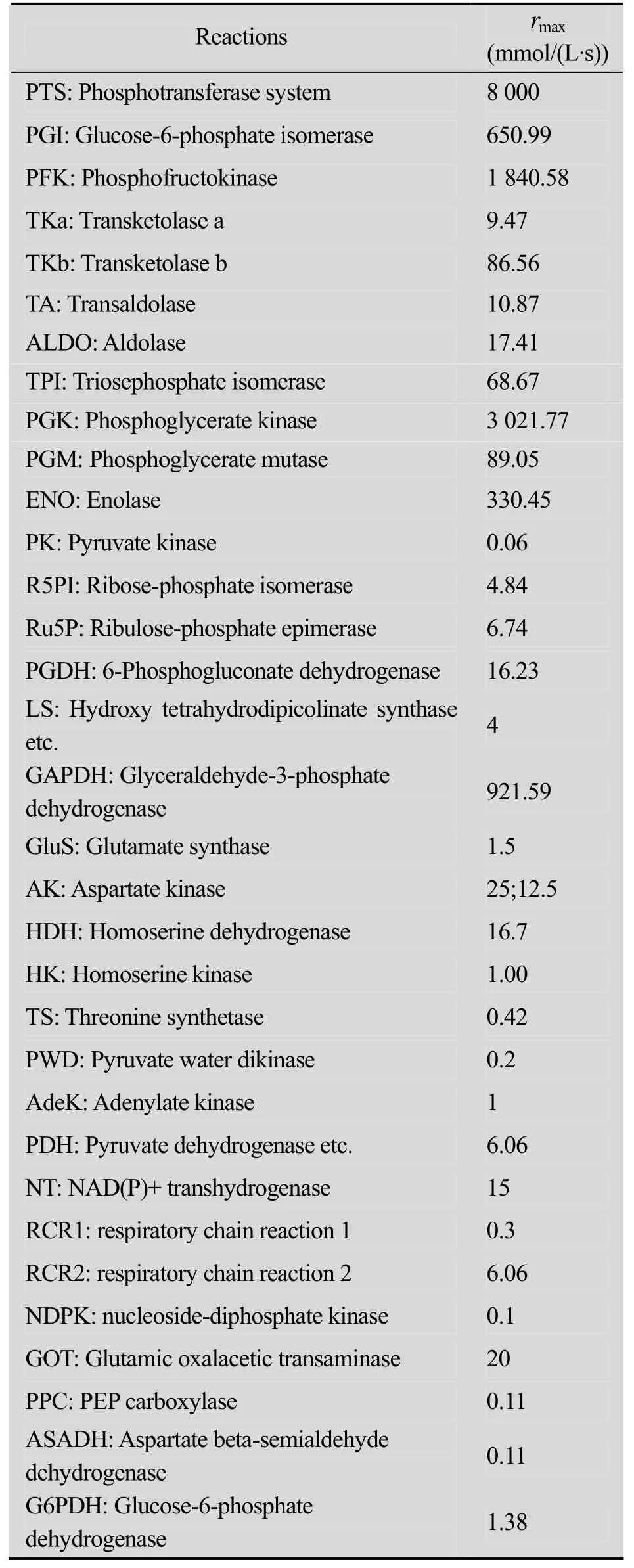
表3 模型中最终设定的rmax值Table 3 Final rmax values in the model
2.2 代谢控制分析
通量控制系数 (Flux control coefficient,FCC)表明了代谢途径中的酶活性改变对途径稳态通量的影响,定义式见方程 (1)[25]。通过比较途径中各酶反应过程对某一途径通量的控制系数的大小,可以确定对代谢途径 (此模型中指对苏氨酸合成通量)起关键控制作用的酶,进而通过调节关键酶的浓度及活性来改变途径中的通量分布[26]。对于FCC为正数的酶反应,表明增加其酶量或酶活,可以提高目标反应的通量;对于FCC为负数的酶反应,增加其酶量或酶活,则对提高目标反应的通量不利。数值越大表明该酶对通量影响越大, 是理想的改造位点。
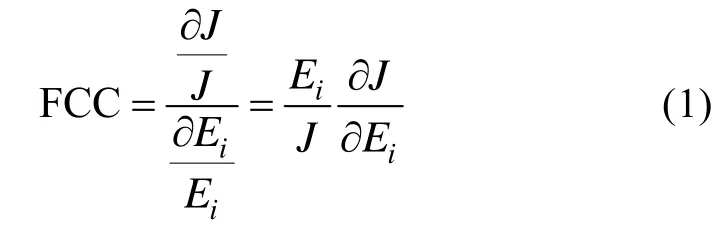
2.2.1 关键酶预测
通过代谢控制分析,我们获得了模型稳态状态下酶与反应间的FCC矩阵。选择FCC矩阵中各酶对苏氨酸合成通量和赖氨酸合成通量的FCC值,分布如图2所示。
由图2可见FCC数值较大的反应均为途径中的不可逆反应或者位于分支点,主要集中在糖酵解过程以及能量和还原力的生成过程,这说明前体、能量和还原力的供给是苏氨酸合成的主要限制因素。图2A表明对苏氨酸合成,控制步骤主要为ASADH和HDH,其中HDH增加对苏氨酸的合成有利而对赖氨酸合成不利,ASADH则正好相反;同时,G6PDH和GAPDH两个脱氢酶对苏氨酸和赖氨酸两个竞争分支的控制作用也是相反的,表明过表达这些酶可以为苏氨酸合成提供更多还原力,使更多碳流从赖氨酸合成转向苏氨酸合成。PPC对苏氨酸合成反应的控制系数为负值,表明 PPC增加对苏氨酸的合成并不总是有利的,这主要是由于反应的复杂非线性特征及产物合成与能量和还原力供给之间的复杂协调机制造成的。这一模拟分析结果与Lee等的实验研究结果一致[3]。
以往的研究结果已充分证实了苏氨酸对其合成途径中的天冬氨酸激酶Ⅰ、高丝氨酸脱氢酶Ⅰ和苏氨酸合成酶的反馈抑制作用[5-6],因此我们在模型中考察了解除苏氨酸抑制对其合成通量和得率的影响,结果见图 2B。结果表明,通过解除苏氨酸的抑制可以有效降低ASADH和HDH对苏氨酸合成的限制作用,更有利于碳流流向苏氨酸的合成。在模型中将苏氨酸抑制解除后,PTS/TS/LS的通量分布由图 1中的 0.268/0.155/0.134变为0.248/0.296/0.027,糖转运系统PTS通量没有明显变化,但是苏氨酸和赖氨酸两个分支的合成通量比值发生明显改变,由原来的 1.16∶1增加至 10.96∶1,苏氨酸相对于葡萄糖的碳摩尔得率从38.56%增加至79.57%,这充分说明了解除苏氨酸抑制对提高苏氨酸通量和得率的必要性。
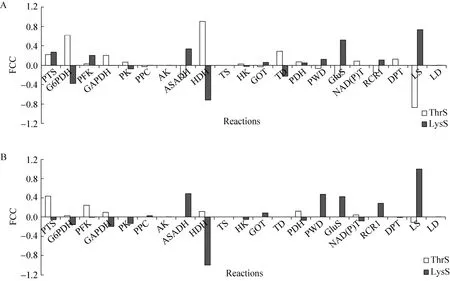
图2 不同酶对苏氨酸和赖氨酸合成反应的通量控制系数 (a: 存在苏氨酸抑制;b: 解除苏氨酸抑制)Fig. 2 Flux control coefficients of enzymes on the threonine and lysine biosynthesis. (A)FCCs of different enzymes at unrelieved threonine inhibition. (B)completely relieve the threonine inhibition.
此外,敲除赖氨酸分支途径,重新计算后,PK对 TS的 FCC为负值,这一结果与Rodríguez-Prados等不考虑赖氨酸分支途径时提出的PK敲除策略并不矛盾[7]。添加了赖氨酸分支途径,考虑了能量和还原力的平衡关系后,在我们的控制步骤预测结果中,PK并不是苏氨酸合成过程的关键酶,敲除PK后对TS的通量影响并不明显。对此我们还需要在后续工作中对模型进一步验证和完善。
2.2.2 关键酶过表达分析
对考虑苏氨酸抑制和赖氨酸分支的模型初始状态进行的控制分析结果表明,PTS、G6PDH和 HDH对苏氨酸合成反应的通量具有较大影响,即改变其酶量可以显著提高苏氨酸合成速率。在我们的动力学模型中酶量体现在参数rmax中,因此我们通过提高PTS、G6PDH和HDH三个反应的 rmax来考察其对提高苏氨酸合成通量的影响,结果如图3所示。从图3A可以看出,随着 PTS反应速率的提高,代表糖转运系统的PTS反应、苏氨酸合成的TS反应和赖氨酸合成的LS反应的通量均增加,但最大反应速率增加至1.5倍后,PTS和TS的合成通量开始下降,只有赖氨酸合成继续增加。图3B中,当G6PDH的最大反应速率增加时,PTS的通量已经趋于平衡,苏氨酸合成通量在上升,赖氨酸合成通量逐渐下降。图3C中,增加HDH的最大反应速率,PTS的通量没有发生明显变化,苏氨酸和赖氨酸的通量分布发生明显改变,通过在模型中解除苏氨酸对HDH的抑制,也可以使苏氨酸和赖氨酸的合成通量比值增加至10.54∶1,说明HDH对苏氨酸合成的限制非常强烈。通量控制系数是系统中的局部概念,通过对上面3个模型初始状态预测的关键酶进行过表达,在一定范围内确实可以增加目标通量,但继续增加效果却适得其反,因此在菌种改造过程中关键酶的过表达并非越多越好,而是有一个最佳范围。我们的模型构建和模拟分析工作都是在专用的生化反应动力学模型工具软件COPASI中进行的[23-24]。
3 结论
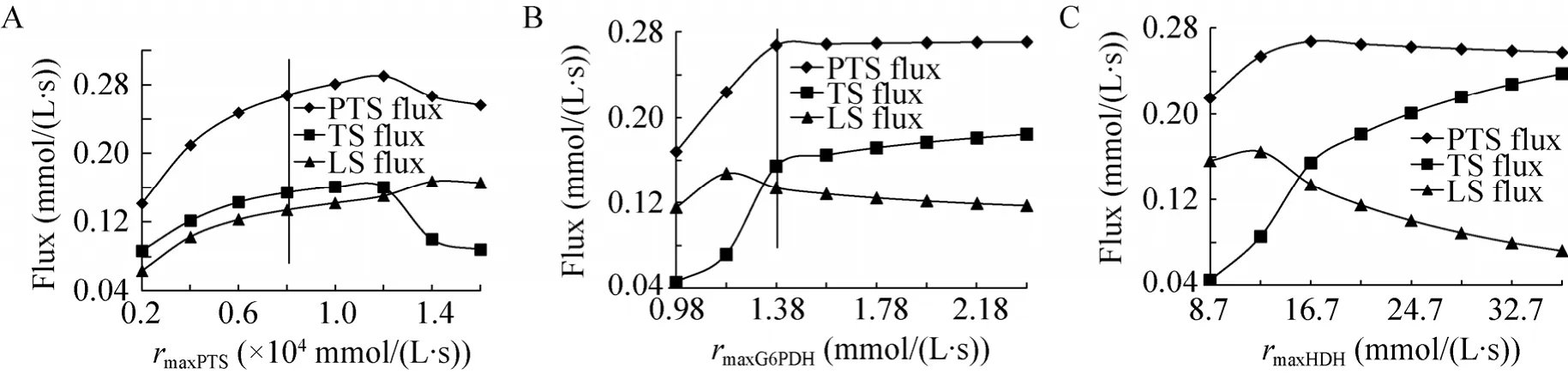
图3 稳态时糖输入通量 (PTS)、苏氨酸合成通量 (TS)和赖氨酸合成通量 (LS)随关键酶过表达的变化Fig. 3 Effect of enzyme amounts on the PTS, threonine synthesis and lysine synthesis fluxes. (A)PTS over expression.(B)G6PDH over expression. (C)HDH over expression.
我们在基因组规模代谢网络分析求得的苏氨酸合成最优途径基础上,整合了已有的中心代谢动力学模型和从天冬氨酸出发的苏氨酸合成动力学模型,借助文献信息对一些新反应补充了动力学方程和参数信息,并考虑了整个途径的能量和还原力平衡关系、从而构建了自洽的从葡萄糖到苏氨酸的完整代谢途径动力学模型。对新的整合模型的代谢控制分析表明控制系数较大的反应均为不可逆反应或者处于途径的分支点,包括前体、能量和还原力的生成和转化过程,这表明苏氨酸合成过程需要前体、能量和还原力的协调分配。分析结果表明解除苏氨酸抑制确实可以显著改变苏氨酸和赖氨酸两个分支间的通量分配,增加苏氨酸的得率。对关键酶如PTS、G6PDH和HDH的分析表明在一定范围内提高其速率确实可以有效提高苏氨酸合成途径的通量,但关键酶的过表达应在一适当范围内,因为随着酶量的增加该酶的控制系数也会减小甚至变为负值,继续过表达反而对产物合成不利。
[1]Wang H, Ma HW, Zhao XM. Progress in genome-scale metabolic network: a review. Chin J Biotech, 2010, 26(10): 1340–1348 (in Chinese).王晖, 马红武, 赵学明. 基因组尺度代谢网络研究进展. 生物工程学报, 2010, 26(10): 1340–1348.
[2]Krömer JO, Wittmann C, Schröder H, et al.Metabolic pathway analysis for rational design of L-methionine production by Escherichia coli and Corynebacterium glutamicum. Metab Engin, 2006,8(4): 353–369.
[3]Lee KH, Park JH, Kim TY, et al. Systems metabolic engineering of Escherichia coli for L-threonine production. Mol Syst Biol, 2007, 3: 149–156.
[4]Liu S, Liang Y, Liu Q, et al. Development of a two-stage feeding strategy based on the kind and level of feeding nutrients for improving fed-batch production of L-threonine by Escherichia coli. Appl Microbiol Biotechnol, 2013, 97(2): 573–583.
[5]Chassagnole C, Raïs B, Quentin E, et al. An integrated study of threonine-pathway enzyme kinetics in Escherichia coli. Biochem J, 2001, 356:415–423.
[6]Chassagnole C, Fell DA, Raïs B, et al. Control of the threonine-synthesis pathway in Escherichia coli:a theoretical and experimental approach. Biochem J,2001, 356: 433–444.
[7]Rodríguez-Prados JC, De Atauri P, Maury J, et al. In silico strategy to rationally engineer metabolite production: a case study for threonine in Escherichia coli. Biotechnol Bioeng, 2009, 103(3): 609–620.
[8]Orth JD, Conrad TM, Na J, et al. A comprehensive genome-scale reconstruction of Escherichia coli metabolism——2011. Mol Syst Biol, 2011, 7: 535.
[9]Chassagnole C, Noisommit-Rizzi N, Schmid JW, et al. Dynamic modeling of the central carbon metabolism of Escherichia coli. Biotechnol Bioeng,2002, 79(1): 53–73.
[10]Le Novère N, Bornstein B, Broicher A, et al.BioModels Database: a free, centralized database of curated, published, quantitative kinetic models of biochemical and cellular systems. Oxford J, 2005,34(suppl 1): 689–691.
[11]Barden RE, Fung CH, Utter MF, et al. Pyruvate carboxylase from chicken liver: steady state kinetic studies indicate a “Two Site” ping-pong mechanism. J Biol Chem, 1972, 247: 1323–1333.
[12]Sung HAC, Tachiki T, Kumagai H, et al. Properties of glutamate synthase from Brevibachterium flavum. J Ferment Technol, 1984, 62: 569–575.
[13]Vanoni MA, Nuzzi L, Rescigno M, et al. The kinetic mechanism of the reactions catalyzed by the glutamate synthase from Azospirillum brasilense.Eur J Biochem, 1991, 202(1): 181–189.
[14]Contador CA, Rizk ML, Asenjo JA, et al. Ensemble modeling for strain development of L-lysine producing Escherichia coli. Metab Eng, 2009, 11:221–233.
[15]Tjaden B, Plagens A, Dörr C, et al.Phosphoenolpyruvate synthetase and pyruvate,phosphate dikinase of Thermoproteus tenax: key pieces in the puzzle of archaeal carbohydrate metabolism. Mol Microbiol, 2006, 60(2): 287–298.
[16]Narindrasorasak S, Bridger WA.Phosphoenolypyruvate synthetase of Escherichia coli: molecular weight, subunit composition, and identification of phosphohistidine in phosphoenzyme intermediate. J Biol Chem, 1977,252(10): 3121–3127.
[17]Freeman H, Shimomura K, Horner E, et al.Nicotinamide nucleotide transhydrogenase: a key role in insulin secretion. Cell Metab, 2006, 3(1):35–45.
[18]Velázquez I, Pardo JP. Kinetic characterization of the rotenone-insensitive internal NADH: ubiquinone oxidoreductase of mitochondria from Saccharomyces cerevisiae. Arch Biochem Biophys,2001, 389(1): 7–14.
[19]Morin-Leisk J, Lee TH. Nucleotide-dependent self-assembly of Nucleoside Diphosphate Kinase(NDPK)in vitro. BBA Proteins Proteom, 2008,1784(12): 2045–2051.
[20]Oberhardt MA, Puchałka J, Martins dos Santos VAP, et al. Reconciliation of genome-scale metabolic reconstructions for comparative systems analysis. PLoS Comput Biol, 2011, 7(3): e1001116.
[21]Pramanik J, Keasling JD. Effect of Escherichia coli biomass composition on central metabolic fluxes predicted by a stoichiometric model.Biotechnol Bioeng, 1998, 60(2): 230–238.
[22]Pramanik J, Keasling JD. Stoichiometric model of Escherichia coli metabolism: incorporation of growth-rate dependent biomass composition and mechanistic energy requirements. Biotechnol Bioeng, 1997, 56(4): 398–421.
[23]Hoops S, Sahle S, Gauges R, et al. COPASI–a complex pathway simulator. Bioinformatics, 2006,22(24): 3067–3074.
[24]Mendes P, Hoops S, Sahle S, et al. Computational modeling of biochemical networks using COPASI.Syst Biology, 2009, 500: 17–59.
[25]Pryor HJ, Smyth JE, Quinlan PT, et al. Evidence that the flux control coefficient of the respiratory chain is high during gluconeogenesis from lactate in hepatocytes from starved rats. Biochem J, 1987,247: 449–457.
[26]Nikolaev EV. The elucidation of metabolic pathways and their improvements using stable optimization of large-scale kinetic models of cellular systems. Metab Eng, 2010, 12: 26–38.

