利用CRISPR/Cas9技术构建定点突变小鼠品系
白敏,李崎,邵艳姣,黄元华,李大力,马燕琳1. 贵阳中医学院研究生院,贵阳 55000;. 海南医学院附属医院,海南省人类生殖与遗传重点实验室,海口 57010;. 华东师范大学生命科学学院,上海 0041
利用CRISPR/Cas9技术构建定点突变小鼠品系
白敏1,2,李崎2,邵艳姣3,黄元华2,李大力3,马燕琳2
1. 贵阳中医学院研究生院,贵阳 550002;2. 海南医学院附属医院,海南省人类生殖与遗传重点实验室,海口 570102;3. 华东师范大学生命科学学院,上海 200241
CRISPR/Cas9技术是新近发展起来的对细胞和动物模型进行基因编辑的重要方法。本文利用DNA双链断裂(Double-strand breaks, DSBs)引起的同源重组(Homologous recombination, HR)依赖与非依赖的修复机制,建立基于 CRISPR/Cas9核酸酶技术构建定点突变小鼠品系的技术体系。针对赖氨酸特异脱甲基化酶 2b(Lysine (K)-specific demethylase 2b, Kdm2b)酶活关键位点对应的基因组DNA序列设计单一导向RNA(Single-guide RNA, sgRNA),通过与Cas9 mRNA共显微注射,分别得到Kdm2b基因发生移码突变的基因失活品系及关键位点氨基酸缺失的酶活突变型小鼠品系。此外,利用HR介导的修复机理,将黄素单加氧酶 3(Flavin containing monooxygenases3, Fmo3)基因的sgRNA序列及对应的点突变单链寡脱氧核苷(Single strand oligonucleotides, ssODN)修复模板共注射到小鼠受精卵雄原核。对F0小鼠基因测序分析显示,成功构建了Fmo3基因移码突变的基因敲除和单碱基定点突变的基因敲入小鼠,这些突变能够稳定遗传给子代。本研究利用CRISPR/Cas9技术,通过同源重组依赖与非依赖两种DNA损伤修复方式,成功构建了特定位点突变的小鼠品系。
基因敲除;基因敲入;酶活突变;同源重组
基因修饰动物模型是在活体动物上开展基因功能研究、寻找合适药物作用靶标的重要工具[1]。小鼠因遗传操作手段丰富,特别是成熟的胚胎干细胞(Embryonic stem cell, ESC)打靶技术的广泛应用[2,3],使其成为研究基因功能和人类疾病发生发展机理最理想的模式动物。然而基于小鼠ESC同源重组的基因打靶技术操作复杂、耗时长、人力和经济成本高,在一定程度上影响了该技术更广泛的应用。近年来基因编辑技术的出现,使基因修饰模型的构建摆脱了ESC的限制,为基因改造提供了方便、快捷、有效的技术体系[4,5]。在现有的基因编辑技术中,一种来源于细菌的成簇的规律间隔的短回文重复序列(Clustered regularly interspaced short palindromic repeat, CRISPR)及临近相关基因(CRISPR-associated, Cas)系统经过改造后,因其操作简便、效率高而成为最热门的基因编辑技术,即CRISPR/Cas9技术[6~14]。与锌指核酶(Zinc finger nucleases, ZFN)和转录激活因子样效应物核酸酶(Transcription activator-like effector nuclease, TALEN)技术相比,CRISPR/Cas9系统具有操作更为简便、靶点选择灵活性强、活性更高等优势。目前,使用最为广泛的是来自细菌 S. pyogenes中的 Cas9基因及经过改造的单向导 RNA (Single-guide RNA, sgRNA)体系。Cas9是一种特殊的核酸内切酶,能与sgRNA结合,sgRNA 3'端20个碱基通过碱基配对原则与目标DNA配对结合,促使Cas9核酸酶对目标DNA进行切割产生双链断裂,从而对基因进行定点编辑[8]。对于靶点选择的唯一要求是 sgRNA所识别配对序列紧紧相邻的目标DNA序列必需是NGG(N代表任一碱基),该序列一般被称为前导间隔相邻基序(Protospacer adjacent motif, PAM)[15~17]。Cas9切割后产生的DNA双链断裂(Double-strand breaks, DSBs)在没有同源重组模板存在的情况下会利用非同源末端连接(Non-homologous end joining, NHEJ)的方式进行修复。由于NHEJ没有修复模板,非常容易出错,造成碱基的确失、插入或者替换,从而使目标DNA发生突变。如果缺失或者插入的碱基数目为 3的非整倍数,则会改变基因的编码框而导致基因的失活;如果缺失为 3的倍数则造成部分氨基酸的缺失。在有同源重组模板的情况下,DSBs则可能通过高保真的同源重组(Homologous recombination, HR)修复方式,将模板DNA序列精确插入到目标DNA序列中。NHEJ的发生率非常高,而HR的修复方式发生概率较低。Cas9在小鼠和大鼠胚胎中具有很高的活性,已有文献证明利用这两种方法都能对特定基因进行编辑[18~20]。但如何快速构建特定位点突变的酶活缺失型小鼠品系却鲜有报道。
Kdm2b是包含JmjC结构域的组蛋白脱甲基化酶,其酶活性需要二价铁离子(Fe2+)和α-酮戊二酸为辅助因子,通过羟基化作用使组蛋白赖氨酸脱甲基[21,22]。细胞实验证明该类脱甲基化酶JmjC结构域中3个氨基酸为铁离子结合所必需,这些氨基酸的突变或者缺失导致其失去酶活性。鱼腥味综合征(Fish odor syndrome)是一种人类遗传疾病,主要由FMO3基因突变使代谢三甲胺的酶功能丧失或者降低导致三甲胺不能排泄出体外,从而体液与气息含有鱼腥味[23]。人类 FMO3基因第 8外显子编码 470位氨基酸的CAG密码子出现无义突变成为TAG而导致Fmo3功能丧失是鱼腥味综合征的常见突变[23]。本研究使用CRISPR/Cas9技术,利用NHEJ和HR两种DNA损伤修复方式,成功地构建了Kdm2b基因敲除、酶活突变的小鼠品系以及模拟人类FMO3基因突变的小鼠品系,为动物模型构建提供了新的思路。
1 材料和方法
1.1 材料
实验所用的小鼠均为C57BL/6J品系,来源于上海斯莱克实验动物有限公司。小鼠饲养于华东师范大学SPF级实验动物中心,恒温恒湿,12 h光照,12 h黑暗,自由采食。动物实验符合华东师范大学动物伦理委员会的规定并被授权。
1.2 方法
1.2.1 RNA体外转录
含有SP6启动子的Cas9表达质粒经NotⅠ限制性内切酶酶切线性化,经酚/氯仿抽提纯化后,溶于无RNase A 的水中,经mMESSAGE mMACHINE®SP6(Life Technologies公司)体外转录试剂盒进行体外转录,通过氯化锂沉淀,纯化得到Cas9 mRNA。利用本课题组已发表的方法[24]构建 sgRNA(靶点序列:Kdm2b,5'-GAACTTCCACATTGACTTTGG-3';Fmo3,5'-GGGCCTACCAGCCGGAAC-3')模板,通过酚/氯仿抽提和乙醇沉淀进行纯化,使用 T7体外转录试剂盒(TaKaRa公司)进行体外转录,并通过酚/氯仿抽提和异丙醇沉淀得到sgRNA。Cas9 mRNA及sgRNA沉淀均用20 µL TE缓冲液重悬。通过琼脂糖凝胶电泳检测质量后测浓度,分装储存于-80℃。具体实验步骤参考文献[24]。
1.2.2 小鼠受精卵显微注射
将体外转录所得的靶向Kdm2b基因的sgRNA与Cas9 mRNA混合,用TE缓冲液稀释,使sgRNA与Cas9 mRNA终浓度分别为12.5 ng/μL和25 ng/μL。使用显微注射仪将其注射到C57 BL/6J小鼠单细胞受精卵的胞浆中。Fmo3点突变小鼠构建与之类似,在注射液中加入10 ng/μL ssODN(5'-AGCCCCTACTAGTTCCGGCTGGTAGGCCCAGGAAAGTGGTCAGGAGCCCGGAACGCCATCCTA-3')作为修复模板,与Cas9/sgRNA共注射于C57 BL/6J小鼠受精卵的原核中。胚胎体外培养 1~2 h后移植入假孕母鼠的输卵管。代孕鼠伤口缝合后饲养待产。
1.2.3 小鼠基因型鉴定
小鼠出生1周后,剪取脚趾或尾尖,置于1.5 mL EP管内。加入500 μL裂解液和1 μL的蛋白酶K贮存液。将上述EP管置于55℃恒温水浴锅水浴过夜,或待脚趾消化完全,用酚/氯仿法抽提基因组DNA。以小鼠基因组DNA为模板,通过PCR扩增sgRNA靶向位点两侧各约200 bp的序列。引物序列如下:
Kdm2b:KF,5'-GTGCTGTCTGTGAGCGTCTG- 3',KR, 5'-ACAACCACCCATAATGAGAT-3';Fmo3:FF, 5'-AGGGCAATGTAGTAAAGCAG-3',FR, 5'-GAGCAAACGGGAATAGAAGT-3'。
PCR产物经纯化、变性、缓慢退火,加入 T7核酸内切酶I (T7 endonuclease I, T7EⅠ)(New England Biolabs),进行琼脂糖凝胶电泳。将T7EⅠ酶切检测阳性小鼠的PCR产物分别连入pMD18-T载体,挑取若干克隆提取质粒并进行测序。
2 结果与分析
2.1 Kdm2b基因敲除及酶活突变小鼠的构建
根据NHEJ修复原理,本文针对组蛋白脱甲基化酶Kdm2b基因的铁离子结合位点区域设计sgRNA靶点(图1A)。将经显微注射的106个胚胎分别移植到5只假孕小鼠中,共有24只F0代小鼠出生。利用引物KF和KR,以小鼠基因组DNA为模板进行PCR扩增 (图1B),同时通过T7EⅠ酶切检测产生基因突变的小鼠(图1C)。从图1中可以看出,24只小鼠中有12只除了野生型条带外还出现了一条新的条带(箭头所示),表明突变效率为 50%。根据 T7EⅠ检测结果,选取阳性小鼠(1#、11#、21#、22#)的PCR产物连接T载体并测序,其中1#小鼠在铁离子结合位点出现缺失12 bp和17 bp两种等位基因突变(图1D)。缺失17 bp将导致Kdm2b基因后续蛋白质产生移码突变导致基因失活。缺失12 bp的突变使铁离子结合位点缺失4个氨基酸(分别是H、I、D、F),虽然没有造成移码但突变的Kdm2b蛋白失去脱甲基化酶活性。21#和22# F0代小鼠虽然也包含12 bp的碱基缺失,但考虑到在铁离子结合位点只缺失了 1个氨基酸,推测可能对酶活造成的影响不大,因此在实验中选取1#小鼠进行传代。将1#小鼠与野生型小鼠进行交配,得到 F1代小鼠后对其进行 PCR扩增和测序(图1E)。其中7#和8#小鼠为酶活突变小鼠,其余7只小鼠为缺失17 bp的基因敲除小鼠(图1E)。以上研究表明,利用CRISPR/Cas9系统通过一次注射成功地构建了Kdm2b基因酶活突变和基因敲除的两种基因修饰小鼠品系。
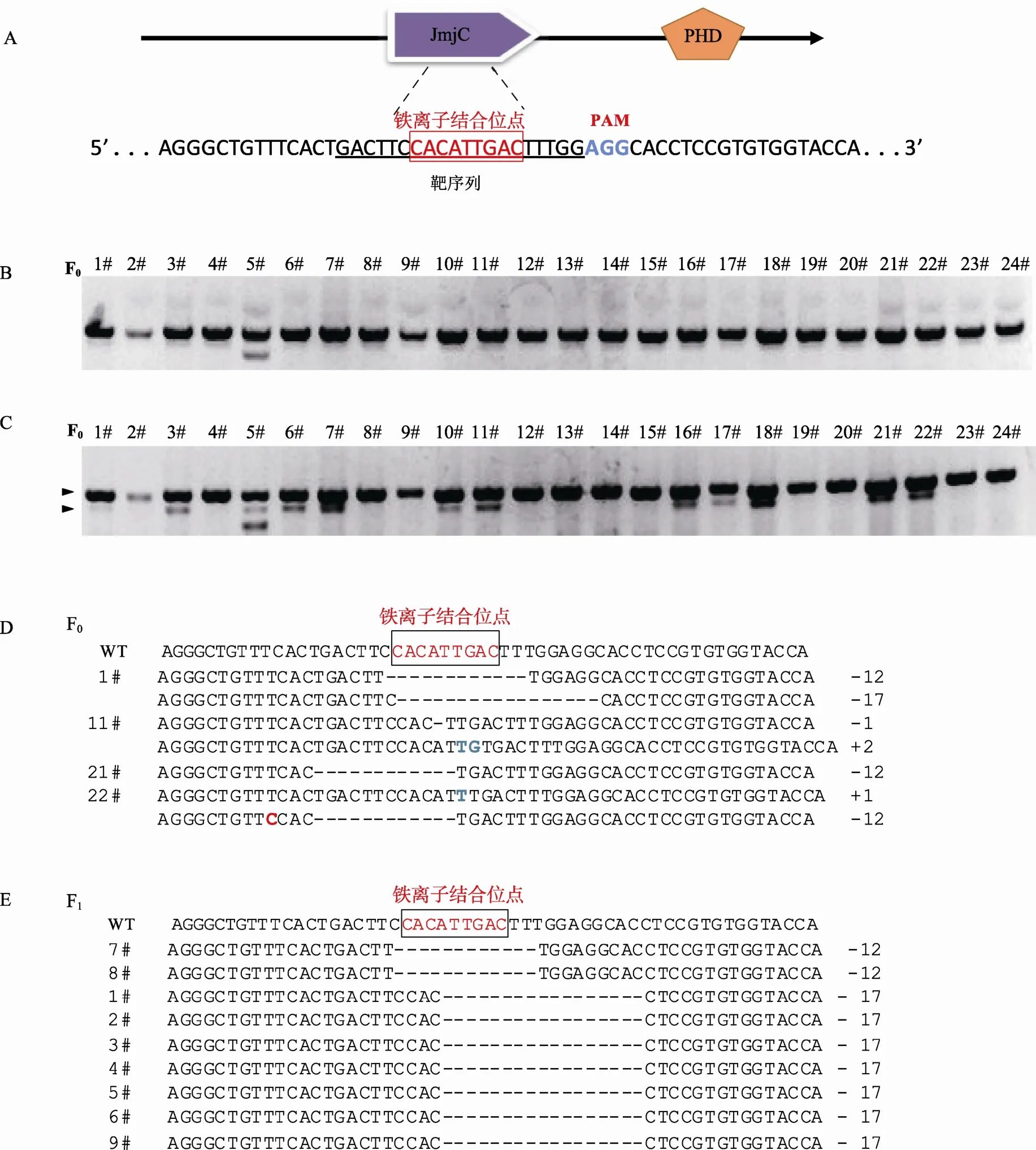
图1 Kdm2B基因突变小鼠构建
2.2 Fmo3基因定点敲入小鼠品系的构建
为构建鱼腥味综合征小鼠模型,本文将靶向小鼠Fmo3基因第8外显子的sgRNA及99 bp的ssODN作为同源修复的模板序列在对应位点引入C>T的突变,使密码子CAG突变为终止密码子TAG(图2A)。显微注射后存活的 65个胚胎移植到 3只假孕小鼠中,共有9只F0代小鼠出生。对应位点的基因组片段经FF、FR引物PCR扩增后测序,结果显示9只小鼠中有 6只发生了突变,突变效率为 66.7%(图 2B)。其中 2#小鼠虽然整合了特定突变,但是在靶点附近出现了6 bp的缺失(图2B),因此不是所需定点敲入小鼠。3#小鼠特定位点的C被置换为T而出现了预期的新的TAG终止密码子(图2:B、C),该突变将造成 Fmo3基因的转录提前终止,能较好地模拟人类鱼腥味综合征的一个突变类型。其余 4只小鼠产生了 NHEJ介导的基因敲除但没有发生特定位点的同源重组。将3#小鼠与野生型小鼠交配,得到F1代小鼠,通过测序发现C>T的点突变能够高效地传递到子代,意味着 Fmo3点突变小鼠建系成功(图 2D)。本研究利用单链寡核苷酸可介导基因发生精确的同源重组,成功构建 Fmo3基因定点敲入小鼠品系。
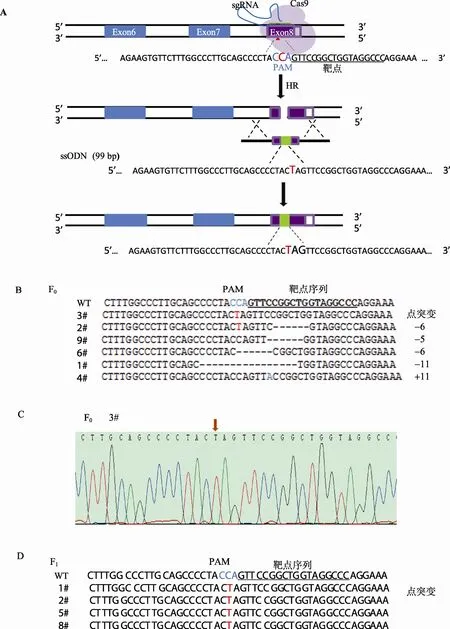
图2 Fmo3基因点突变(CAG>TAG, Q470X)小鼠构建
3 讨 论
基因敲除技术作为一种重要基因工程手段在研究基因功能方面发挥着重要作用。19世纪80年代,Thompson等[23]首次建立了基于ES细胞自发同源重组的基因打靶技术,成功地构建了基因敲除小鼠模型。基于ES细胞基因打靶技术成为一种强有力的哺乳动物精确遗传修饰技术,极大地推动了基础研究的发展[25]。然而,目前,还有许多哺乳动物的 ESC培养体系没有建立,极大地限制了该技术的广泛应用。
近年兴起的以ZFNs、TALENs和CRISPR/Cas[26~31]技术为代表的基因编辑技术在动植物基因改造方面展现出了巨大的潜力,得到了广泛的应用。由于基因编辑技术几乎在任何细胞中都有活性,摆脱了细胞类型的限制,使其应用更广、更灵活。与ES细胞打靶技术不同的是,基因编辑技术可利用 NHEJ和HDR两种修复方式对目的基因组DNA进行操作。NHEJ方式具有随机性,所以可以看到F0代阳性小鼠的基因型多种多样,具有一定的不可控性(图1D、图 2B)。本研究利用这种不可控性实现了一次注射同时得到Kdm2b移码突变的基因敲除小鼠和甲基转移酶酶活突变的小鼠,对于在动物体内研究该基因功能,特别是酶活性功能具有重要意义。虽然 NHEJ修复方式有一定的概率得到 3的整倍数碱基缺失的个体,但毕竟不是精确突变,具有很大的局限性。因此本研究利用ssODN作为修复模板进行了精确突变的尝试,并成功获得了模拟人类突变的 Fmo3基因突变小鼠。虽然 ssODN作为修复模板相对双链DNA模板具有更高的修复效率[31],但是本文也发现ssODN作为模板修复的精确度不能达到 100%。例如,Fmo3点突变F0代2#小鼠,虽然引入了所需的C>T突变,但同时也出现了意料之外的6个碱基的缺失(图2B)。总之,ssODN介导的正确修复效率还不到 10%。如何发展 CRISPR/Cas技术实现高效、精确的基因组编辑是今后相关研究的重要方向。
[1] 傅继梁, 王铸钢. 基因工程小鼠. 上海: 上海科学技术出版社, 2006: 5-18.
[2] Thomas KR, Capecchi MR. Site-directed mutagenesis by gene targeting in mouse embryo-derived stem cells. Cell, 1987, 51(3): 503-512.
[3] Doetschman T, Gregg RG, Maeda N, Hooper ML, Melton DW, Thompson S, Smithies O. Targetted correction of a mutant HPRT gene in mouse embryonic stem cells. Nature, 1987, 330(6148): 576-578.
[4] Mali P, Yang LH, Esvelt KM, Aach J, Guell M, DiCarlo JE, Norville JE, Church GM. RNA-guided human genome engineering via Cas9. Science, 2013, 339(6121): 823-826.
[5] Jinek M, Chylinski K, Fonfara I, Hauer M, Doudna JA, Charpentier E. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science, 2012, 337(6096): 816-821.
[6] Auer TO, Duroure K, De Cian A, Concordet JP, Del Bene F. Highly efficient CRISPR/Cas9-mediated knock-in in zebrafish by homology-independent DNA repair. Genome Res, 2014, 24(1): 142-153.
[7] Burgess DJ. Technology: a CRISPR genome-editing tool. Nat Rev Genet, 2013, 14(2): 80-81.
[8] Cong L, Ran FA, Cox D, Lin SL, Barretto R, Habib N, Hsu PD, Wu XB, Jiang WY, Marraffini LA, Zhang F. Multiplex genome engineering using CRISPR/Cas systems. Science, 2013, 339(6121): 819-823.
[9] Li W, Teng F, Li TD, Zhou Q. Simultaneous generation and germline transmission of multiple gene mutations in rat using CRISPR-Cas systems. Nat Biotechnol, 2013, 31(8): 684-686.
[10] Hou ZG, Zhang Y, Propson NE, Howden SE, Chu LF, Sontheimer EJ, Thomson JA. Efficient genome engineering in human pluripotent stem cells using Cas9 from Neisseria meningitidis. Proc Natl Acad Sci USA, 2013, 110(39): 15644-15649.
[11] Shen B, Zhang J, Wu HY, Wang JY, Ma K, Li Z, Zhang XG, Zhang PM, Huang XX. Generation of gene-modified mice via Cas9/RNA-mediated gene targeting. Cell Res, 2013, 23(5): 720-723.
[12] Ma YW, Zhang X, Shen B, Lu YD, Chen W, Ma J, Bai L, Huang XX, Zhang LF. Generating rats with conditional alleles using CRISPR/Cas9. Cell Res, 2014, 24(1): 122-125.
[13] Wang HY, Yang H, Shivalila CS, Dawlaty MM, Cheng AW, Zhang F, Jaenisch R. One-step generation of mice carrying mutations in multiple genes by CRISPR/Cas-mediated genome engineering. Cell, 2013, 153(4): 910-918.
[14] Taylor GK, Heiter DF, Pietrokovski S, Stoddard BL. Activity, specificity and structure of I-Bth0305I: a representative of a new homing endonuclease family. Nucleic Acids Res, 2011, 39(22): 9705-9719.
[15] Sapranauskas R, Gasiunas G, Fremaux C, Barrangou R, Horvath P, Siksnys V. The Streptococcus thermophilus CRISPR/Cas system provides immunity in Escherichia coli. Nucleic Acids Res, 2011, 39(21): 9275-9282.
[16] Makarova KS, Aravind L, Wolf YI, Koonin EV. Unification of Cas protein families and a simple scenario for the origin and evolution of CRISPR-Cas systems. Biol Direct, 2011, 6: 38.
[17] Platt RJ, Chen SD, Zhou Y, Yim MJ, Swiech L, Kempton HR, Dahlman JE, Parnas O, Eisenhaure TM, Jovanovic M, Graham DB, Jhunjhunwala S, Heidenreich M, Xavier RJ, Langer R, Anderson DG, Hacohen N, Regev A, Feng GP, Sharp PA, Zhang F. CRISPR-Cas9 knockin mice for genome editing and cancer modeling. Cell, 2014, 159(2): 440-455.
[18] Shen B, Zhang WS, Zhang J, Zhou JK, Wang JY, Chen L, Wang L, Hodgkins A, Iyer V, Huang XX, Skarnes WC. Efficient genome modification by CRISPR-Cas9 nickase with minimal off-target effects. Nat Methods, 2014, 11(4): 399-402. [19] Yang H, Wang HY, Shivalila CS, Cheng AW, Shi LY, Jaenisch R. One-step generation of mice carrying reporter and conditional alleles by CRISPR/Cas-mediated genome engineering. Cell, 2013, 154(6): 1370-1379.
[20] Tsukada Y, Fang J, Erdjument-Bromage H, Warren ME, Borchers CH, Tempst P, Zhang Y. Histone demethylation by a family of JmjC domain-containing proteins. Nature, 2006, 439(7078): 811-816.
[21] Han ZF, Liu PY, Gu LC, Zhang Y, Li H, Chen S, Chai JJ. Structural basis for histone demethylation by JHDM1. Front Sci, 2007, (1): 52-61.
[22] Phillips IR, Shephard EA. Flavin-containing monooxygenases: mutations, disease and drug response. Trends Pharmacol Sci, 2008, 29(6): 294-301.
[23] Yamazaki H, Shimizu M. Genetic polymorphism of the flavin-containing monooxygenase 3 (FMO3) associated with trimethylaminuria (fish odor syndrome): observations from Japanese patients. Curr Drug Metab, 2007, 8(5): 487-491.
[24] Li DL, Qiu ZW, Shao YJ, Chen YT, Guan YT, Liu MZ, Li YM, Gao N, Wang LR, Lu XL, Zhao YX, Liu MY. Heritable gene targeting in the mouse and rat using a CRISPR-Cas system. Nat Biotechnol, 2013, 31(8): 681-683.
[25] Skarnes WC, Rosen B, West AP, Koutsourakis M, Bushell W, Iyer V, Mujica AO, Thomas M, Harrow J, Cox T, Jackson D, Severin J, Biggs P, Fu J, Nefedov M, de Jong PJ, Stewart AF, Bradley A. A conditional knockout resource for the genome-wide study of mouse gene function. Nature, 2011, 474(7351): 337-342.
[26] Li H, Haurigot V, Doyon Y, Li TJ, Wong SY, Bhagwat AS, Malani N, Anguela XM, Sharma R, Ivanciu L, Murphy SL, Finn JD, Khazi FR, Zhou SZ, Paschon DE, Rebar EJ, Bushman FD, Gregory PD, Holmes MC, High KA. In vivo genome editing restores haemostasis in a mouse model of haemophilia. Nature, 2011, 475(7355): 217-221.
[27] Merlin C, Beaver LE, Taylor OR, Wolfe SA, Reppert SM. Efficient targeted mutagenesis in the monarch butterfly using zinc-finger nucleases. Genome Res, 2013, 23(1): 159-168. [28] Geurts AM, Cost GJ, Freyvert Y, Zeitler B, Miller JC, Choi VM, Jenkins SS, Wood A, Cui XX, Meng XD, Vincent A, Lam S, Michalkiewicz M, Schilling R, Foeckler J, Kalloway S, Weiler H, Ménoret S, Anegon I, Davis GD, Zhang L, Rebar EJ, Gregory PD, Urnov FD, Jacob HJ, Buelow R. Knockout rats via embryo microinjection of zine-finger nucleases. Science, 2009, 325(5939): 433.
[29] Brown AJ, Fisher DA, Kouranova E, McCoy A, Forbes K, Wu YM, Henry R, Ji D, Chambers A, Warren J, Shu WG, Weinstein EJ, Cui XX. Whole-rat conditional gene knockout via genome editing. Nat Methods, 2013, 10(7): 638-640. [30] Niu YY, Shen B, Cui YQ, Chen YC, Wang JY, Wang L, Kang Y, Zhao XY, Si W, Li W, Xiang AP, Zhou JK, Guo XJ, Bi Y, Si CY, Hu B, Dong GY, Wang H, Zhou ZM, Li TQ, Tan T, Pu XQ, Wang F, Ji SH, Zhou Q, Huang XX, Ji WZ, Sha JH. Generation of gene-modified cynomolgus monkey via Cas9/RNA-mediated gene targeting in one-cell embryos. Cell, 2014, 156(4): 836-843.
[31] Shen B, Zhang X, Du YN, Wang JY, Gong J, Zhang XD, Tate PH, Li HL, Huang XX, Zhang WS. Efficient knockin mouse generation by ssDNA oligonucleotides and zinc-finger nuclease assisted homologous recombination in zygotes. PLoS One, 2013, 8(10): e77696.
(责任编委: 张博)
Generation of site-specific mutant mice using the CRISPR/Cas9 system
Min Bai1,2, Qi Li2, Yanjiao Shao3, Yuanhua Huang2, Dali Li3, Yanlin Ma2
1. Guiyang College of Traditional Chinese Medicine, Guiyang 550002, China; 2. Hainan Provincial Key Laboratory for Human Reproductive Medicine and Genetic Research, Hainan Reproductive Medical Center, the Affiliated Hospital of Hainan Medical University, Haikou 570102, China;3. Shanghai Key Laboratory of Regulatory Biology, Institute of Biomedical Sciences and School of Life Sciences, East China Normal University, Shanghai 200241, China
The CRISPR/Cas9 system is a recently developed important technology for genome editing in cellular and animal models. Here we established a CRISPR/Cas9-based system of generating site-specific mutant mice using DNA double-strand breaks (DSBs) induced homologous recombination (HR)-dependent or independent repair me-chanism. Through co-microinjection of Cas9 mRNA and single-guide RNA (sgRNA) targeting genomic DNA sequence corresponding to enzyme activity of lysine (K)-specific demethylase 2b (Kdm2b), both a frame-shifted Kdm2b null mutant and a Kdm2b enzyme activity disrupted mouse strain were obtained simultaneously. Moreover, sgRNA targeting flavin containing monooxygenases3 (Fmo3) gene and the corresponding single strand oligonucleotides (ssODN) donor template with point mutation were co-injected into the male pronucleus of one-cell mouse embryos stimulated HR-mediated repair mechanism. Genomic sequence analysis of F0mice showed that frame-shifted Fmo3 knockout mouse and site-specific Fmo3 knock-in mouse with single base substitution were successfully generated, and these mutations could be stably transmitted to the next generation. Therefore, we successfully generated mouse strains containing site-specific mutations through HR-dependent and -independent DSB repair using the CRISPR/Cas9 system.
knockout; knock-in; enzyme activity mutant; homologous recombination
2015-03-26;
2015-07-21
国家重点基础研究发展计划项目(973项目)(编号:2012CB966502),国家国际科技合作专项(编号:2014DFA30180),国家自然科学基金项目(编号:81060175,30860103,81460034,81260032,81060016,31140021),海南省重大科技项目(编号:ZDZX2013003),海南省国际科技合作专项(编号:GJXM20100004,GJXM201106,KJHZ2014-11),人力资源和社会保障部2011年度留学人员科技活动择优资助项目和教育部归国留学人员启动基金项目资助
白敏,硕士研究生,专业方向:生殖医学。Tel: 0898-66776091;E-mail: whizzard@163.com
马燕琳,博士,副教授,研究方向:生殖医学与医学遗传学。E-mail: mayanlinma@hotmail.com
李大力,博士,研究员,研究方向:发育生物学。E-mail: dalidalili@yahoo.com
10.16288/j.yczz.15-127
时间:2015-8-12 9:06:31
URL:http://www.cnki.net/kcms/detail/11.1913.R.20150812.0906.002.html

