基因组编辑技术——CRISPR/Cas系统
陈勇龙,黄华荣,唐平平,羊雪芹,李斐雪,许 杰,张遵义
(1.杭州师范大学发育与再生研究所,浙江 杭州 310036;2.石门中心卫生院,浙江 嘉兴 314512)
基因组编辑技术——CRISPR/Cas系统
陈勇龙1,黄华荣1,唐平平2,羊雪芹1,李斐雪1,许杰1,张遵义1
(1.杭州师范大学发育与再生研究所,浙江 杭州 310036;2.石门中心卫生院,浙江 嘉兴 314512)
摘要:基因定点编辑技术包括基于胚胎干细胞及同源基因片段重组的基因打靶技术、锌指结构(ZFN)、类转录激活因子效应物(TALEN)以及CRISPR/Cas系统.CRISPR/Cas系统具有操作简单、突变率高、成本低,同时可针对多个基因等优点;该技术可进行定点修饰, 如敲除、插入、替换等.目前CRISPR/Cas系统已成功应用到小鼠、人类细胞、线虫、果蝇、斑马鱼、拟南芥、水稻和猴等.文章将对基因修饰技术发展脉络做统一梳理,并将系统阐述CRISPR/Cas系统的原理、应用以及该技术的优缺点.
关键词:基因修饰;CRISPR/Cas系统;基因编辑
从20世纪50年代沃森和克里克揭示了DNA分子的双螺旋结构开始,人们真正从分子水平认识了基因,开启了通过改造基因来改造生物的科学实践.1980年,Gordon利用显微注射的方法获得了第一例转基因小鼠,标志着动物转基因技术的建立,也是基因编辑技术的开始[1].
基因编辑技术是人们开启认识基因功能的钥匙.早期研究基因功能主要是利用随机插入方式将外源基因整合到生物体内或是通过同源重组方式删除目的基因(图1),以获得遗传动物修饰模型,这些技术有诸多不利如周期长、成本高、技术体系复杂等;随着人类基因组计划的完成,将已知基因的序列与功能联系在一起的功能基因组学研究成为后基因组时代的一大研究热点.2012入选《Science》十大重要科学进展之一——基因组编辑技术[2],方便了研究者构建基因修饰模型生物;这些技术可分为三类:锌指核酸酶(Zinc finger nucleases, ZFN)、类转录激活因子效应物核酸酶(Transcription activator-like effector nucleases, TALEN)和成簇规律间隔短回文重复与Cas蛋白(Clustered regularly interspaced short palindromic repeats, CRISPR/Cas).这些技术的应用提高了基因编辑的效率,大大缩短模型生物建立的周期,方便对基因功能的研究.
1传统基因修饰技术
传统的基因修饰技术主要是利用随机导入方式将目的基因整合到基因组上而产生转基因机体[1],以及基于胚胎干细胞与同源基因片段重组的基因打靶方式敲除目的基因而得到转基因机体[3](图1).该技术是过去几十年并延续至今的研究基因功能及基因产物与关联疾病机理的手段.但是这种基因修饰技术对实验技术要求高,时间和资金投入大等缺点限制了其发展,而近些年来兴起的基因组编辑技术可对目的基因进行定点插入或删除,同时操作步骤更简化,周期和费用大大降低.
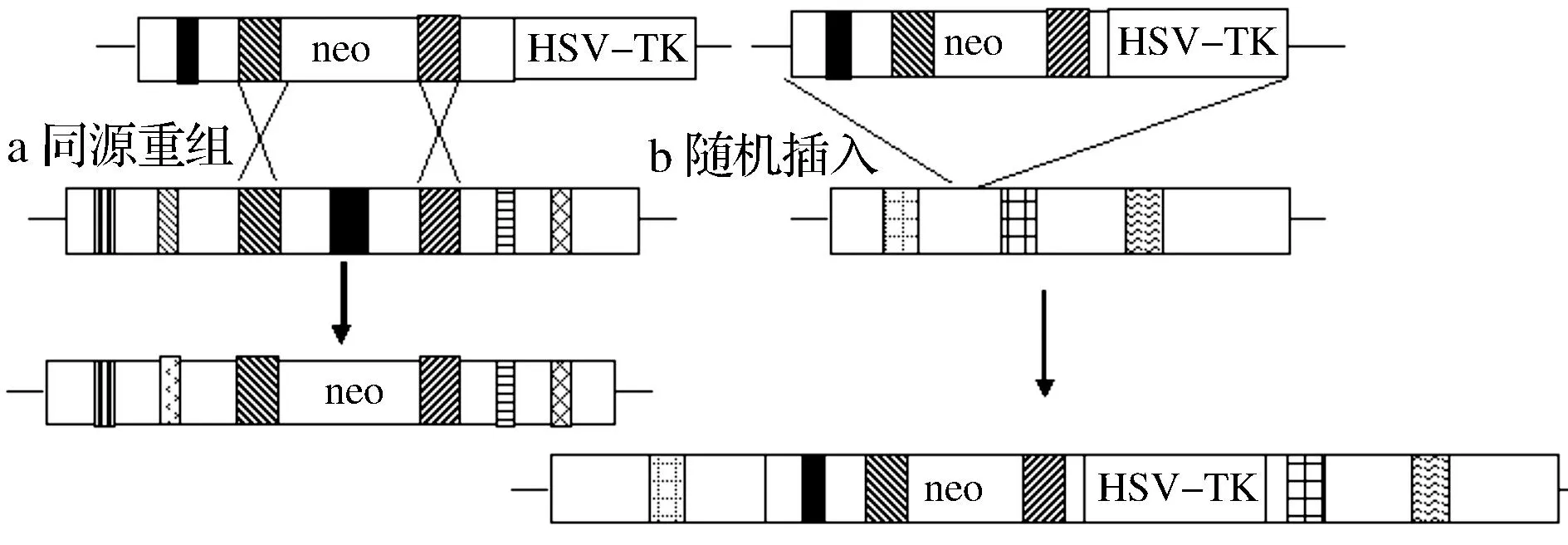
a.同源重组方式删除目的基因;b.随机插入方式整合到基因组图1 传统转基因技术原理Fig. 1 The theory of traditional transgenic technology
2基因组编辑技术
基因组编辑技术主要是通过核酸酶在DNA靶位点引起DNA双链断裂,激活细胞内DNA两种修复机制:非同源末端连接(Non-homologous End Joining, NHEJ)和同源重组(Homologous Recombination, HR),对靶点DNA进行修复.核酸酶主要依赖于蛋白质或RNA与非特异性内切酶的组合对目的DNA序列进行切割.目前已经成熟应用的基因组编辑核酸酶有锌指核酸酶(ZFNs)和类转录激活因子效应物(TALENs),以及2013年以来一项新兴的基因组编辑技术——CRISPR/Cas系统,该系统已逐渐成熟并成功应用到多种生物,促进了基因功能的研究.
ZFN和TALEN两个系统的核酸酶分别依靠于锌指蛋白和类转录激活因子蛋白的特异性识别DNA序列,然后依靠非特异性的FokI内切酶切割DNA.Yang等利用ZFNs技术研究导致动脉硬化症的ApoCIII基因,设计了8对针对ApoCIII基因的第二个外显子2的 ZFNs,将mRNA注射到兔的受精卵,体外培养结果显示ApoCIII基因突变高达50%,体内验证ApoCIII突变达到23.8%[4].Luo等针对中国黄牛的肌生成抑制蛋白基因MSTN设计了一对ZFNs,靶点位于MSTN的第一个外显子上,胚胎鉴定结果显示,靶点突变率达20%,等位基因发生突变达8.3%[5].Wilson 等使用SSA技术针对人端粒反转录酶基因hTERT设计了5对特异性的ZFNs,将700 ng ZFN表达质粒转染到293细胞内,靶点突变达18.5%[6].ZFN不仅可以修饰目的基因,还可以用于疾病的治疗.Xavier等利用借助腺病毒将ZFN和同源序列注射到小鼠腹腔,通过DNA同源重组修复机制将突变基因hF9修复,以达到治愈乙型血友病[7].同时Soldner在干细胞水平利用ZFN技术将突变的α-突触核蛋白基因功能恢复正常,该基因的点突变会造成帕金森综合症[8].Höher等利用ZFN技术成功使皮肤起泡致病基因失活,并且发现高剂量的ZFN对皮肤干细胞没有伤害同时仍具有再生活力[9].由此可看出,ZFN在人类疾病的治疗方面具有很大的潜力;目前,ZFN进行基因治疗已应用到临床阶段.ZFN在基因编辑方面表现出色,但自身缺点限制了其发展(表1),因此,需要一种更为方便的技术解决ZFN的缺点.

表1 ZFN、TALEN和CRISPR/Cas系统特点
TALEN蛋白和真核生物的转录因子类似,识别特定的DNA序列调控基因的表达.欧光朔等利用TALEN技术修饰线虫表皮胶原蛋白基因dpy-5,导致线虫体型较小[10].马三垣等利用TALEN技术修饰家蚕的BmBlos2,发现对目的基因的修饰高达60%,同时还发现利用一对TALEN同时注射可以删除大片段的核苷酸序列[11].刘海亮等针对关联Rett综合症发病基因Mecp2设计TALEN,将TALEN质粒注射到猴受精卵内成功将该基因修饰,得到表型与人的Rett综合症一致[12].Takada等利用TALEN技术成功将miR-10a敲除,该技术是在microRNA的前体上进行修饰,造成成熟的microRNA功能丧失[13].TALEN技术已经应用到大多数模式生物,例如猴[12]、果蝇[14]、小鼠[7]、线虫[10]和斑马鱼[15]等等,可见该技术没有物种特异性.虽然无论从时间还是资金方面,TALEN比ZFN较具有优势(见表1),但是TALEN也存在着不足之处:识别目的基因的成分是蛋白质,构建识别20 bpDNA的TALEN蛋白可能需要上千个氨基酸,对机体来说可能会产生免疫反应降低TALEN效率.如何高效、廉价、快速地构建研究模型,是很多研究者共同关心的话题.CRISPR/Cas9技术的出现满足了研究者的这一需求.
3CRISPR/Cas系统
3.1 CRISPR发现历史及类型
CRISPR/Cas系统发现于1987年,日本研究者在研究EcoliK12碱性磷酸酶序列附近发现了成簇的短的间隔重复序列[16];Grissa等研究表明,大约有40%的细菌和90%的古生菌的基因组都存在这种序列,定位于染色体或质粒上[17].细菌和古生菌内的CRISPR/Cas系统主要用于抵御病毒或噬菌体的入侵,是它们进化形成的一种适应性免疫系统.CRISPR/Cas的组成序列很保守,这些短的重复序列被短的可变序列所间隔.CRISPR/Cas系统内,Cas蛋白将入侵的DNA切割为短的序列并整合到细菌或古生菌的基因组内,这些序列插入到重复序列之间,当病毒或噬菌体再次入侵时,细菌或古生菌以整合到基因组的序列为模板转录为crRNA, crRNA引导Cas蛋白切割入侵的细菌或古生菌的DNA序列.目前已知的Cas蛋白家族已超过40个,他们在crRNA形成、外源DNA的整合以及剪切外源DNA发挥重要作用.根据Cas基因序列及蛋白结构,可将CRISPR/Cas系统分为3种类型:I型、II型和III型.I型和III型较复杂,需要多个cas蛋白形成复合物才能切割靶DNA链,而II型只需要一个Cas9蛋白即可对DNA链进行切割,因此II型也是目前被成功改造的人工核酸酶[18].本文将以II型CRISPR/Cas系统进行重点阐述:II型CRISPR/Cas系统仅含有一个Cas9蛋白和一段20 bp的sgRNA序列,sgRNA用于识别目的序列,Cas9指导前体crRNA的成熟和外源DNA或质粒的降解.Cas9蛋白由1 409个氨基酸组成的多结构域蛋白质,包括N端的类Ruc核酸酶结构域和中部NHN核酸酶结构域,NHN结构域可切割与crRNA互补配对的模板链,切割位点在PAM序列上游4 bp的位置;Ruc结构域可对另一条链进行切割,切割位点在PAM序列上游3~8 bp(图2).
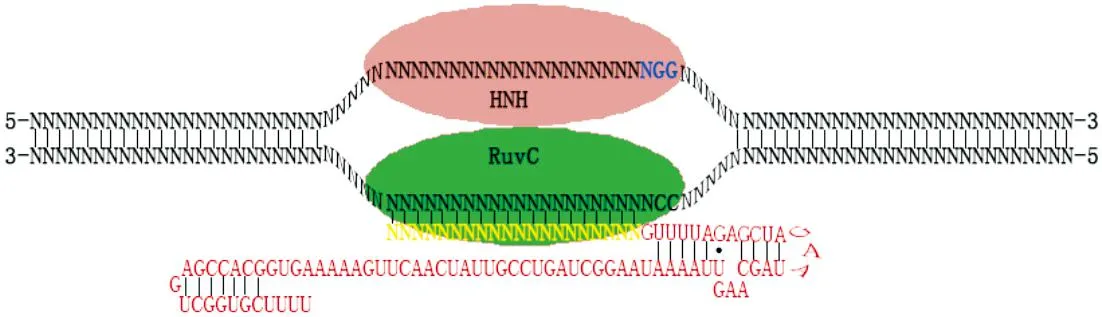
注:绿色部分为RuvC结构域,粉色部分为HNH结构域;↓为DNA序列断裂位点;黄色序列为sgRNA,蓝色为PAM序列,红色序列为chimeric guide RNA折叠序列图2 CRISPR/Cas9 模式图Fig. 2 The CRISPR/Cas9 model
3.2 CRISPR/Cas9作用原理
II型CRISPR/Cas系统编码tracrRNA,tracrRNA指导RNaseIII和crRNA前体的成熟;成熟的crRNA与tracrRNA互补配对形成RNA二聚体,介导Cas9蛋白对靶位点进行切割,造成DNA双链的断裂,引起体内细胞修复机制包括插入/缺失引起的末端连接和同源重组修复机制(图2).插入/缺失机制会造成原序列缺少一些碱基或在原序列引入一些碱基,无论何种情况都会引起目的基因突变,可能使该基因失去功能.而同源重组介导的DNA修复机制,利用CRISPR/Cas系统将DNA双链切断,通过引入的一段与目的基因同源序列进行同源交换以达到修复目的基因.大量实验证明,CRISPR/Cas9是最行之有效的基因编辑工具,已广泛应用于基因组编辑、基因治疗以及转录调控.
3.3 CRISPR/Cas9系统应用
CRISPR/Cas系统在基因组编辑方面具有优势,就II型系统来说仅需要一段20 bp的DNA序列和Cas9蛋白.目前,已有商品化的质粒供研究者选择,仅需将20 bp的sgRNA克隆到CRISPR/Cas9质粒即可.Wang等利用该技术将Cas9 mRNA和sgRNA注射到小鼠的受精卵,结果显示80%都发生了等位基因的突变;CRISPR/Cas9系统打破了在基因组编辑方面一次仅能编辑一个基因的格局,该文章作者设计5个不同基因的sgRNA的质粒同时转染到小鼠胚胎干细胞内,结果发现Tet1、Tet2、Tet3和Sry、Uty同时被编辑且效率很高,这对于研究基因家族成员的功能相关性至关重要[19].同样的结果在斑马鱼[20]、人体细胞[21]、线虫[22]、大鼠[23]和猴[24]等也得到了验证.单奇伟等利用CRISPR/Cas技术对水稻OsPDS、OsMPK2 以及OsBADH2基因进行改造得到了这3个基因的突变株,这对于作物性状改良及分子定向育种非同寻常[25].
基因能否正常发挥功能,取决于在转录水平能否正常转录.在转录水平研究基因,对了解功能及调控网络非常有意义.由于CRISPR/Cas系统具有定向识别作用,当sgRNA和Cas蛋白形成复合物结合到DNA序列上后,复合体的结合解开DNA双链可能导致RNA酶及转录因子无法结合到DNA序列使得转录受阻.Lei等利用此原理设计两个sgRNA,一个结合在编码红色荧光蛋白的序列,另一个结合在编码绿色荧光蛋白的序列,两个蛋白表达互不干扰的情况下,将两个sgRNA和Cas9质粒共转染,结果显示发现两种蛋白荧光强度均大幅度下调[26].Gilbert等将抑制因子及激活因子和Cas9蛋白共同表达,结果显示在人的细胞系和酵母细胞内基因的转录明显受到抑制或激活;将转录抑制因子和Cas9蛋白耦合表达,发现很多基因表达受到抑制[27].因此,iCRISPR (interfere CRISPR )系统是个特异性极强的工具,在不改变传统遗传物质的基础上调控基因的表达.
CRISPR/Cas不仅可以调控基因的表达,还可校正突变基因.Urnov等利用ZFN技术在细胞水平校正了镰刀状贫血病和乙型血友病的突变基因片段,尽管致病基因得到校正,但是通过腺病毒感染人细胞从而将ZFN和同源序列引入细胞[28].然而,CRISPR/Cas9系统为基因治疗提供了一个新的、有力的技术手段.CRISPR/Cas9系统进行基因治疗的原理在于同源介导的修复机制,将正确的序列替换下突变的序列,从而治愈疾病.李劲松课题组利用CRISPR/Cas9系统成功将小鼠白内障遗传病治愈[29]; Schwank等利用该技术校正人干细胞中一种与囊肿性纤维化相关联的基因缺陷[30].Ebina等利用该技术将HIV-1启动子沉默,在感染CRISPR/Cas9质粒的人干细胞细胞中发现HIV-1表达明显下降[31].因此,可以利用CRISPR/Cas9系统对一些难以治愈的疾病进行基因修复,以恢复其正常功能.
3.4 CRSIPR/Cas系统利弊
人工核酸酶最吸引人的地方在于个性化的定制及对基因组进行定点修饰.目前这三种人工核酸酶在基因组改造方面都有着很高的编辑效率.CRISPR/Cas系统相较于ZFN和TALEN有着明显的优势:1.CRISPR/Cas系统比ZFN和TALEN在基因组中分布范围广,每8 bp就有一个靶点,而TALEN 1/125 bp,ZFN 1/500 bp;2.CRISPR/Cas系统可以一次对多个基因进行定点修饰,而ZFN和TALEN则需要多对;3.CRISPR/Cas系统性价比高,仅需将一段20 bp的sgRNA克隆到表达质粒上即可,ZFN和TALEN目前都被公司垄断——成本高,且需要各种组装——操作复杂;4.II型CRISPR/Cas系统易于改造,将Cas9蛋白功能域NHN结构域和RuvC结构域中的一个进行突变,突变的Cas9蛋白只能切割DNA单链,增加缺陷基因被修复的概率;双突变可以在转录水平调控基因表达;还可将其他功能蛋白加到Cas9蛋白末端,例如将转录抑制因子连接到Cas9蛋白末端等,用于抑制基因转录[27].因此,研究人员有待对CRISPR/Cas系统进行深入研究,使其具有更广泛的实用性及高效性.
基因组编辑技术在基因修饰方面有着重要作用,特别是CRISPR/Cas系统在生物模型构建方面的优势更是传统转基因技术无法达到的.但无论ZFN还是TALEN,以及性价比很高的CRISPR/Cas系统都无法避开一个问题:脱靶效应[32-33].由于II型CRISPR/Cas系统的特异性取决于PAM前20 bp的sgRNA,理论上脱靶效率会很高,目前很多实验证实CRISPR/Cas系统的脱靶效率低于ZFN和TALEN,特异性很高.但针对不同生物、不同组织、不同细胞CRISPR/Cas系统作用效率还有差别.CRISPR/Cas系统还面临另一个问题——很多器官组织及原代细胞难转染,张锋课题组构建出了慢病毒CRISPR/Cas9,可以高效转染组织和细胞[34].在原核注射方面,sgRNA和Cas9 RNA的浓度越高,作用效率越高,但是理论上来说可能会给胚胎带来高毒性; Wang等研究表明,高浓度的RNA给胚胎带来的毒性很低[19].因此,需要研究者继续优化以开发出低脱靶、高效率的CRISPR/Cas系统.
4展望
CRISPR/Cas系统对生命科学的发展起着革命性的作用,极大地拓展了研究者对模式生物的研究能力,同时也为探索治愈遗传性疾病提供了一条路径.该系统在基因改造方面具有极强的潜力和灵活性,但是,PAM序列的存在限制了II型CRISPR/cas系统的应用.如何对 II型CRISPR/Cas系统Cas9蛋白改造最终摒弃PAM序列,以拓展其应用范围?目前,研究者在研究强烈火球菌和硫磺矿硫化叶菌时发现Cas RAMP模块(Cmr)系统,由蛋白质和30~40 nt碱基组成,修饰对象为RNA;该系统具有高效率、低脱靶[35-36]特点.该技术还主要集中在对原核生物的研究,相信不久将来,将会应用到哺乳类等真核生物.可以预见,CRISPR/Cas系统在基础研究、疾病治疗以及育种方面具有广阔的应用前景,将产生深远的意义.
参考文献:
[1] Jon W G, George A S, Diane J P,etal. Genetic transformation of mouse embryos by microinjection of purified DNA[J]. PNAS,1980,7(12):7380-7384.
[2] Breakthrough of the Year. The runners-up[J]. Science,2012,338(6114):1525-1532.
[3] Plump A S, Smith J D, Hayek T,etal. Severe hypercholesterolemia and atherosclerosis in apolipoprotein E-deficient mice created by homologous recombination in ES cells[J]. Cell,1992,71(2):343-353.
[4] Yang D S, Zhang J F, Xu J,etal. Production of apolipoprotein C-III knockout rabbits using zinc finger nucleases[J]. J Vis Exp,2013,81:e50957.
[5] Luo J J, Song Z Y, Yu S L,etal. Efficient generation of myostatin (MSTN) biallelic mutations in cattle using zinc finger nucleases[J]. PLoS One,2014,9(4):e95225.
[6] Wilson K A, Chateau M L, Porteus M H. Design and development of artificial zinc finger transcription factors and zinc finger nucleases to thehTERTLocus[J]. Molecular Therapy-Nucleic Acids,2013,2(4):e87.
[7] Xavier M A, Rajiv S, Yannick D,etal. Robust ZFN-mediated genome editing in adult hemophilic mice[J]. Blood,2013,122(19):3283-3287.
[8] Soldner F, Laganière J, Cheng A W,etal. Generation of isogenic pluripotent stem cells differing exclusively at two early onset Parkinson point mutations[J]. Cell,2011,146(2):318-331.
[9] Höher T, Wallace L, Khan K,etal. Highly efficient zinc-finger nuclease-mediated disruption of an eGFP transgene in keratinocyte stem cells without impairment of stem cell properties[J]. Stem Cell Rev,2011,8(2):426-434.
[10] Cheng Z, Yi P S, Wang X M,etal. Conditional targeted genome editing using somatically expressed TALENs inC.elegans[J]. Nature Biotechnology,2013,31(10):934-937.
[11] Ma S Y, Zhang S L, Wang F,etal. Highly efficient and specific genome editing in silkworm using custom TALENs[J]. PLoS One,2012,7(9):e45035.
[12] Liu H L, Chen Y C, Niu Y Y,etal. TALEN-mediated gene mutagenesis in rhesus and cynomolgus monkeys[J]. Cell Stem Cell,2014,14(3):323-328.
[13] Takada S, Sato T, Ito Y,etal. Targeted gene deletion of miRNAs in mice by TALEN system[J]. PLoS One,2013,8(10):e76004.
[14] Takefumi K, Tetsushi S, Housei W,etal. TALEN-induced gene knock out inDrosophila[J]. Develop Growth Differ,2014,56(1):86-91.
[15] Zu Y, Tong X J, Wang Z X,etal. TALEN-mediated precise genome modification by homologous recombination in zebrafish[J]. Nature Methods,2013,10(4):329-331.
[16] Ishino Y, Shinagawa H, Makino K,etal. Nucleotide sequence of theiapgene, responsible for alkaline phosphatase isozyme conversion inEscherichiacoli, and identification of the gene product[J]. J Bacteriol,1987,169(12):5429-5433.
[17] Grissa I, Vergnaud G, Pourcel C. The CRISPRdb database and tools to display CRISPRs and to generate dictionaries of spacers and repeats[J]. BMC Bioinformatics,2007,8(1):172.
[18] Deltcheva E, Chylinski K, Sharma C M,etal. CRISPR RNA maturation by trans-encoded small RNA and host factor RNaseIII[J]. Nature,2011,471(7340):602-607.
[19] Wang H Y, Yang H, Shivalila C S,etal. One-step generation of mice carrying mutations in multiple genes by CRISPR/Cas-mediated genome engineering[J]. Cell,2013,154(6):1370-1379.
[20] Chang N N, Sun C H, Gao L,etal. Genome editing with RNA-guided Cas9 nuclease in zebrafish embryos[J]. Cell Res,2013,23(4):465-472.
[21] Mali P, Yang L H, Esvelt K M,etal. RNA-guided human genome engineering via Cas9[J]. Science,2013,339(6121):823-826.
[22] Friedland A E, Tzur Y B, Esvelt K M,etal. Heritable genome editing inC.elegansvia a CRISPR-Cas9 system[J].Nat Methods,2013,10(8):741-743.
[23] Li D L, Qiu Z W, Shao Y J,etal. Heritable gene targeting in the mouse and rat using a CRISPR-Cas system[J]. Nat Biotechnology,2013,31(8):681-683.
[24] Niu Y Y, Shen B, Cui Y Q,etal. Generation of gene-modifiedcynomolgusmonkey via Cas9/RNA-mediated gene targeting in one-cell embryos[J]. Cell,2014,156(4):836-843.
[25] Shan Q W, Wang Y P, Li J,etal. Targeted genome modification of crop plants using a CRISPR-Cas system[J]. Nature Biotechnology,2013,31(8):686-688.
[26] Lei S Q, Matthew H L, Luke A G,etal. Repurposing CRISPR as an RNA-Guided platform for sequence specific control of gene expression[J]. Cell,2013,152(5):1173-1183.
[27] Gilbert L A, Larson M H, Morsut L,etal. CRISPR-Mediated modular RNA-Guided regulation of transcription in eukaryotes[J]. Cell,2013,154(2):442-451.
[28] Urnov F D, Miller J C, Lee Y L,etal. Highly efficient endogenous human gene correction using designed zinc-finger nucleases[J]. Nature,2005,435(7042):646-651.
[29] Wu Y X, Liang D,Wang Y H,etal. Correction of a genetic disease in mouse via use of CRISPR-Cas9[J]. Cell Stem Cell,2013,13(6):659-662.
[30] Schwank G, Koo B K, Sasselli V,etal. Functional repair of CFTR by CRISPR/Cas9 in intestinal stem cell organoids of cystic fibrosis patients[J]. Cell Stem Cell,2013,13(6):653-658.
[31] Ebina H, Misawa N, Kanemura Y,etal. Harnessing the CRISPR/Cas9 system to disrupt latentHIV-1 provirus[J]. Sci Rep,2013(3):2510.doi:10.1038/srep02510.
[32] Kuscu C, Arslan S, Singh R,etal. Genome-wide analysis reveals characteristics of off-target sites bound by the Cas9 endonuclease[J]. Nat Biotechnology,2014,32(7):677-683.
[33] Lin Y N, Cradick T J, Brown M T,etal. CRISPR/Cas9 systems have off-target activity with insertions or deletions between target DNA and guide RNA sequences[J].Nucleic Acids Res,2014,42(11):7473-7485.
[34] Yin H, Xue W, Chen S D,etal. Genome editing with Cas9 in adult mice corrects a disease mutation and phenotype[J]. Nat Biotechnology,2014,32:551-553.
[35] Hale C R, Zhao P, Olson S,etal. RNA-guided RNA cleavage by a CRISPRRNA-Cas protein complex[J]. Cell,2009,139(5):945-956.
[36] Hale C R., Majumdar S, Elmore J,etal. Essential features and rational design of CRISPR RNAs that function with the Cas RAMP module complex to cleave RNAs[J]. Mol Cell,2012,45(3):292-302.
Genome Editing Techniques—CRISPR/Cas System
CHEN Yonglong1, HUANG Huarong1, TANG Pingping2, YANG Xueqin1, LI Feixue1, XU Jie1, ZHANG Zunyi1
(1.Institute of Developmental and Regenerative Biology, Hangzhou Normal University, Hangzhou 310036, China;
2.Shimen Central Hospital, Jiaxing 314512, China)
Abstract:Genome editing technologies include gene targeting based on embryonic stem cells and homologous gene fragments recombination, zinc-finger nucleases(ZFN), transcription activator-like effectors nuclease(TALEN) and CRISPR/Cas system. CRISPR/Cas system has merits of operating simply, high mutation rate with low-cost and editing multiple genes simultaneously. It can fixed-point modify, such as knockout, insert and replace genes. This system has successfully applied to a variety of organisms such as mouse, human cells, nematode, fruit fly, zebra fish, arabidopsis, rice and monkey. The paper outlines the development of gene modificative technology, and illustrates the principle, application as well as the merits and demerits of CRISPR/Cas system.
Key words:genomic modification; CRISPR/cas system; genome editing

第14卷第1期2015年1月杭州师范大学学报(自然科学版)JournalofHangzhouNormalUniversity(NaturalScienceEdition)Vol.14No.1Jan.2015
文章编号:1674-232X(2015)01-0060-06
中图分类号:Q33;Q78
文献标志码:A
doi:10.3969/j.issn.1674-232X.2015.01.011
通信作者:张遵义(1957—),男,教授,博士,主要从事动物头面部器官发育及新生缺陷形成中的遗传学机理研究.E-mail:zunyizhang@idrbio.org
基金项目:浙江省大学生科技创新活动计划暨新苗人才计划项目(2014R421071);杭州师范大学大学生科研项目(1283XXM141);浙江省科技计划项目(2013C37024);杭州师范大学2014研究生创新基金项目.
收稿日期:2014-07-31

