DNA甲基化芯片检测技术在恶性肿瘤诊疗中的应用
王 娟(综述),张艳亮,段 勇(审校)
(昆明医科大学第一附属医院检验科,昆明 650032)
DNA甲基化芯片检测技术在恶性肿瘤诊疗中的应用
王娟△(综述),张艳亮※,段勇(审校)
(昆明医科大学第一附属医院检验科,昆明 650032)
摘要:表观遗传学的改变普遍存在于各种恶性肿瘤中,DNA甲基化是表观遗传学改变的主要形式之一。DNA甲基化状态的改变可显著影响基因的表达,抑癌基因高甲基化及癌基因低甲基化是导致恶性肿瘤发生、发展的重要机制。因此,检测恶性肿瘤DNA甲基化状态的变化对于揭示其发病机制意义重大。近年来,DNA甲基化检测技术尤其是DNA甲基化芯片检测技术在恶性肿瘤发病机制、临床诊疗等方面得到越来越广泛的应用。
关键词:甲基化芯片技术;恶性肿瘤;去甲基化药物
近年来随着基因检测技术的发展,研究人员发现,除了基因序列改变外,基因的表观遗传学改变也是导致恶性肿瘤发生、发展的重要机制[1]。DNA甲基化是基因表观遗传学改变的主要形式之一,所谓DNA甲基化,是指通过DNA甲基化转移酶(DNA methyltransferases,DNMTs)将S腺苷甲硫氨酸上的甲基转移至胞嘧啶的碳5′位上。大于500 bp 的DNA序列,GC含量大于55%的DNA区域称为CpG岛[2]。哺乳动物50%~60%的启动子区位于CpG岛上[3]。正常组织中,CpG岛一般没有甲基化,CpG岛外的CG序列可发生甲基化;当CpG岛的胞嘧啶发生甲基化或CpG岛外CG序列发生低甲基化时可导致肿瘤的发生[4]。
1检测DNA甲基化的方法
检测基因组DNA甲基化水平的方法很多,包括亚硫酸氢盐测序法、限制性内切酶酶切法、甲基化酶温育法、高压液相色谱法、免疫法、氯乙醛法等;检测特定DNA片段甲基化水平的方法包括限制酶聚合酶链反应法、甲基化特异性聚合酶链反应、甲基化敏感性单链核苷酸引物延伸、聚合酶链反应荧光溶解曲线分析等。常用的检测DNA甲基化的方法是亚硫酸氢盐测序,该方法最精确,可定量检测全基因组的甲基化水平,但该法需要大量的克隆测序,过程较为繁琐、耗时且费用昂贵[5];甲基化特异性聚合酶链反应是一种快速、特异性好、灵敏度高、所需样本量少的DNA甲基化检测方法,但其只能定性而不能准确定量[6]。由于上述检测方法的缺陷,有必要开发一种快捷、灵敏、相对廉价且可定量的检测的技术。DNA甲基化芯片检测技术具有上述优点,可用于恶性肿瘤发病机制的研究,而且在恶性肿瘤的诊断、分类、分期、预后判断及药物疗效预测等诸多方面均发挥重要作用。
2DNA甲基化芯片检测技术
甲基化免疫沉淀联合芯片的检测技术是将待测DNA通过酶切降解后,用甲基化胞嘧啶抗体免疫沉淀有甲基化修饰的胞嘧啶。对沉淀产物和非沉淀产物进行聚合酶链反应扩增。利用双色荧光分别标记两组扩增产物,再在基因芯片上杂交,最后通过荧光密度比值的高低筛选出差异甲基化基因。该技术可在整个基因组范围内扫描差异甲基化,也可针对某个特定基因的甲基化进行检测。甲基化芯片检测技术具有高通量性、自动化、微型性的优点,可用于大规模研究[7]。
3DNA甲基化芯片检测技术在恶性肿瘤诊疗中的应用
3.1筛查患癌高危人群及早期诊断对于恶性肿瘤患者来说,早期治疗可以提高患者生存期,减小手术创面,提高患者生存质量。如乳腺癌早期诊断的存活率为98%,晚期诊断存活率仅为27%[8]。目前,CT或磁共振成像能发现直径>1 cm 的实体肿瘤,肿瘤标志物,如糖类肿瘤标志物CA199只能在肿瘤的中晚期才能检测到。目前研究人员可从基因及基因表观遗传学的水平检测肿瘤标志物,因为在实体肿瘤出现前,基因表观遗传学就有可能发生明显的改变。如在多个类型的肿瘤形成前,多梳蛋白组靶基因就已经出现高甲基化,所以其可作为一个诊断潜在肿瘤的标志物[9]。针对恶性肿瘤早期生物学标志物的研究是很有必要的。Bethqe等[10]应用芯片技术在480例淋巴瘤患者中筛查表观遗传学药物治疗前后基因的表达情况发现,治疗后表达上调的基因有233个;在18个淋巴瘤细胞株中用甲基化特异性聚合酶链反应检测233个基因中表达上调最高的30个基因发现,有7个基因在>70%的细胞株中出现甲基化;再在37例淋巴瘤病例中检测这7个基因,其中的36例患者中牙本质涎蛋白基因、人卷曲蛋白8基因、电压门控钾通道H2基因及人类蛋白磷酸酶1调节亚基14A基因启动子区的甲基化率分别为28%、67%、22%和78%,把它们结合起来的阳性率高达89%,表明这些基因很有可能是诊断和监测B细胞非霍奇金淋巴瘤的标志物。Kim等[11]应用甲基化芯片技术发现,人中心体蛋白1基因在前列腺癌和胰腺癌中的高表达与其启动子区甲基化有关。Kuhmann等[12]应用DNA甲基化芯片技术检测大肠癌患者中231个DNA修复基因,发现在不同人种间大肠癌患者的基质金属蛋白酶9基因、DNA甲基化转移酶3A基因及连接酶4基因均出现高甲基化,在51%的患者中连接酶4基因启动子区的高甲基化达60%。连接酶4基因启动子甲基化是控制连接酶Ⅳ表达的新机制,其可能是诊断大肠癌的一个新的表观遗传学标志物[12]。随着研究的开展,有望找出特定肿瘤的特异性标志物,从而早期诊断肿瘤。常见的高甲基化基因及其相关的恶性肿瘤类型见表1。

表1 肿瘤中常见的高甲基化基因及其相关的恶性肿瘤类型
RASSF1A:相关区域家族1A基因;APC:大肠息肉基因;BRCA1:乳腺癌1基因;CDH1:钙黏蛋白1基因;CDKN2A:细胞周期蛋白依赖性激酶抑制剂2A基因;GSTP1:谷胱甘肽S转移酶1基因;MGMT:甲基鸟嘌呤-DNA甲基转移酶基因;DAPK1:死亡相关蛋白激酶 1基因
3.2评估预后应用甲基化芯片技术检测癌组织、癌旁组织、癌转移组织及正常组织的甲基化水平,并通过对比患者生存期、复发率等指标,寻找与预后相关的异常甲基化基因,以指导临床诊断,避免不必要的侵入性治疗。Sandoval等[13]使用甲基化芯片法检测444例非小细胞肺癌患者45万个CpG点的甲基化水平发现,组蛋白H4基因、 原钙黏蛋白亚族B6基因、黑白神经肽受体1基因、四氧嘧啶基因及同源异形盒A9基因的高甲基化可增加非小细胞肺癌的复发率。Zhen等[14]通过甲基化芯片分析69对鼻咽癌组织与正常鼻咽部组织的结缔组织生长因子基因的甲基化水平发现,结缔组织生长因子基因高甲基化导致该基因表达受抑制,使黏着斑激酶/磷脂酰肌醇三羟基激酶/蛋白激酶B途径活化并调节下游的细胞周期,最终导致肿瘤增殖、转移及侵袭。Hou等[15]应用甲基化芯片方法检测45对肝癌及癌旁组织和6例正常人肝组织的甲基化水平,并分析其中的高甲基化基因与无病生存期和总生存期的关系发现,无眼基因4的甲基化水平与无病生存期及总生存期呈反比,无眼基因4基因的表达水平与肿瘤的大小呈负相关,因此无眼基因4基因甲基化水平的检测有助于判断肝癌预后。上述研究表明,基因甲基化水平的改变与恶性肿瘤的预后相关,找出其关联程度可更有效地判断预后。
3.3预测药物疗效及靶向治疗DNA甲基化水平的检测可帮助临床医师判断恶性肿瘤患者对抗癌药物的反应,从而优化治疗,实现靶向及个体化治疗,减少疗程并评估药物不良反应。Moutinho等[16]应用甲基化芯片技术发现,大肠癌患者中致敏红细胞基因的高甲基化预示着对奥沙利铂的抵抗。在Karakoula等[17]的研究中,应用芯片技术检测颅内室管膜瘤经5-氮杂-2-脱氧胞苷(5-aza-2′-deoxycytidine,5-Aza-dC)治疗前后多个基因的甲基化改变发现,因高甲基化而失活的脑组织表达X1基因可作为靶向治疗的潜在靶标。Huang等[18]研究发现,骨髓增生异常综合征的患者磷酸酶张力蛋白同源基因高甲基化使磷酸酶张力蛋白同源基因表达降低、甲基化转移酶表达升高,应用尿多酸肽治疗后,可使磷酸酶张力蛋白同源基因逆甲基化。因此,基因的甲基化水平可预测抗肿瘤药物的疗效。常见的基因甲基化与药物疗效关系,见表2。
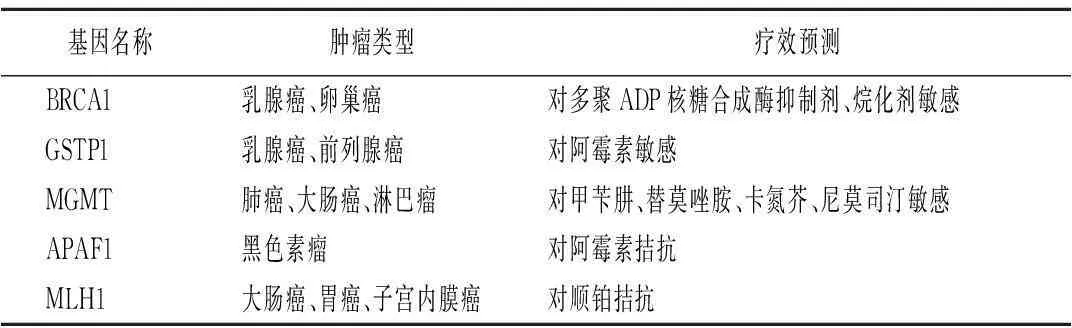
表2 常见基因高甲基化与抗肿瘤药物疗效预测
BRCA1:乳腺癌1基因;GSTP1:谷胱甘肽S转移酶1基因;MGMT:O-6-甲基鸟嘌呤-DNA甲基转移酶基因;APAF1:凋亡肽酶活化因子1基因;MLH1:mutL同系物1基因
4逆转甲基化的药物
DNA甲基化相对于基因的改变来说是可逆的,可使用DNMTs抑制剂对肿瘤进行靶向治疗。逆转甲基化的药物主要分为核苷类似物和非核苷类似物。
4.1核苷类似物核苷类似物有5氮杂胞苷(5-azacytidine,5-Aza-CR)和5-Aza-dC,这两种药物整合进入DNA,干扰DNA与DNMTs的相互作用,通过降解DNMTs改变嘧啶起作用。但两者也略有不同:5-Aza-CR下调p53,导致细胞凋亡;而5-Aza-dC可致细胞衰老[19]。这两种药物已被美国食品药物管理局批准用于骨髓增生异常综合征、恶性间皮瘤、白血病、乳腺癌以及鼻咽癌等疾病的治疗。但这两种药物细胞毒性大、易水解、不稳定。嘧啶酮-B-核苷是一种新的DNMTs抑制剂,可抑制DNMTs和胞嘧啶脱氨酶的作用,而且还可使抑癌基因再活化,且具有较低的细胞毒性和较高的肿瘤敏感性[20],其通过细胞周期阻滞以及caspase依赖的细胞凋亡和细胞死亡途径抑制肺癌细胞生长[21],其与甲氨蝶呤联合治疗急性淋巴细胞白血病具有很好的协同效应[20]。2′-脱氧-N4[2-(4-硝基苯基)乙氧羰基]-5-氮杂胞苷是一种5-氮杂-2-脱氧胞苷的前体药物,其可整合到DNA中,从而抑制DNMTs;2′-脱氧-N4[2-(4-硝基苯基)乙氧羰基]-5-氮杂胞苷可保护5-Aza-dC不被脱氨基,但疗效弱于5-Aza-dC,而且其活性依赖于羧酸酯酶[22]。RG108是一个小分子药物,其通过作用于DNA甲基化转移酶1,使其失活而发挥逆甲基化的作用[23]。
4.2非核苷类似物这类药物不需要整合进入DNA,所以细胞毒性较小。在实体肿瘤中逆甲基化的药物目前有肼苯达嗪和普鲁卡因胺[24]。
5展望
DNA甲基化芯片检测技术在恶性肿瘤的检测中具有重要作用,且DNA甲基化芯片检测技术较其他甲基化检测技术而言具有高通量、快捷、简便、可定量的优势,可广泛应用于科学研究中。该技术不仅可早期诊断恶性肿瘤,而且能监测肿瘤发展、评价药物疗效、评估预后以及在此基础上研究新药等。众多研究表明,甲基化标志物在临床应用是可行的,但目前仍存在诸多困难,如甲基化芯片检测技术仍需通过其他方式验证、特定肿瘤甲基化标志物的确定等[25]。
参考文献
[1]Stefansson OA,Esteller M.Epigenetic modifications in breast cancer and their role in personalized medicine[J].Am J Pathol,2013,183(4):1052-1063.
[2]Suzuki MM,Bird A.DNA methylation landscapes: provocative insights from epigenomics[J].Nat Rev Genet,2008,9(6):465-476.
[3]Jones PA,Liang G.Rethinking how DNA methylation patterns are maintained[J].Nat Rev Genet,2009,10(11):805-811.
[4]Pradhan MP,Desai A,Palakal MJ.Systems biology approach to stage-wise characterization of epigenetic genes in lung adenocarcinoma[J].BMC Syst Biol,2013,7:141.
[5]Lee YK,Jin S,Duan S,etal.Improved reduced representation bisulfite sequencing for epigenomic profiling of clinical samples[J].Biol Proced Online,2014,16(1):1.
[6]陆晓筱,葛明华,凌志强,等.甲状腺乳头状癌组织中hMLH1基因启动子甲基化及其临床意义[J].中华肿瘤杂志,2013,35(11):833-836.
[7]De Braekeleer E,Douet-Guilbert N,De Braekeleer M.Genetic diagnosis in malignant hemopathies:from cytogenetics to next-generation sequencing[J].Expert Rev Mol Diagn,2014,14(2):127-129.
[8]Xiang TX,Yuan Y,Li LL,etal.Aberrant promoter CpG methylation and its translational applications in breast cancer[J].Chin J Cancer,2013,32(1):12-20.
[9]Zhuang J,Jones A,Lee SH,etal.The dynamics and prognostic potential of DNA methylation changes at stem cell gene loci in women′s cancer[J].PLoS Genet,2012,8(2):e1002517.
[10]Bethqe N,Honne H,Hilden V,etal.Identification of highly methylated genes across various types of B-cell non-hodgkin lymp-homa[J].PLoS One,2013,8(11):e79602.
[11]Kim JJ,Rajagopalan K,Hussain B,etal.CETN1 is a cancer testis antigen with expression in prostate and pancreatic cancers[J].Biomark Res,2013,1(1):22.
[12]Kuhmann C,Li C,Kloor M,etal.Altered regulation of DNA ligase Ⅳ activity by aberrant promoter DNA methylation and gene amplification in colorectal cancer[J].Hum Mol Genet,2014,23(8):2043-2054.
[13]Sandoval J,Mendez-Gonzalez J,Nadal E,etal.A prognostic DNA methylation signature for stage Ⅰ non-small-cell lung cancer[J].J Clin Oncol,2013,31(32):4140-4147.
[14]Zhen Y,Ye Y,Yu X,etal.Reduced CTGF expression promotes cell growth,migration,and invasion in nasopharyngeal carcinoma[J].PLoS One,2013,8(6):e64976.
[15]Hou X,Peng JX,Hao XY,etal.DNA Methylation profiling identifies EYA4 gene as a prognostic molecular marker in hepatocellular carcinoma[J].Ann Surg Oncol,2014,21(12):3891-3899.
[16]Moutinho C,Martinez-Cardús A,Santos C,etal.Epigenetic inactivation of the BRCA1 interactor SRBC and resistance to oxaliplatin in colorectal cancer[J].J Natl Cancer Inst,2014,106(1):djt322.
[17]Karakoula K,Jacques TS,Phipps KP,etal.Epigenetic genome-wide analysis identifies BEX1 as a candidate tumour suppressor gene in paediatric intracranial ependymoma[J].Cancer Lett,2014,346(1):34-44.
[18]黄健,杨敏,金洁.尿多酸肽对骨髓增生异常综合征PTEN基因甲基化的影响及相关机制研究[J].中华血液学杂志,2013,34(7):600-605.
[19]Venturelli S,Berger A,Weiland T,etal.Differential induction of apoptosis and senescence by the DNA methyltransferase inhibitors 5-azacytidine and 5-aza-2′-deoxycytidine in solid tumor cells[J].Mol Cancer Ther,2013,12(10):2226-2236.
[20]Andrade AF,Borges KS,Castro-Gamero AM,etal.Zebularine induces chemosensitization to methotrexate and efficiently decreases AhR gene methylation in childhood acute lymphoblastic leukemia cells[J].Anticancer Drugs,2014,25(1):72-81.
[21]You BR,Park WH.Zebularine-induced apoptosis in Calu-6 lung cancer cells is influenced by ROS and GSH level changes[J].Tumour Biol,2013,34(2):1145-1153.
[22]Yang X,Lay F,Han H,etal.Targeting DNA methylation for epigenetic therapy[J].Trends Pharmacol Sci,2010,31(11):536-546.
[23]Asgatay S,Champion C,Marloie G,etal.Synthesis and evaluation of analogues of N-phthaloyl-l-tryptophan (RG108) as inhibitors of DNA methyltransferase 1[J].J Med Chem,2014,57(2):421-434.
[24]Castellano S,Kuck D,Sala M.etal.Constrained analogues of procaine as novel small molecule inhibitors of DNA methyltrans-ferase-1[J].J Med Chem,2008,51(7):2321-2325.
[25]Jain S,Wojdacz TK,Su YH.etal.Challenges for the application of DNA methylation biomarkers in molecular diagnostic testing for cancer[J].Expert Rev Mol Diagn,2013,13(3):283-294.
DNA Methylation Microarray Technology in Diagnosis and Treatment Malignant Tumor
WANGJuan,ZHANGYan-liang,DUANYong.
(DepartmentofClinicalLaboratory,theFirstAffiliatedHospitalofKunmingMedicalUniversity,Kunming650032,China)
Abstract:Epigenetic changes generally exist in various malignant tumors.DNA methylation is one of the main forms of epigenetic changes.Change of DNA methylation can significantly affect expression of gene.Antioncogenes hypermethylation and oncogene hypomethylation may be the important mechanism for the occurrence and development of malignant tumor.Therefore,it is significant that detecting the changes of malignant tumor DNA methylation status can reveal the pathogenesis of malignant tumor.In recent years,DNA methylation detection technology especially DNA methylation microarray technology has been widely used in the study of the pathogenesis,clinical diagnosis and treatment of malignant tumor.
Key words:Methylation microarray technology; Malignant tumour; Demethylation drugs
收稿日期:2014-05-12修回日期:2014-08-22编辑:辛欣
基金项目:国家自然科学基金(81160292)
doi:10.3969/j.issn.1006-2084.2015.07.046
中图分类号:R733
文献标识码:A
文章编号:1006-2084(2015)07-1270-03

