长牡蛎(Crassostrea gigas)中ChIP-seq方法的建立*
展 望 王 文 施威扬
(同济大学生命科学与技术学院 上海 200092)
长牡蛎又称太平洋牡蛎,是我国最重要的海水养殖对象之一,也是世界主要海水养殖贝类。培育高质量、高产的新品种一直是牡蛎养殖业的重要目标,这使得牡蛎成为了研究最为深入的贝类之一(Qin et al,2012)。长牡蛎全基因组测序的完成(Zhang et al,2012),为解密其遗传信息打开了大门,并使其成为最主要的海洋贝类模式动物。通过对长牡蛎全基因组数据的分析,已经初步揭示了一些其基因组的重要特性(Xu et al,2014;Zhou et al,2014;Li et al,2015;She et al,2015;Zhang et al,2015),但是更细致的组学功能研究还有待深入开展。本文通过在牡蛎中建立完整的染色质免疫共沉淀-测序技术(ChIP-seq),在全基因组水平上获得了组蛋白修饰分布的信息,并结合后期生物信息学分析,初步阐释了牡蛎表观遗传修饰的特点。同时,牡蛎中完整的 ChIP技术的建立也可以更好地应用于转录因子或其它表观遗传修饰信息对发育的调控的研究,从而为改良育种提供高效和正确的遗传信息。
通过DNA测序快速并准确获得各种生物体的遗传信息对生命科学研究具有重大意义,而测序技术的进步极大推动了生物学的发展。测序技术最早可以追溯到20世纪50年代,Whitfeld等(1954)报道了用化学降解的方法测定多聚核糖核苷酸序列;Sanger等(1975,1977)发明的双脱氧核苷酸末端终止法和Gilbert等(Maxam et al,1977;Gilbert,1981)发明的化学降解法,标志着第一代测序技术的诞生。从此,DNA测序走向了大规模实用化的道路。而高通量测序技术(high-throughput sequencing)的出现,将生物学研究推向了新的高度。这些方法的应用和普及,使人们能够更加全面地深入了解各种生物学过程,以及如何在外界因素的影响下发生相应的动态变化(Pepke et al,2009)。
目前染色质免疫共沉淀技术(chromatin immunoprecipitation,ChIP)与测序(sequencing)相结合的ChIP-seq技术(Johnson et al,2007)是在基因组层次研究体内蛋白质与 DNA相互作用的有力工具,ChIP-seq技术能够高效地在全基因组范围内检测与组蛋白、转录因子等互作的DNA区段(Mikkelsen et al,2007;Robertson et al,2007)。ChIP-Seq 的原理是: 利用目的蛋白特异性的抗体通过免疫共沉淀对与目的蛋白结合的 DNA片段进行特异性的富集,然后进行下一步的 DNA纯化与测序文库构建(Park,2009;Chen et al,2012),最后进行高通量测序。随后利用生物信息学软件将获得的数百万条序列定位到基因组上,在全基因组范围内鉴别DNA结合蛋白结合位点、组蛋白修饰、核小体定位等信息。ChIP-seq技术可以应用到任何基因组序列已知的物种,通过测序完整揭示免疫沉淀富集的DNA片段包含的蛋白定位信息(Schuster,2008)。近些年来,ChIP-seq技术已经发展成为研究转录因子结合位点和组蛋白修饰的最有力的实验手段,而基于多种高通量测序平台的 ChIP-seq在基因调控网络研究中也发挥着重要作用。
1 材料与方法
1.1 实验材料
长牡蛎(Crassostrea gigas)取自山东省青岛市胶南海水养殖场;挑选性腺饱满的一龄贝,镜检雄贝精子活力旺盛,雌贝卵子呈现梨形或者圆形;空运至上海后,随即放置在安装有增氧泵、水循环过滤系统以及水温控制冷却系统的人工海水水族箱内;人工海水盐度约28—30,水温恒定在16°C。
挑选出雌性和雄性牡蛎若干,解剖分离出精子和卵子,分别用海水漂洗 5—6次,获得干净的卵细胞和精子。受精前,精子需要在海水中活化10min左右,卵细胞活化约 1h。向活化的卵细胞中添加精子,精子密度为5—6个精子包围1个卵子为宜。在水温25°C,装有氧装置的培养箱中培养,保持搅拌(刘鹏超等,2011)。加入精子后30min用500目滤网滤去精子,更换新鲜海水。显微镜观察胚胎发育同步性,对于在 4细胞期发育同步性好的样品在培养箱中继续培养,收集16细胞期(约受精后3h 10min,25°C)胚胎样品;1000r/min离心5min,用2.9×室温PBS重悬后加入终浓度为 1%的甲醛室温下固定 10min。加入终浓度为 0.125mol/L的甘氨酸终止反应 5min。1000r/min 4°C 离心 5min,弃上清;用 2.9×预冷的PBS重悬,再次离心后弃上清;用冷的2.9×PBS重悬后,分装至EP管中;3000r/min 4°C离心1min,弃上清,液氮速冻,–80°C 冰箱保存。
1.2 实验方法
1.2.1 Western-Blot验证 H3K4me3抗体 收集精子、16细胞期胚胎和闭壳肌组织,加入适当体积的RIPA细胞裂解液(10mmol Tris-HCl,1%脱氧胆酸钠,l% NP-40,150mmol NaCl,0.1% SDS,0.2mmol PMSF)裂解细胞,冰上裂解30min后,超声破碎细胞。最后,将裂解复合物 12000×g离心 20min,吸取上清,弃沉淀,–80°C冻存备用。含总蛋白为20µg的样品溶液加入5×上样缓冲液,100°C沸水加热5min。热变性后的样品进行浓度为 12%的 SDS-PAGE电泳。湿法转膜(PVDF膜,0.22µm,Millipore)100mA恒流转膜2h。5%脱脂奶粉液室温封闭1h后,将滤膜与由5%脱脂乳粉液稀释 1000倍的第一抗体(rabbit-anti-H3K4me3,Millpore,07-473)4°C孵育过夜,然后用TBST(50mmol Tris,150mmol NaCl,0.05% Tween 20)漂洗 3次,每次10min。用1 : 5000稀释的辣根过氧化物酶标记的免疫球蛋白(Santa cruz)与滤膜温育,在摇床上摇动孵育1h。然后用TBST漂洗3次,每次10min。用ECL显色法(碧云天超敏ECL化学发光试剂盒)进行显色。
1.2.2 胚胎的染色质免疫共沉淀(ChIP) 取湿重约1g的固定的胚胎用膜裂解液(20mmol Tris,85mmol KCl,0.5% NP-40,0.5%脱氧胆酸钠)裂解,高速离心收集细胞核。用800µL微球菌核酸酶缓冲液(10mmol Tris-HCl,15mmol NaCl,60mmol KCl,2mmol CaCl2,0.15mmol精胺,0.5mmol亚精胺)重悬;加入1000U的微球菌核酸酶(micrococcal nuclease,NEB)37°C消化10min;期间每隔2min轻弹混匀一次;加入终浓度为10mmol的EDTA和终浓度0.1% SDS终止反应,置于冰上冷却;用Bioruptor进行超声;能量H;间隔30s,工作 30s,时间 6—7min。14000r/min 4°C离心 15min,收集上清。用30kDa超滤管(Amicon Ultra-0.5)将上清浓缩至体积约 100—200µL;取浓缩液体积的 1%作为input,存放–80°C;其余部分加入 800µL IP dilution buffer (2mmol EDTA,150mmol NaCl,20mmol Tris-HCl);加入约5μg ChIP抗体(rabbit-anti-H3K4me3,Millipore,07-473)和 Dynabeads Protein A/Protein G混合均匀,在4°C孵育过夜。次日分别TSⅠ液(0.1% SDS,1% TritonX-100,2mmol EDTA,20mmol Tris-HCl,150mmol NaCl)、TSⅡ液(0.1% SDS,1% TritonX-100,2mmol EDTA,20mmol Tris-HCl,500mmol NaCl)、BufferⅢ液(0.25mol/L LiCl,1% NP-40,1%脱氧胆酸钠,1mmol EDTA,10mmol Tris-HCl)、TE各洗 3次,每次5min;洗涤完成后弃上清;加入IP洗脱液(1% SDS,0.1mol/L NaHCO3)200µL,放置在 65°C 金属浴振荡1h;加入RNase A去除 RNA;加入蛋白酶K、NaCl置于65°C解交联过夜。用酚氯仿法抽提-乙醇沉淀纯化DNA,TE溶解DNA后随即构建文库。
1.2.3 构建文库 取 input DNA(1μg)和 ChIP DNA,按照NEBNext DNA超快速文库制备试剂盒(E7370S)的说明书指示制备测序文库。文库构建成功后送往华大基因公司进行PE 50双端测序。
1.2.4 数据分析 使用 bowtie(version 1.0.1)将测序读长回贴到牡蛎的基因组(从 http: //oysterdb.cn/下载)上,参数设置为比对时种子长度为 28bp,最多允许2个错配的默认值。对回贴所得的结果BAM文件用 MACS(1.4.2 20120305)以默认参数进行处理。对MACS所得结果使用CEAS (Package Version 1.0.2)进行处理,所用的Gene信息为从http: //oysterdb.cn/下载后转换格式所得。
2 实验结果
2.1 抗体验证
ChIP实验的成功首先需要能够在所实验的物种中工作,且特异性较好的抗体;但由于牡蛎是一个比较新的模式动物,基本上所有商品化的抗体都没有在牡蛎中验证抗体有效性的资料;为了验证本实验所用抗体的有效性,作者收集了牡蛎精子、胚胎(16细胞)以及闭壳肌三种样品,采用 Western-Blot的方法验证所用的H3K4me3抗体在牡蛎中工作效果。图1中显示在精子、胚胎和肌肉中都能检测到信号,且条带较为特异,没有其它干扰条带;但从图1中可以发现在精子中 H3K4me3的信号较弱,暗示精子中可能缺少组蛋白的修饰。因为H3K4甲基化通常被认为是基因的活化信号,如 H3K4me3在转录起始位点的富集通常表明着基因的转录激活(Li et al,2007)。综合三种样品中的实验结果表明本研究所选的抗体能够应用于牡蛎中,而且识别 H3K4me3比较特异,可以用于下一步的ChIP实验。
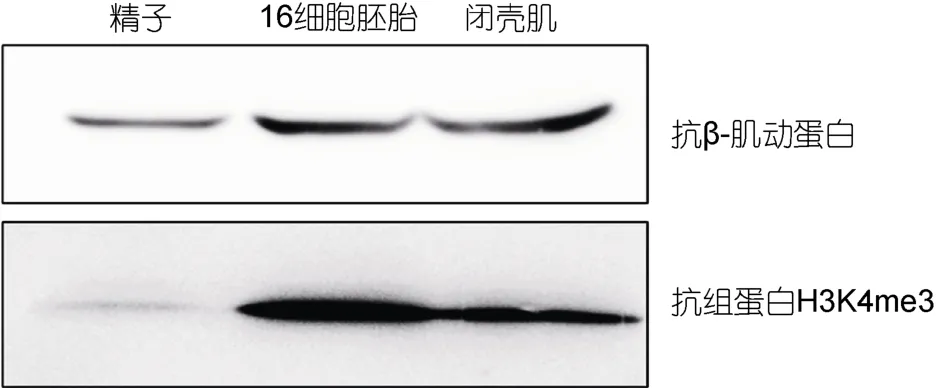
图1 牡蛎中H3K4me3抗体验证Fig.1 Verification of anti-H3K4me3 antibody in the oyster
2.2 ChIP-seq文库构建结果
早期胚胎细胞分裂活动旺盛,染色质较为疏松,而且含有大量的细胞质,若使用传统的超声方法制备染色质,裂解液中会溶解大量的细胞质蛋白,对后一步免疫沉淀过程产生影响。因此,针对早期胚胎的结构特点,作者利用微球菌核酸酶(Micrococcal Nuclease)将染色质酶切成单核小体。
作者前期摸索了不同时间梯度下酶切的效果,以加入酶为零点,分别在0、5、10、20、30和40min取等量的反应液,经过解交联纯化 DNA后电泳;如图2a显示,随着酶切时间的延长,所获得的核小体从起始点呈现阶梯状分布逐渐到 40min时候大部分可见的单核小体(150bp)状态;酶切40min后,绝大部分染色质已经变成单核小体甚至更小的片段;根据上述条件,最终确定酶切 10min使得染色质绝大部分是单核小体(150bp),但双核小体(300bp)依然可见(图2b 所示)。
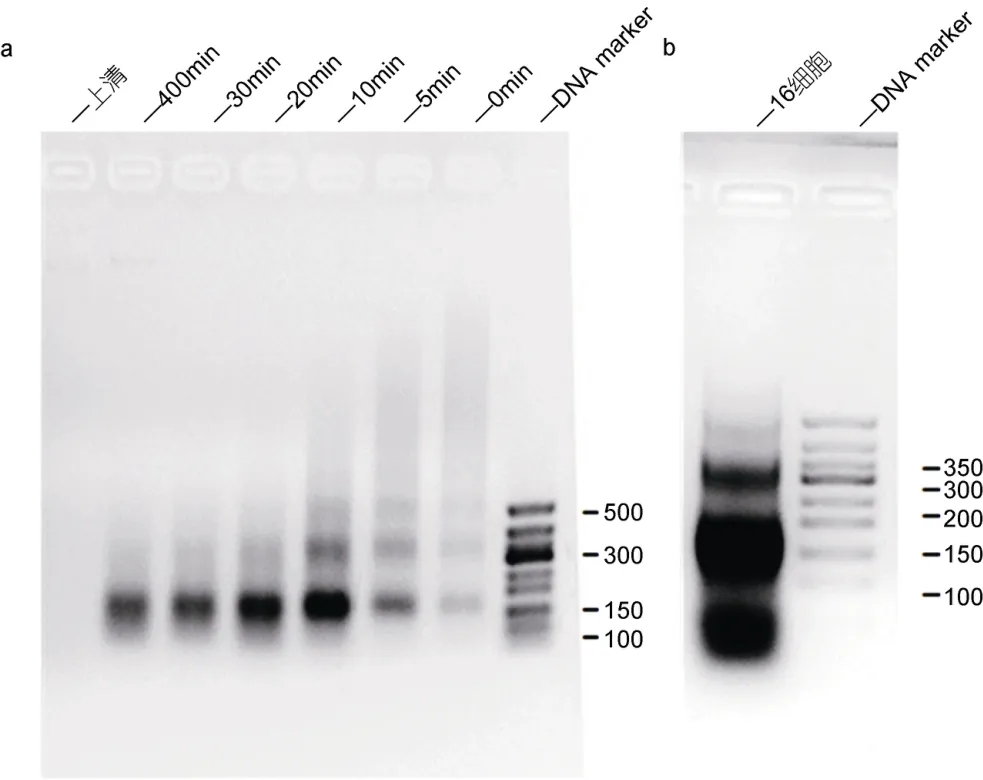
图2 不同时间点酶切效果检测Fig.2 The effect of enzyme digestion in time series
随后将按照上述条件制备的DNA-组蛋白复合物经ChIP后纯化DNA,使用NEBNext DNA超快速文库制备试剂盒构建文库。通常来讲,ChIP-seq对样品的要求是DNA总量大于10ng以上,片段主峰分布在100—500bp之间。使用Agilent 2100检测构建成功的测序文库的实际分布和浓度(图3);其中主峰片段大小为279bp,浓度为17.5 nmol/L,符合上机测序要求;将样本交于华大基因公司测序,测序完成后返回原始数据,进行数据的处理。
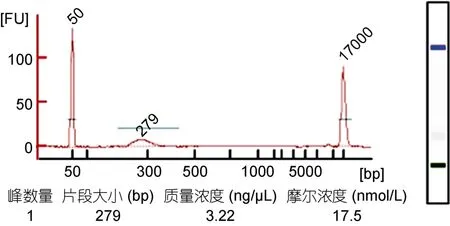
图3 使用Agilent 2100分析ChIP-seq文库结果Fig.3 Agilent 2100 analysis of ChIP-seq library using MNase digestion
2.3 ChIP-seq结果分析
为了进一步检验按照本文描述的方法所构建的文库,将所测的数据首先利用数据质控软件 FastQC进行测序数据的质量控制;FastQC通过分析原始数据的每一个碱基来显示整体样品的测序质量。图4中选取展示了 FastQC结果中的 4条结果,a图中全部reads的评分均在30以上,说明测序结果偏差较小;b图中统计了全部reads的CG分布比较均一;仅在5’端出现由于机器导致的偏差;c图中表明测序结果中表示测序结果中不能分辨的碱基N的比例很小;d图表明了测序结果中的重复序列出现的次数较低;结果显示利用本文的ChIP方法所获得的数据质量比较好,可以用于下一步的分析。
所得数据reads总数为17885715,reads双端比对到基因组上的数量为8258488,比对效率46.17%。这和其它物种中 ChIP-seq实验的一般比对效率(40%—70%)近似(Park,2009;Wei et al,2013)。接下来使用CEAS软件(Ji et al,2006)对ChIP-Seq数据的初步分析,图5a显示相对于全基因组,ChIP-seq实验组在基因组上富集于启动子区域,同时在转录起始位点(TSS)有一个峰的集中分布(图5b),从而可以确定 ChIP-Seq实验是成功的。后续工作开始对这些富集峰进行准确的注释,以了解更多信息。
3 讨论
目前染色质免疫共沉淀技术(ChIP)与测序(sequencing)相结合的 ChIP-seq技术是在基因组层次研究体内蛋白质与 DNA相互作用的有力工具,能够高效地在全基因组范围内检测与组蛋白、转录因子等互作的DNA区段。然而目前在牡蛎等贝类中尚未见相关报道。
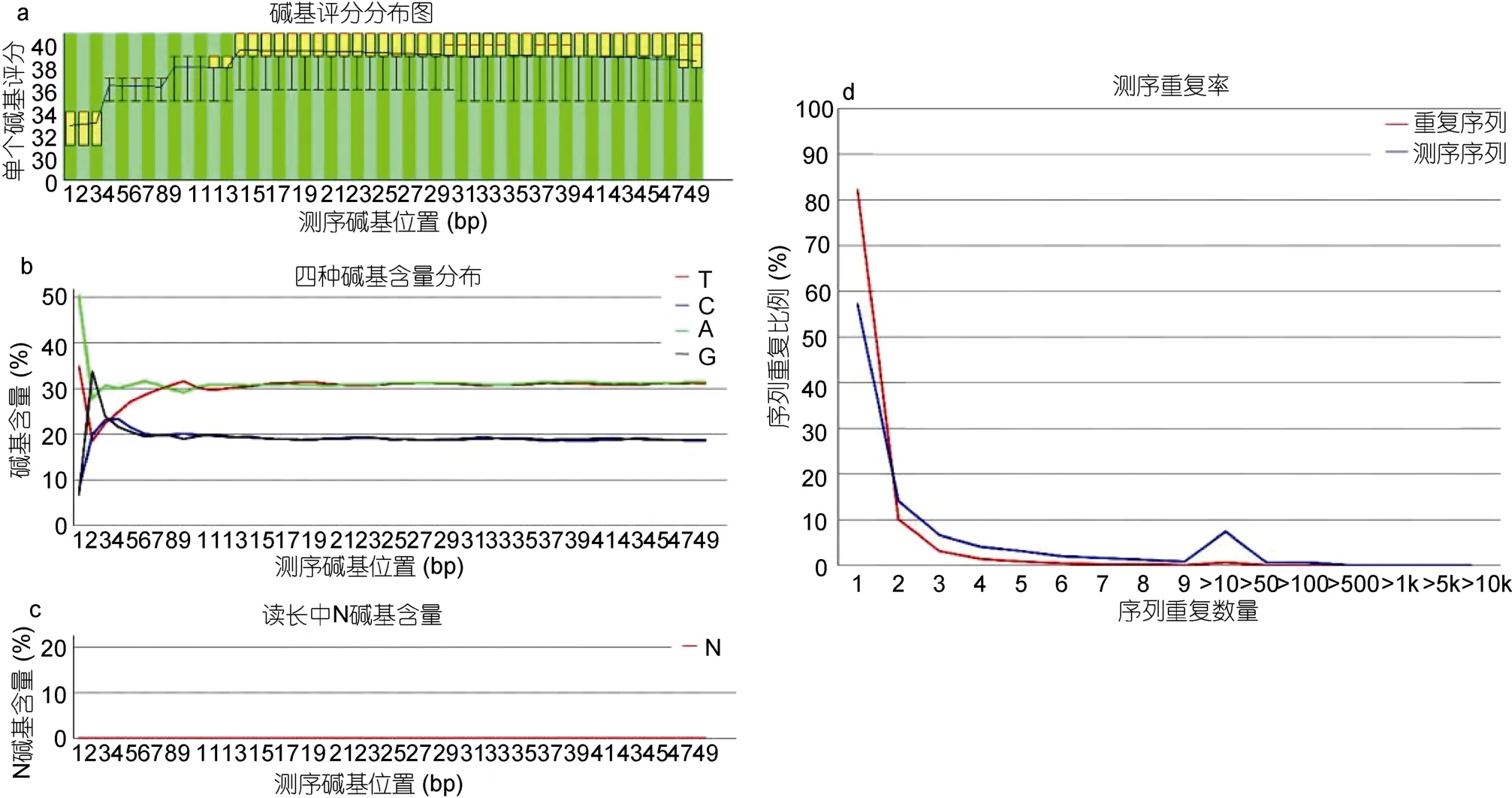
图4 FastQC质控结果Fig.4 Result of quality control with FastQC
进行ChIP实验的关键因素是特异性的抗体和高质量的染色质制备。作者尝试了可以广泛识别真核生物保守度 H3K4me3修饰的抗体并证实其可以用于ChIP。在染色质制备上面,尝试了超声和酶切消化两种方法。超声有重复性差、需要样品多等劣势,根据牡蛎胚胎的细胞性质,采用了微球菌核酸酶消化的方式制备了高质量的单核小体染色质。此外,为了获得发育同步性高度早期胚胎,通过缩短受精时间和洗除精子等方法,收集了发育同步性较高的早期胚胎;探索出了一套稳定性好、重复性高而且对于各个时期的胚胎都适宜的 ChIP方法;最后,将制备的ChIP样品在实验室自行建库,克服了商业化公司构建文库成本高、风险大等因素。测序结果说明在文库质量、基因组比对效率等方面都和模式生物中的ChIP-seq结果类似,说明作者建立的牡蛎ChIP-seq试验体系可以用于研究组蛋白的表观遗传修饰。本研究的结果显示与其它模式生物类似,牡蛎的 H3K4me3修饰主要分布在基因转录其实位点附近,具体的基因数据需要进一步的分析处理。这些信息可以为揭示牡蛎胚胎发育的表观遗传调控机制、解析基因调控网络提供巨大帮助。
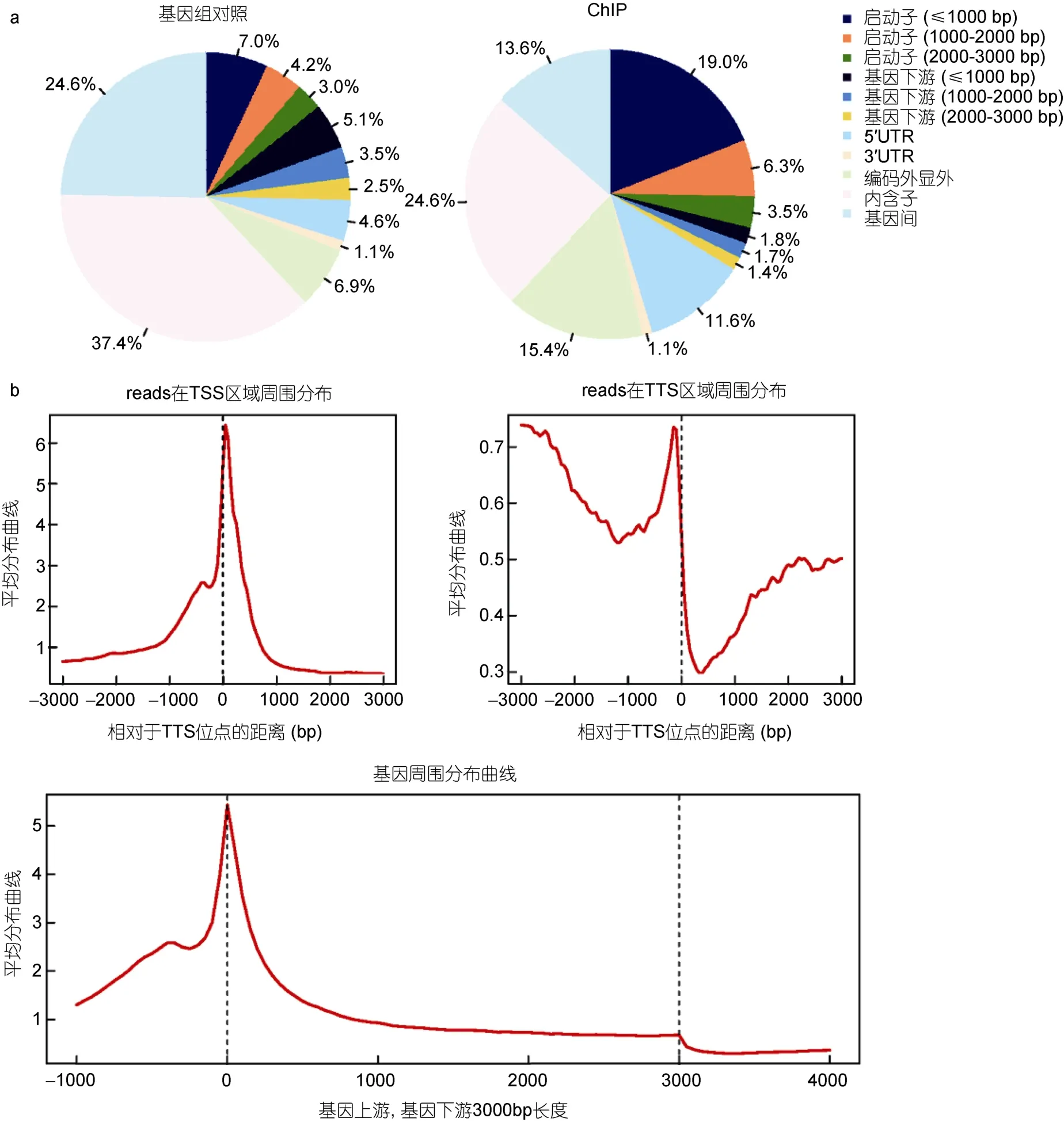
图5 CEAS分析结果Fig.5 Results of analysis with CEAS
4 结语
将ChIP应用于一种新的动物系统是很大的挑战,物种特异的实验步骤及实验方案的设计都少有前人工作可以参考。以牡蛎为样品的表观遗传方法的建立可以为牡蛎中的多种研究奠定方法学基础。本文的结果证实了在牡蛎中进行 ChIP实验的可行性,并且揭示了在牡蛎早期配套中存在着广泛的组蛋白H3k4me3的修饰,这一技术也可以更好地应用于研究转录因子或其它表观遗传修饰的信息,并为改良育种提供高效和正确的遗传信息。作为第一个完成基因组测序的经济贝类动物,对其进行多方面的组学研究,势必有助于了解海洋中种类最丰富、但研究甚少的海洋生物。
刘鹏超,阙华勇,张国范,2011.长牡蛎早期胚胎发育中脂肪酸和氨基酸组成变化的研究.海洋科学,35(10): 16—21
Chen Y W,Negre N,Li Q H et al,2012.Systematic evaluation of factors influencing ChIP-seq fidelity.Nat Methods,9(6):609—614
Gilbert W,1981.DNA sequencing and gene structure.Science,214(4527): 1305—1312
Ji X W,Li W,Song J et al,2006.CEAS: cis-regulatory element annotation system.Nucleic Acids Res,34(S2):W551—W554,http: //dx.doi.org/10.1093/nar/gkl322
Johnson D S,Mortazavi A,Myers R M et al,2007.Genome-wide mapping of in vivo protein-DNA interactions.Science,316(5830): 1497—1502
Li B,Carey M,Workman J L,2007.The role of chromatin during transcription.Cell,128(4): 707—719
Li H,Zhang H,Jiang S et al,2015.A single-CRD C-type lectin from oyster Crassostrea gigas mediates immune recognition and pathogen elimination with a potential role in the activation of complement system.Fish Shellfish Immunol,44(2): 566—575
Maxam A,Gilbert W,1977.A new method for sequencing DNA.Proc Natl Acad Sci U S A,74(2): 560—564
Mikkelsen T S,Ku M,Jaffe D B et al,2007.Genome-wide maps of chromatin state in pluripotent and lineage-committed cells.Nature,448(7153): 553—560
Park P J,2009.ChIP-seq: advantages and challenges of a maturing technology.Nat Rev Genet,10(10): 669—680
Pepke S,Wold B,Mortazavi A,2009.Computation for ChIP-seq and RNA-seq studies.Nat Methods,6(11S): S22—S32,doi:10.1038/nmeth.1371
Qin J,Huang Z X,Chen J et al,2012.Sequencing and de novo analysis of Crassostrea angulata (Fujian oyster)from 8 different developing phases using 454 GSFlx.PLoS One,7(8):e43653,http: //dx.doi.org/10.1371/journal.pone.0043653
Robertson G,Hirst M,Bainbridge M et al,2007.Genome-wide profiles of STAT1 DNA association using chromatin immunoprecipitation and massively parallel sequencing.Nat Methods,4(8): 651—657
Sanger F,Coulson A R,1975.A rapid method for determining sequences in DNA by primed synthesis with DNA polymerase.J Mol Biol,94(3): 441—448
Sanger F,Nicklen S,Coulson A R,1977.DNA sequencing with chain-terminating inhibitors.Proc Natl Acad Sci U S A,74(12): 5463—5467
Schuster S C,2008.Next-generation sequencing transforms today's biology.Nat Methods,5(1): 16—18
She Z C,Li L,Qi H G et al,2015.Candidate gene polymorphisms and their association with glycogen content in the pacific oyster Crassostrea gigas.PLoS One,10(5):e124401,http: //dx.doi.org/10.1371/journal.pone.0124401
Wei Y,Chen R,Dimicoli S et al,2013.Global H3K4me3 genome mapping reveals alterations of innate immunity signaling and overexpression of JMJD3 in human myelodysplastic syndrome CD34+ cells.Leukemia,27(11): 2177—2186
Whitfeld P R,1954.A method for the determination of nucleotide sequence in polyribonucleotides.Biochem J,58(3): 390—396
Xu F,Wang X T,Feng Y et al,2014.Identification of conserved and novel microRNAs in the Pacific oyster Crassostrea gigas by deep sequencing.PLoS One,9(8): e104371,http: //doi: 10.1371/journal.pone.0104371
Zhang G F,Fang X D,Guo X M et al,2012.The oyster genome reveals stress adaptation and complexity of shell formation.Nature,490(7418): 49—54
Zhang L L,Li L,Guo X M et al,2015.Massive expansion and functional divergence of innate immune genes in a protostome.Sci Rep,5: Article number: 8693
Zhou Z,Wang L L,Song L S et al,2014.The identification and characteristics of immune-related microRNAs in haemocytes of oyster Crassostrea gigas.PLoS One,9(2): e88397

