中国鲎基因组微卫星富集文库的构建及分析
宁晔凤 , 黎中宝 , 戴 刚 , 上官静波 , 李清荟
(1.集美大学 水产学院, 福建 厦门 361021; 2.福建省海洋渔业资源与生态环境重点实验室, 福建 厦门361021)
中国鲎(Tachypleus tridentatus)又名三刺鲎、小海鲎或东方鲎, 是大型海洋底栖动物, 其祖先可追溯至4亿多年前, 是动物界少数现存珍贵的“活化石”[1]。它隶属于节肢动物门(Arthropoda)、肢口纲(Merostomata)、剑尾目(Xiphosura)、鲎科(Limulidae)、鲎属(Tachypleus), 主要分布于中国长江口以南、日本九州岛以南、爪哇岛以北海域[2]。中国鲎具有极高的经济、药用及科学研究价值, 其血液制品鲎试剂是目前检测内毒素应用最广、最有效的试剂[3], 且其微妙的视神经系统使得它成为生理学和仿生学研究的重要模型[4]。然而近些年来, 栖息地生境的破坏、人类大规模滥捕滥杀等原因造成了中国鲎数量锐减, 野生资源几近枯竭[5], 加之其生长周期从卵细胞受精至成熟需要 13~15年, 中国鲎的保育工作迫在眉捷[6]。目前,福建、浙江、广东、海南及广西省已将中国鲎列为省级重点保护动物, 因此, 进行中国鲎资源保护及开展相关基础研究显得十分需要。
微卫星即为简单重复序列(simple sequence repeat, SSR)又称为短串联重复序列(Short Tandem Repeat, STR), 是由中央核心序列和两侧保守侧翼序列组成的, 核心序列为少数几个核苷酸(2~6个)组成的串联重复核苷酸序列。由于微卫星分子遗传标记技术所具有的易分离、共显性、多态性高、信息含量多、关联性好等优点[7], 使得其在物种遗传多样性分析研究上比其他DNA标记技术更适合, 并因此在濒危动物种群遗传学及保护遗传学中得到越来越多的应用[8-9]。中国鲎作为重要的野生保护动物之一, 目前国内对中国鲎的研究多见于资源分布及保育方面[10-11],中国鲎微卫星标记分离方面的研究还有很大空缺,目前相关研究仅见于Li等[12]开发出7对多态微卫星引物。
本研究采用生物素标记的(GT)15、(CT)15混合探针杂交与磁珠富集法相结合从中国鲎基因组中构建微卫星富集文库, 通过克隆、测序、设计引物和PCR检测来获得多态性微卫星标记, 为进一步研究中国鲎群体遗传多样性, 种群遗传结构的分析奠定基础,且为其野生种质资源保护及评估提供科学依据。
1 材料与方法
1.1 实验材料
中国鲎样品采自福建省厦门市集美水产品市场,平均体长为35.6 cm。剪取样品附肢肌肉于95%乙醇中固定, 并于–20℃保存。
1.2 基因组 DNA 的提取与检测
基因组 DNA采用试剂盒提取法。具体操作步骤按照天根血液/细胞/组织基因组DNA提取试剂盒-离心柱型/DP304(天根生化科技有限公司, 北京)的说明书进行。经K5500超微紫外分光光度计测定所提基因组 DNA 的纯度和浓度后, 取 2 μL 基因组DNA 进行 1%琼脂糖凝胶小样量样品电泳检测其DNA完整性, –20℃保存备用。
1.3 酶切及接头连接
基因组DNA选用FastDigest Tru1I酶(Fermentas)进行快速酶切, 并选取400 bp~1 000 bp片段进行连接。等摩尔比混合两组寡核苷酸链 LinkerA(5'-GACGATGAGTCCTGAG-3')及LinkerB (5'-TACTCA GGACTCAT-3'), 95℃变性10 min, 37℃10 min, 冰置10 min, 形成终浓度为 10 μmol/L 的双链接头供连接体系使用。
建立30 μL的连接体系, 其中包括酶切产物20 μL,双链接头 5μL (10μmol/L), T4 连接酶(5 U/μL)1 μL,10×T4 buffer 4 μL, 于 22℃连接过夜。
1.4 微卫星文库的构建
1.4.1 杂交
将10 μL接头连接产物于95℃变性 10 min, 迅速置于冰上使双链分开。取变性后的连接产物与生物素标记的 Bio-(CT)15、Bio-(GT)15混合探针进行杂交反应, 反应体系为 50 μL , 其中包括 25 μL 已预热至55℃的缓冲液2×Hyb buffer, 10 μL 接头连接产物, 5μL Bio-(CT)15(10μmol/L), 5 μL Bio-(GT)15(10 μmol/L) 以及 5 μL ddH2O。61℃杂交 1 h, 放置 4℃备用。
1.4.2 磁珠富集纯化
1.4.2.1 平衡磁珠
取150 μL混匀后的磁珠(Promega, USA)原液于1.5 mL离心管。而后置于磁力架上吸附磁珠, 轻轻吸出上清盐保护液。用150 μL(等比例)0.5×SSC于室温下洗涤磁珠两遍, 最后加入150 μL 1×Hyb buffer重悬磁珠。
1.4.2.2 磁珠的富集
于已平衡好的重悬磁珠液中加入 50 μL杂交产物混匀, 室温下放置30 min, 期间每5 min用移液枪轻轻吹打混匀 1 次, 30 min 后将离心管置于磁力架上,吸去上清液。之后将离心管放置于磁力架上于室温下依次用 400 μL 洗脱液 I(2×SSC, 0.1% SDS)和400 μL 洗脱液Ⅱ(1×SSC, 0.1% SDS)各洗脱2次。最后再用已提前预热至 50℃的 洗脱液Ⅱ(1×SSC, 0.1%SDS)400 μL快速洗脱两次。即可基本将不含微卫星的以及与探针结合不牢固的片段去除干净。
1.4.2.3 捕获微卫星片段
洗脱完成后, 在洗脱液中加入200 μL TE混合均匀, 95℃温育 10 min后, 将释放出含有单链微卫星序列的片段, 迅速将其置于磁力架上吸取上清液转至新离心管中。
1.4.2.4 纯化富集微卫星片段
在上述制备好的含有微卫星片段的溶液中加入400 μL, –20 ℃ 预冷的无水乙醇, 并加入 22 μL NaAc/EDTA(pH8.0)螯合剂助沉, 冰置15 min后4℃, 12 000 r/min 离心 10 min, 弃上清。再加入 500 μL, –20℃预冷的 75%乙醇, 冰置 15 min后4℃, 12 000 r/min离心10 min, 弃上清, 室温风干样品。最后用25 μL TE溶液溶解微卫星DNA, 放置4℃过1~2 d待微卫星 DNA 充分溶解后方可使用。
1.5 产物扩增及纯化
建立 25 μL 扩增体系, 用 MseI primer(MseI adapterA)为引物对富集纯化所得微卫星 DNA 片段进行 PCR 扩增。扩增体系包括精制产物2 μL, 10×Taq Buffer(含 Mg2+) 4 μL, dNTPs(10mmol/L)0.4 μL, Taq聚合酶(5U/μL)0.2 μL, MseI primer 引物 1.4 μL(10 μmol/L), 17 μL ddH2O。PCR 扩增条件为: 95℃预变性 2 min, 32×(95℃ 30 s, 53℃ 30 s, 72℃ 40 s),最后 72℃, 20 min 完成扩增片段末端加poly A。用1%琼脂糖凝胶电泳检测扩增产物片段。选取片段400 bp~1 000 bp的产物, 用 GenCleanPCR回收试剂盒(上海捷瑞)纯化, 去除多余的dNTPs 和接头等杂质。
1.6 载体连接、富集文库的筛选及测序
取2 μL纯化产物与pMD19-T载体(Takara, Japan)于16℃连接3.5 h, 将连接产物转化至感受态大肠杆菌 DH5α中培养, 得到中国鲎微卫星基因组文库。挑选阳性单克隆菌接种于含有氨苄青霉素的 LB液体培养基中, 37℃振荡培养 3.5 h。随后用 M13F/R通用引物进行 PCR 扩增筛选检测, 用 1%琼脂糖凝胶电泳检测PCR扩增产物, 挑取片段为400 bp~1 000 bp的阳性克隆菌液送至上海美吉生物医药科技有限公司进行测序。
1.7 序列分析与引物设计
利用 NCBI载体查找工具 VecScreen 去除两侧的载体引物, 汇总所有输出序列后再利用微卫星查找软件SSRHunter 1.3 对重复序列进行查找。获得的微卫星序列按完美型, 非完美型和复合型人工细化分析。并用 Primer Premier version 5.0对所得的微卫星序列进行引物设计。
2 结果与分析
2.1 克隆与测序结果
经PCR检测筛选, 本研究共获得334个阳性克隆,从中随机选取196个片段大小为400 bp~1 000 bp的阳性克隆送出测序。图1为部分筛选结果的电泳图。
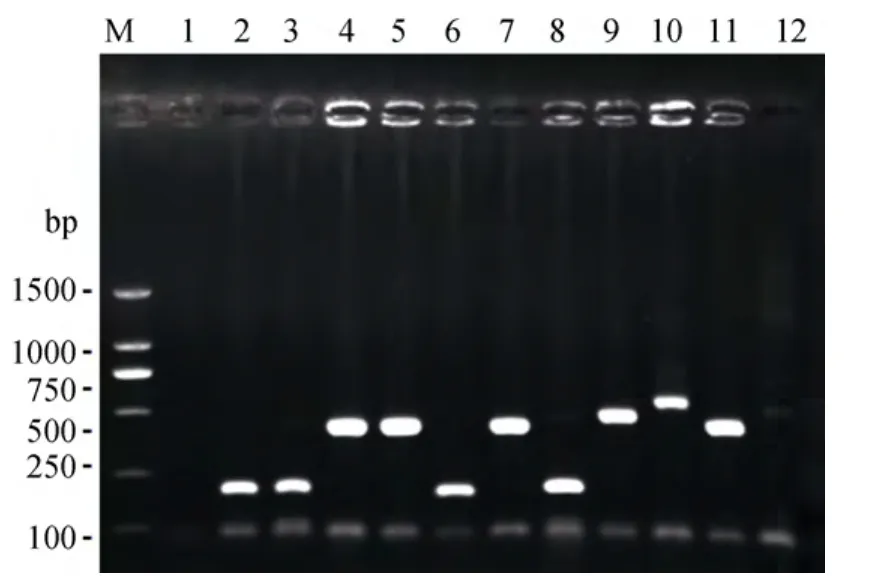
图1 单克隆检测结果Fig.1 Partial results of the positive monoclonal PCR detection
2.2 微卫星重复次数分析
在送出的 196个阳性克隆的测序过程中, 有 18条微卫星序列测序出现中断, 测序失败, 占所有克隆的9.08%。这部分的测序结果显示, 在单个克隆中有多处微卫星重复序列, 有的甚至高达 6 处, 并且重复次数通常大于30次, 复杂的碱基重复可能是导致测序过程出现信号衰减、测序峰图紊乱、测序中断的原因。图2为单向测序测通的中国鲎基因组中微卫星的特征序列部分峰图。
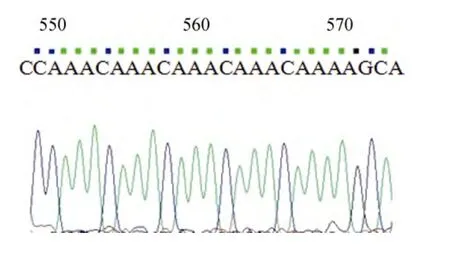
图2 中国鲎基因组中微卫星特征序列(CAAA)nFig.2 Diagram of (CAAA)n repeat sequence in Tachypleus tridentatus
本研究利用磁珠富集获得的中国鲎基因组微卫星最大重复次数为 98次, 大多集中在 7~35次, 占82.6%; 最高在5~10次, 为29.5%, 多数是二核苷酸碱基重复7次以上的微卫星序列; 50次以上的频率为10.2%(图3)。
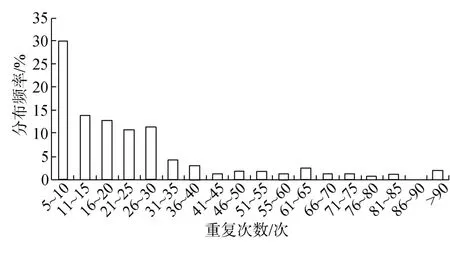
图3 中国鲎微卫星重复次数与分布频率Fig.3 Distribution frequency of microsatellite repeat times in Tachypleus tridentatus
2.3 微卫星序列结果及分类
利用SSR hunter 1.3软件对去除载体引物后的序列进行微卫星序列的查找, 使用的标准依据Linda[13]对微卫星的划定标准: 单碱基重复大于 14次, 双碱基重复大于7次, 三碱基重复大于5次, 四碱基及五碱基重复大于4次。结果显示, 在178个成功测序的阳性克隆中共获得 127个微卫星序列, 其中完美型占 69.3%; 非完美型占 11.0%; 复合型 占 19.7%(表1)。除探针使用的GT和CT的重复序列外, 还筛选到多碱基 GCT、TGG、AAAC、ACAA、GATTT、TTTTA 的重复序列。部分微卫星详细信息可见表2。
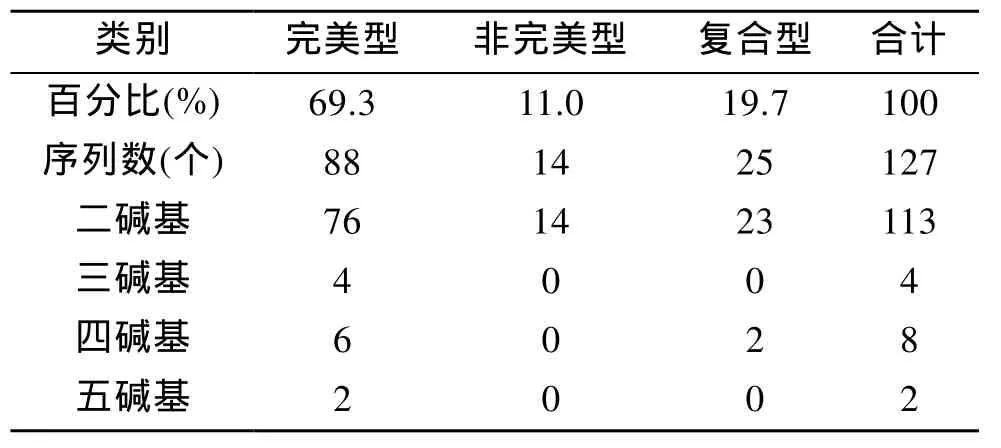
表1 中国鲎基因组微卫星序列分类情况Tab.1 Systematization of microsatellites in T.tridentatus
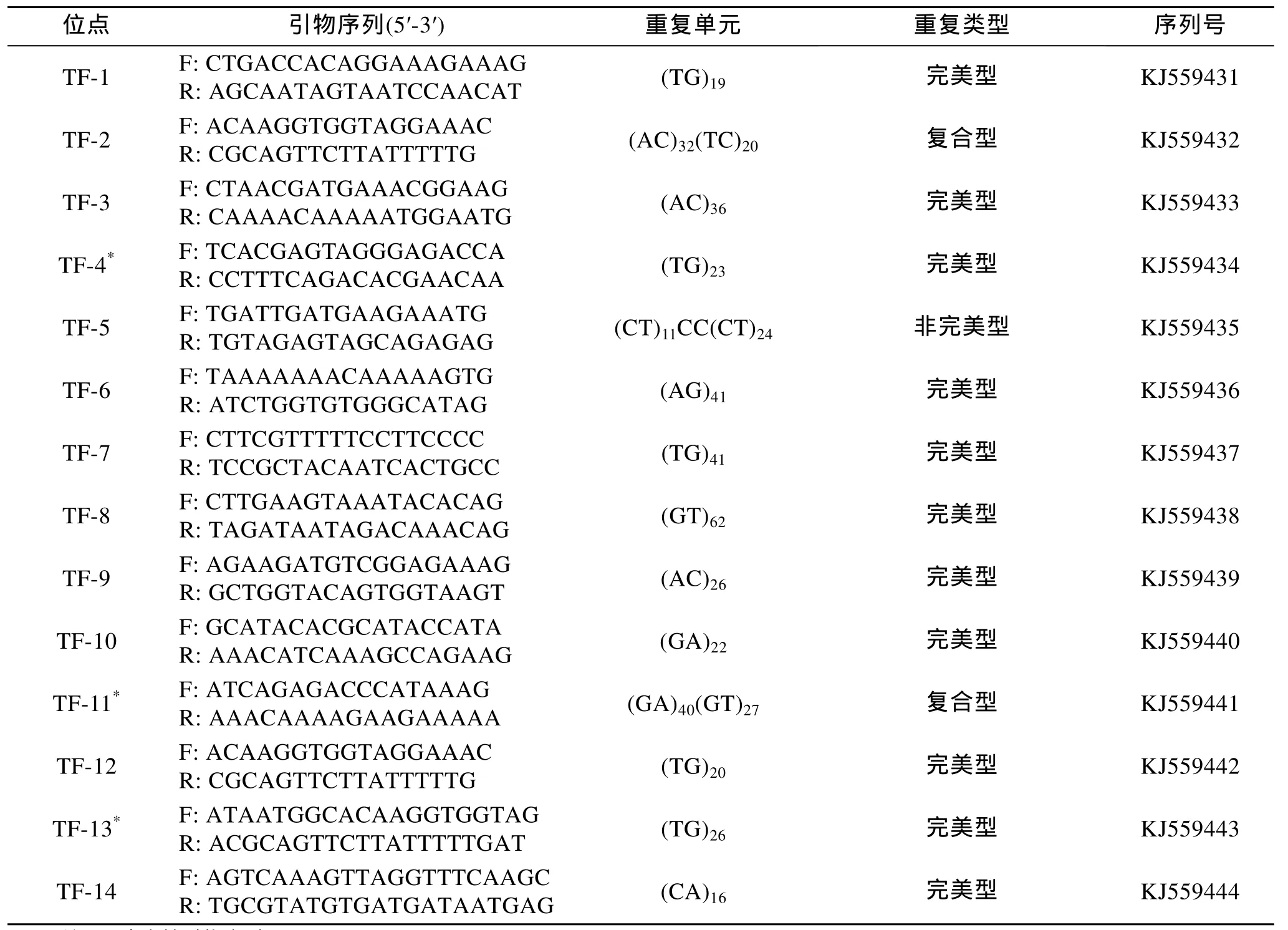
表2 部分微卫星位点引物及其他相关信息Tab.2 Relevant information of part of microsatellite primer sequences of T.tridentatus
2.4 引物设计及扩增结果分析
利用引物设计软件 Primer Premier version 5.0对所得到的序列设计引物, 在 127个微卫星序列中,有43个微卫星座位由于位于序列的末端或没有足够的侧翼序列无法设计, 在有侧翼的 84个序列中, 然后为选定的序列设计 PCR引物, 在 moderate stringency条件下[温度: 45.0~70.0℃, GC含量: 40%~60%; 引物长度: (21±4) bp; PCR产物大小: 100~330 bp], 有56个序列可设计出引物, 本研究选择合成40对, 其中有19对可扩增出清晰目的条带, 16对表现出单一条带, 3对表现出多态性。图4为TF-4位点扩增图谱。目的片段为237bp处, 显示出多态性。

图4 中国鲎TF-4位点扩增图谱Fig.4 Electrophoretogram of TF-4 locus in T.tridentatus
3 讨论与结论
3.1 酶切片段的大小及酶的种类
构建微卫星富集文库文库时, 使用的酶切片段大小应适宜, 过长的片段易导致亲和捕捉不到, 后续难以扩增, 影响测序结果, 增加实验成本; 过短的片段则很难保证微卫星侧翼序列长度, 影响引物的设计, 进而影响多态性引物的筛选。从便于提高实验有效性角度来看, 本实验选用400~1 000 bp大小的酶切片段是合理的。在选择限制性内切酶上, 本实验选用FastDigest Tru1I酶(Fermentas)是由于其酶切位点在动物基因组中分布广泛, 可以将基因组DNA均匀地切成大小不同的片段, 且反应速度很快, 大大缩减了实验用时, 既保证了实验准确性、微卫星位点随机性, 又保证了实验结果的高效性和真实性。
3.2 探针的选择
在所有动物基因组中(CA)n或(GT)n微卫星的含量最为丰富, 其次是(CT)n[14]。本研究选用(GT)n为探针进行富集和杂交。为了提高微卫星的富集效率,Edwards[15]等提出用多种探针来筛选文库被广泛应用。Stamati[16]等利用(CA)15、(GA)8、(AAG)8和(ATG)8为探针构建了Salix lanata基因组文库, 李齐发等[17]选择(CA)12、(CCG)8、(CAG)8和(TTTC)8混合探针富集牦牛微卫星, 均证实了应用多种探针混合进行杂交分离微卫星片段是增加微卫星种类的理想选择。因此本研究在(GT)15的基础上又采用了(CT)15探针,以期获得更多的微卫星标记位点。此外, 探针选用时碱基数要适中, 探针碱基对的增加会直接影响到杂交的效率, 碱基对越多越难杂交。所以在以后的研究中, 根据研究对象特点选用相应合适的混合探针,是成功获得更多微卫星种类的必要条件。
3.3 微卫星序列特征及扩增结果分析
本研究中获得的微卫星序列中, 完美型微卫星占69.3%、不完美型占11.0%、复合的占19.7% 其中完美型微卫星比例最高, 此结果与日本盘鲍(Paralichthys olivaceus)[18]、细角螺(Hemifusus ternatanus)[19]、中国对虾(Fenneropenaeus chinensis)[20]、剑尾鱼(Xiphophorus helleri)[21]、海湾扇贝(Argopecten irradians)[22]以及大珠母贝(Pinctada maxima)[23]等水产动物的微卫星序列特点基本相符。微卫星核心序列相对较高的突变率, 是微卫星多态性的基础。根据Weber等[24]的观点, 重复次数高的微卫星在种群中表现出的多态性高。本研究所得序列重复次数多集中在7~35, 占82.6%, 最高达到98次。但是高重复性序列往往周围侧翼序列不足, 或是1个克隆中出现多处微卫星序列, 复杂的碱基重复, 导致测序峰图紊乱以至于引物设计困难。
本实验研究显示, 合成的引物 40对经扩增后,有19对可扩增出清晰目的条带。其中有16对表现出单一条带, 3对具有多态性。其余的因存在无扩增产物, 扩增产物与目的条带长度不一致, 扩增图谱个体缺失严重等导致无法判定。出现这些情况的原因可能是由于某些引物序列特异性不好, 无法扩增出产物, 某些引物与基因组中序列错配, 导致扩增产物长度改变, 或是有些个体基因突变, 无法与引物结合。本实验筛选出的微卫星引物可为中国鲎遗传多样性分析的进一步研究奠定基础, 为以后中国鲎的遗传育种、种苗放流等提供基础理论依据, 这对于后期实现其种质资源保护及科学的管理具有重要意义。
[1] 翁朝红, 谢仰杰, 洪水根, 鲎血淋巴系统的特点及其功能[J].集美大学学报(自然科学版)[J].2003, 8(1): 16-21.
[2] Sekiguchi K.Biology of horseshoe crabs[M].Tokyo:Science House CO., Ltd.1988: 1-9.
[3] Swan B L.A unique medial product (LAL) from the horseshoe crab and monitoring the Delaware Bay horseshore crab population.In: Limulus in the limelight[M].New York: Kluwer Academic/Plenum Publishers, 2001: 53-62.
[4] 洪水根.中国鲎生物学研究[M].厦门: 厦门大学出版社, 2011: 10-11.
[5] 黄勤, 林能锋, 陈英禄, 等.平潭中国鲎保护区规划建议[J].福建环境, 2003, 20(4): 35-38.
[6] 李琼珍.中国鲎保育工作研究进展[J].生物学杂志,2010, 27(4): 71.
[7] 王伟, 尤峰, 高天翔, 等.鱼类微卫星标记的研究进展[J].海洋科学, 2006, 30(10): 81-86.
[8] Cao Y Y, Li Z B, Li Q H, et al.Development and characterization of microsatellite loci forFenneropenaeus penicillatusAlock[J].African Journal of Biotechnology,2012, 11(48): 10831-10833.
[9] Dai G, Li Y Y, Li Z B, et al.Isolation and characterization of twelve novel polymorphic microsatellite loci in theBranchiostoma belcheriGray (Amphioxus)[J].Conservation Genet Resource, 2013, 5(4): 1115-1116.
[10] 廖永岩, 李晓梅.中国海域鲎资源现状及保护策略[J].资源科学, 2001, 23(2): 53-57.
[11] 翁朝红, 肖志群, 谢仰杰, 等.创建厦门海域中国鲎自然保护区[J].集美大学学报(自然科学版), 2008,13(1): 40-44.
[12] Li Q Z, Li Q, Liu J, et al.Isolation and characterization of microsatellite loci in the horseshoe crab (Tachypleus tridentatus)[J].Conservation Genet Resource, 2009, 10:1879-1881.
[13] Linda C, Luke R, Dan M, et al.Computational and experimental characterization of physically clustered simple sequence repeats in plants [J].Genetics, 2000,156(2): 847-854.
[14] Brenner S, Elgar G, Sandford R, et al.Characterization of the pufferfish(Fugu)genome as a compact model vertebrate genome[J].Nature, 1993, 366: 265-268.
[15] Edwards K J, Baker J H, Daly A, et al.Microsatellite libraries enriched for several microsatellite sequences in plants[J].Biotechniques, 1996, 20: 758-760.
[16] Stamati K, Blackie S, Brown J W S, et al.A set of polymorphic SSR loci for subarctic williow(Salix Lanata,S.lappommandS.herbacea)[J].Molecular Ecology Notes, 2003, 2: 280-282.
[17] 李齐发, 赵兴波, 罗晓林, 等.牦牛基因组微卫星富集文库的构建与分析[J].遗传学报, 2004, 31(5): 489-494.
[18] Sekino M, Hara M.Isolation and characterization microsatellite DNA loci in Japan flounderParalichthys olivaceus[J].Molecular Ecology, 2000, 9: 2201-2203.
[19] 李清荟, 陈晓姣, 黎中宝, 等.细角螺微卫星 DNA富集文库构建及特征分析[J].海洋科学, 2013, 37(4): 1.
[20] 徐鹏, 周岭华, 相建海, 中国对虾微卫星 DNA 的筛选[J].海洋与湖沼, 2001, 32(3): 225-259.
[21] 李霞, 白俊杰, 吴淑勤, 等.剑尾鱼微卫星 DNA的筛选[J].中国水产科学, 2004, 11(3): 196-201.
[22] 李宏俊, 刘晓, 杜雪地, 等.海湾扇贝微卫星标记开发及其分离方式分析[J].海洋科学, 2009 , 33(12): 4-8.
[23] 柳明, 喻达辉, 黄桂菊.大珠母贝微卫星 DNA 标记的分离与筛选[J].海洋科学, 2010, 34(8): 1-5.
[24] Weber J L.Informativeness of human (dC-dA)n (dG-Dt)n ploymorphisms[J].Genomics, 1990, 7(3): 524-530.

