绿僵菌非核糖体肽合成酶的生物信息学分析
张 杰
病原真菌中非核糖体肽合成酶的产物由5~10个苯环或开环连接而成的环状四肽所构成的毒素类物质[1,2].非核糖体肽合成酶由腺苷酰化结构域、巯基化结构域和缩合结构域所构成的模块组成[3],在蓝绿藻和丝状真菌中产生的毒素具有宿主特异性[4-6].非核糖体肽是微生物代谢产物的衍生物[5],在植物、昆虫和哺乳动物中非核糖体肽是组蛋白脱乙酰基酶(HDACs)的抑制因子之一,然而,病原菌侵染宿主时抑制宿主HDACs致病和生物合成的机制目前尚不清楚[7],但有报道认为其为莽草酸途径的分支[8].
利用生物信息学技术对绿僵菌非核糖体肽合成酶的进化 、二级结构、三级结构、跨膜结构域及物理化学性质分析,可以为绿僵菌非核糖体肽合成酶生物学功能的进一步研究提供科学依据.
1 材料与方法
1.1 材料
以NCBI(http://www.ncbi.nlm.nih.gov/)数据库中的绿僵菌基因组数据为材料.
1.2 方法
1.2.1 序列的查找
通过NCBI数据库搜索非核糖体肽合成酶的蛋白质序列,再通过绿僵菌数据库NCBI/BLAST/tblastn搜索绿僵菌的核酸序列,进而获得非核糖体肽合成酶基因的全长序列.
1.2.2 序列的生物信息学分析
用DNAman软件对非核糖体肽合成酶蛋白序列运行clustal W,对亲水性、疏水性和跨膜结构进行分析;应用MEGA 5.0构建系统发育树;用The PSIPRED Protein Sequence Analysis Workbench和COILS on line分析二级结构;用NCBI blastp在线分析可能的结构域;用swissmodel(http://swissmodel.expasy.org/)数据库在线建模和三级结构分析[9];用SignalP 4.1 Server预测蛋白质序列的信号肽剪切位点;蛋白质结构域的预测用在线SMART(http://smart.embl-heidelberg.de/);利用KEGG数据库分析其所在的信号通路;利用ProtParam(http://web.expasy.org/cgi-bin/protparam/protparam)数据库在线预测分析其物理和化学性质.
2 结果与分析
2.1 非核糖体肽合成酶一级结构和结构域
在一级结构上对非核糖体肽合成酶(绿僵菌毒素合成酶、罗氏绿僵菌毒素合成酶、蛹虫草毒素合成酶、白僵菌毒素合成酶、丽蝇蛹集金小蜂毒素合成酶、牡蛎毒素合成酶、深裂眶锯雀鲷毒素合成酶、女王燕尾毒素合成酶和牛目朴丽鱼毒素合成酶)的氨基酸序列运行clustal W和MEGA5.0软件.由图1A可知绿僵菌非核糖体肽合成酶在一级结构上保守性极低,在不同的病原菌中只有一个共同的甘氨酸位点 (黑色、灰色和浅灰色域分别表示相同性高于90%,75%和50%).由图1B可知绿僵菌的非核糖体肽合成酶与冬虫夏草和半知菌类的虫生真菌(白僵菌)的毒素合成酶具有较高亲缘性,其次是昆虫类毒素合成酶,而与水生动物类的毒素合成酶亲缘关系较远.通过在线比对预测到该基因编码的蛋白具有酰基激活酶、乙酰辅酶A绑定位点、腺苷一磷酸绑定位点,见图1C.
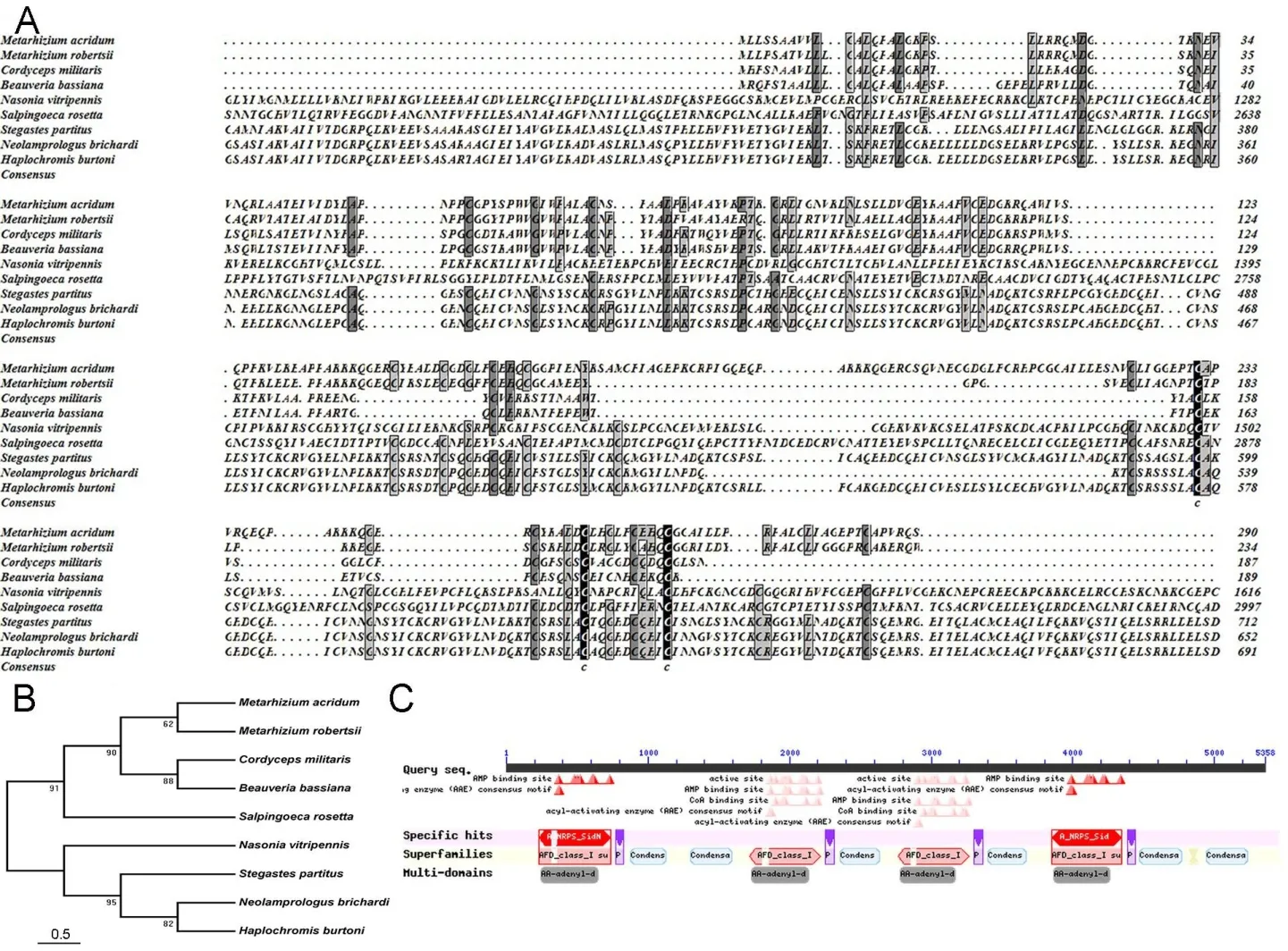
图1 (A)酰基毒素合成酶多序列比对,(B)非核糖体肽合成酶的系统进化树,(C)绿僵菌非核糖体肽合成酶的结构域
2.2 非核糖体肽合成酶二级结构预测
在一级结构的基础上用COILS在线分析了非核糖体肽合成酶的二级结构(见图2).由图2可知:非核糖体肽合成酶有15个α螺旋结构,在蛋白质中的含量为34.6%;9个β折叠,其含量为19%;无规则卷曲最多,其含量为46.4%.

图2 非核糖体肽合成酶的二级结构图
2.3 非核糖体肽合成酶亲水性、疏水性、跨膜结构和下游产物分子结构的预测
由图3A可知非核糖体肽合成酶最高亲水性分值为2.30,而最低分值为-2.12;由图3B可知非核糖体肽合成酶疏水性不显著,其疏水性最高分值为4.20,最低值为-3.01,总体亲水性为0.002(定义为序列中所有氨基酸亲水值的总和与氨基酸数量的比值,负值越大表示亲水性越好,正值越大表示疏水性越强),推测其可能是非水溶性蛋白.由图3C可知非核糖体肽合成酶具有三次跨膜结构.图3D是预测到的非核糖体肽合成酶合成物的分子结构,其由三个苯环构成.用SignalP 4.1 Server对其一级结构上的信号肽进行预测,由图3E可知该蛋白质无信号肽剪切位点.
2.4 非核糖体肽合成酶三维建模
由图4A和4B可知在SWISS-MODE数据库中搜索到了50个非核糖体肽合成酶的模板,其中4gr5.1.A模板与靶蛋白的相似性评估得分为26.32%,正常的QMEAN4分值为-4.52,靶蛋白与模型氨基酸的对应序列如图4C所示,其中第484位氨基酸可能由Val突变为ILe.蛋白建模序列的覆盖度如图4E所示,最有益建模的区域在Psi值-60度至180度区域,不可接受区域为0度,该结果表明该蛋白质的三维结构与上述预测结果相一致.

图3 (A)非核糖体肽合成酶亲水性分析,(B)非核糖体肽合成酶疏水性分析,(C)非核糖体肽合成酶跨膜结构分析,(D)非核糖体肽合成酶合成产物分子结构,(E)信号肽预测图
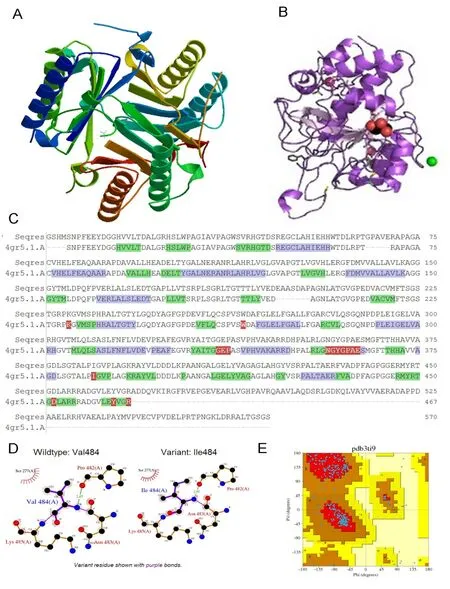
图4 (A)非核糖体肽合成酶三维结构,(B)非核糖体肽合成酶三维建模,(C)非核糖体肽合成酶三维建模序列,(D)非核糖体肽合成酶可变氨基酸,(E)非核糖体肽合成酶三维建模评分
2.5 非核糖体肽合成酶抗原性片段、物理和化学性质分析
用DNAman软件预测到该非核糖体肽合成酶含有24个抗原肽段,其分值和长度(下划线字母表示最大分值位点)分别如下:
[1]分值=1.266,长度=20,277-TNIVLVLIPVSFVLQSVGLS-296.[2]分值=1.210,长度=36,42RYLSLSHNCCDWVVFSVIPTGVPYAMKVLSRLLKLQ-77.[3]分值=1.167,长度=13,25-LGGVAPFLALLGE-37.[4]分值=1.155,长度=12,321-RKEVPPEFVLRL-332.[5]分值=1.150,长度=9,227-AKVVWVQRQ-235.[6]分值=1.149,长度=16,299-HWAVQTWQFIALVFMF-314.[7]分值=1.149,长度=10,356-DKAVHVIEHD-365.[8]分值=1.148,长度=21,195-FKRSLSLLYTAKSLTLPLKVR-215.[9]分值=1.134,长度=11,395-VDDVTAVVNKI-405.[10]分值=1.124,长度=10,265-ALSHHWCFES-274.[11]分值=1.119,长度=9,379-TVQKIVANT-387.[12]分值=1.114,长度=9,341-IQGILAGSP-349.[13]分值=1.111,长度=15,93-EKDLLSTCSKEICEI-107.[14]分值=1.107,长度=8,186-PNLLLNLT-193.[15]分值=1.104,长度=8,253-EAEVLSAH-260.[16]分值=1.103,长度=10,140-PPELLPSKIA-149.[17]分值=1.086,长度=8,127-IREYIYLP-134.[18]分值=1.082,长度=8,407-GGPSLLDE-414.[19]分值=1.070,长度=8,244-EAYILAKN-251.[20]分值=1.068,长度=6,171-PIGLGS-176.[21]分值=1.049,长度=7,113-VERVEKT-119.[22]分值=1.045,长度=6,162-PWLQPS-167.[23]分值=1.031,长度=6,13-SPSLTP-18.[24]分值=1.028,长度=7,85-YTSYRKA-91.
其物理化学性质用ProtParam在线预测得知该蛋白质的等电点为4.82,分子量为34 119.1 D,40个负电荷残基(Asp+Glu),26个正电荷残基(Arg+Lys),分子式为C1520H2424N404O461S12.假定所有的半胱氨酸形成二硫键,其在水中280 nm的消光系数为29 700 M-1cm1.其氮端的起始氨基酸为Met,预测到该蛋白质在哺乳动物网状细胞中,离体状态下的半衰期为30小时;在酵母活体中的半衰期为大于20小时;在细菌活体中的半衰期大于10小时.其不稳定性指数为30.19,属于稳定性蛋白质.其脂肪族指数为103.11.
3 讨论
利用生物信息学的方法对研究的目的基因进行正确的分析是现代分子生物学研究的有效途径之一.微生物的毒素类物质的研究起始于50年前[10],但是没有一种微生物具有全部毒素种类,而只是含有其中的一种毒素或类似物.非核糖体肽合成酶的同源序列比对及系统进化树分析的结果与Brosch G等的报道相一致[11],说明了生物信息学的预测结果是可靠的.同时用本地软件与在线对信号肽段预测结果相一致,说明在生物信息学分析中用多种软件互相印证结果的可靠性是必要的.非核糖体肽合成酶的二级结构和三级结构的预测与Stachelhaus T等报道的合成抗生素的酶类结构相一致[12].非核糖体肽合成酶的物理化学性质及抗原片段尚未见报道.总体来说,绿僵菌非核糖体肽合成酶结构及功能和微生物分泌的抗生素及类似物的合成酶类相似,其与植物病原菌分泌的毒素因子的异同尚需实验进一步证明,其物理化学性质也需进一步地验证.生物信息学与其他生物学技术相结合是生物学研究的首要途径之一.
参考文献:
[1]Evans B S,Robinson S J,Kelleher N L.Surveys of non-ribosomal peptide and polyketide assembly lines in fungi and prospects for their analysis in vitro and in vivo[J].Fungal Genetics and Biology,2011,48(1):49-61.
[2]Challis G L,Ravel J,Townsend C A.Predictive,structure-based model of amino acid recognition by nonribosomal peptide synthetase adenylation domains[J].Chemistry&Biology,2000,7(3):211-224.
[3]Wang B,Kang Q,Lu Y,et al.Unveiling the biosynthetic puzzle of destruxins in Metarhizium species[J].Proceedings of the national academy of sciences of the united states of america,2012,109(4):1287-1292.
[4]Heussner A H,Mazija L,Fastner J,et al.Toxin content and cytotoxicity of algal dietary supplements[J].Toxicology and Applied Pharmacology,2012,265(2):263-271.
[5]Fox E M,Howlett B J.Secondary metabolism:regulation and role in fungal biology[J].Current Opinion in Microbiology,2008,11(6):481-487.
[6]Cramer Jr.R A,Stajich J E,Yamanaka Y,et al.Phylogenomic analysis of non-ribosomal peptide synthetases in the genus Aspergillus[J].Gene,2006,383(15):24-32.
[7]Martín J F,Casqueiro J,Liras P.Secretion systems for secondary metabolites:how producer cells send out messages of intercellular communication[J].Current O-pinion in Microbiology,2005,8(3):282-293.
[8]Doekel S,Marahiel M A.Biosynthesis of natural products on modular peptide synthetases[J].Metabolic Engineering,2001,3(1):64-77.
[9]Wu B,Luo L,Li C,et al.Comprehensive bioinformation analysis of the MRNA profile of fascin knockdown in esophageal squamous cell carcinoma[J].Asian pacific journal of cancer prevention,2013,14(12):7221-7227.
[10]Waxman S,Corcino J,Herbert V.Drugs,toxins and dietary amino acids affecting vitamin B12 or folic acid absorption or utilization[J].The American Journal of Medicine,1970,48(5):599-608.
[11]Brosch G,Ramsom R,Lechner T,et al.Inhibition of maize histone deacetylases by hc toxin,the host-selective toxin of cochliobolus-carbonum[J].Plant cell,1995,7(11):1941-1950.
[12]Stachelhaus T,Schneider A,Marahiel M A.Engineered biosynthesis of peptide antibiotics[J].Biochemical Pharmacology,1996,52(2):177-186.

