γ-氨基丁酸受体基因的系统性调控网络
赵云燕,李 中,陈丹妮,雷清锋,何 露,韦 睿
1)中山大学附属第六医院神经科,广州510655;2)深圳大学光电工程学院,深圳518060

【生物工程 / Bioengineering】
γ-氨基丁酸受体基因的系统性调控网络
赵云燕1,李 中1,陈丹妮2,雷清锋1,何 露1,韦 睿1
1)中山大学附属第六医院神经科,广州510655;2)深圳大学光电工程学院,深圳518060
提出一种多学科交叉结合的策略,试图初步获得系统性调控γ-氨基丁酸(gamma-aminobutyric acid, GABA)受体基因的转录因子线索. 利用基于功能基因组学方法的DNA元件百科全书计划已发表的数据,系统性地获得GABA受体基因的开放染色质序列,并以此作为固相化探针,捕获与其特异性相互作用的蛋白质分子,并利用质谱分析鉴定蛋白. 结果发现,对不同脑区获得的核蛋白,探针都能捕获到同样的特异性条带,然而,作为对照的天门冬氨酸(N-methyl-D-aspartate, NMDA)受体基因相关的开放染色质探针,在不同脑区中并未检获上述特异信号,说明结合蛋白是特异的. 质谱分析表明,与GABA受体基因相关的开放染色质特异相互作用的蛋白是核膜血影重复蛋白-1(nuclear envelop spectrin repeatprotein-1, Nesprin-1),又称synaptic nuclear envelope-1(SYNE-1). 进一步的调控网络生物信息学分析表明,Nesprin-1可能与MAFA、IRX2、BCL6、CEBPA以及RP58等转录因子形成复合物,并与GABA受体基因GABRA5、GABRA6、GABBR1和GABBR2等共表达. 表明GABA受体基因在不同脑区是通过相同的转录调控机制进行表达的,Nesprin-1可能与MAFA、IRX2、BCL6、CEBPA以及RP58等转录因子形成复合物进而调控GABA受体基因表达,该特异的转录因子调控网络有望用于诱导多能干细胞或是前体细胞直接分化为GABA能神经元.
分子生物学;γ-氨基丁酸受体;开放性染色体;转录调控;转录因子;核膜血影重复蛋白
γ-氨基丁酸(gamma-aminobutyric acid, GABA) 能系统是哺乳动物中枢神经系统内最主要的抑制性神经递质系统. 在中枢神经系统中,大约有20%~50%的突触为GABA能突触. GABA受体作为该系统最重要的组成部分,参与了中枢神经系统内正常生理功能的维持[1]. 在神经、精神病理情况下都可以观察到GABA受体的分布或功能的异常,如癫痫、卒中、阿尔茨海默症、精神分裂症、抑郁和焦虑等[2]. GABA受体是临床药理上重要的药物靶标,应用于癫痫、焦虑、抑郁、精神分裂、强迫症、成瘾症以及疼痛等疾病[3]. 目前,多数的研究主要着眼于GABA受体的分子结构以及药理特性等方面,对于GABA受体基因表达调控机制方面却较少研究. GABA受体基因的正确表达有赖于精确的调控系统,从而使GABA受体在特定的部位表达特定的数量. GABA受体相关转录因子与转录因子结合部位的相互作用是这一系统的重要组成部分,不同转录因子对外界环境的各种刺激或不同发育阶段的各种信号做出反应,结合于转录调控元件,激活或抑制该基因的转录,从而调控GABA受体基因表达的部位以及水平. 目前仍未确定可精确调控GABA受体基因特异性表达的转录因子,因此,本研究将对GABA受体基因调控机制进行研究.

图1 实验设计及流程Fig.1 Design and flow chart of experiment
随着2012年DNA元件百科全书(encyclopedia of DNA elements, ENCODE)数据库的发布,彻底颠覆了“人类基因组中90%以上的序列都是垃圾DNA”的结论,使得基因组中“垃圾DNA”片段处于活性状态时所处染色质区域结构变得开放可及,形成对DNase I超敏感的开放染色质结构(open chromatin),直接反映了基因组上的活性功能区域,并可作为标志物来挖掘不同状态下细胞的基因或功能元件[4]. 基于功能基因组学数据,将由生物信息学预测得到的DNA序列通过最新的DNA偶联蛋白富集实验(DNA-pull down assay)探索其相互作用蛋白(图1). 相比传统的基因转录调控方法,如电泳迁移率实验(electrophoretic mobility shift assay,EMSA)和染色质免疫共沉淀测序(CHIP-chip/seq),DNA-pull down assay法更便捷可信,且具可重复性[5].
1 实验材料
1.1 主要试剂
HEPES、氯化钾、氯化镁、氯化钠、乙二胺四乙酸(ethylene diamine tetraacetic acid,EDTA)、苯甲基磺酰氟(phenylmethanesulfonyl fluoride,PMSF)、蛋白酶抑制剂混合剂(protease inhibitor cocktail,PIC)、聚脱氧肌苷-脱氧胞苷酸钠盐(poly (deoxyinosinic-deoxycytidylic) acid sodium salt,Poly dI dC)、TEMED和甘油为美国Sigma公司生产;二硫苏糖醇(dithiotreitol,DTT)由Invitrogen公司生产;二喹啉甲酸(bicinchoninic acid,BCA)蛋白浓度测定试剂盒、链亲和素琼脂糖树脂由Pierce公司生产;QIAamp®血液DNA提取试剂盒由QIAgen公司生产;pMD-T Vector 试剂盒由Takara公司生产;丙烯酰胺和甲叉双丙烯酰胺由Bio-Rad公司生产.
1.2 实验仪器
荧光/化学发光分析仪(酶标仪)由美国Bio-Tek公司生产;PCR仪由美国Thermo公司生产;凝胶扫描成像系统由美国Bio-Rad公司生产.
1.3 实验动物
野生型正常BALB/c小鼠3只,3月龄,雌性,由香港大学宋又强教授惠赠.
2 实验方法
2.1 生物信息学与基因组学预测分析
为发掘潜在的GABA受体基因转录控制因子,基于生物信息学与基因组学的最新进展,在最新的ENCODE数据库中进行数据挖掘,采取以下步骤进行分析:① 根据在神经元细胞中不同的组蛋白修饰情况确定核心启动区域;② 根据在神经组织细胞中open chromatin以及绝缘子的分布情况,在GABA受体基因转录起始位点周围确定最近端的调控区;③ 参考不同细胞系中在此区段上各种转录因子的结合情况(ChIP-seq数据)确定可能的转录因子结合区间;④ 作为对照组,运用上述方法对NMDA受体基因表达的转录因子进行预测分析.
2.2 小鼠不同脑区核蛋白提取
颈椎脱臼法处死小鼠后剪刀断头,用镊子掀开头盖骨后将脑移到盛有预冷PBS的培养皿(直径为60 mm)中. 剥离大脑膜,冲洗干净后在解剖镜下沿中线切开大脑,精细镊分离脑区:前额叶皮质、海马纹状体及小脑. 将解剖得到的小鼠不同脑区(前额叶皮质和小脑)各约0.5 g置于EP管中,洗去多余血迹后,每管加入500 μL胞浆蛋白裂解液(含50 μL 100 mmol/L HEPES(pH=7.9)、50 μL 100 mmol/L KCl、50 μL 10 mmol/L MgCl2、50 μL 1 mmol/L EDTA、5 μL 100 mmol/L DTT、50 μL 10 mmol/L PMSF、5 μL 100× PIC及240 μL双蒸水(distillation-distillation H2O, ddH2O)并充分磨碎组织. 将组织匀浆放置冰上并加入NP-40 裂解液50 μL,强烈震荡10 s后,使用胰岛素针上下抽吸组织匀浆20次. 4 000 r/min离心1 min后,弃上清,加入500 μL预冷PBS溶液,重悬,取3 μL混合物滴于载玻片上,在高倍显微镜下观察细胞是否膜裂解完全. 再以4 000 r/min离心1 min,弃上清后加入500 μL胞浆蛋白裂解液,重复3次. 重悬细胞核后加入100 μL核裂解液(含20 μL 100 mmol/L HEPES(pH=7.9)、10 μL 4.2 mol/L NaCl、10 μL 15 mmol/L MgCl2、20 μL 1 mmol/L EDTA、25 μL甘油、0.5 μL 100 mmol/L DTT、5 μL 10 mmol/L PMSF、1 μL 100× PIC及8.5 μL ddH2O),置于冰上30 min后高速(13 000 r/min)离心1 min. 将上清移至新的1.5 mL EP管后各取5 μL 上清用于BCA法蛋白浓度测定;剩余的核蛋白中加入体积分数为15%的甘油,混合充分,按10 μL/管分装,冻存于-80 ℃,避免反复冻融破坏核蛋白活性.
2.3 GABA和NMDA受体基因探针合成及生物素标记
使用Primer Premier 5.0软件对预测得到的转录因子结合区间DNA序列设计相应引物,分别在上游/下游引物5′端加入4个腺嘌呤脱氧核苷酸/胸腺嘧啶脱氧核苷酸,引物序列见表2,对PCR条件进行优化后(退火温度60 ℃,38个循环反应),将得到的DNA片段分别克隆进pMD20-T Vector,经由大肠杆菌DH5α表达后获得包含有DNA探针序列的质粒. 将7个GABA能探针质粒及5个NMDA能探针质粒分别等量混合,并以此作为模板,加入共同引物(pMD®20-T Vector_上游引物5′-TTTTTGGATCTACTAGTCATATGGA-3′,pMD®20-T Vector_下游引物5′-TTTTTCTCGGTACCCGGGGATCCGA-3′)在PCR扩增过程中(退火温度60 ℃,38个循环反应)进行Biotin-14-dATP标记,使得同种受体探针在同一体系中标记. 采用DNAmate核酸共沉淀剂试剂盒对上述PCR产物纯化,以便进行后续DNA pull-down实验.
2.4 DNA pull-down实验
将80 μL的链亲和素琼脂糖树脂与1 mL DNA-链亲和素琼脂糖树脂结合缓冲液(10 mmol/L Tris溶液、50 mmol/L KCl,pH=7.4)混匀后,3 000 r/min离心10 s(重复1次),弃上清,再加入100 μL DNA-链亲和素琼脂糖树脂结合缓冲液,重悬后平均分为2份,各3 000 r/min离心10 s(重复1次),然后弃上清,每管分别加入40 μL DNA-链亲和素琼脂糖树脂结合缓冲液重悬. 之后,将生物素标记并纯化后的GABA受体探针和NMDA受体探针各10 μL分别加入上述悬液中,55 ℃反应过夜. 取出上述两管过夜反应生物素探针-链亲和素琼脂糖树脂混合物3 000 r/min离心10 s(重复1次),弃上清,各加入80 μL 10 g/L BSA后冰浴30 min. 之后再次3 000 r/min离心10 s(重复1次),弃上清,分别加入200 μL结合缓冲液(8 mmol/L HEPES、100 mmol/L KCl、0.08 mmol/L EDTA、2 mmol/L MgCl2,pH=7.9)重悬后将每管悬液各均分为两管,3 000 r/min离心10s(重复1次)后弃上清,按照不同的组别分别配制100 μL反应体系(每个反应体系含20 μL PolydIdC、0.5 μL DTT、10 μL 500 mmol/L Protease Inhibitor Cocktail、8 μL Glycerol、6.6 μL核蛋白抽提物及54.9 μL ddH2O),之后4 ℃滚动反应过夜. 取出上述过夜反应混合物,3 000 r/min离心10 s(重复1次)后弃上清,各加入1 mL 1×结合缓冲液,重悬后3 000 r/min离心10 s (重复1次),弃上清,将上述洗涤过程重复2次.然后,用胰岛素针将残余于管内的缓冲液吸尽,再加入15 μL 3×loading buffer重悬. 100 ℃水浴15 min后,14 500 r/min离心5 min,将上清移至新的1.5 mL管内,进行十二烷基硫酸钠-聚丙烯酰胺凝胶电泳(sodium dodecyl sulfate-polyacrylamide gelelectrophoresis, SDS-PAGE)及银染检测.
2.5 质谱分析
将银染后SDS-PAGE胶的目的条带在解剖显微镜下分离出来,送至香港大学李嘉诚医学院基因研究中心进行质谱分析检测.
2.6 调控网络生物信息学分析
基于生物信息学方法,采用Transfac/JASPAR在线数据库对DNA探针区段进行预测,确定可能的转录因子结合位点(transcription factor binding site,TFBS)及与之相作用的转录因子(transcription factor).通过文献挖掘,利用已有知识确定上述因子可能与GABA调控以及信号通路之间的关系,并除去非神经组织表达的转录因子. 然后利用HPRD/BIOGRID中的蛋白质-蛋白质相互作用(protein-protein interaction,PPI)网络进行扩展分析,挖掘能够与候选转录因子直接相互作用的中间因子,从而构建出完整的转录调控网络.
3 实验结果
3.1 生物信息学与基因组学预测分析结果
依据生物信息学与基因组学的最新进展,采用2014年3月发布的ENCODE数据,分析比较神经元与其他非神经元细胞基因,得到在神经元中特异性表达的GABA受体以及NMDA受体基因,如表1.

表1 神经元内特异表达的GABA和NMDA受体基因
针对这两组基因,结合组蛋白修饰情况、Open Chromatin数据和ChIP-seq数据等进行生物信息学分析后,对中枢神经系统神经细胞内GABA受体以及NMDA受体基因开放性染色体上可能的转录调控因子结合位点进行预测,得到特异在中枢神经元内起作用的转录因子结合位点,如表2. 本研究预测的7个GABA受体基因探针在不同脑区的分布情况不同,因此,将选择分布探针最多的前额叶皮质区(5个)与分布探针最少的小脑区(1个)进行后续实验研究.

表2 GABA和NMDA受体基因转录因子结合位点预测结果及相应引物序列
3.2 小鼠不同脑区核蛋白提取
细胞核蛋白的抽提过程中,在细胞膜破裂后,高倍光镜下进行观察,发现细胞膜破裂完全,仅剩细胞核. 提出核蛋白后,测得抽提物蛋白质量浓度分别为:前额叶皮质4.98 g/L,小脑5.8 g/L.
3.3 GABA和NMDA受体基因探针合成及生物素标记
将7条GABA受体及5条NMDA受体基因转录因子结合区间进行PCR扩增后,分别成功克隆λpMD20-T Vector后可得到如图2(a)所示包含DNA探针序列的质粒. 将这些质粒作为模板,Biotin-14-dATP作为底物进行PCR扩增后可得如图2(b)所示的生物素标记后探针.
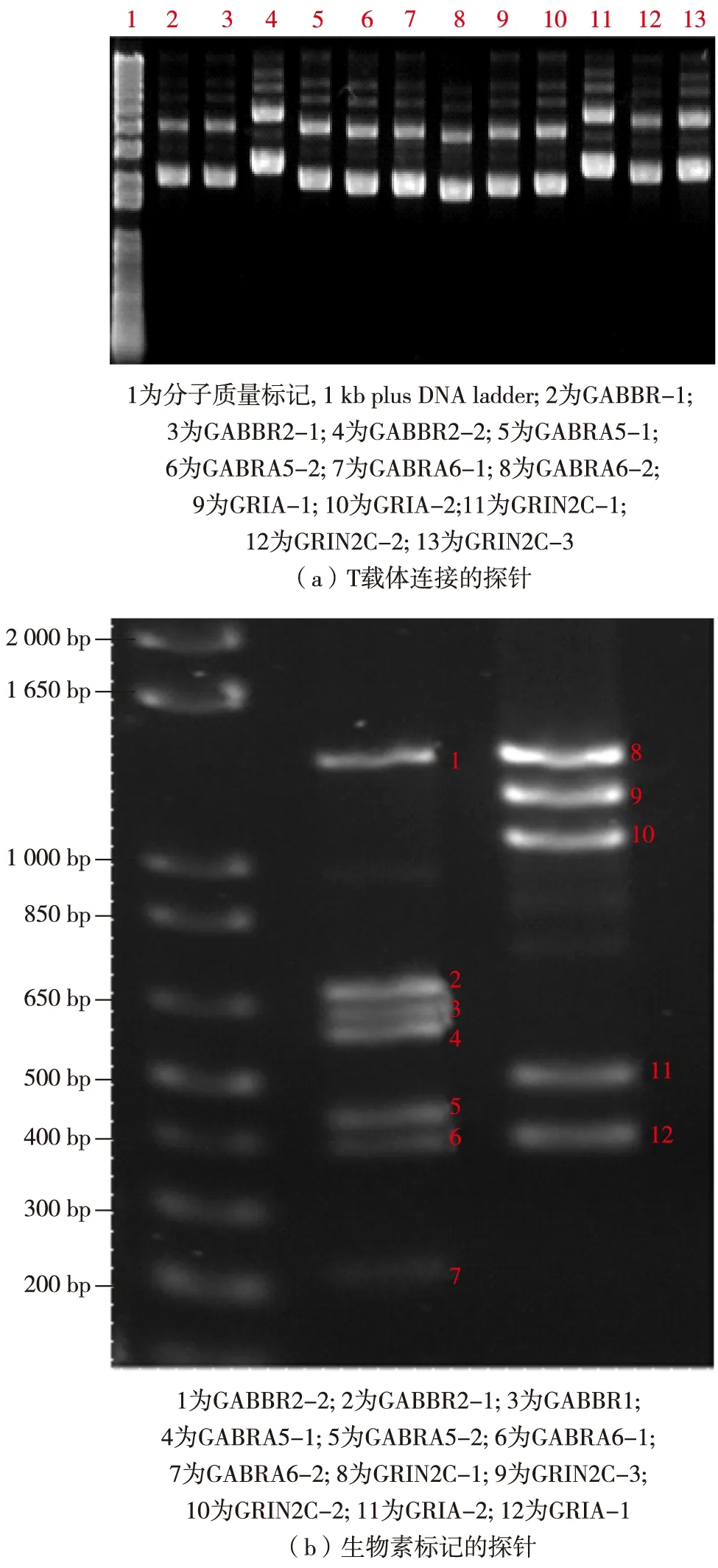
图2 GABA和NMDA受体基因探针合成及生物素标记Fig.2 Synthesis and biotin labeling of GABA and NMDA receptor gene probes
3.4 DNA pull-down实验
标记后的DNA探针与链亲和素琼脂糖树脂反应24 h后,取上清进行琼脂糖胶电泳并未看到DNA探针条带,表明标记后的探针已与链亲和素琼脂糖树脂充分结合. 前额叶皮质区与小脑区的核蛋白抽提物分别与GABA受体探针及NMDA受体探针行DNA pull-down实验后发现,GABA受体DNA探针在前额叶皮质和小脑神经元细胞核蛋白中分别都有相同的特异性结合条带,大小约为34 kDa(如图3红色箭头所示),而在NMDA受体DNA探针中未观察到相应的结合条带.

图3 GABA受体以及NMDA受体DNA探针在前额叶皮质、小脑神经元细胞核蛋白中的相互作用Fig.3 Interaction of GABA and NMDA receptor gene probes in nuclear proteins of prefrontal cortex and cerebellum
3.5 质谱分析结果
特异与GABA受体DNA探针结合的约34 kDa的蛋白条带,其质谱分析结果为核膜血影重复蛋白-1 (nuclear envelop spectrin repeatprotein-1, Nesprin-1),又称synaptic nuclear envelope-1 (SYNE-1).
3.6 调控网络生物信息学分析
通过上述DNA pull-down实验,GABA受体DNA探针可特异性地与Nesprin-1结合. 然而,作为核内重要蛋白的Nesprin-1并不是转录因子,故Nesprin-1应该通过与其他转录因子相互作用形成复合物进而调控GABA受体基因表达.
首先,通过运用Transfac/JASPAR在线数据库分析,7条GABA受体基因探针上共找到112个转录因子结合位点(transcription factor binding site,TFBS)及75个转录因子(transcription factor). 进一步将75个转录因子依据可与其作用的GABA受体基因探针数目进行排序.其中,出现频率大于2次的转录因子共25个(表3),再经过文献挖掘,筛选出神经组织表达的转录因子共5个,分别是MAFA、IRX2、BCL6、CEBPA和RP58.

表3 GABA受体基因相互作用的可能转录因子
在HPRD/BIOGRID数据库的蛋白质-蛋白质相互作用(protein-protein interaction,PPI)网络中进行扩展分析,Nesprin-1与上述5个可能转录因子之间分别可通过≤2个中间因子构成相互作用网络(图4).

图4 GABA受体基因与NESPRIN-1相互作用调控网络中可能的转录因子Fig.4 Possible transcriptional factors in the regulatory network between NESPRIN-1 and GABA receptors
对所得到的相互作用网络继续进行拓展分析,即分别对5个转录因子、中间因子以及NESPRIN-1与已知GABA相关蛋白相互作用的情况进行综合分析,得到更加精确的GABA受体基因的调控网络,如图5.

图5 GABA受体基因通过CEBPA或IRX2与NESPRIN-1相互作用调控拓展网络Fig.5 Expanded interaction reaction network between GABA receptors and NESPRIN-1 via CEBPA or IRX2
4 讨 论
作为中枢神经系统内最主要的抑制性神经递质受体,GABA受体参与了许多中枢神经系统的生理活动,故GABA受体数量、分布或功能的异常与多种神经系统疾病密切相关,如癫痫、卒中、阿尔茨海默症、精神分裂症、抑郁和焦虑等. 目前,多数的研究主要着眼于GABA受体的分子结构组成以及药理特性等方面,但对于GABA受体基因表达调控机制方面的研究鲜有报道. 为探究GABA受体基因的转录调控机制,发掘潜在的GABA受体基因转录控制因子,本研究基于生物信息学与基因组学的最新进展对GABA受体可能的转录因子结合部位进行了预测,以此作为DNA探针在神经元核蛋白中识别与之特异结合的核蛋白Nesprin-1. 然而作为一种核内重要的蛋白,Nesprin-1并不是转录因子. Nesprins家族是最近新发现的一组在细胞内广泛表达的含多个血影蛋白重复序列的细胞骨架蛋白家族[6],最初是Zhang等[7]在对血管平滑肌细胞分化标记物的研究中发现,由于此类蛋白结构上具有聚合血影蛋白重复序列、双组分核定位序列、保守的C-端以及单跨膜区域等特点,故将之命名为Nesprin(nuclear envelope spectrin repeat). Nesprin-1 是Nesprin 家族中研究较多的蛋白之一,其基因位于染色体的6q25.Nesprin-1蛋白的基本结构为:N端是串联成对的钙调蛋白同源区( CH-domain) 的肌动蛋白结合区( actin-binding domain,ABD),中间是多个血影蛋白重复序列形成的杆状区域,其上有多种蛋白结合位点,C端为一跨膜区,是果蝇属及秀丽隐杆线虫的Klarischt 同源,称 KLS区. 钙调蛋白同源区能连接细胞骨架肌动蛋白,而KLS 区是Nesprin 定位于核膜上所必需的,并能参与核膜定位. Nesprin-1主要定位在核膜,参与维持核形态、连接细胞核到细胞骨架,使细胞核固定在细胞的特定位置,其突变将使细胞核无法维持正常形态和空间构象,进而导致细胞内的信号传递异常[6].
近年来,研究者发现Nesprin-1/2参与了神经发育以及神经元迁移过程[8]. Zhang等[8-12]发现在大脑皮层细胞核迁移(interkinetic nuclear migration,INM)和神经元迁移过程中,Nesprin-1/2通过与动力蛋白Dynein/Dynactin和驱动蛋白(kinesin)相互作用进而介导中心体和细胞核的连接. 在动物实验中,文献[8]指出对Nesprin-1/2突变后可引起严重的学习以及记忆方面的损害. 另外,Gros-Louis等[13]报道了一个新发现的常染色体隐性遗传小脑共济失调(autosomal recessive cerebellar ataxia,ARCA)致病基因——Nesprin-1. 由此可知,Nesprin-1不仅参与了神经系统发育过程,且其基因的突变可引起严重的神经系统疾病.
综上可知,Nesprin-1与中枢神经系统病理生理过程有密切关系,本研究通过DNA pulldown实验发现其可与GABA受体基因探针特异性结合,因此,Nesprin-1可能通过与转录因子形成复合物从而参与GABA受体基因的转录调控. 综合Transfac/JASPAR在线数据库以及HPRD/BIOGRID数据库,进一步分析并描绘了Nesprin-1与可能的转录因子MAFA、IRX2、BCL6、CEBPA以及RP58形成的调控网络,并推测,在中枢神经组织中,细胞通过该调控网络调控GABA受体在特定的部位、特定的时间进行表达.
结 语
本研究采用的DNA-PullDown方法中,将预测所得的同种受体(GABA能或NMDA受体)基因调控序列分别混合起来,再将之与不同脑区神经元核抽提物进行反应,这种体外反应模式模拟了细胞内染色质的三维作用. 真核细胞的染色体是由一系列各自独立、高度有序的染色质结构域构成的,这些结构域是独立的基因表达调控单位,在真核生物的基因表达调控中起重要作用,比如DNA拓扑结构变化、DNA碱基修饰变化和组蛋白变化,因此,研究者一直致力于研究染色质构架在基因调控中的作用. Zhang等[14]证实了染色质通过DNA的三维折叠帮助距离远的区域调控基因表达,这项发现揭示了人类基因组中的基因虽然相隔甚远,但相关基因可以通过长距离的染色体相互作用、高度有序的染色体构架,有序地进行转录调控. 本研究将预测所得的同种受体基因调控序列结合起来进行实验,很好地模拟了细胞核内真实的基因表达调控模式.
在对GABA受体基因转录调控机制的探索中,目前的工作仅是一个开端. 通过上述实验分析,初步建立了基于神经细胞特异性功能基因组数据库探索调控GABA受体基因的分子控制模式,并且该分子控制模式是调节GABA受体在特定部位、特定时间表达的关键调控网络. 后续实验中,将与iNc转录因子相结合后,以期能够应用于诱导多能干细胞或是体细胞直接分化为GABA能神经元,为将来构建与GABA能神经元相关的疾病模型或再生医学治疗提供研究基础.
/ References:
[1] Mary Chebib, Graham A R J. The ‘ABC’ of GABA receptors: a brief review[J]. Clinical and Experimental Pharmacology and Physiology, 1999, 26(11): 937-940.
[2] Ni Jiazuan,Chen Ping,Liu Qiong,et al. Advance research on strategies for the prevention of Alzheimer’s disease [J].Journal of Shenzhen University Science and Engineering,2013,30(4):331-348.(in Chinese) 倪嘉缵, 陈 平, 刘 琼, 等. 阿尔茨海默病的防治策略研究进展[J]. 深圳大学学报理工版, 2013, 30(4): 331-348.
[3] Li Jing, Du Yongping, Zhang Yueping. Effects of hypoxia on glutamatergic and GABAergic synaptic transmission[J]. Chinese Journal of Pathophysiology, 2013, 29(2):371-375.(in Chinese) 李 晶, 杜永平, 张月萍. 缺氧对谷氨酸能和GABA能突触传递的影响[J]. 中国病理生理杂志, 2013, 29(2):371-375.
[4] Song Lingyun, Zhang Zhancheng, Grasfeder L L, et al. Open chromatin defined by DNaseI and FAIRE identifies regulatory elements that shape cell-type identity[J]. Genome Research, 2011, 21(10):1757-1767.
[5] Jutras B L, Verma A, Stevenson B. Identification of novel DNA-binding proteins using DNA affinity chromatography-pull down[J]. Current Protocols in Microbiology, 2012, 24:1F.1.1-1F.1.13.
[6] Warren D T, Zhang Qiuping, Weissberg P L, et al. Nesprins: intracellular scaffolds that maintain cell architecture and coordinate cell function[J]. Expert Reviews in Molecular Medicine, 2005, 7(11):1-15.
[7] Zhang Qiuping, Skepper J N, Yang Fangtang, et al. Nesprins: a novel family of spectrin-repeat-containing proteins that localize to the nuclear membrane in multiple tissues[J]. Journal of Cell Science, 2001, 114(Pt 24):4485-4498.
[8] Zhang Xiaochang,Lei Kai,Yuan Xiaobing,et al.SUN1/2 and Syne/Nesprin-1/2 complexes connect centrosome to the nucleus during neurogenesis and neuronal migration in mice[J]. Neuron, 2009, 64(2):173-187.
[9] Feng Yuanyi, Walsh C A. Protein-protein interactions, cytoskeletal regulation and neuronal migration[J]. Nature Reviews Neuroscience, 2001, 2(6):408-416.
[10] Tsai J W, Bremner K H, Vallee R B. Dual subcellular roles for LIS1 and dynein in radial neuronal migration in live brain tissue[J]. Nature Neuroscience, 2007, 10(8):970-979.
[11] Tsai L H, Gleeson J G. Nucleokinesis in neuronal migration[J]. Neuron, 2005, 46(3):383-388.
[12] Wynshaw-Boris A. Lissencephaly and LIS1: insights into the molecular mechanisms of neuronal migration and development[J]. Clinical Genetics, 2007, 72(4):296-304.
[13] Gros-Louis F, Dupre N, Dion P, et al. Mutations in SYNE1 lead to a newly discovered form of autosomal recessive cerebellar ataxia[J]. Nature Genetics, 2007, 39(1):80-85.
[14] Zhang Jingyao, Poh H M, Peh S Q, et al. ChIA-PET analysis of transcriptional chromatin interactions[J]. Methods, 2012, 58(3):289-299.
【中文责编:晨 兮;英文责编:艾 琳】
Systematic regulatory network of gamma-aminobutyric acid receptor genes
Zhao Yunyan1, Li Zhong1†, Chen Danni2, Lei Qingfeng1,He Lu1, and Wei Rui1
1) Department of Neurology, The Sixth Affiliated Hospital, Sun Yat-sen University, Guangzhou 510655, P.R.China 2) College of Optoelectronic Engineering, Shenzhen University, Shenzhen 518060, P.R.China
By using a multidisciplinary strategy, we try to systematically find out the transcription factors that regulate gamma-aminobutyric acid (GABA) receptor expression. Based on the published data in the encyclopedia of DNA elements (ENCODE), we obtain open chromatin sequences of GABA receptor genes. These sequences are used as solid phase probes to capture the specific proteins that could interact with the sequences directly. The captured protein is identified with mass spectrometry for further regulatory network analysis. The experimental results show that the GABA receptor gene probes can capture one specific band in different brain domains while the N-methyl-D-aspartate (NMDA) gene probes, which are designed as negative control, could not capture this specific band. Mass spectrometry results indicate that the protein, which could specifically bind with GABA receptor gene open chromatin sequence, is nuclear envelop spectrin repeatprotein-1(Nesprin-1)(synaptic nuclear envelope-1, SYNE-1). Further bioinformatics analysis results suggest that Nesprin-1 could form a complex with transcription factors such as MAFA, IRX2, BCL6, CEBPA and RP58 and could co-express with GABA receptor genes such as GABRA5, GABRA6, GABBR1 and GABBR2. In conclusion, GABA receptor genes are regulated through the same transcriptional regulation mechanism in different brain domains. Nesprin-1 could interact with transcription factors such as MAFA, IRX2, BCL6, CEBPA and RP58 to form a regulatory complex that regulates the expression of GABA receptor genes. This specific regulatory network can be used as a tool for further inductions of the differentiation of embryonic stem cells or other stem cells into GABA receptor expressing neurons.
molecular biology; gamma-aminobutyric acid (GABA) receptor; open chromatin; transcription regulation; transcription factor; nuclear envelop spectrin repeatprotein-1 (Nesprin-1)
:Zhao Yunyan, Li Zhong, Chen Danni, et al.Systematic regulatory network of gamma-aminobutyric acid receptor genes[J]. Journal of Shenzhen University Science and Engineering, 2015, 32(2): 128-136.(in Chinese)
R 338.2; Q 426
A
10.3724/SP.J.1249.2015.02128
广东省医学科学技术研究基金资助项目(A2012211);广州市天河区科技计划重点资助项目(201404KW028)
赵云燕(1986—),女(汉族),福建省福州市人,中山大学硕士研究生,E-mail:zoezhao7898@gmail.com
Received:2014-11-03;Accepted:2014-12-26
Foundation:Guangdong Medical Science and Technology Research Foundation(A2012211);Guangzhou Tianhe District Science and Technology Key Project (201404KW028)
† Corresponding author:Chief physician Li Zhong.E-mail: zslyjohn@163.com
引 文:赵云燕,李 中,陈丹妮,等.γ-氨基丁酸受体基因的系统性调控网络[J]. 深圳大学学报理工版,2015,32(2):128-136.

