线粒体蛋白质组学研究进展
许静怡 陈超翔 韩锦艳 杭纬 颜晓梅

摘要 线粒体是细胞能量代谢、生物合成和细胞死亡的调控中心,其功能异常会导致许多疾病的产生。线粒体蛋白质组学研究为解析线粒体的生理功能、探索线粒体相关疾病的发病机制及促进线粒体为靶向的药物研发提供重要理论依据。本文对线粒体蛋白质组学的研究方法,尤其是近年的技术发展以及线粒体蛋白质组学的性质及其应用进行总结,并对其未来的发展前景和挑战进行展望。
关键词 线粒体; 蛋白质组学; 质谱; 疾病; 评述
1 引 言
线粒体是真核细胞重要的细胞器,其在细胞能量代谢[1]、生物合成和细胞死亡[2,3](包括细胞凋亡和细胞程序性坏死)的调控中起关键作用。此外,线粒体还参与三羧酸循环、脂肪酸和氨基酸氧化、钙离子稳态调节等重要生理过程[4]。由于线粒体在维持生命稳态中发挥重要作用,因此其功能异常与许多人类疾病相关,如神经退行性疾病、癌症、心脏病和糖尿病等[5~7]。由于线粒体蛋白质表达异常是其功能紊乱的主要原因,而线粒体蛋白质组学正是从整体角度,分析线粒体的蛋白质组成、表达水平与修饰状态等的动态变化,旨在阐明线粒体内全部蛋白质的表达模式和功能模式,包括蛋白质的表达与存在方式(修饰方式)、结构与功能、以及蛋白质相互作用等,从而在蛋白质水平探索线粒体生理功能和相关疾病的联系[8,9]。通过线粒体蛋白质组学可系统研究正常和病变组织中线粒体蛋白分布与表达的差异,从而为研究线粒体相关疾病的分子机制和以线粒体为靶标的药物研发奠定理论基础[10]。近年来,人类基因组测序的完成、串级质谱和蛋白质数据库的发展加速了线粒体蛋白组学的研究。本文对线粒体蛋白质组学的研究方法,尤其是近年的技术发展进行总结,分析并讨论蛋白质组学的性质及其应用。
2 线粒体蛋白质组学的研究方法与技术
2.1 线粒体蛋白质的提取策略
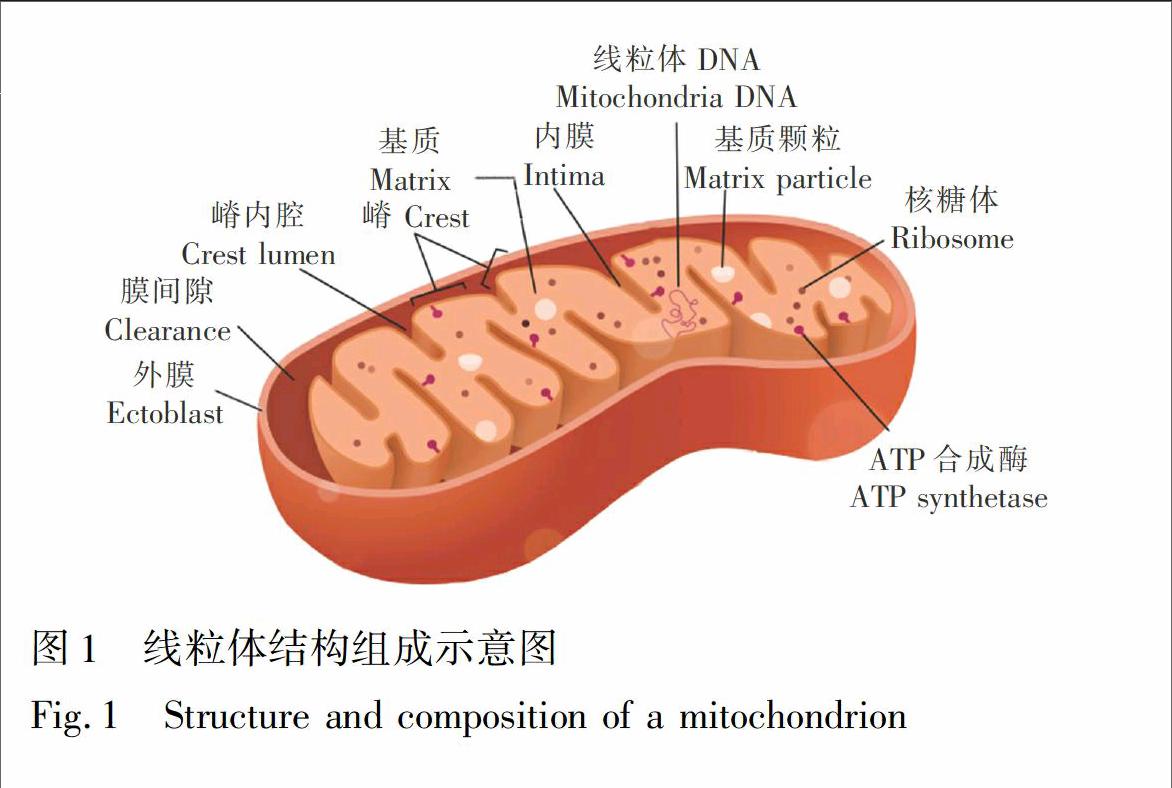
提取大量高纯度的线粒体蛋白质是开展线粒体蛋白质组学研究的重要前提[11],线粒体的纯度及其结构的完整性是保障实验结果准确的关键所在。如图1所示,线粒体由外膜、膜间隙、内膜和基质构成,基质或者膜间隙蛋白质组学可促进线粒体蛋白质的定位和功能研究,但同时也对提取策略的选择性和特异性提出了更高要求[12]。
差速离心结合密度梯度离心是分离纯化线粒体的经典方法[13]。差速离心用于分离不同大小、体积和重量的细胞器[14]。通过差速离心可初步分离得到线粒体,为尽量避免其它细胞器组分的干扰,需通过密度梯度离心对得到的线粒体进一步分离纯化[15]。密度梯度离心是在离心管内通过特定介质形成连续或不连续的密度梯度,将差速离心得到的线粒体悬液置于介质顶部,悬液通过重力或离心力场作用进一步分层和分离。线粒体密度梯度离心常用介质有氯化铯、蔗糖、Ficoll、Percoll及碘化介质(如Nycodenz)等。其中蔗糖黏度大且渗透性高, 易使细胞器破裂;而Percoll和Nycodenz介质形成的梯度十分稳定,且不易穿透生物膜造成细胞器损伤。例如,2010年Ferreira等利用Percoll为介质的密度梯度离心法提取小鼠的腓肠骨骼肌中肌膜和肌纤维线粒体进行线粒体蛋白质组学研究[16]。2014年Chen等利用以Nycodenz为介质的密度梯度离心法对卵巢癌中有阿霉素抗性与无阿霉素抗性的两个细胞系分别提取线粒体,分离鉴定后发现,122种蛋白质在有阿霉素抗性的细胞系中表达量显著变化[17]。提取线粒体的纯度和完整性通常采用形态学观察、特定抗体检测或特定酶活性检测等方法进行分析。电镜可直观地观察线粒体形态及结构的完整性,采用免疫印迹法分析线粒体标志性蛋白如外膜VDAC、膜间隙细胞色素C蛋白的丰度可进一步证明膜结构的完整性。此外,通过测定线粒体标志性酶如琥珀酸脱氢酶的活性及与其它细胞器标志酶的活性比较可检测提取线粒体的纯度。近年来,本课题组采用线粒体内膜心磷脂特异性荧光探针(NAO)和核酸探针(SYTO 62)(与线粒体DNA结合)对线粒体进行标记,利用其自行研发的超高灵敏流式检测装置(High sensitivity flow cytometer, HSFCM)在单颗粒水平对双荧光标记的线粒体进行检测,通过散射光通道与两个荧光通道信号的时间相关性可定量分析所提取线粒体的纯度和结构完整性并能从单细胞器水平揭示线粒体的异质性[18]。
然而,通过上述分级分离技术获得的线粒体蛋白不仅由于细胞器粘连导致纯度较低,容易损失或受到污染,而且难以特异地检测线粒体各个亚区间(基质、膜间隙)的蛋白质[11]。为了实现线粒体亚区间蛋白质的特异提取,美国MIT大学的Ting课题组对抗坏血酸过氧化物酶(APEX)进行基因改造,可特异地“钓到”膜间隙或基质蛋白质[19]。如图2所示,基因工程改造的APEX可靶向线粒体基质、氧化多种苯酚衍生物产生羟基自由基,这些自由基寿命短(<1 ms)[20],标记半径小(<20 nm)[21,22],可与富电子氨基酸例如Tyr, Trp, His等共价结合,从而标记邻近蛋白质[23]。通过该方法可鉴定HEK细胞线粒体基质的495种蛋白质,其中31种是新发现的蛋白质[24]。采用该方法捕获的基质蛋白组覆盖率和特异性分别达到85%和94%,覆盖率较低的原因可能是一些蛋白质侧链被包埋太致密而无法与羟基自由基作用。该课题组又进一步通过APEX结合细胞培养稳定同位素标记方法(SILAC)[25] 提取和鉴定线粒体膜间隙蛋白组,共鉴定了127种膜间隙蛋白质(其中9种蛋白为新发现的线粒体蛋白),特异性高达94%,实现了对线粒体膜间隙蛋白质组的高效测定[26]。
图2 标记活细胞的线粒体基质蛋白质组[24]
Fig.2 Labeling the mitochondrial matrix proteome in living cells[24] (Adapted from Ref.[24], with kind permission from Science)
综上所述,获得线粒体总蛋白质的经典方法是采用差速离心结合密度梯度离心提取线粒体,裂解之后进行分离和质谱分析,该提取策略促进了线粒体总蛋白质“目录”的建立。Ting课题组创新性地通过APEX与基质或膜间隙靶向序列的融合表达,特异获取线粒体基质或膜间隙蛋白组,省略了提取细胞器这一繁琐步骤,实现对线粒体亚区间蛋白质的特异性提取和鉴定,促进了新蛋白的发现、蛋白质的亚线粒体定位和亚区间蛋白质功能研究,使蛋白质组学迈入了 “细胞器亚区间”时代。
2.2 线粒体蛋白质的分离策略
对于线粒体蛋白质组学研究,无论通过上述何种策略获得线粒体蛋白质组,都需要对蛋白质混合物进行分离,分离后酶解成多肽片段,再通过质谱进行鉴定。分离线粒体蛋白质混合物的方法主要是电泳和色谱法。
二维电泳是分离线粒体蛋白质最广泛最经典的方法,它是一种高分辨率的蛋白质分离技术。二维凝胶电泳的第一相常为等电聚焦电泳(IEF),根据蛋白质等电点的不同进行分离;第二相是SDS聚丙烯酰胺凝胶电泳(SDSPAGE),根据蛋白质分子量的不同进行分离。1998年Rabilloud等首先利用IEF/SDSPAGE分离人胚胎组织线粒体蛋白质并结合肽指纹图谱鉴定了46种线粒体蛋白质[27]。王媛等通过双向凝胶电泳在小鼠心脏、肝脏和肾脏3种组织中发现了87种组织特异的差异蛋白[28]。此外,有报道采用双向荧光差异凝胶电泳(2DDIGE)分析蛋白表达量的变化,即利用不同荧光染料标记不同处理方法的蛋白质,再等量混合标记不同荧光染料的蛋白质进行双向电泳,通过荧光强度的变化可测定蛋白表达量的变化。如van Laar等提取小鼠脑线粒体并分成有无多巴胺醌处理的两组,通过2DDIGE结合质谱分析发现18种蛋白质的表达量发生显著变化[29]。RamírezTorres等分别提取两组膳食中“含”与“不含”鲨烯的小鼠肝脏线粒体,利用2DDIGE分离蛋白质结合质谱分析,发现鲨烯处理的小鼠肝脏线粒体中有18种蛋白质表达量发生改变[30]。然而, IEF/SDSPAGE技术难以分离强酸性、强碱性、相对分子质量过大或过小的蛋白质或者膜蛋白和一些低丰度蛋白质等。通过蓝绿温和非变性凝胶电泳(BNPAGE)结合SDSPAGE可以弥补IEF/SDSPAGE的技术缺陷,可分离线粒体膜蛋白复合物等。该技术是在非变性状态下根据相对分子质量分离膜蛋白复合体且不改变膜蛋白复合物的生理活性和结合状态,再通过SDSPAGE将各个复合物的亚基或蛋白质分离出来。Ferreira等通过综合使用2DIEF/SDSPAGE/MS/MS和2DBN/SDSPAGE/MS/MS对腓肠骨骼肌中肌膜和肌纤维的线粒体蛋白质进行分离和鉴定,识别325种线粒体蛋白质[16]。Chou等综合BN/SDSPAGE和IEF/SDSPAGE两种分离方法对小鼠的大脑皮质线粒体蛋白质进行分离并结合质谱分析,发现23种与三羧酸循环(TCA)相关蛋白质在AD模型小鼠中表达失调[31]。
色谱是将线粒体蛋白质用蛋白酶水解后,根据不同多肽在流动相和固定相之间的分配系数不同,使其在两相间反复多次分配,以不同速度迁移,从而分离不同肽段。多维液相色谱组合了两种或两种以上不同分离机制,根据样品不同特性(如分子大小、亲水性、等电点等)对混合多肽进行分离。多维液相色谱具有快速、高通量、自动化、重现性高等优点,且能够检测疏水性以及低丰度蛋白质,但难以提供翻译后修饰等信息[32]。
通过将凝胶电泳与色谱两种策略以合理方式组合,可使混合物达到更好分离效果。即先将线粒体的蛋白混合物通过SDSPAGE分离并切割条带水解成多肽,最后用一维或多维液相色谱串联质谱来鉴定水解得到的肽段。例如,Techritz等从鼠脑、骨骼肌、心脏、肝脏和肾五种组织中提取线粒体并纯化,通过IEF/SDSPAGE和LC/ESIMS及MS/MS分离鉴定,发现有48种蛋白质在不同组织中存在不同表达程度[33]。
2.3 线粒体蛋白质的鉴定
质谱是鉴定蛋白质的首选方法,其可以准确测量肽段和蛋白质的分子量、氨基酸序列及翻译后修饰等信息。目前,根据蛋白质或多肽分子离子化的方式不同可分为电喷雾离子化质谱(ESIMS)和基质辅助激光解吸离子化质谱(MALDIMS)。MALDI产生的离子可以用飞行时间质量分析器(TOF)检测,即基质辅助激光解吸飞行时间质谱(MALDITOFMS)。MALDITOFMS可以检测肽段的分子量,获得蛋白质的肽质量指纹图谱(PMF),通过与相应的线粒体蛋白质数据库比对来鉴定蛋白质[34]。此外,通过串级质谱技术(MS/MS)可对质谱分析的肽段进行碎裂再用质谱分析,从而得到肽段序列信息。质谱技术还可与双向凝胶电泳、二维液相色谱等蛋白质或肽段分离技术联用,实现“在线”式分离鉴定蛋白质,具有灵敏度高、准确度高和自动化程度高的优点。如Taylor等通过蔗糖密度梯度法提取人心脏线粒体,利用SDSPAGE结合质谱和生物信息学分析,识别615种线粒体蛋白质,这些蛋白质主要参与生物合成,离子转运及脂代谢[35]。Lamb等通过LCMS/MS发现40种以上线粒体蛋白质在癌干细胞中表达量无限上调[36]。Mootha等通过串级质谱技术LCMS/MS鉴定来自小鼠脑、心脏、肾、肝脏的399种线粒体蛋白质[37]。但这些检测到的大部分是高丰度蛋白质,随着串联质谱技术灵敏度等的提高,也促进了低丰度蛋白质的检测。如Pagliarini等提取小鼠的14种组织结合MS/MS技术鉴定了3881种线粒体蛋白质[38]。因为2DDIGE在样品处理上费时费力,不利于大规模的蛋白质组定量,而通过同位素标记、化学标记(ICAT或iTRAQ)或label free方法,质谱也可定量分析线粒体蛋白组的改变。例如,Bugger等通过label free方法和LCMS/MS发现心衰过程中脂肪酸氧化酶表达量减少,而丙酮酸脱氢酶亚基和两种TCA循环关键酶表达量均上调,实现对线粒体蛋白组的定量分析[39]。为了探索氧含量对神经元线粒体功能的影响,Villeneuve等分别提取5%和21%氧含量培养SHSY5Y(神经母细胞瘤细胞)的线粒体,利用同位素标记结合IEFPAGE及LCMS/MS分离鉴定蛋白质,鉴定出714种线粒体蛋白质,且5%氧含量培养的细胞的线粒体呼吸链复合物亚基等显著高于21%氧含量培养的细胞,前者所产生的活性氧水平也低于后者,说明SHSY5Y更适合在5%氧含量条件下培养[40]。线粒体蛋白质的合成与降解过程之间的动态平衡是维持线粒体稳态的重要因素,因此Kim等通过重水标记策略结合SDSPAGE, LCMS, MS/MS分离鉴定来自鼠心脏和肝脏485种“转换蛋白质”。实验结果表明, 肝线粒体蛋白质转换速率快于心脏组织,这些信息可以促进线粒体蛋白翻译及稳态维持的机理研究[41]。
显微镜技术是与质谱互补的蛋白质鉴定方法,其主要作用是对线粒体蛋白质进行定位分析,即通过荧光标记抗体或者转染荧光蛋白(如GFP)等进行观察,已成功运用于酵母菌的线粒体蛋白质组学研究[42,43]。2013年Techritz等[33]从鼠脑、骨骼肌、心脏、肝脏和肾5种组织中提取线粒体,质谱鉴定后再结合显微镜观察GFP标记的蛋白质,识别了6种新的线粒体蛋白质。该技术的缺陷在于高质量抗体的数量和种类有限,转染实验周期较长且引入外源蛋白质,只能对少量几种蛋白进行观察,无法实现高通量分析[44]。
总之,质谱和显微镜技术的发展促进了线粒体蛋白质组学的研究,既可进行高通量分析,也可选择性地观察目标蛋白,促进未知线粒体蛋白的发现,完善线粒体蛋白组的“目录”,也使线粒体蛋白质组学在相关疾病的研究中得以应用。
3 线粒体蛋白质组学的性质及其应用
随着线粒体蛋白质组提取、分离和鉴定技术的发展,目前已建立了较为完善的线粒体蛋白质“目录”。已检测到1000多种人类线粒体蛋白质[8],并发现线粒体蛋白质的丰度变化非常大,最丰富的内膜和外膜蛋白分别是ANT1和VDAC[45]。以下对线粒体蛋白质组的蛋白数目、丰度、定位、翻译后修饰、组织分布、生化途径归属及其应用进行介绍。
在线粒体蛋白质的定位方面,一部分蛋白质只存在于线粒体中,而另一部分线粒体蛋白质可以“双重”定位,即同一基因编码的蛋白质除了定位于线粒体,还出现在胞质或其它细胞器中[46]。如在凋亡信号刺激下,tBID被凋亡蛋白酶8切割后转位到线粒体外膜上[47];线粒体膜间隙细胞色素C借助凋亡蛋白Bax等形成的孔道转位到胞浆[48]。
在线粒体蛋白质的翻译后修饰方面,主要采用3种方式:乙酰化、磷酸化和羟基化。SIRT3是依赖NAD+的线粒体去乙酰化酶,为了解其作用底物,Rardin等利用Label free定量方法检测到小鼠肝脏组织中483种乙酰化线粒体蛋白质,2187个赖氨酸乙酰化位点,且发现136种蛋白质中283个乙酰化位点在SIRT3基因敲除小鼠模型中显著上调,说明SITR3可对多种蛋白质以及同一种蛋白质上的多个位点去乙酰化。进一步分析发现, 这些乙酰化线粒体蛋白质主要参与脂肪酸氧化、TCA循环等,表明SITR3是一种广谱的线粒体蛋白质去乙酰化酶[49]。Grimsrud等通过对不同生理状态小鼠肝脏磷酸化线粒体蛋白组进行多参数分析,结合iTRAQ定量标记策略及LCMS/MS技术分离鉴定磷酸化多肽,共识别295种磷酸化线粒体蛋白质,包括811个磷酸化位点,且这些线粒体蛋白质的磷酸化在肥胖症及T2D相关的酮体合成中起重要调节作用[50]。Deng等提取T2D模型小鼠的肝脏线粒体,通过LCMS/MS技术分离鉴定出355种羟基化线粒体蛋白质,且羟基化程度在T2D病变过程中显著提高,这种高程度的羟基化与ROS过量产生和氧化应激相关,可加速凋亡速率,这些蛋白质组学研究也促进了T2D发病机理探索和药物研发[51]。总之,线粒体蛋白质组学技术与方法的发展促进动态可逆的蛋白质翻译后修饰的研究。
在线粒体蛋白质的组织分布方面,由于不同组织对线粒体的功能需求不同,如肝脏主要需要生物合成功能,而心脏主要需要能量代谢功能,因而线粒体蛋白质在不同组织中分布不同。如Johson等分别提取小鼠的脑、肝脏、心脏和肾脏线粒体,通过LCMS鉴定识别1162种高信任度线粒体蛋白质,然而任意两种组织间有1149种蛋白质显著不同,4种组织中仅有13种蛋白质相同,说明线粒体蛋白质组的组织分布差异巨大[52]。Lotz等分别提取人心肌、小鼠心肌、小鼠肝脏组织及果蝇线粒体,利用SDSPAGE、LCMS/MS和光谱分析分别识别1398, 1620, 1733和1015种线粒体蛋白质,4种体系中仅有419种蛋白质高度保守,且这些蛋白质表达量动态范围大(跨越5个数量级),高丰度蛋白主要与氧化磷酸化、代谢、信号转导和蛋白转运等相关[53]。且研究发现,呼吸链复合物Ⅰ, Ⅱ, Ⅲ和Ⅴ在所有组织中高丰度分布,然而复合物IV在不同组织中分布不同[54]。总之,线粒体蛋白质组学的研究揭示了线粒体蛋白质在不同组织或种属间分布差异大,不同组织的线粒体蛋白质组学研究可以促进相关疾病的研究和治疗。
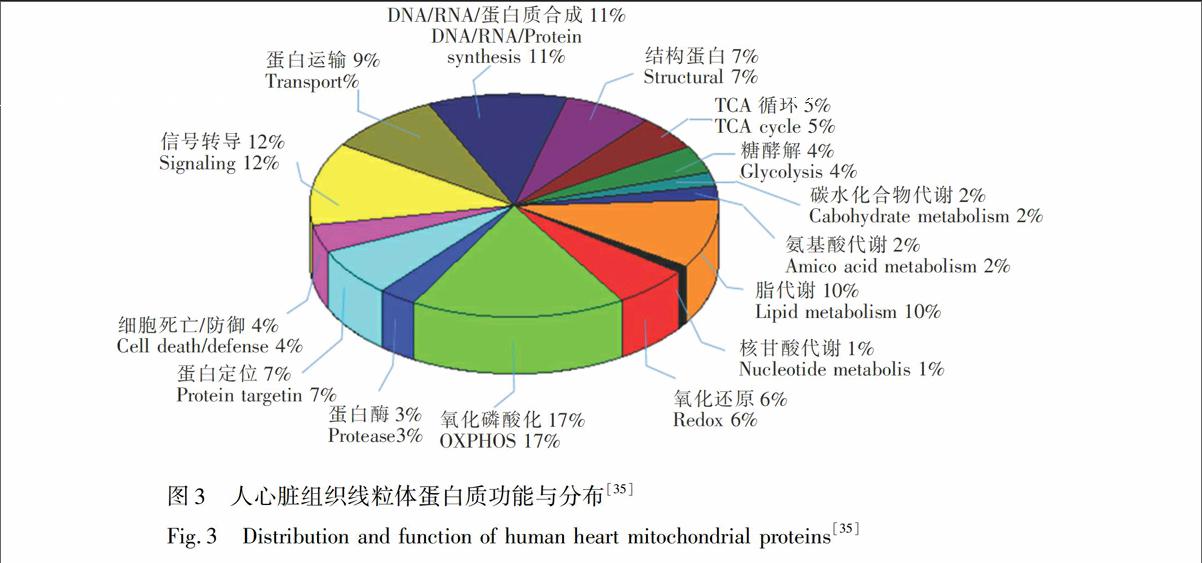
在线粒体蛋白质的生化途径归属方面,以人的正常心脏组织为例(如图3所示)[35],在所识别的498种线粒体蛋白质中,与氧化磷酸化功能相关蛋白质数目最多,占17%,信号转导相关的占12%,生物合成相关的占11%,与核苷酸代谢功能相关的数目最少,占1%,说明线粒体在能量代谢、生物合成及信号转导中起着重要的调控作用。
图3 人心脏组织线粒体蛋白质功能与分布[35]
Fig.3 Distribution and function of human heart mitochondrial proteins[35]
线粒体蛋白质组学的发展对线粒体相关疾病的研究起到了积极的推动作用。例如,线粒体氧化磷酸化受阻,将导致呼吸链疾病(RCD),患病率约0.02%,临床特征包含骨骼肌病变、心肌症、癫痫、中风、失明和内分泌紊乱等,通过线粒体蛋白组学研究已发现92种蛋白质突变与RCD相关[55]。在退行性疾病方面,通过线粒体蛋白质组学研究发现复合物I功能失调与帕金森疾病(PD)相关,其参与蛋白折叠和细胞凋亡,促进了PD发病机制的研究[56]。在癌症方面,Kim等在人胃癌细胞系AGS线粒体蛋白组研究中发现4种高表达的线粒体蛋白质[57];Chen等利用2DDIGE结合MALDITOFMS对不同的乳腺癌细胞系的线粒体蛋白组进行研究[58],促进了肿瘤细胞线粒体生物标记物的发现。在心血管疾病方面,通过线粒体蛋白质组学研究可阐明压力负荷诱导心衰[39]、缺血性心衰、以及慢性缺氧疾病的发病机制[59, 60]。在肝病方面,Eccleston等发现高脂肪膳食导致线粒体蛋白组改变,其与脂肪变性、NO合成及代谢有关[61]。在衰老方面,Alves等通过电泳和质谱串联技术对有无运动的骨骼肌线粒体蛋白组研究,说明运动是改善衰老和维持线粒体功能的重要调节因素[62]。
在药物研发方面,由于癌细胞抗药性的产生使癌症治疗效果大大降低。Chen等通过SILAC和LCMS/MS技术发现122种线粒体蛋白质在对阿霉素抗性的卵巢癌细胞系中表达量显著改变,分析表明线粒体是改善抗药性的重要靶点[17]。Chappell等通过线粒体蛋白质组学研究发现抗药性细胞系中线粒体蛋白质表达量的改变与凋亡规避、肿瘤侵染和转移等生理活动相关[63]。线粒体蛋白质组学也可用于药物副作用的研究,Lee等通过iTRAQ定量并结合2DLC分离、MALDITOF/TOF MS/MS鉴定线粒体蛋白质,说明曲格列酮会损伤线粒体功能[64]。综上所述,线粒体蛋白质组学的发展促进了线粒体相关疾病的发病机制的系统研究和药物研发。
4 结论与展望
随着线粒体蛋白质组提取、分离和鉴定技术的发展,目前既能对整个线粒体蛋白质组进行分析,也可针对膜间隙或基质亚区间蛋白质组分析,进一步完善线粒体蛋白质组学的“目录”。蛋白质组学的发展为我们提供了大量线粒体基因和蛋白信息,通过结合临床特征,有利于发现与疾病相关的基因和蛋白,为我们系统研究线粒体与人类疾病的关系奠定基础。线粒体蛋白质组学需要在以下几个方向进一步发展:第一,进一步完善线粒体蛋白组目录。一些蛋白质可能由于丰度太低无法使用传统方法检测,一些蛋白质由于侧链包埋于封闭结构中而无法对其进行生物素化检测。因此需要改进实验技术与方法,提高线粒体蛋白质提取方法的覆盖率和特异性,鉴定、识别之前难以检测的蛋白质;第二,除鉴定线粒体蛋白质的氨基酸序列,还需完善蛋白质翻译后修饰和线粒体亚区间定位等信息,因为这些信息与蛋白质自身功能紧密相关;第三,通过RNA干扰、蛋白蛋白相互作用、理论计算结合数据库等方法研究蛋白质在线粒体能量代谢、生物合成、细胞死亡中所起的作用(功能预测);最后,借助生物及化学的新技术、新方法和数据库资源等将线粒体蛋白质组学中蛋白质序列突变等信息与线粒体的生理功能和相关疾病的临床特征及特定表型关联起来,这不仅对于线粒体相关疾病发病机理的研究和药物研发意义重大,而且也正是发展线粒体蛋白质组学的目的所在。总而言之,线粒体蛋白质组学对线粒体全部蛋白质的“破译”将会为人类疾病研究和线粒体生物学开辟一个崭新时代。
References
1 Wagner B K, Kitami T, Gilbert T J, Peck D, Ramanathan A, Schreiber S L, Golub T R, Mootha V K. Nat. Biotechnol., 2008, 26: 343-351
2 Soriano M E, Scorrano L. Cell, 2011, 145(1): 15-17
3 Hengartner M O. Nature, 2000, 407: 770-776
4 Detmer S A, Chan D C. Nat. Rev. Mol. Cell Biol., 2007, 8(11): 870-879
5 Heller A, Brockhoff G, Goepferich A. Eur. J. Pharm. Biopharm., 2012, 82(1): 1-18
6 Abramov A Y, SmuldersSrinivasan T K, Kirby D M, AcinPerez R, Enriquez J A, Lightowlers R N, Duchen M R, Turnbull D M. Brain, 2010, 133: 797-807
7 Weinberg S E, Chandel N S. Nat. Chem. Biol., 2015, 11(1): 9-15
8 Calvo S E, Mootha V K. Annu. Rev. Genom. Hum. G, 2010, 11: 25-44
9 Wallace D C, Fan W, Procaccio V. Annu. Rev. Pathol. Mech., 2010, 5: 297-348
10 Jiang Y J, Wang X. J. Hematol. Oncol., 2012, 5: 1-13
11 Forner F, Foster L J, Campanaro S, Valle G, Mann M. Mol. Cell Proteomics, 2006, 5(4): 608-619
12 Bardel J, Louwagie M, Jaquinod M, Jourdain A, Luche S, Rabilloud T, Macherel D, Garin J, Bourguignon J. Proteomics, 2002, 2(7): 880-898
13 Stimpson S E, Coorssen J R, Myers S J. Anal. Biochem., 2015, 475: 1-3
14 Foster L J, de Hoog C L, Zhang Y, Zhang Y, Xie X, Mootha V K, Mann M. Cell, 2006, 125(1): 187-199
15 Hartwig S, Feckler C, Lehr S, Wallbrecht K, Wolgast H, MüllerWieland D, Kotzka J. Proteomics, 2009, 9(11): 3209-3214
16 Ferreira R, Vitorino R, Alves R M, Appell H J, Powers S K, Duarte J A, Amado F. Proteomics, 2010, 10(17): 3142-3154
17 Chen X L, Wei S S, Ma Y, Lu J, Niu G, Xue Y H, Chen X Y, Yang F Q. Theranostics, 2014, 4(12): 1164-1175
18 Zhang S Y, Zhu S B, Yang L L, Zheng Y, Gao M, Wang S, Zeng J Z, Yan X M. Anal. Chem., 2012, 84(15): 6421-6428
19 Martell J D, Deerinck T J, Sancak Y, Poulos T L, Mootha V K, Sosinsky G E, Ellisman M H, Ting A Y. Nat. Biotechnol., 2012, 30: 1143-1148
20 Mortensen A, Skibsted L H. J. Agr. Food Chem., 1997, 45(8): 2970-2977
21 Bendayan M. Science, 2001, 291: 1363-1365
22 Mayer G, Bendayan M. J. Histochem. Cytochem., 1997, 45: 1449-1454
23 Minamihata K, Goto M, Kamiya N. Bioconjugate. Chem., 2011, 22(11): 2332-2338
24 Rhee H W, Zou P, Udeshi N D, Martell J D, Mootha V K, Carr S A, Ting A Y. Science, 2013, 339: 1328-1331
25 Geiger T, Cox J, Ostasiewicz P, Wisniewski J R, Mann M. Nat. Methods, 2010, 7: 383-385
26 Hung V, Zou P, Rhee H W, Udeshi N D, Cracan V, Svinkina T, Carr S A, Mootha V K, Ting A Y. Mol. Cell, 2014, 55: 332-341
27 Rabilloud T, Kieffer S, Procaccio V, Louwagie M, Courchesne P L, Patterson S D, Martinez P, Garin J, Lunardi J. Electrophoresis, 1998, 19(6): 1006-1014
28 WANG Yuan, SUN HaiDan, RU YaWei, YIN Liang, LIU SiQi. Science China: Lift Sci., 2010, 9: 820-833
王 媛, 孙海丹, 茹雅维, 尹松月, 阴 亮, 刘斯奇. 中国科学: 生命科学, 2010, 9: 820-833
29 van Laar V S, Dukes A A, Cascio M, Hastings T G. Neurobiol. Dis., 2008, 29(3): 477-489
30 RamirezTorres A, BarceloBatllori S, FernandezVizarra E, Navarro M A, Arnal C, Guillen N, Acin S, Osada J. J. Proteomics, 2012, 75(9): 2563-2575
31 Chou J L, Shenoy D V, Thomas N, Choudhary P K, Laferla F M, Goodman S R, Breen G A. J. Proteomics, 2011, 74(4): 466-479
32 Wu Q, Yuan H, Zhang L, Zhang Y. Anal. Chim. Acta, 2012, 731: 1-10
33 Techritz S, Lutzkendorf S, Bazant E, Becker S, Klose J, Schuelke M. Proteomics, 2013, 13(1): 179-195
34 Chang H W, Chuang L Y, Cheng Y H, Gu D L, Huang H W, Yang C H. Genetic Variation. Springer Press, 2010: 259-274
35 Taylor S W, Fahy E, Zhang B, Glenn G M, Warnock D E, Wiley S, Murphy A N, Gaucher S P, Capaldi R A, Gibson B W, Ghosh S S. Nat. Biotechnol., 2003, 21: 281-286
36 Lamb R, Harrison H, Hulit J, Smith D L, Lisanti M P, Sotgia F. Oncotarget, 2014, 5: 11029-11037
37 Mootha V K, Bunkenborg J, Olsen JV, Hjerrild M, Wisniewski J R, Stahl E, Bolouri M S, Ray H N, Sihag S, Kamal M, Patterson N, Lander ES, Mann M. Cell, 2003, 115(5): 629-640
38 Pagliarini D J, Calvo S E, Chang B, Sheth S A, Vafai S B, Ong S E, Walford G A, Sugiana C, Boneh A, Chen W K, Hill D E, Vidal M, Evans J G, Thorburn D R, Carr S A, Mootha V K. Cell, 2008, 134(1): 112-123
39 Bugger H, Schwarzer M, Chen D, Schrepper A, Amorim PA, Schoepe M, Nguyen T D, Mohr F W, Khalimonchuk O, Weimer B C, Doenst T. Cardiovasc Res., 2010, 85(2): 376-384
40 Villeneuve L, Tiede L M, Morsey B, Fox H S. J. Proteome. Res., 2013, 12(10): 4599-4606
41 Kim T Y, Wang D, Kim A K, Lau E, Lin A J, Liem D A, Zhang J, Zong N C, Lam M P, Ping P. Mol. Cell Proteomics, 2012, 11(12): 1586-1594
42 Kumar A, Agarwal S, Heyman J A, Matson S, Heidtman M, Piccirillo S, Umansky L, Drawid A, Jansen R, Liu Y, Cheung K H, Miller P, Gerstein M, Roeder GS, Snyder M. Gene Dev., 2002, 16(6): 707-719
43 Huh W K, Falvo J V, Gerke L C, Carroll A S, Howson R W, Weissman J S, O'Shea E K. Nature, 2003, 425: 686-691
44 Ozawa T, Sako Y, Sato M, Kitamura T, Umezawa Y. Nat. Biotechnol., 2003, 21: 287-293
45 Vieira H L, Haouzi D, El Hamel C, Jacotot E, Belzacq A S, Brenner C, Kroemer G. Cell Death Differ., 2000, 7(12): 1146-1154
46 Tong W H, Rouault T. EMBO J, 2000, 19: 5692-5700
47 Zaltsman Y, Shachnai L, YivgiOhana N, Schwarz M, Maryanovich M, Houtkooper R H, Vaz F M, De Leonardis F, Fiermonte G, Palmieri F, Gillissen B, Daniel P T, Jimenez E, Walsh S, Koehler C M, Roy S S, Walter L, Hajnoczky G, Gross A. Nat. Cell Biol., 2010, 12(6): 553-562
48 Chen T T, Tian X, Liu C L, Ge J, Chu X, Li Y F. J. Am. Chem. Soc., 2015, 137(2): 982-989
49 Rardin M J, Newman J C, Held J M, Cusack M P, Sorensen D J, Li B, Schilling B, Mooney S D, Kahn C R, Verdin E, Gibson B W. Proc. Natl. Acad. Sci. USA, 2013, 110(16): 6601-6606
50 Grimsrud P A, Carson J J, Hebert A S, Hubler S L, Niemi N M, Bailey D J, Jochem A, Stapleton D S, Keller M P, Westphall M S, Yandell B S, Attie A D, Coon J J, Pagliarini D J. Cell Metab., 2012, 16(5): 672-683
51 Deng W J, Nie S, Dai J, Wu J R, Zeng R. Mol. Cell Proteomics, 2010, 9(1): 100-116
52 Johnson D T, Harris R A, French S, Blair PV, You J, Bemis K G, Wang M, Balaban R S. Am. J. PhysiolCell Ph., 2007, 292(2): C689-C697
53 Lotz C, Lin A J, Black C M, Zhang J, Lau E, Deng N, Wang Y, Zong NC, Choi J H, Xu T, Liem D A, Korge P, Weiss J N, Hermjakob H, Yates JR, 3rd, Apweiler R, Ping P. J. Proteome Res., 2014, 13(2): 433-446
54 Capaldi R A, Halphen D G, Zhang Y Z, Yanamura W. J Bioenerg. Biomembr., 1988, 20(3): 291-311
55 Kirby D M, Thorburn D R. Twin Res. Hum. Genet., 2008, 11(4): 395-411
56 Burte F, de Girolamo L A, Hargreaves A J, Billett E E. J. Proteome. Res., 2011, 10(4): 1974-1986
57 Kim H K, Park W S, Kang S H, Warda M, Kim N, Ko J H, Prince Ael B, Han J. Am. J. PhysiolCell Ph., 2007, 293(2): C761-C771
58 Chen Y W, Chou H C, Lyu P C, Yin H S, Huang F L, Chang W S, Fan C Y, Tu I F, Lai T C, Lin S T, Lu Y C, Wu C L, Huang S H, Chan HL. Funct. Integr. Genomic., 2011, 11(2): 225-239
59 Vigano A, Vasso M, Caretti A, Bravata V, Terraneo L, Fania C, Capitanio D, Samaja M, Gelfi C. Proteomics, 2011, 11: 4202-4217
60 Liu T, Chen L, Kim E, Tran D, Phinney B S, Knowlton A A. Life Sci., 2014, 101: 27-36
61 Eccleston H B, Andringa K K, Betancourt A M, King A L, Mantena S K, Swain T M, Tinsley H N, Nolte R N, Nagy T R, Abrams G A, Bailey S M. Antioxid. Redox. Sign., 2011, 15(2): 447-459
62 Alves R M, Vitorino R, Figueiredo P, Duarte J A, Ferreira R, Amado F. J. Gerontol. A Biol. Sci. Med. Sci., 2010, 65(8): 832-842
63 Chappell N P, Teng P N, Hood B L, Wang G, Darcy K M, Hamilton C A, Maxwell G L, Conrads T P. J. Proteome. Res., 2012, 11(9): 4605-4614
64 Lee Y H, Bin Goh W W, Ng C K, Raida M, Wong L, Lin Q S, Boelsterli U A, Chung M C M. J. Proteome. Res., 2013, 12(6): 2933-2945

