启动子陷阱技术在植物启动子克隆研究中的应用
李季++黄天带++华玉伟++黄华孙
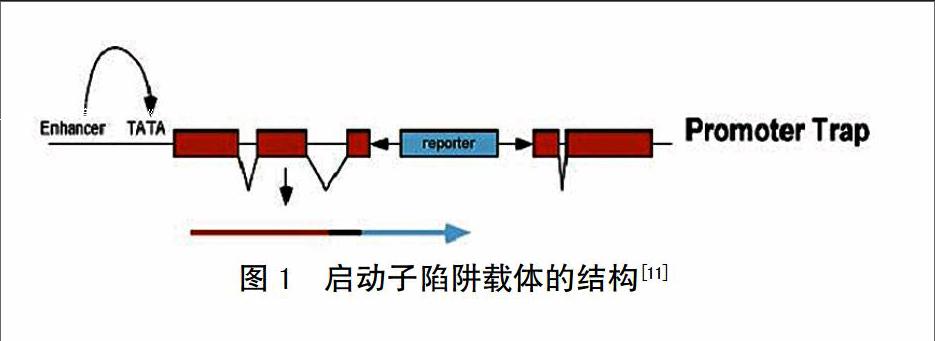
摘 要 启动子陷阱技术是一种报告基因的随机整合技术,现已成为基因功能研究中的重要手段。从启动子陷阱技术的原理、启动子陷阱报告基因的种类、启动子陷阱在植物中的应用及在应用过程中存在的问题等方面进行综述,并针对有关问题进行讨论和展望。
关键词 启动子陷阱 ;T-DNA ;报告基因 ;功能研究
分类号 Q75
Application of Promoter-trap in Promoter Cloning Research of Plants
LI Ji HUANG Tiandai HUA Yuwei HUANG Huasun
(Rubber Research Institute, CATAS /
Key Laboratory of Rubber Biology, Ministry of Agriculture, Danzhou, Hainan 571737)
Abstract Promoter-trap is a random integration of a reporter gene constructs, which provides extremely powerful tools for gene functional research. In this review, the cloning method of promoter, the principle of promoter trap, the choice of reporter gene, and the applications and the questions of promoter-trap were described in detail, In the end, the related problems with prospect were discussed.
Keywords promoter-trap ; T-DNA ; reporter gene ; functional research
启动子是RNA聚合酶能够识别并与之结合,从起始基因转录的一段DNA序列,通常位于基因的上游。目前常用的启动子克隆方法有利用启动子探针载体筛选启动子[1]、PCR法克隆启动子[2-3]及启动子陷阱技术[4-6]等。虽然启动子探针法不需知道具体的基因序列,并且能获得大量的启动子片段,但整个过程工作量大,费时费力。PCR法克隆启动子操作简单、便捷,但必须以已知的基因序列或基因片段为前提。启动子陷阱方法依赖于报告基因的表达来鉴定基因及其启动子,因此不需要已知突变表型和基因序列,广泛应用于细菌、酵母、动物和植物中[7-10]。通过建立高效稳定的启动子捕获系统,构建包含报告基因随机插入整个基因组的个体突变体库,已成为当前未知序列基因启动子克隆的常用方法之一。
1 启动子陷阱技术的原理
启动子陷阱(Promoter-trap)是将一个无启动子的报告基因随机插入到基因组DNA的外显子上,一旦发现其与细胞基因组中的基因被共同转录或表达,则表明该报告基因附近有启动子,从而起到以之为诱饵、发现启动子的目的。报告基因与被插入的基因及其调控序列紧密连锁,这样通过载体上的序列就很容易分离相应的启动子,而报告基因的表达模式也就反映出被插入基因所具有调控序列的特性[11],该技术的利用依赖于报告基因的高频率、多模式表达,为在限制细胞类型表达的基因或在发育过程中短暂时间表达的基因鉴定和克隆提供了强有力的工具[9,12]。启动子陷阱的构建需要建立报告基因插入基因组的个体突变库,主要采取T-DNA和转座子系统2种方式[13-14],而T-DNA介导的遗传转化是主要方法。构建启动子陷阱载体时,一般优先将无启动子的报告基因与T-DNA的右边界相邻,研究结果发现,具有功能的转录基因融合频率为54%,而翻译融合频率为1.6%[15],左边界的无启动子gusA基因在不同植物中的表达频率不同[4]。转座子系统中目前只有Ac/Ds可用于基因陷阱的构建,该系统同样利用T-DNA介导的遗传转化将Ac(活化因子)插入到植物基因组中,Ac/Ds转座子具有偏好向邻近连锁位置进行转座的特性[16],因此,不仅增加了构建突变库的工作量,而且不适合杂交困难的植物。
2 启动子陷阱的报告基因
启动子陷阱中常用的报告基因有GUS(β-葡萄糖醛酸酶)、luc(荧光素酶)、GFP(绿色荧光蛋白)和Lc(花青素调节基因)等。目前,GUS基因是最常用的报告基因,GUS蛋白相当稳定且灵敏,但底物价格较贵,且其在染色和脱色的过程中会破坏组织,不能在活体组织中进行分析[17],这就限制了其在多种植物大规模筛选中的应用。luc或GFP作为一种非破坏性的报告基因已成为基因捕获系统中常见的报告基因[18-19],其无需反应底物和辅助因子,直接通过适当的光源进行荧光检测,可在活细胞中进行多次非破坏性的检测,对植物本身无伤害,但某些植物体内具有高水平的自发荧光[20],且当GFP蛋白含量较高时,转化细胞的再生能力下降,使GFP基因在用于筛选转基因植物时受到局限[21],同时需借助荧光显微镜或激光共聚焦显微镜等比较昂贵的大型设备才能准确的检测。Lc作为玉米Myc-like转录因子的R基因家族成员之一,调控花青素的生物合成,该报告基因不需要昂贵的底物,并且不具有破坏性,在大规模、田间栽培植物中更加具有应用前景,但高表达Lc编码的转录因子会造成表型的影响[22]。
3 启动子陷阱技术的应用
起初启动子陷阱技术主要应用于动物与微生物的基因或启动子克隆方面[23-24],也取得了较大进展[25]。而在植物中起步相对较晚,早期主要是以克隆新的基因为主。Topping等[26]利用启动子陷阱载体研究早代胚胎发生的基因,其中鉴定了一个株系对应的cDNA,表明这个标签序列被转录。随后,Topping等[27]为研究拟南芥胚和籽苗中建立的极性构成机制,利用启动子陷阱鉴定了在根尖顶端至基部表达的3个分子标记。Eastmond等[28]通过启动子陷阱成功的分离了一个新的拟南芥酰基辅酶A氧化酶基因AtACX3,在acx3突变体籽苗中,中链的酰基辅酶A氧化酶活性降低了95%,长链及短链的活性未改变。Meissner等[29]利用玉米的Ac/Ds转座子系统获得了番茄的Ds因子转座并稳定的家系,为高通量分析番茄中的功能基因和启动子的分离鉴定奠定基础,与此同时,利用荧光素酶基因作为报告基因优化了启动子陷阱载体。Tanaka等[30]从NASC提供的启动子陷阱系中重新分离了一个N35株系,其GUS报告基因在下胚轴表达,并且表现为依赖于远红外光处理。分析结果发现,报告基因插入到拟南芥同源的大豆GH3基因,RNA blot分析进一步证实,该基因确实对光照存在反应。
与此同时,利用启动子陷阱技术克隆有功能的植物自身启动子的策略是可行的[31]。Jeon等[32]尝试用无启动子的GUS基因构建在T-DNA上,通过T-DNA的随机插入,来分离水稻中的启动子。Bade等[33]将一个无启动子的gus::nptII陷阱载体转化甘蓝型油菜,选择3个T-DNA插入单拷贝的株系进行了启动子的分离,通过瞬时和稳定的GUS活性检测证明这些新的序列具有启动子活性。Alvarado等[18]利用携带有无启动子的luc报告基因的启动子陷阱载体pTluc转化拟南芥,获得具有组成型、组织特异型和胁迫诱导型的荧光素酶表达模式的转基因株系。Jin等[5]研究结果初步证明启动子陷阱技术在棉花功能基因组学研究中的可行性,并提出建立的启动子陷阱系统中GUS基因高频率、多模式和时空特异性表达,这为分离基因及其调控序列,开展棉花功能基因组学奠定研究基础。Santos等[19]设计并应用一种高通量的real-time结合荧光素酶依赖的启动子陷阱载体的体外筛选平台,鉴定和分离香蕉低温诱导表达的启动子,该系统或类似的系统可成功的应用于具有较好体外再生体系的物种。魏小慧等[34]利用启动子捕获技术建立了橡胶树炭疽病菌的遗传转化体系,提出启动子捕获技术更适合开展植物病原菌致病分子生物学的研究。同时抗性宿主基因型中类似转录激活效应子识别位点最常用策略为利用启动子陷阱调取与执行类型抗性基因(E基因)相结合的启动子,如水稻的Xa27基因和胡椒的Bs3基因[6]。在木本植物橡胶树中,也首次通过启动子陷阱技术初步获得了1个组成型表达启动子和1个根特异性表达启动子[35]。
4 启动子陷阱在基因组学研究中的问题及展望
4.1 问题
4.1.1 启动子或基因的确定较困难且耗时较长
通过启动子陷阱载体可挖掘相应的启动子或基因,但通过克隆和筛选融合转录物获融合蛋白费时费力。目前较为常见的是通过Tail-PCR方法来获得其插入的侧翼序列,然后通过生物信息学分析获得启动子或基因,但Tail-PCR方法在反应过程中需要较多的引物组合。由于AD(Arbitrary degenerate,随机简并)引物存在有限的结合位点,对个别的侧翼序列,即便用不同的简并引物也难以扩增到阳性结果。目前提出用RAPD引物替代AD引物的观点,由于RAPD引物数量很多,所以可解决AD引物存在有限的结合位点的不足问题,此外,Tail-PCR法对引物和模板纯度的要求较高且扩增的片段为高度重复序列时,该方法无法步移。
4.1.2 可导致基因的失活或报告基因表型丧失
启动子陷阱载体要求插入在一个基因的外显子上,可能会导致基因失活。启动子捕获载体具有DNA插入的偏向性,且只能捕获有转录活性的基因[36]。其需要插入的方向正确,而在T-DNA的左右两侧分别插入1个无启动子的报告基因,是否可能会避免由于T-DNA插入方向的不正确造成报告基因无法表达的问题还有待于进一步验证[35]。若报告基因未按预期加以整合,或者报告基因被剪接时整个被切除,都有可能使报告基因的表型丧失[37]。
4.1.3 依赖于转基因体系的建立
启动子陷阱技术成功应用的先决条件是具备有效的植物转化机制。目前T-DNA介导的转化仍是常用方法,模式植物的遗传转化体系比较成熟,但木本植物的转化起步较晚,其遗传转化体系还不够完善,转化效率较低,这对于由启动子陷阱的遗传转化获得阳性植株带来一定的困难。
4.1.4 多拷贝植株分析的复杂性
利用T-DNA介导的遗传转化技术转化启动子陷阱载体,在获得的转化植株中,通常可检测到一个位点包含多个T-DNA插入或者多个位点包含有多个插入,甚至发生T-DNA的正向或反向重复,以及与比邻的染色体发生重排等,这些都给后期的分子分析带来较大的复杂性,增加了启动子的分离难度[9,38]。
4.2 展望
自启动子陷阱技术出现后,用其进行启动子及基因的克隆一直受到研究者的青睐。传统遗传学方法并不是对所有的重要功能基因都能进行研究,其中包括低水平表达的、在发育各个阶段均发挥重要作用的重要基因,突变后可能会导致在早期致死或高度多效性及在实验室条件下功能上融合表达,基因缺失后,成为表型变化不明显的功能冗余基因。而启动子陷阱技术可很好的解决上述基因的研究,提高标记基因插入基因组的机率,并不需要产生可见的功能缺失突变体,可很好的分离特异性表达的基因,具有更为广泛的应用前景。其次,利用启动子陷阱技术构建的突变体库也将为利用反向遗传学研究基因功能奠定基础,获得的突变体将为分子生物学研究提供良好材料。近期的研究结果表明,内源启动子与受体系统兼容性较好,在目的基因高效、稳定及持久表达等方面较外源启动子更具优势[39]。而启动子陷阱技术是挖掘内源启动子强有力的工具,将会对内源组成型启动子、组织特异型启动子或诱导型启动子的挖掘等方面研究发挥更大的作用。
参考文献
[1] Donna M W, Elizabeth J D, Paul S L. Cloning restriction fragments that promote expression of a gene in Bacillus subtilis[J]. J Bacterol, 1981, 146(6): 1 162-1 165.
[2] Kim M J, Kim H, Shin J S, et al. Seed-specific expression of sesame microsomal oleic acid desaturase is controlled by combinatorial properties between negative cis-regulatory elements in the SeFAD2 promoter and enhancers in the 5′-UTR intron[J]. Mo1ecular Genetics and Genomics, 2006, 276(4): 351-368.
[3] Zhang J, Guo D, Chang Y X, et al. Non-random distribution of T-DNA insertions at various levels of the genome hierarchy as revealed by analyzing 13804 T-DNA flanking sequences from an enhancer-trap mutant library[J]. Plant J, 2007, 49(5): 947-959.
[4] Topping J F, Wei W, Lindsey K. Functional tagging of regulatory elements in the plant genome[J]. Development, 1991, 112(5): 1 009-1 019.
[5] Jin S X, Zhang X L, Nie Y C, et al. Functional analysis of promoter trapping system after inserted into cotton(Gossypium hirsutum L.)genome[J]. Acta Genetica Sinica, 2005, 32(12): 1 266-1 274.
[6] Schornack S, Moscou M J, Ward E R, et al. Engineering plant disease resistance based on TAL effectors[J]. Annu Rev Phytopathol, 2013, 51(2):383-406.
[7] Moore D, Wu J H, Kathir P, et al. Analysis of transfer genes and gene products with the traB-traC region of the Escherichia coli fertility factor, F[J]. J Bacteriol, 1987, 169: 3 994-4 002.
[8] Vida T A, Graham T R, Emr S D. In vitro reconstitution of intercompartmental protein transport to the yeast vacuole[J]. J Cell Biol, 1990, 111: 2 781-2 884.
[9] Lindsey K, Wei W, Clarke M C, et al. Tagging genomics sequences that direct transgene expression by activation of a promoter trap in plants[J]. Transgenic Res, 1993, 2(1): 33-47.
[10] Bellen H J. Ten years of enhancer detection:Lessons from the fly[J]. Plant Cell, 1999, 11(11): 2 271-2 281.
[11] Springer P S. Gene traps tools for plant development and genomics[J]. Plant Cell, 2000, 12, (7): 1 007-1 020.
[12] 胡昌泉,刘华清,李 素,等. 水稻T-DNA插入启动子捕获系的初步筛选[J]. 福建农业学报, 2011, 26(2):143-147.
[13] Koncz C, Martini N, Mayerhofer R. High frequency T-DNA-mediated gene tagging in plants[J]. Proc Natl Acad Sci USA, 1989, 86: 8 467-8 471.
[14] Fedoroff N V, Smith D L. A versatile system for detecting transposition in Arabidopsis[J]. The Plant Journal, 1993, 3(2): 273-289.
[15] Kertbundit S, De Greve H, Deboeck F. In vivo random beta-glucuronidase gene fusions in Arabidopsis thaliana[J]. Proc Natl Acad Sci, 1991, 88(12): 5 212-5 216.
[16] Greenblatt I M. A chromosome replication pattern deduced from pericarp phenotypes resulting from movements of the transposable element Modulator in maize[J]. Genetics, 1984, 108(3): 471-485.
[17] Jefferson R A, Kavanagh T A, Bevan M W. GUS fusions:β-glucuronidase as a sensitive and versatile gene fusion marker in higher plants[J]. EMBO J, 1987, 6: 3 901-3 907.
[18] Alvarado M C, Zsigmond L M, Kovács I, et al. Gene trapping with firefly luciferase in Arabidopsis. Tagging of stress-responsive genes[J]. Plant Physiology, 2004, 134(1): 18-27.
[19] Santos E, Remy S, Thiry E, et al. Characterization and isolation of a T-DNA tagged banana promoter active during in vitro culture and low temperature stress[J]. BMC Plant Biology, 2009, 9(1): 77-92.
[20] 谭德冠,吴 煜,张家明. 巴西橡胶树离体材料的绿色荧光背景研究[J]. 热带作物学报,2008,29(1):23-26.
[21] 宫 硖,杨凤萍,薛 静,等. 花青素合成转录因子基因在玉米中的表达研究:一种新型基因可视化跟踪表达系统[J]. 科学通报,2012,57(24):2 285-2 291.
[22] Goldsbrough A P, Tong Y, Yoder J I. Lc as a non-destructive visual reporter and transposition excision marker gone for tomato[J]. The Plant Journal, 1996, 9(6): 927-933.
[23] Skarnes W C. Entrapment vectors:A new tool for mammalian genetics[J]. Bio Technology, 1990, 8(5): 827-831.
[24] Chang W T, Gross J D, Newell P C. Trapping development promoters in Dictyostelium[J]. Plasmid, 1995, 34(1): 175-183.
[25] Yadav A, Sood S, Shrivastava R. Promoter trap strategy for gene expression analysis under stress conditions of M. tuberculosis latency[J]. BMC Infectious Diseases, 2014, 14(S 3):13. DOI:10.1186/1471-2334-14-S3-O13.
[26] Topping J F, Agyeman F, Henricot B. Identification of molecular markers of embryogenesis in Arabidopsis thaliana by promoter trap[J]. The Plant Journal, 1994, 5(6):895-903.
[27] Topping J F, Lindsey K. Promoter trap markers differentiate structural and positional components of polar development[J]. The Plant Cell, 1997, 9(8): 1 713-1 725.
[28] Eastmond P J, Hooks M A, Williams D. Promoter trapping of a novel medium-chain Acyl-CoA oxidase, which is induced transcriptionally during Arabiopsis seed germination[J]. The Journal of Biological Chemistry, 2000, 275:34 375-34 381.
[29] Meissner R, Chague V, Zhu Q H, et al. A high throughput system for transposon tagging and promoter trapping in tomato[J]. The Plant Joural, 2000, 22(3): 265-274.
[30] Tanaka S, Mochizuki N, Nagatani A. Expression of the AtGH3a gene Arabidopsis homologue of the soybean GH3 gene, is regulated by phytochrome B[J]. Plant Cell Physiology, 2002, 43(3):281-289.
[31] Girijashankar V, Sharma H C, Sharma K K, et al. Development of transgenic sorghum for insect resistance against the spotted stem borer(Chilo partellus)[J]. Plant Cell Rep, 2005, 24(3): 513-522.
[32] Jeon J S, Lee S, Jung K H, et al. T-DNA insertional mutagenesis for functional genomics in rice[J]. Plant J, 2000, 22(6): 561-570.
[33] Bade J, van Grinsven E, Custer J, et al. T-DNA tagging in Brassica napus as an efficient tool for the isolation of new promoters for selectable marker genes[J]. Plant Molecular Biology, 2003, 52(1): 53-68.
[34] 魏小慧,林春花,郑肖兰, 等. 利用启动子捕获技术构建橡胶炭疽病菌突变体库[J]. 热带作物学报,2009,30(6):804-810.
[35] 华玉伟,杨加伟,黄天带,等. 启动子陷阱技术在橡胶树中的应用研究 [J]. 热带作物学报,2013,34(8):1-7.
[36] 王明科,孙慧勤,粟永萍,等. 基因捕获技术的现状及应用[J]. 中国生物工程杂志,2014,34(12):107-111.
[37] 方 芳,胡英考,张飞雄. 基因陷 [J]. 生命的化学,2003,23(3):208-210.
[38] Laufs P, Autran D, Trims J. A chromosomal paracentric inversion associated with T-DNA integration in Arabidopsis [J]. Plant J, l999, 18: 131-139.
[39] Hirata R, Jeong Won-Joong, Saga N, et al. Heterologous activation of the Porphyra tenera HSP70 promoter in Bangiophycean algal cells[J]. Bioengineered, 2011, 2(5): 271-274.

