单克隆抗体依那西普融合蛋白的活性结构保护
潘 琦,李代禧,郭柏松,杨春生,杨 智
( 1.上海理工大学食品科学与工程研究所,上海200093;2.上海东富龙科技股份有限公司冻干工艺研究室,上海201108)
单克隆抗体依那西普融合蛋白的活性结构保护
潘琦1,李代禧1,郭柏松2,杨春生2,杨智2
( 1.上海理工大学食品科学与工程研究所,上海200093;
2.上海东富龙科技股份有限公司冻干工艺研究室,上海201108)
摘要采用拉伸模拟和伞状采样的方法,利用Gromacs软件和Amber99sb-ildn分子力场研究了不同浓度的海藻糖体系和海藻糖-甘露醇复合体系中单克隆依那西普融合蛋白二聚体的解离过程.结果表明,海藻糖能显著增强依那西普二聚体活性结构的稳定性,且海藻糖-甘露醇的复合保护优于海藻糖的单一保护;保护剂的种类及其选择性吸附保护的特定位置对依那西普蛋白二聚体活性结构保护效果的影响显著.
关键词依那西普;拉伸模拟;伞状采样;海藻糖
依那西普( Etanercept)是1998年由美国食品药品监管局( FDA)批准用于类风湿性关节炎[1]、银屑病性关节炎[2]、强直性脊柱炎[3]和幼年慢性关节炎[4]等疾病的抗体药物.该药物是以中国仓鼠细胞表达系统产生的人肿瘤坏死因子( TNF-α)受体P75与IgG Fc段结合的二聚体融合蛋白,由931个氨基酸组成,相对分子质量为1. 5×105[5].其作用机制是通过与TNF-α竞争性结合,抑制其生物活性,从而阻断TNF-α介导的细胞反应[6].
市售的依那西普一般为无菌、白色、不含防腐剂的冻干粉末,其储存温度为2~8℃,溶解后应立即使用;如果未立即使用,应将溶解后的依那西普注射液贮存于2~8℃,最长可保存6 h.依那西普抗体蛋白复溶后的稳定性差,容易分解成单体.
在恶劣条件下,为稳定抗体药物分子的活性结构,通常会加入一些糖类保护剂,如海藻糖( THL)、蔗糖( SUC)、甘露醇( MAN)和山梨醇( SOR)等作为辅料[7],但保护机理尚不清楚.为此,本文以依那西普为研究对象,以海藻糖和甘露醇为保护剂,研究了低温下保护剂对单克隆抗体融合蛋白活性结构的保护作用与规律,分析了不同保护剂的保护效果和作用机理,以期为抗体蛋白辅料配方的筛选和优化提供技术支持.
1 实验部分
1.1结构准备
采用上海理工大学计算生物学实验室的五舟高性能服务器集群作为高性能并行计算的计算平台.集群共有24个计算节点,每个计算节点有2颗Intel XeonE5-2650处理器( 2. 0 GHz,每颗8核心).
选择依那西普晶体结构( PDB ID: 2DTQ)溶剂化后的模型作为保护体系的初始模型.其晶体结构由X射线衍射分析得到,分辨率为0. 2 nm,获取地址为http: / /www.rcsb.org/pdb/files/2dtq.pdb.结构[图1( A)]尾部2个单体由3个二硫键相连,通常二硫键易发生可逆氧化还原反应,属于氧化还原保护.因此,在建模处理时剔除了二硫键相连的部分.保护剂海藻糖[图1( B)]和甘露醇[图1( C)]的晶体结构采用Gaussion09软件包在B3LYP/6-31G*方法下进行结构优化,优化后的结构为最终结构[8].
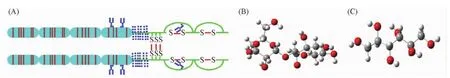
Fig.1 Sketch of monoclonal antibody fusion protein etanercept( A) and the structure of trehalose( B) and mannitol( C)
1.2计算过程
利用Gromacs4.6生物大分子模拟软件[9]采用拉伸模拟( SMD)[10~12]和伞状采样( US)[13,14]方法,在恒温恒容条件下完成保护剂对依那西普融合蛋白的保护过程.在研究海藻糖单一保护时,不同体系中的海藻糖按线性增加的趋势添加,而复合保护中保护剂的比例参考糖和多元醇结合的依那西普稳定配方专利[15],每个体系的组成如表1所示.其中依那西普采用Amber99sb-ildn分子力场[16],海藻糖和甘露醇采用Amber通用分子力场[17],水分子采用TIP3P模型[18].范德华力和静电相互作用的截距分别设为1. 4和1. 0 nm,为了修正范德华力的远程相互作用,将邻近作用半径设为1. 0~1. 4 nm.另外,在模拟中采用Particle Mesh Ewald( PME)方法[19]用于静电相互作用的修正.
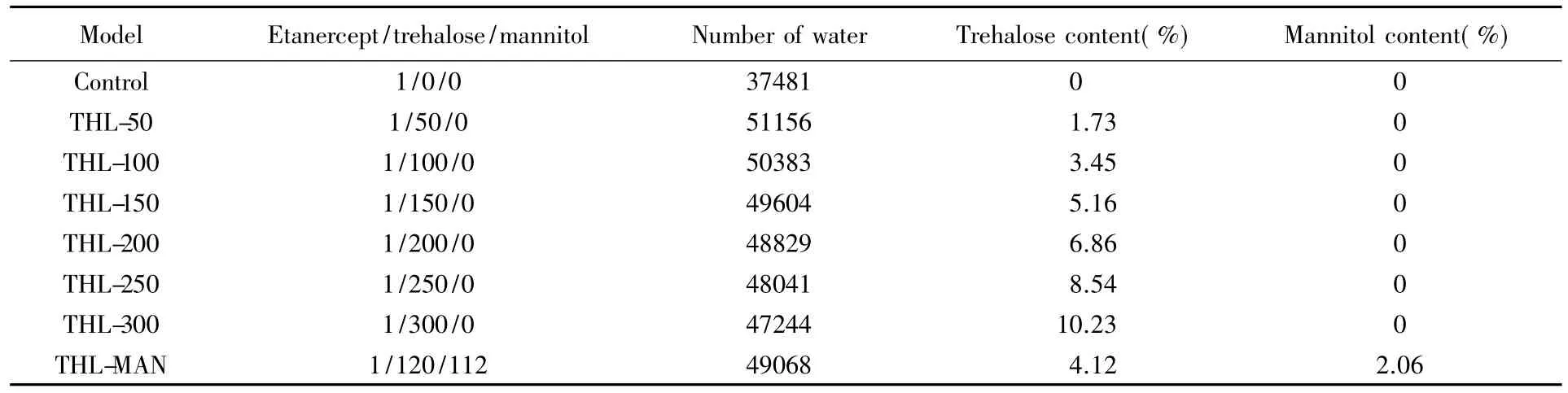
Table 1 Models for simulation and the detail components
首先,建立20. 2825 nm×10. 2579 nm×7. 8697 nm的周期性体系.每个体系分别进行0. 2 ns的结构最小化,再以LINear Constraint Solver( LINCS)方法[20]限制所有化学键的键长,利用Maxwell分布方法[21]设置体系各原子的初始速度,并通过Velocity-rescale方法[22]控制体系的温度为300 K,耦合常数为0. 1,同时采用Berendsen方法[23]设定体系的压强耦合常数为0. 5.对水溶液体系进行0. 5 ns升温模拟[24]和5 ns的预平衡,再平衡40 ns,得到均匀稳定的依那西普融合蛋白的水溶液体系.然后设定时间步长为1 fs,拉伸速率为0. 5 nm/ns,简谐力常数为1000 kJ/( mol·nm2),在恒温恒容条件下进行x轴方向的拉伸解离.根据拉伸解离的轨迹,每隔0. 1 nm提取1个体系构象,每个体系提取100个体系构象.针对每个构象分别进行伞状采样模拟,利用加权直方图分析方法( WHAM)[25]得到依那西普融合蛋白二聚体的合理Potential of Mean Force( PMF)解离自由能[26].
2 结果与讨论
2.1海藻糖和甘露醇在依那西普蛋白表面的吸附作用
海藻糖是一种优良的生物活性稳定剂.从溶液结构上看,在添加有依那西普融合蛋白的海藻糖均匀溶液中,经过系统平衡后,海藻糖分子会自动吸附在依那西普抗体的蛋白表面,以实现其稳定和保护蛋白活性结构的功能.
通常,形成有效氢键的最大距离是0. 35 nm,能够发生较强分子间库仑作用和范德华力的最长距离分别是1. 00和1. 40 nm.基于此,分别统计了各体系中距离依那西普蛋白表面0. 35,1. 00和1. 40 nm范围内吸附的保护剂分子数目,如图2所示.
在海藻糖单一保护体系中,厚度为0. 35 nm保护剂层内的保护剂分子与蛋白表面有直接作用,其吸附效果和数目对蛋白药物活性结构的保护起着重要作用.由统计数据可知,各模拟体系中保护层内保护剂的数目变化不大,平均吸附分子个数仅为16个.这说明大部分蛋白表面直接被水分子占据.而在复合保护体系中,该层吸附海藻糖分子10个,甘露醇分子17个,共27个保护剂分子与蛋白表面直接作用.复合保护体系中直接吸附的保护剂分子数目和蛋白表面都明显增加,致使复合保护效果得到有效提高.
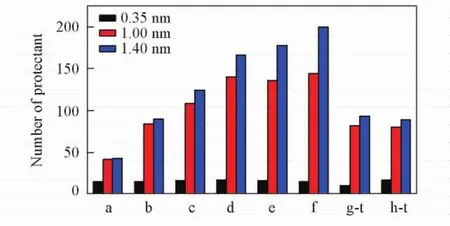
Fig.2 Numbers of protection in different thickness of protective layer around atanercept in each systema-f respectively represents the etanercept solvation systems with 50,100,150,200,250 and 300 molecular numbers of trehalose.g-t and g-m respectively represents trehalose and mannitol in complex protection system.
在厚度为1. 00 nm保护层内的保护剂分子数目随着保护剂的添加呈现先增大后稳定的趋势;而在厚度为1. 40 nm保护层内的保护剂分子数目随着保护剂添加数量的增加而增大.此结果表明,不同厚度的保护层可容纳的保护剂分子数目有限.
在海藻糖-甘露醇复合保护体系中,距离依那西普蛋白1. 00~1. 40 nm范围内的2种保护剂的吸附量基本相当,海藻糖和甘露醇分子的平均吸附个数分别是81和91.复合保护体系中,120个海藻糖分子和112个甘露醇分子的总吸附量比加入250个海藻糖分子单一保护体系的吸附量多,进一步表明复合保护的优势.
在不同保护剂浓度的依那西普溶液体系中,保护剂分子对抗体蛋白表面的选择性吸附情况如图3所示.可知,大量的保护剂分子可以直接或间接地吸附在依那西普抗体的蛋白表面.在海藻糖单一保护的体系中,随着海藻糖的添加,保护剂分子对抗体蛋白的吸附面积逐渐增加[图3( A)~( D)].当添加的海藻糖较多时,海藻糖分子会在抗体蛋白部分表面产生聚集[图3( E)和( F)],而不是全面吸附和覆盖在整个蛋白表面.说明保护剂在蛋白药物分子表面的吸附保护是有区域选择性的.
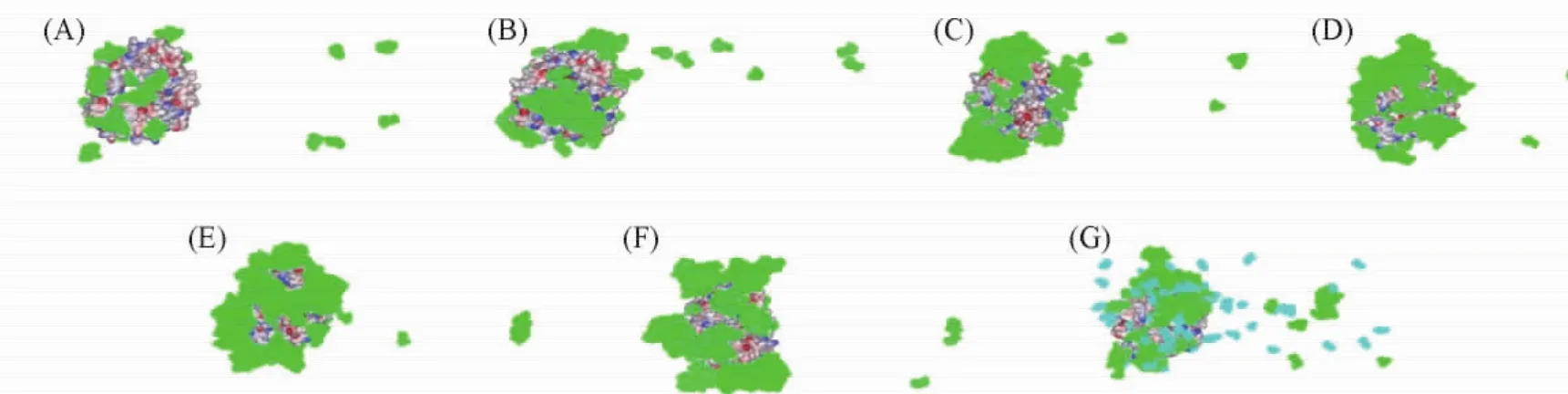
Fig.3 Regional selective adsorption of protectant onto the surface of etanercept in different systems( A)—( F) represent the etanercept solvation systems with 50,100,150,200,250 and 300 molecular numbers of trehalose.( G) is trehalose and mannitol complex protection system.Closed electrostatic potential surface: etanercept; green: trehalose; cyan: mannitol.In order to show clearly,the molecular of water in systems is removed.
当用海藻糖和甘露醇对依那西普进行复合保护时,由于2种保护剂在蛋白表面的选择性吸附区域不同,会交叉协同地吸附在蛋白表面[图3( G)],因此吸附面积更大,分子间的相互作用会更强,实验结果表明其保护效果也更好[27].
2.2依那西普融合蛋白二聚体在不同体系中的稳定性
依那西普融合蛋白二聚体在不同溶液体系中解离时2个单体间拉力随着解离时间的变化曲线见图4,2个单体间最大拉力的柱形图见图5.由图5可知,与未加保护剂的体系相比,在加入保护剂的溶液体系中依那西普蛋白解离所需的最大拉力明显增大.这表明添加的海藻糖对依那西普蛋白二聚体活性结构的稳定性起到了保护效果.在海藻糖单一保护的溶液体系中,随着海藻糖的加入,2个单体解离所需的最大拉力增加,表明抗体蛋白的稳定性随着保护剂的增加而增大.在加入200个海藻糖保护剂分子(即海藻糖的质量分数为6. 86%)之后,2个单体解离所需的最大拉力不再明显增加,这一趋势与厚度为1. 00 nm保护层内的保护剂分子吸附个数随着保护剂添加量的变化趋势一致.此结果表明,海藻糖保护剂的添加基本达到了保护效果的上限,继续添加海藻糖不能提升保护效果.
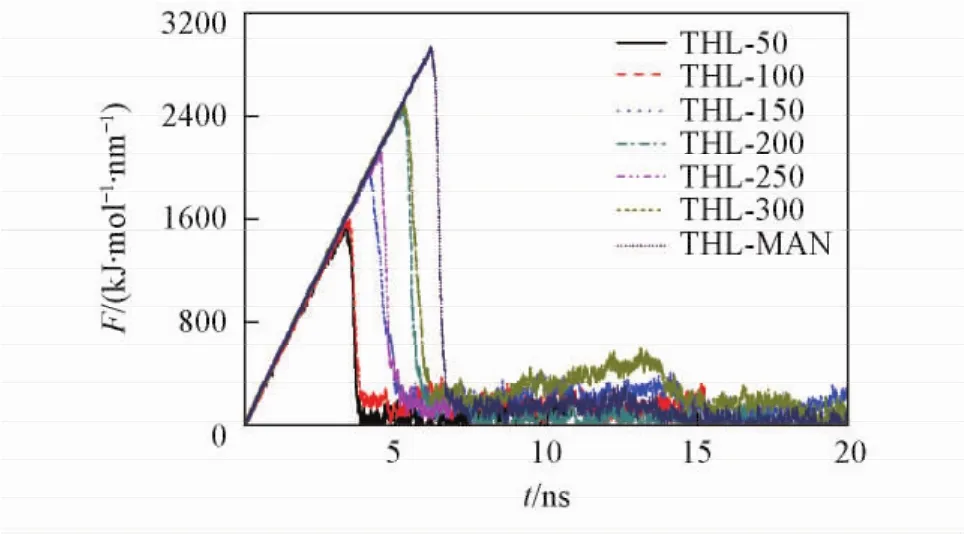
Fig.4 Curves of the pull force for dissociating etanercept dimer in different systems
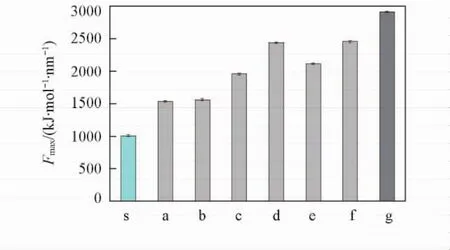
Fig.5 Maximum pull force for dissociating etanercept dimer in different systems.THL-0; a.THL-50; b.THL-100; c.THL-150; d.THL-200; e.THL-250; f.THL-300; g.THL-MAN.
将海藻糖单一保护与海藻糖-甘露醇复合保护相比,依那西普蛋白二聚体在复合保护体系中解离所需的最大拉力明显高于单一保护体系,表明复合保护效果要优于单一保护效果.这是因为在海藻糖单一保护时,与抗体蛋白有效作用范围内的保护剂分子数目有限所致.当保护剂与蛋白表面残基的直接保护作用达到饱和时,继续增加保护剂的数目,保护剂则会在抗体蛋白部分表面聚集,使保护剂层增厚,保护剂对抗体蛋白的间接保护作用开始显现,但保护效果不再明显提高.但是在复合保护时,2种保护剂的分子结构和大小存在差异,保护剂会交叉和协同地吸附在抗体蛋白表面,选择性吸附的面积同样增大,保护剂与抗体蛋白表面直接作用的残基数增加,因而复合保护效果明显强于单一保护效果.
2.3依那西普蛋白二聚体在不同保护体系中的PMF解离自由能
依那西普蛋白二聚体在不同保护体系中的PMF解离自由能变化图和平均PMF解离自由能分别示于图6和表2.
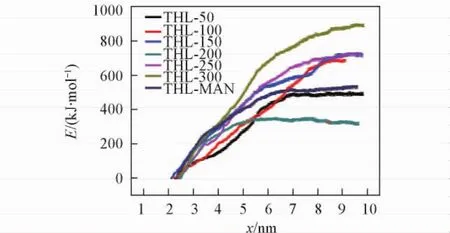
Fig.6 PMF free energy of etanercept dimer during dissociation in different systems
由图6可知,在海藻糖单一保护体系中,随着海藻糖的添加,依那西普蛋白二聚体的解离自由能逐渐提高,与解离所需最大拉力的变化趋势一致,但是在超过了150个海藻糖分子添加量的体系中(即海藻糖保护剂的浓度为5. 16%),依那西普抗体的PMF解离自由能出现一个平台,这表明海藻糖对于依那西普抗体的有效保护存在最佳浓度范围.
由表2可知,添加海藻糖保护的依那西普的PMF解离自由能明显高于参照体系,表明海藻糖能显著提高依那西普蛋白二聚体的热力学稳定性,起到保护依那西普活性结构的效果.而从复合保护体系的PMF解离自由能来看,复合保护的依那西普蛋白二聚体的热力学稳定性反而降低,这可能是由于在复合保护中,在有效保护范围内的保护剂中约1/2为甘露醇,并且甘露醇对依那西普蛋白二聚体的热力学稳定性的影响作用不大.
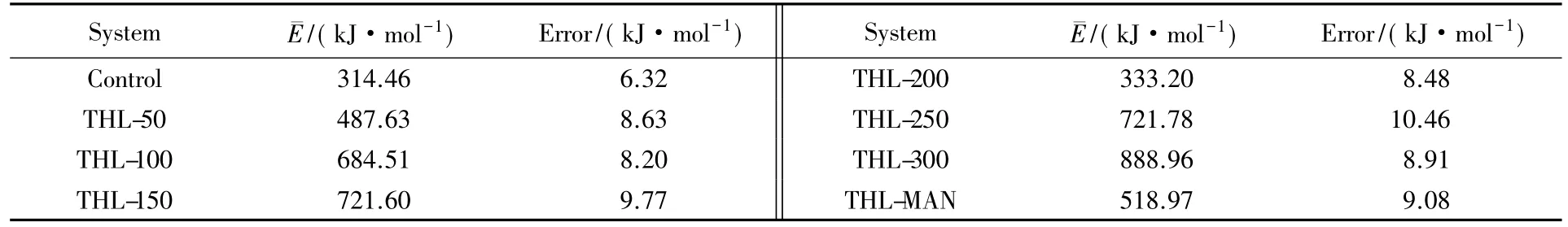
Table 2 Average PMF free energies and errors in etanercept dissociation in different systems
在添加了200个海藻糖分子的单一保护体系中,依那西普蛋白二聚体的解离自由能反而明显下降,可能由于保护剂在依那西普蛋白二聚体分子表面的吸附位置不同所致.
2.4保护剂的吸附位置对依那西普蛋白二聚体解离自由能的影响
为了探究依那西普蛋白二聚体在添加了200个海藻糖分子的溶液体系中解离自由能不增反降的原因,进一步分析了保护剂的吸附位置对依那西普蛋白二聚体解离自由能的影响.在海藻糖单一保护体系和海藻糖-甘露醇复合保护体系中,保护剂在距离依那西普抗体表面0. 35 nm范围内的吸附情况如图7所示.可见,每个体系中保护剂可以吸附在抗体蛋白2个单体相互接触的界面处或吸附在抗体蛋白单体分子表面.当保护剂吸附在2个单体相互接触的界面处时,对二聚体稳定性影响最佳.在添加200个海藻糖分子的体系中海藻糖虽然吸附在抗体蛋白的表面,但并未有效吸附在2个单体之间接触的部位[图7( D)],导致其PMF解离自由能无明显提高.

Fig.7 Adsorption of protectant within the range of 0. 35 nm from etanercept dimer surface( A)—( F) represent the etanercept solvation systems with 50,100,150,200,250 and 300 molecular numbers of trehalose.( G) is trehalose and mannitol complex protection system.Dimer with scarlet and indigo: etanercept; green: trehalose; cyan: mannitol.In order to show clearly,the molecular of water in systems is removed.
另外设计了200个海藻糖保护体系,在此体系中依那西普2个单体之间的交界处有海藻糖的吸附,并通过相同计算加以验证.将海藻糖吸附在2个单体的界面处[图8( A)],其平均解离自由能得到明显提高[708. 85 kJ/mol,见图7( B)].比第一个体系( 333. 20 kJ/mol)提高约375. 65 kJ/mol.可见,保护剂的吸附位置显著影响依那西普抗体蛋白的热力学稳定性.
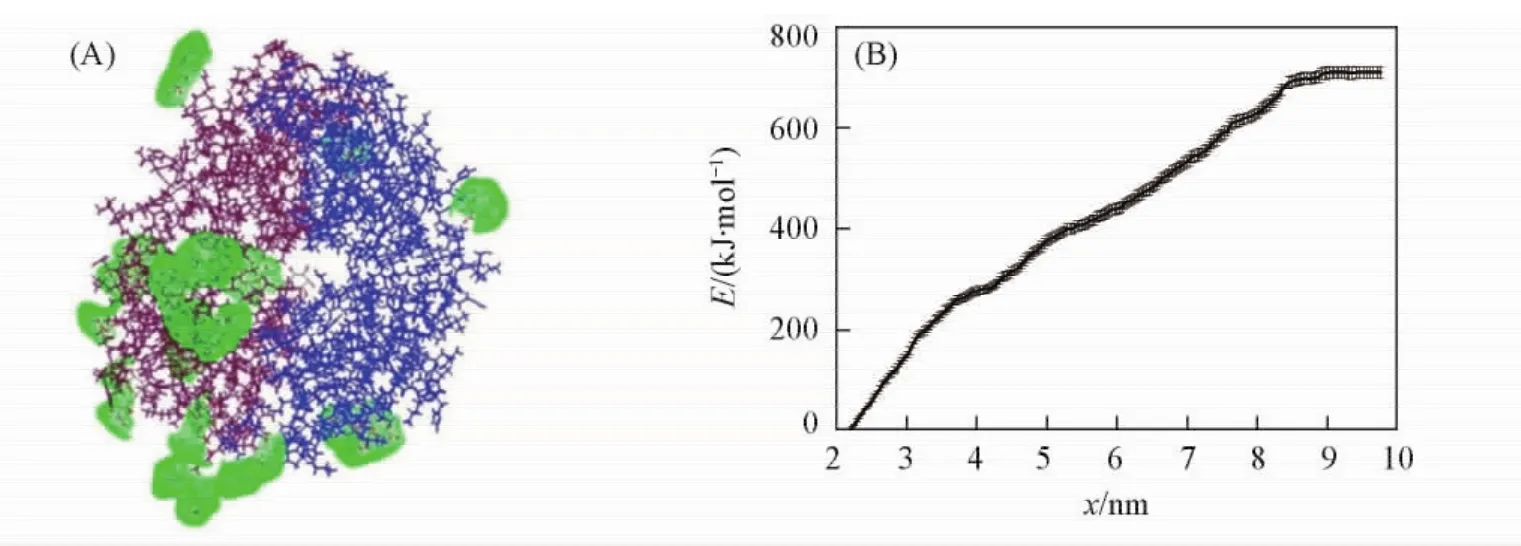
Fig.8 Effect of trehalose adsorptive location on the structural stability of etanercept dimer( A) Adsorption of trehalose within the range of 0. 35 nm from the etanercept dimer surface;( B) PMF free energy of etanercept dissociation in new system.
在海藻糖-甘露醇复合保护体系中,在吸附依那西普抗体2个单体相接触界面上较多的保护剂是甘露醇而不是海藻糖[图7( G)].为考察在关键吸附位置保护剂的种类对依那西普抗体蛋白热力学稳定性的影响,将依那西普抗体2个单体交界处换为海藻糖[图9( A)],用相同方法计算得到新复合保护体系中依那西普的平均解离自由能值为593. 97 kJ/mol[图8( B)].与未更换保护剂种类前相比( 518. 97 kJ/mol,表2),解离自由能提高约75 kJ/mol.在厚度为0. 35 nm的吸附保护层中,海藻糖与甘露醇的吸附分子数目分别是11和18个,与第一个复合保护体系相比,海藻糖与甘露醇的吸附数目均有所增加.这表明保护剂的种类和在抗体蛋白表面的吸附位置对蛋白药物活性结构稳定性的保护效果具有重要影响.
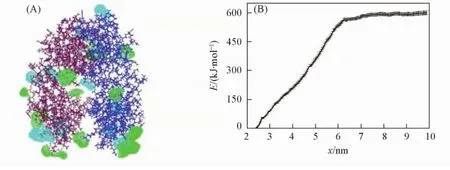
Fig.9 Effect of adsorptive location on the structural stability of etanercept dimer( A) Adsorption of protectant within the range of 0. 35 nm from the etanercept dimer surface;( B) the PMF free energy of etanercept dissociation in new complex system.
采用拉伸模拟和伞状采样方法研究不同保护体系中依那西普二聚体的解离过程可以直观有效及定量地揭示保护剂数目、种类和吸附位置对抗体蛋白活性结构热力学稳定性的影响.相对于解离自由能,最大解离拉力更能有效地判断蛋白药物活性结构的稳定性.动力学比热力学更能有效地判断活性结构保护剂对蛋白药物的保护作用.同时,该理论研究结果和前期依那西普稳定配方专利[15]的实验结果一致.上述结果表明,运用拉伸模拟和伞状采样方法研究结果综合判定活性结构保护剂的保护效果是可行的.
3 结 论
将拉伸模拟和伞状采样方法相结合研究了不同数量的海藻糖单一保护体系和海藻糖-甘露醇复合保护体系中依那西普蛋白二聚体的解离过程.结果表明,海藻糖能保护依那西普二聚体活性结构,且海藻糖-甘露醇复合保护的效果优于海藻糖单一保护;活性结构保护剂对蛋白药物的保护作用是通过对特定关键部分的选择性非亲和弱吸附作用的体现;动力学比热力学更能直接和有效地评价与判断活性结构保护剂对蛋白药物的保护作用.
参考文献
[1]Goldenberg M.M.,Clin.Ther.,1999,21,75—87
[2]Mease P.J.,Goffe B.S.,Metz J.,Vanderstoep A.,Finck B.,Burge D.J.,Lancet,2000,356,385—390
[3]Heijde D.V.D.,Silva J.C.D.,Dougados M.,Geher P.,Horst-Bruinsma I.V.D.,Juanola X.,Olivieri I.,Raeman F.,Settas L.,Sieper J.,Szechinski J.,Walker D.,Boussuge M.P.,Wajdula J.S.,Paolozzi L.,Fatenejad S.,Ann.Rheum.Dis.,2006,65( 12),1572—1577
[4]Pirjo T.,Lindahl P.,Honkanen V.,Lahdenne P.,Kotaniemi K.,Ann.Rheum.Dis.,2007,66( 4),548—550
[5]Goffe B.,Cather J.C.,J.Am.Acad.Dermatol.,2003,49( 2S),S105—S111
[6]Wong M.,Ziring D.,Korin Y.,Desai S.,Kim S.,Lin J.,Gjertson D.,Braun J.,Reed E.,Singh R.R.,Clin.Immunol.,2008,126 ( 2),121—136
[7]Feofilova E.P.,Usov A.I.,Mysyakina I.S.,Kochkina G.A.,Microbiology,2014,83( 3),271—283
[8]Wu Y.J.,Cui Y.L.,Zheng Q.C.,Zhang H.X.,Chem.J.Chinese Universities,2014,34( 12),2605—2611(吴云剑,崔颖璐,郑清川,张红星.高等学校化学学报,2014,34( 12),2605—2611)
[9]Spoel D.V.D.,Lindahl E.,Hess B.,Groenhof G.,Mark A.E.,Berendsen H.J.C.,J.Comput.Chem.,2005,26( 16),1701—1718
[10]Mu G.,Wilmanns M.,Schulten K.,Biophys.J.,2002,83( 6),3435—3445
[11]Isralewitz B.,Baudry J.,Gullingsrud J.,Kosztin D.,Schulten K.,J.Mol.Graph.Model.,2001,19( 1),13—25
[12]Shen L.L.,Shen J.H.,Luo X.M.,Cheng F.,Xu Y.C.,Chen K.X.,Arnold E.,Ding J.P.,Jiang H.L.,Biophys.J.,2003,84( 6),3547—3563
[13]Jani V.,Sonavane U.B.,Joshi R.,J.Mol.Model.,2014,20( 6),1—14
[14]Kim I.,Allen T.W.,J.Chem.Phys.,2012,136( 16),164103
[15]Manning M.,Murphy B.,Etanercept Formulations Stabilized with Combination of Sugars and Polyols,UP 0101640A1,2013-04-25
[16]Lindorff L.K.,Piana S.K.,Maragakis P.,Klepeis J.L.,Dror R.O.,Shaw D.E.,Proteins: Struc.Func.&Bioinf.,2010,78( 8),1950—1958
[17]Hornak V.,Abel R.,Okur A.,Strockbine B.,Roitberg A.,Simmerling C.,Proteins,2006,65( 3),712—725
[18]Price D.J.,J.Chem.Phys.,2004,121( 20),10096—10103
[19]Wu Y.J.,Zheng Q.C.,Xu Y.,Chu W.T.,Cui Y.L.,Wang Y.,Zhang H.X.,Chem.Res.Chinese Universities,2014,30( 6),1011—1017
[20]Lan H.N.,Wang Y.X.,Zhang M.Z.,Han W.W.,Zheng X.,Chem.Res.Chinese Universities,2013,29( 1),139—143
[21]Thurtell J.K.,Thurtell G.W.,J.Chem.Phys.,1988,88( 10),6641—6646
[22]Bussi G.,Donadio D.,Parrinello M.,J.Chem.Phys.,2007,126( 1),14101—14107
[23]Waichman K.,Barmashenko B.D.,Rosenwaks S.,J.Chem.Phys.,2010,133( 8),362—364
[24]Wang Y.H.,Li W.,Chem.Res.Chinese Universities,2005,21( 1),73—77
[25]Souaile M.,Roux B.,Comp.Phys.Comm.,2001,135( 1),40—57
[26]Roux B.,Comp.Phys.Comm.,1995,19( 1—3),275—282
[27]Kumar V.,Sharma V.K.,Kalonia D.S.,Int.J.Pharm.,2009,3661( 1/2),88—98
Active Structure Protection of Monoclonal Antibody Fusion Protein Etanercept†
PAN Qi1,LI Daixi1*,GUO Baisong2,YANG Chunsheng2,YANG Zhi2
( 1.Institute of Food Science and Engineering,University of Shanghai for Science and Technology,Shanghai 200093,China;
2.Institute of Freeze-drying Technology,Shanghai Tofflon Science and Technology Co,Ltd.,Shanghai 201108,China)
Abstract The thermodynamic stability of etanercept in different concentrations of the protective agent was investigated.The approaches of umbrella sampling and steered molecular dynamic simulation were adopted to study the dissociation of dimer etanercept with Gromacs software and amber99sb-ildn united atomic force field.The results indicated that the trehalose has ability to enhance the stability of dimer etanercept,and the protective effect of etanercept with trehalose and mannitol is better than with pure trehalose.In addition,the type of protective agent and alternative specific adsorption position can impact the effect of protecting active structure of etanercept observably.
Keywords Etanercept; Steered molecular dynamic; Umbrella sampling; Trehalose
( Ed.: P,H,S,K)
†Supported by the National Natural Science Foundation of China( No.51076108),the National Science Foundation of Shanghai,China ( No.12ZR1420400),the Innovation Funds for International Cooperation of the Shanghai Committee of Science and Technology,China ( No.12430702000) and the Alliance Program in Shanghai,China.
基金项目:国家自然科学基金(批准号: 51076108)、上海市自然科学基金(批准号: 12ZR1420400)、上海市“创新行动计划”国际科技合作项目(批准号: 12430702000)和上海市联盟计划项目资助.
收稿日期:2015-09-08.网络出版日期: 2016-01-13.
doi:10.7503/cjcu20150695
中图分类号O629; Q341
文献标志码A
联系人简介:李代禧,男,博士,副教授,主要从事计算生物学方面的研究.E-mail: dxli75@ 126.com

