餐厨垃圾降解细菌的Biology鉴定及系统发育分析
陈泽斌,郑 旴,夏体渊*,李 冰,徐胜光,任 禛,张永福,牛燕芬
(1.昆明学院农学院,云南 昆明 650214;2.云南省都市特色农业工程技术研究中心,云南 昆明 650214;3.中国科学院昆明动物研究所,云南 昆明 650223)
餐厨垃圾降解细菌的Biology鉴定及系统发育分析
陈泽斌1,2,郑 旴1*,夏体渊1**,李 冰3,徐胜光1,任 禛1,张永福1,牛燕芬1
(1.昆明学院农学院,云南 昆明 650214;2.云南省都市特色农业工程技术研究中心,云南 昆明 650214;3.中国科学院昆明动物研究所,云南 昆明 650223)
采用Biology和16S rDNA序列分析法对2株餐厨垃圾降解细菌进行了鉴定。菌株经纯化培养,提取基因组DNA,采用16S rDNA通用引物,进行了16S rDNA序列PCR扩增,扩增产物纯化后进行测序,序列经人工校对后用Megalign 7.0软件进行比对分析,构建系统发育树,表明C95可能归属于苍白杆菌属(Ochrobactrum)或寡养单胞菌属(Stenotrophomonas),C132归属于Pseudomonasputida。用Biology微生物鉴定系统进行了鉴定,确定C95为Ochrobactrumgrignonense,C132为Pseudomonasputida。
餐厨垃圾;降解;细菌;Biolog鉴定;系统发育分析
餐厨垃圾是指宾馆饭店等餐饮业单位和企事业单位食堂以及家庭生活在食物加工和用餐过程中产生的食物下料和剩余食物,它是目前城市生活垃圾的主要来源,在有些城市餐厨垃圾的比例甚至高达50 %以上[1-4]。以上海为例,每天产生的餐厨垃圾约达1100 t,在春节期间至少还要增加50 %以上。目前餐厨垃圾的处理,主要采用填埋、焚烧、堆肥、热解、蚯蚓床法处理,都不够安全有效,在应用中也存在各自的局限性[5-6]。对于如何安全有效地处理餐厨垃圾,少数发达的大城市已经引起了足够的重视,从环保和资源化的观点出发,利用生物技术处理城市生活垃圾正渐渐受到了人们的青睐。
好氧降解是在通气的条件下,以好氧微生物为主将大分子的餐厨垃圾分解为小分子的有机物,微生物吸收、利用中间的代谢产物合成自身的细胞物质,生长繁殖[7-10]。垃圾中微生物种群极其丰富,尤其是含有大量对难降解污染物具有强烈降解作用的降解型微生物,是优良的生物处理载体和复杂生物处理菌种的来源。人们已从垃圾填埋场中分离出高效纤维素分解菌、能降解DBP的菌株、具有明显除臭效果的菌株等[11-12]。
前期研究以昆明学院润泽苑教工餐厅的餐厨垃圾为对象,对餐厨垃圾的各组分进行了研究,采用多种培养基广泛分离菌种,选取与餐厨垃圾各营养成分对应的选择性培养基,进行定性初筛和定量复筛,通过测定液体培养48 h后的菌悬液OD600值比较其对蛋白质、脂肪、淀粉、纤维素的分解能力,得到了2株有效降解细菌株。接下来降解细菌的鉴定工作是今后进行扩大化培养的必要前提。细菌的常规鉴定依赖于形态特征观察及生理生化测定,虽然方法准确可靠,但费时、费力。基于16S rDNA序列分析的分子生物学鉴定快速灵敏,但不能作为最终的鉴定依据,常用于目标菌的初步检测[13]。
Biology全自动微生物自动鉴定系统Gen III微孔板可以对微生物进行94种表型测试,其中包括:71 种碳源利用测试(图3)和23种化学敏感性测试(图3)。它主要根据四唑类氧化还原染料的色度变化来指示微生物对碳源的利用程度和对化学物质的敏感程度。微生物在测试板上所显示出的特定“表型指纹”被用来在种的水平上进行鉴定[14-15]。文章分别通过Biolog微生物鉴定系统及16S rDNA序列分析的方法对2株餐厨垃圾降解细菌进行了快速鉴定,以期为餐厨垃圾降解细菌剂的开发奠定基础。
1 材料与方法
1.1 供试菌株
餐厨垃圾降解细菌C95和C132为2014年从昆明学院润泽苑教工餐厅的餐厨垃圾分离筛选所得,保存于昆明学院农学院植物生产试验中心实验室。
1.2 培养基
NA培养基用于供试菌株的复壮和纯化培养,LB培养基用于提取DNA前的液体培养,BUG + B培养基用于Biology鉴定中菌株的培养。
1.3 试剂和PCR引物
BUG + B培养基、GEN III微孔板(MicroPlate)、浊度仪、8 通道电动移液器等均购自Biology公司;细菌基因组DNA提取试剂盒、DNA Marker购自大连宝生物工程有限公司;Taq酶、dNTP、GoldView购自北京鼎国生物技术有限公司。细菌16S rDNA扩增通用引物[16]由上海桑尼生物科技有限公司合成(正向引物27F:5’-AGAGTTTGATCCTGGCTCAG-3’;反向引物1492R:5’-AAGGAGGTGATCCACCC-3’)。
1.4 16S rDNA系统发育分析
用TaKaRa MiniBEST Bacterial Genomic DNA Extraction Kit Ver.2.0提取细菌基因组DNA,方法参见试剂盒说明书。PCR反应体系(50 μl)组成:10×PCR缓冲液5 μl,dNTPs (10 mmol/L) 4 μl,引物F27/R1492[16](10 μmol/L) 2 μl,模板DNA (约50 g/L) 1μl,Taq酶2.5 U,双蒸水35.5 μl。PCR扩增按Sambrook方法进行[17]。扩增产物经TaKaRa Agarose Gel DNA Purification Kit Ver.2.0回收后,送上海桑尼生物科技有限公司进行序列测定。将测得的16S rDNA全序列用EzTaxon Server version 2.1(http://www.ezbiocloud.net/)进行同源性搜索[18],下载相似序列作为参比序列。用Megalign 7.0软件,将这些序列用Clustal W法进行多序列比对,构建系统发育树。
1.5 Biology鉴定
试验在云南农业大学农业生物多样性应用技术国家工程研究中心完成。试验程序按Biology公司GEN III的操作指南进行[19],所有菌株在Biology推荐的培养基(BUG + B)上33 ℃培养24 h。按照操作规程将微生物细胞浓度调至90 %~98 %(T为Biology浊度仪所测得的透光率),将菌悬液倒入V型加样槽中,用8通道电动移液器按每孔100 μl的量将菌悬液按顺序加入微孔板的所有孔中,33 ℃黑暗培养,24 h后记录结果。所有菌株均采用OmniLog读数仪读板,读数结果直接与数据库进行比较获得鉴定结果。
2 结果与分析
2.1 16S rDNA系统发育分析
将C95和C132在NA培养基复壮纯化(图1),挑取单菌落在LB培养基中28 ℃液体培养12 h后,分别提取细菌基因组DNA,用通用引物27F/1492R对2株细菌的16S rDNA进行PCR扩增,得到1.5 kb的PCR产物,送测序后,将序列提交到GenBank核酸数据库中,C95和C132登录号分别为KP771804和KP771807。将其与12个相似参比菌株的16S rDNA序列(表1)进行多重序列对齐后,构建系统发育树(图2)和系统发育进化距离图(图3)。结果显示,C95与KF844052Ochrobactrumpseudogrignonense、KJ784477Stenotrophomonasmaltophilia、FJ266319Ochrobactrumanthropi、AB680343Ochrobactrumsp.聚于同一系统发育分支,同源性均为100.0 %;C132与FJ588702Pseudomonasfluorescens聚于同一系统发育分支,同源性为99.9 %。通过分子生物学方法尚无法确定C95的分类地位,C95可能归属于苍白杆菌属 (Ochrobactrum),也可能归属于寡养单胞菌属(Stenotrophomonas);将C132确定为荧光假单胞菌(Pseudomonasfluorescens)。
a:C95;b:C132图1 供试菌株在NA培养基上的形态特征Fig.1 The morphology of strains on NA culture medium
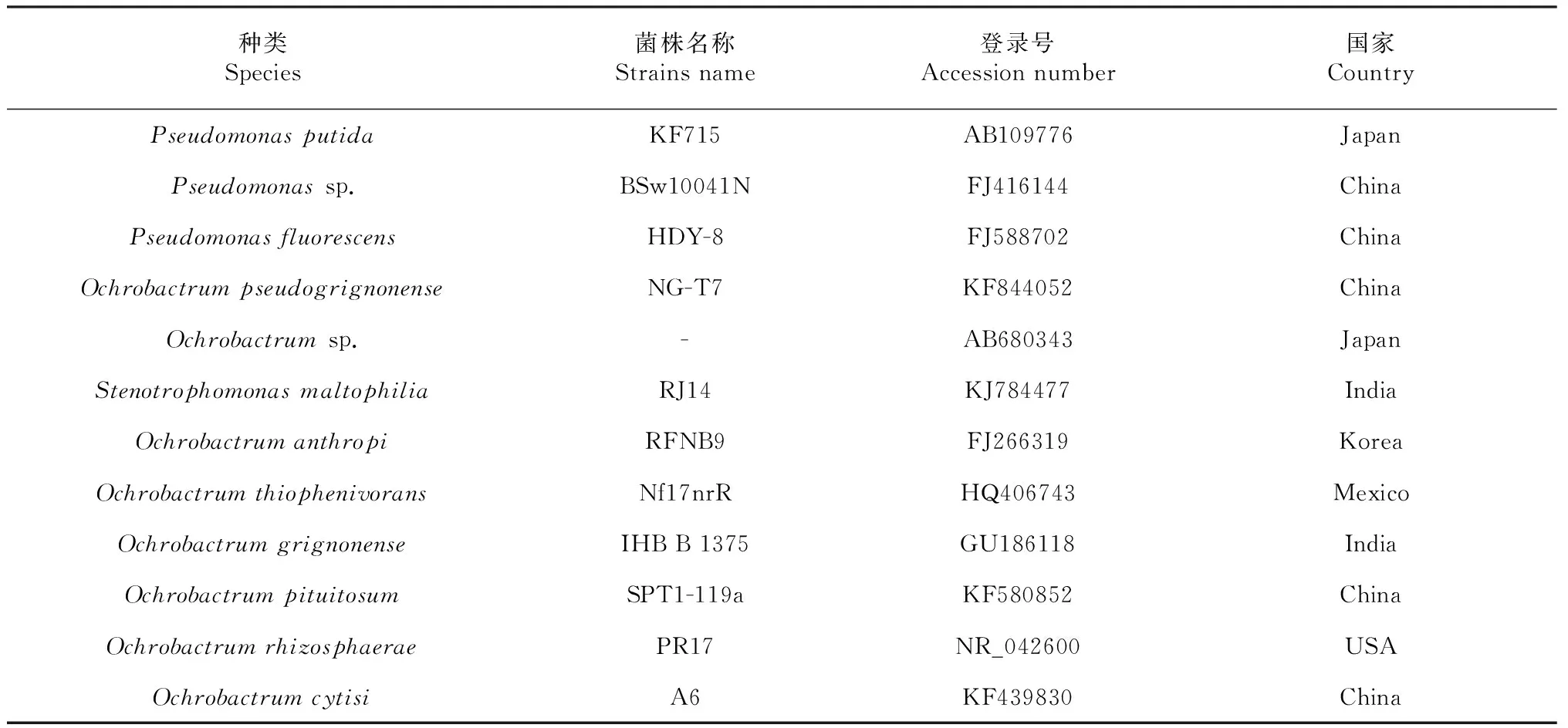
种类Species菌株名称Strainsname登录号Accessionnumber国家CountryPseudomonasputidaKF715AB109776JapanPseudomonassp.BSw10041NFJ416144ChinaPseudomonasfluorescensHDY-8FJ588702ChinaOchrobactrumpseudogrignonenseNG-T7KF844052ChinaOchrobactrumsp.-AB680343JapanStenotrophomonasmaltophiliaRJ14KJ784477IndiaOchrobactrumanthropiRFNB9FJ266319KoreaOchrobactrumthiophenivoransNf17nrRHQ406743MexicoOchrobactrumgrignonenseIHBB1375GU186118IndiaOchrobactrumpituitosumSPT1-119aKF580852ChinaOchrobactrumrhizosphaeraePR17NR_042600USAOchrobactrumcytisiA6KF439830China
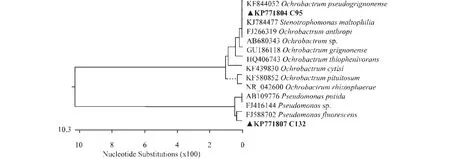
标尺为核酸替换率bar-subsitutions per nucleotide position图2 基于16S rDNA序列的系统发育树Fig.2 Phylogenetic tree based on 16S rDNA sequence

图3 系统发育进化距离Fig.3 Sequence distance based on 16S rDNA full-length sequence
2.2 Biology鉴定结果
GenIII Microplate测试布局见图4,2株餐厨垃圾降解细菌在GenIII鉴定板上的测试表现见图5。以A1孔为阴性对照对GenIII微孔板1~9列中的碳源利用情况进行测试。所有视觉上看起来与A1孔类似的反应定义为“阴性”反应(-),看起来成紫色(深于A1)的定为“阳性”反应(+)。孔中所显的颜色非常浅,或者有紫色的小色斑,或者有菌块或其他块状物,则将其判定为“边界值”()。以A-10孔为阳性对照对列10~12进行化学敏感性测试,所有颜色不到A10孔一半,且容易被该化学物质抑制的反应被定为“阴性”反应(-),显紫色或接近紫色(与A10孔类似)的反应被定为“阳性”反应(+)。统计结果见表2。
图4 GEN Ⅲ微孔板布局Fig.4 The layout of GenIII microplate
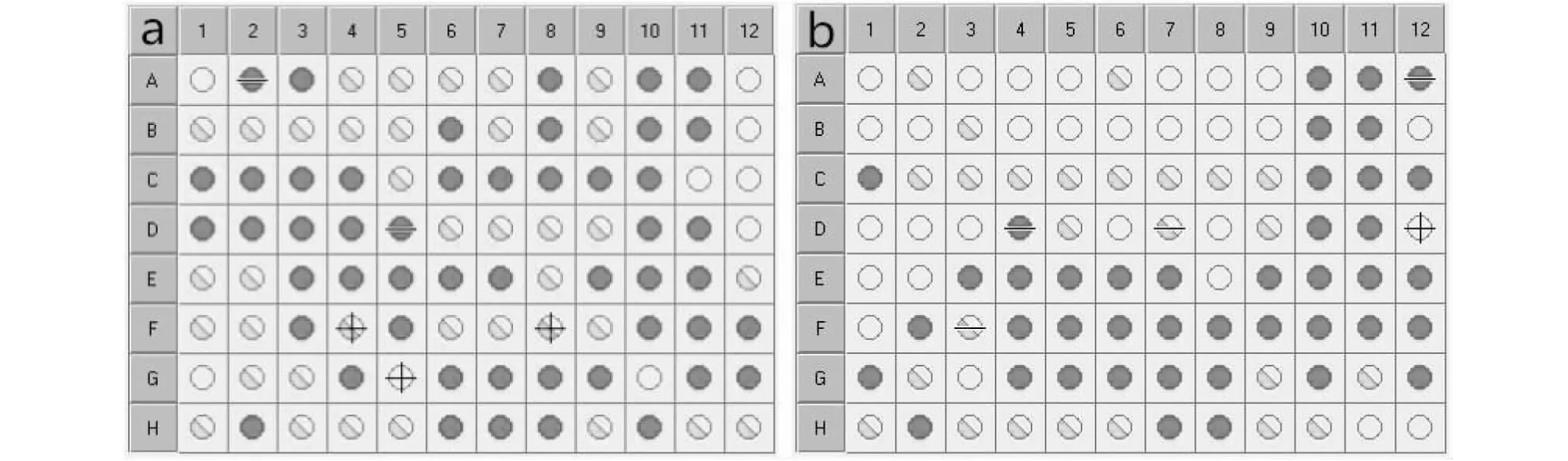
a-C95;b-C132图5 供试菌株在GEN Ⅲ鉴定板上的表现Fig.5 The performance of strains on GenIII microplate

微孔编号MicroporeNo.碳源CarbonsourcesC95C132A1阴性对照--A2糊精+A3D-麦芽糖+-A4D-海藻糖-A5D-纤维二糖-A6龙胆二糖\A7蔗糖-A8松二糖+-A9水苏糖-A10阳性对照++A11pH6++A12pH5-+B1蜜三糖,棉子糖-B2α-D-乳糖-B3蜜二糖\B4β-甲酰-D-葡糖苷-B5D-水杨苷-B6N-乙酰-D-葡糖胺+-B7N-乙酰-β-D-甘露糖胺-B8N-乙酰-D-半乳糖胺+-B9N-乙酰神经氨酸-B101%NaCl++B114%NaCl++B128%NaCl--C1α-D-葡糖++C2D-甘露糖+C3D-果糖+C4D-半乳糖+C53-甲酰葡糖\C6D-果糖+C7L-果糖+C8L-鼠李糖+C9肌苷+C10乳酸钠++C11梭链孢酸-+C12D-丝氨酸-+D1D-山梨醇+-D2D-甘露醇+-D3D-阿拉伯醇+-D4肌醇++D5甘油+
续表2 Continued table 2

微孔编号MicroporeNo.碳源CarbonsourcesC95C132D6D-葡糖-6-磷酸-D7D-果糖-6-磷酸\D8D-天冬氨酸-D9D-丝氨酸\D10醋竹桃霉素++D11利福霉素SV++D12二甲胺四环素--E1明胶-E2氨基乙酰-L-脯氨酸-E3L-丙氨酸++E4L-精氨酸++E5L-天冬氨酸++E6L-谷氨酸++E7L-组胺++E8L-焦谷氨酸-E9L-丝氨酸++E10林肯霉素,洁霉素++E11盐酸胍++E12硫酸四癸钠+F1果胶-F2D-半乳糖醛酸+F3L-半乳糖醛酸内酯+F4D-葡糖酸+F5D-葡糖醛酸++F6葡糖醛酰胺+F7粘酸;粘液酸+F8奎宁酸+F9糖质酸+F10万古霉素++F11四唑紫++F12四唑蓝++G1p-羟基-苯乙酸-+G2丙酮酸甲酯\G3D-乳酸甲酯-G4L-乳酸++G5柠檬酸-+G6α-酮-戊二酸++G7D-苹果酸++G8L-苹果酸++G9溴-丁二酸+G10萘啶酮酸-+G11氯化锂+
续表2 Continued table 2
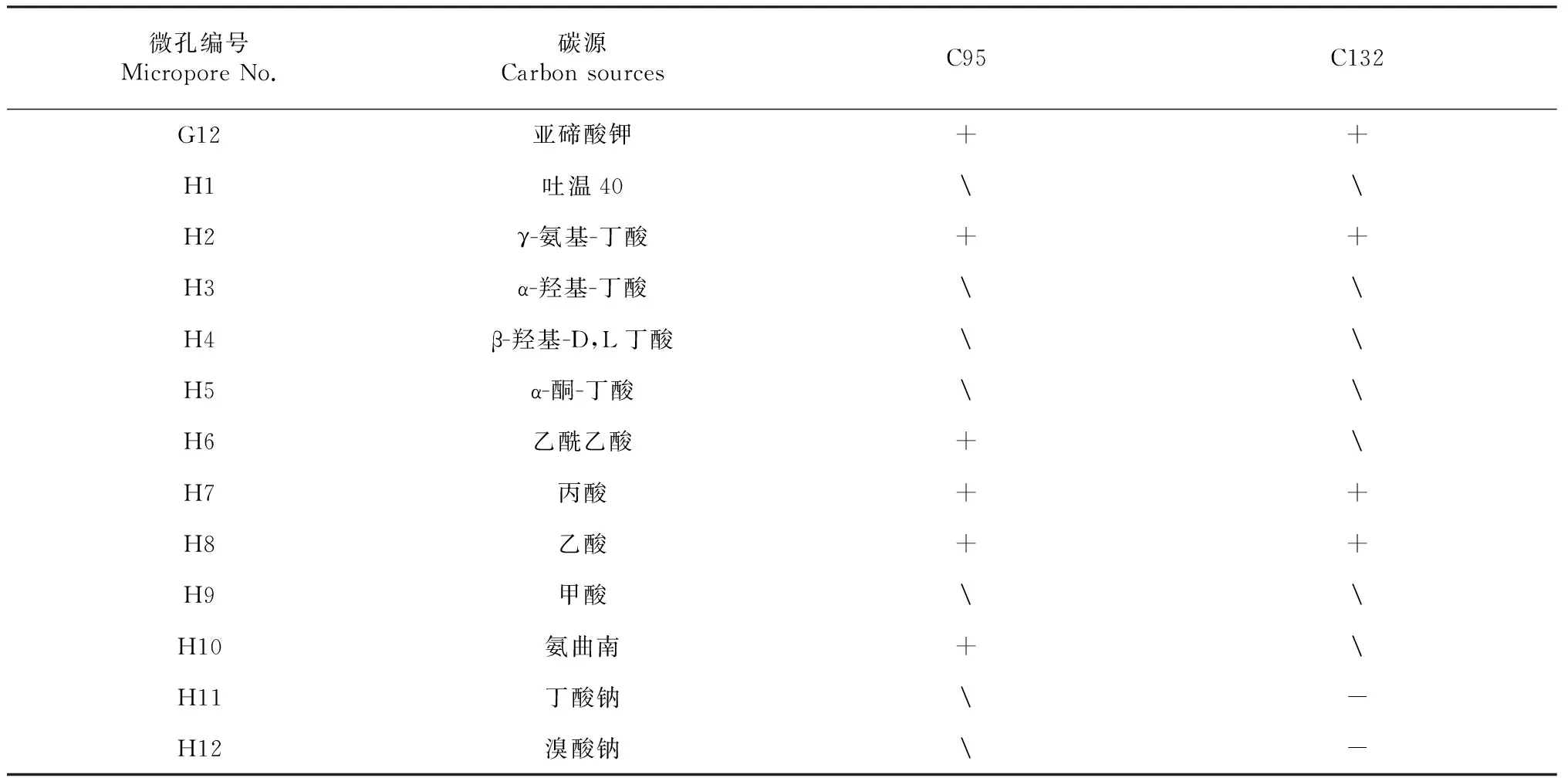
微孔编号MicroporeNo.碳源CarbonsourcesC95C132G12亚碲酸钾++H1吐温40\H2γ-氨基-丁酸++H3α-羟基-丁酸\H4β-羟基-D,L丁酸\H5α-酮-丁酸\H6乙酰乙酸+H7丙酸++H8乙酸++H9甲酸\H10氨曲南+H11丁酸钠-H12溴酸钠-

表3 供试菌株经Biology GEN III 微孔板鉴定结果
2个供试菌株的Biolog鉴定结果见表3,其中相似性(SIM)和位距(DIS)是2个重要的参数,表示测试结果与数据库相应数据的匹配程度。SIM值越接近于1,鉴定结果的可靠性越强。通常在SIM值大于0.5时,就可以确定菌株的分类地位。从表3可以看出,2个供试菌株均得到了准确鉴定,均为革兰氏阴性菌,C95为Ochrobactrumgrignonense,C132为Pseudomonasputida,相似性分别为0.624和0.612。
3 讨 论
采用Biolog鉴定和16S rDNA序列分析法对2株餐厨垃圾降解细菌进行了鉴定,发现分子生物学方法有局限性,从系统发育树(图2)来看,C95与KF844052Ochrobactrumpseudogrignonense、KJ784477Stenotrophomonasmaltophilia、FJ266319Ochrobactrumanthropi、AB680343Ochrobactrumsp.聚于同一系统发育分支,同源性均为100.0 %,表明通过分子生物学方法尚无法确定C95的分类地位,C95可能归属于苍白杆菌属 (Ochrobactrum),也可能归属于寡养单胞菌属(Stenotrophomonas),结合Biology鉴定结果,才将C95鉴定为Ochrobactrumgrignonense。由此可见,基于16S rDNA的分子生物学鉴定方法并不能完全作为细菌鉴定的依据,还有许多细菌不能用这种方法区分开。它在属的水平上效果是比较好的,鉴定到种还需要其他生理生化方法和寄主分析来缩小鉴定范围。Biology 细菌鉴定系统可以将细菌鉴定到种的水平[20]。两者相互验证,互相补充。随着Biology 数据库的增大和培养条件的优化,其鉴定结果会更加简便与准确,GEN III微孔板在细菌的鉴定和分类中具有广阔的应用前景。
C95和C132分别归属于Ochrobactrumgrignonense和Pseudomonasputida2类细菌都曾被报道具有降解活性,Pseudomonasputida可降解萘、苯酚、磷[21-23];Ochrobactrum属细菌可降解烟碱、石油、润滑油、苯胺、喹啉、百菌清[24-26],其生物学特性及对温度、pH等条件是否具有比较广泛的适应性将是下一步研究的重点。
[1]de Souza S N, Horttanainen M, Antonelli J, et al. Technical potential of electricity production from municipal solid waste disposed in the biggest cities in Brazil:landfill gas, biogas and thermal treatment[J]. Waste Manag Res., 2014, 32(10):1015-1023.
[2]Ghanavati H, Nahvi I, Karimi K. Organic fraction of municipal solid waste as a suitable feedstock for the production of lipid by oleaginous yeast Cryptococcus aerius[J]. Waste Manag., 2015, 38:141-148.
[3]Probst M, Walde J, Pümpel T, et al. A closed loop for municipal organic solid waste by lactic acid fermentation[J]. Bioresour Technol., 2014, 175:142-151.
[4]施军营,房 明,乔如林,等. 高温蒸煮除油对餐厨垃圾厌氧消化的影响[J]. 安徽农业科学, 2014(7):2101-2103.
[5]Vilavert L, Figueras MJ, Schuhmacher M, et al. Formaldehyde:a chemical of concern in the vicinity of MBT plants of municipal solid waste[J]. Environ Res., 2014, 133:27-35.
[6]金 雷,陈 瑜,严忠雍,等. 邻苯二甲酸二丁酯高效降解细菌H-2的分离鉴定及其降解特性[J]. 食品科学,2014,15:202-206.
[7]赵燕茹,谢 强,张永兴,等. 城市生活垃圾降解-压缩特性试验研究[J]. 岩土工程学报,2014,10:1863-1871.
[8]孙玉宇,孙晓菲,葛毅鹏. 纤维素降解细菌的筛选及在城市生活垃圾处理中的应用[J]. 洛阳理工学院学报(自然科学版),2014(3):17-20.
[9]郭建华,奚家勤,宋纪真,等. 纤维素降解菌哈茨木霉TC10-13产酶条件优化[J]. 南方农业学报, 2015, 46(1):79-84.
[10]李海红,巩雪松,同 帜. 多元线性回归预测模型在农村生活垃圾产量预测中的应用[J]. 西南农业学报,2010(4):1325-1328.
[11]谢 武,潘 琼,王金菊. 催化型微电解对垃圾渗滤液深度处理的研究[J]. 西南农业学报,2011(5):1943-1947.
[12]连 宾,周正乾,杨秀军,等. 利用生活垃圾制造有机复合肥研究报告[J]. 西南农业学报,1994(4):116-118.
[13]Sogin M L,Gunderson J H. Structural diversity of eukaryotic small subunit ribosomal RNAs evolutionary implications[J]. Academy of Science, 1987, 503(1):125-139.
[14]Bochner B R. Sleuthing out Bacterial Identities[J]. Nature, 1989, 339:157-158.
[15]Bochner B R. ‘Breathprints’at the Microbial Level[J]. ASM News, 1989, 55:536-539.
[16]Li Y W, Lee S A, Kim S B, et al. Diversity of denitrifying bacteria isolated from daejeon sewage treatment plant[J]. The Journal of Microbiology, 2005, 43(5):383-390.
[17]Sambrook J, Fritsch EF, Maniatis T. 分子克隆实验指南[M]. 第2版. 金冬燕, 黎孟枫, 侯云德, 等译. 北京:科学出版社, 1996:682.
[18]陈泽斌,陈兴位,夏体渊,等. 食用玫瑰花瓣可培养内生细菌的分离及多样性分析[J]. 西南农业学报,2016(6):1408-1413.
[19]June C M, Roach K, Levett P N, et al. Identification ofStreptococcusiniaeby commercial bacterial identification systems[J]. Journal of Microbiology Methods, 2006, 67:20-26.
[20]Tang Y W, Ellis N M, HopKins M K, et al. Comparison of phenotypic and genotypic techniques for identification of unusual aerobic pathogenic gram-negative bacilli[J]. Journal of Clinical Microbiology, 1998, 36 (12):3674-3679.
[21]李宝明,阮志勇,姜瑞波. 石油降解菌的筛选、鉴定及菌群构建[J]. 中国土壤与肥料,2007(3):68-72.
[22]史秀珍,李世东,郭荣君,等.OchrobactrumlupiniTP-D1降解百菌清产物的研究[J]. 北京化工大学学报(自然科学版),2007(S2):118-121.
[23]洪 璇,张晓君,孟智奇,等. 废水反硝化生物反应器中喹啉降解细菌的分离与特性[J]. 应用与环境生物学报,2008(6):803-808.
[24]张 楠,陈波水,杨致邦,等. 润滑油降解菌的分离、鉴定及降解特性[J]. 环境科学研究,2010(6):748-753.
[25]刘宪斌,任丽君,田胜艳,等. 稠油降解微生物的筛选鉴定及其降解特性[J]. 环境工程学报,2014(7):3069-3074.
[26]刘 真,邵宗泽. 南海深海沉积物烷烃降解菌的富集分离与多样性初步分析[J]. 微生物学报,2007(5):869-873.
(责任编辑 王家银)
Biology Identification and Phylogenetic Analysis of Efficient Bacteria for Aerobic Degradation on Kitchen Waste
CHEN Ze-bin1,2, ZHENG Xu1*, XIA Ti-yuan1**, LI Bing3, XU Sheng-guang1, REN Zhen1, ZHANG Yong-fu1, NIU Yan-fen1
(1.Agriculture College, Kunming University, Yunnan Kunming 650214, China;2.Engineering Research Center for Characteristics Agriculture of Yunnan Province, Yunnan Kunming 650214, China;3.Kunming Institute of Zoology, the Chinese Academy of Sciences, Yunnan Kunming 650223, China)
2 strains of efficient bacteria for the aerobic degradation on kitchen waste were identified by biology microbes identification system and 16S rDNA gene sequence analysis. After the genomic DNA was extracted, the sequences of 16S rRNA gene were amplified by PCR with the bacterium universal primers and the purified PCR products were directly sequenced. The corrected sequences were aligned with Megalign 7.0 and the phylogenetic tree was constructed. The results of phylogenetic analysis showed that the C95 belonged toOchrobactrumorStenotrophomonas; C132 belonged toPseudomonasputida. The results of biology tests showed that the 2 strains wereOchrobactrumgrignonenseandPseudomonasputida,respectively.
Kitchen waste; Degradation; Bacteria; Biology identification; Phylogenetic analysis
1001-4829(2016)10-2488-08
10.16213/j.cnki.scjas.2016.10.043
2015-03-29
云南省都市特色农业工程技术研究中心项目(TSNY0201);云南省教育厅科学研究基金项目(2014Y390);云南省应用基础研究青年项目(2013FD040);昆明学院人才引进项目(YJL14005);昆明学院人才引进基金项目(YJL12010);昆明学院校立重点基金项目(XJL12020);昆明学院大学生科研项目(XJD15054);云南中烟品牌省内原料产地生态与主栽品种合理布局研究项目;云南省高校优势特色重点学科(生态学)建设项目资助(05000511311);昆明市农业局滇池流域农业面源污染综合防控对策研究(2016JC01)
陈泽斌(1985-),男,云南昆明人,博士,副教授,主要从事环境微生物的教学及科研工作,E-mail:zbchenkmu@163.com,*为共同第一作者,**为通讯作者,E-mail:xiatiyuan@sohu.com。
S182
A

