水稻开花时间基因OsDTH 10的过表达分析
李莹莹 熊炜 宋婷 曹振华 栾维江*
(天津师范大学生命科学学院/天津市动植物抗性重点实验室,天津300387;*通讯联系人,E-mail:lwjzsq@163.com)
水稻开花时间基因OsDTH 10的过表达分析
李莹莹 熊炜 宋婷 曹振华 栾维江*
(天津师范大学生命科学学院/天津市动植物抗性重点实验室,天津300387;*通讯联系人,E-mail:lwjzsq@163.com)
李莹莹,熊炜,宋婷,等.水稻开花时间基因OsDTH10的过表达分析.中国水稻科学,2016,30(2):121-126.
摘 要:开花时间(抽穗期)是农作物一个重要的农艺性状,与农作物的产量息息相关。调节农作物的开花抽穗时间是提高农作物产量的一条重要途径。为了揭示水稻开花时间基因OsDTH10在水稻抽穗期调控中的功能,利用反向遗传学方法构建了水稻OsDTH10基因过量表达载体,获得转基因植株,分析目的基因在过量表达条件下的功能。结果表明,过量表达OsDTH10导致水稻的抽穗期推迟;RT-PCR检测结果表明OsDTH10的表达量在晚抽穗的转基因株系中明显提高,但在没有表型的转基因株系及野生型中表达量较低,说明晚抽穗的表型是由于OsDTH10过量表达所致。组织特异性表达结果表明OsDTH10在植物的不同器官中都有表达,但在水稻的茎、叶鞘中表达量较高。分析OsDTH10在不同叶龄叶片中的表达,结果表明OsDTH10在未完全展开的剑叶叶片及倒2叶中的表达量较高,但在下部较老的倒3叶及倒4叶中的表达量下降。另外,分析OsDTH10在不同光周期条件下的昼夜节律性表达,结果表明无论在短日照还是长日照条件下,OsDTH10都在白天(光期)有表达高峰,在夜晚(暗期)表达降低,表明OsDTH10可能涉及光周期调控通路调节水稻的抽穗期。
关键词:水稻;OsDTH10;过量表达;表达分析
植物开花转变受其内部基因及外界环境因素共同影响。光周期(日照长短)是植物开花转变和开花时间的主要环境决定因素。植物通过内部基因网络识别和测量日照长短,并对光周期作出反应来调控植物开花的时间[1-3]。根据光周期对植物开花的影响将其划分为短日照植物、长日照植物和日中性植物[4]。水稻是典型的短日照植物,短日照条件下水稻的抽穗期提早,而长日照条件下水稻的抽穗期延迟。现已明确短日照条件下水稻抽穗提早主要通过两条途径实现:一条是与模式植物拟南芥CO-FT途径保守的Hd1-Hd3a途径;另一条是水稻所特有的Ehd1-Hd3a途径[5-7]。在这两条途径中,有几个调控基因起重要作用。Hd1编码一个锌指蛋白,通过调控其下游的Hd3a基因,在短日条件下促进水稻的开花,但在长日条件下Hd1通过抑制Hd3a的表达延迟水稻的开花[8-9]。Hd3a是水稻抽穗开花的激活子,它可以促进水稻成花,是水稻的成花素[10],Hd3a在短日照条件下有较高的水平,但在长日照条件下表达水平很低。RFT1是Hd3a的同源基因,也是水稻开花的激活子。在短日照条件下与Hd3a功能冗余,但在长日照条件下可以促进水稻开花,是水稻在长日照条件下的成花素[11]。Ehd1编码一个B型的应答效应因子,在短日照条件下通过诱导FT-like基因(如Hd3a、RFT1等)的表达而促进水稻开花[12]。而在长日照条件下水稻开花延迟主要通过两条途径实现:一条途径是通过Hd1的抑制作用,负向调控Hd3a的表达从而推迟水稻的抽穗开花时间;另一条途径是通过开花的抑制子Ghd7在长日照条件下抑制Ehd1的表达,进而抑制Hd3a和RFT1的表达,从而推迟水稻的抽穗[13]。这两条途径最终都通过成花素基因Hd3a和RFT1的表达调控调节水稻抽穗开花。成花素基因编码一个磷酯酰乙醇胺结合蛋白(Phosphatidylethanolaminebinding protein,PEBP)家族[14],拟南芥中已发现该家族成员在功能上已有了分化,除了促进拟南芥成花的FT基因外,还发现一个与FT基因同源但功能却完全相反的TFL1家族成员,它能够抑制拟南芥开花[15]。水稻中该家族有13个成员[16],Hd3a和RFT1是两个功能明确的成员,但其他成员功能仍然未知。为了进一步研究水稻中成花素家族成员的功能,我们对其他未知的家族成员进行反向遗传学分析,发现其中一个家族成员OsDTH10(Os06g35940)与Hd3a和RFT1在水稻抽穗开花上具有相反的功能,过量表达Os-DTH10延迟了水稻抽穗。另外还揭示了Os-DTH10的表达规律,表明OsDTH10可能涉及光周期调控通路调控水稻的抽穗期。
1 材料与方法
1.1 材料
粳稻品种中花11(Oryza sativa L.ssp.japonica cv.Zhonghua 11)及其转基因植株用于本实验,材料种植于天津市农业科学院试验田。田间常规管理。
1.2 OsDTH10过表达载体的构建
OsDTH10目的基因用RT-PCR方法获得,总RNA从40d的水稻叶片中提取,用M-MLV反转录酶反转录合成cDNA,以它为模板,用基因特异性引物OsDTH10F(5′-ATTggtaccGCTTAGCTTCA GCTCACACC-3′,小写字母为酶切位点)和OsDTH10R(5′-ATTgtcgacGCTGGCTAGACACA CACTGCAT-3′,小写字母为酶切位点)进行PCR扩增,获得目的基因完整的ORF(开放阅读框)。PCR运行程序如下:95℃下1 min;95℃下30s,60℃下30s,72℃下30s,35次循环;之后72℃下延伸5min。凝胶检测后回收纯化目的片段,将获得的目的片段用相应酶切位点连入空载体pCAMBIA2300中,获得过表达融合载体。对融合载体进行测序验证,正确无误后用农杆菌介导的遗传转化方法获得转基因植株,并用空载体产生转基因植株作对照,具体程序参照文献[17]。
1.3 DNA的提取及转基因植株的分子检测
用CTAB法[18]从水稻叶片中提取基因组DNA。然后用载体中的新霉素磷酸转移酶抗性基因(NPTⅡ)对获得的过表达转基因植株进行PCR检测。所用引物为NPTⅡF(5′-TTCTCACTGAA GCGGGAAGGG-3′)和NPTⅡR(5′-GCGATACC GTAAAGCACCAGG-3′)。反应条件如下:94℃下4min;94℃下30s,57℃下30s,72℃下30s,30次循环;之后72℃下延伸5min。
1.4 目的基因的表达分析
在过表达转基因植株的表达分析中,总RNA 从60d水稻植株的叶片中提取;在水稻组织特异性表达分析中,总RNA分别从水稻根、茎、不同部位的叶片、茎顶端分生组织、不同时期的幼穗中提取。提取的总RNA用DNaseⅠ进行处理,消化基因DNA后用M-MLV反转录酶反转录合成cDNA后进行RT-PCR。引物序列为OsDTH10F(5′-CTGT GATGTATGATGGGAGG-3′)和OsDTH10R(5′-AAGGTTGGGTTGCTGGGATT-3′)。运行程序如下:94℃下4min;94℃下30s,58℃下30s,72℃下30s,32次循环;之后72℃下延伸5min。并以水稻OsActin1为内参进行相对定量分析。引物为OsActin1F(5′-GACTCTGGTGATGGTGTCAGC-3′)和OsActin1R(5′-GGCTGGAAGAGGACCTC AGG-3′)。反应条件为94℃下4min;94℃下30s,60℃下30s,72℃下30s,24次循环;之后72℃下延伸5min。
1.5 光周期材料的处理及表达分析
田间生长40d、健壮一致的野生型中花11移入大型人工气候箱中,分别用两个光周期条件进行处理,即长日照(LD,15h光照/9h黑暗)和短日照(SD,11h光照/13h黑暗)。温度为28℃。处理25 d后,按24h节律性,每隔3h取样提取总RNA,并用DNaseⅠ进行处理,消化基因DNA后反转录合成cDNA,利用qRT-PCR分析目的基因在不同光周期条件下的表达模式。qRT-PCR按照SYBR Green PCR master mix(Vazyme,南京)试剂盒说明操作,在7500Fast实时PCR System(ABi-7500,美国)荧光定量PCR仪上进行扩增反应,并用2-△△Ct方法进行相对定量分析,每个反应重复3次。所用引物同1.4所述。
2 结果与分析
2.1 OsDTH10过表达载体的构建
用RT-PCR方法从水稻叶片中获得OsDTH10的全长ORF,目的片段符合预期设计(图1-B)。纯化回收后用限制性内切酶KpnⅠ和BamHⅠ对目的片段和空质粒pCAMBIA2300进行酶切,纯化回收后用T4DNA连接酶进行连接,获得重组载体。对重组质粒进行酶切检测,发现切出了预期的目的片段(图1-C),表明目的片段已成功连入空载体中,获得最终的过表达载体(图1-A)。将构建成功的重组质粒进行测序验证后,转化到农杆菌EHA105中,用于后续的转基因实验。
2.2 转基因植株的表型分析
通过农杆菌介导的遗传转化,将重组载体pCAMBIA2300-Os DTH10导入野生型中花11中,最终获得了11个独立株系的98株T0代转基因阳性植株。我们选取了5个株系种植到大田里,获得T1代转基因株系。并检测了转基因植株中目的基因的表达,发现其中3个株系目的基因表达水平明显提高(3~5号株系;图2-B),其他两个株系与野生型相比没有明显差异(1号和2号株系;图2-B),表明所构建的过表达载体正常工作。表型观察发现这3个表达量明显提高株系(3~5号株系)的抽穗期比野生型对照分别推迟了4、7和16d(图2-A,C),而1号和2号株系的抽穗期与野生型没有明显差异,说明转基因植株延迟抽穗是由OsDTH10基因过量表达所致(图2-C)。

A-过表达载体结构;LB为T-DNA左边界;NPTⅡ-新霉素磷酸转移酶抗性筛选标记;p2×35S-2个串连的花椰菜花叶病毒(CaMV)35S启动子;RB为T-DNA右边界。B-OsDTH10全长开放阅读框的PCR扩增。C-过表达载体的酶切鉴定。1~3为3个不同单克隆;M-DNA标记。A,Constructure of overexpression vetor;LB,Left border of TDNA;NPTⅡ,Neomycin phosphotransferaseⅡ;p2×35S,Double cauliflower mosaic virus(CaMV)35Spromoter;RB,Right border of T-DNA;B,PCR amplification of OsDTH10 full open reading frame;C,Digesting identification of recombinant plasmid;1 -3,Three independent clones of recombinant plasmid;M,DNA marker.图1 过表达载体的构建Fig.1.Construction of overexpression vetor.
2.3 OsDTH10基因的组织特异性表达分析
为了确定OsDTH10在不同组织器官中的表达,分别提取了野生型水稻中花11的茎顶端分生组织、根、叶片、叶鞘、茎和不同时期幼穗的总RNA,反转录合成cDNA,通过RT-PCR检测目的基因在水稻不同器官中的表达差异。结果显示OsDTH10在水稻各个组织器官中均有表达,但表达量有差异。比较而言,在茎和叶鞘中的表达量最高(图3-A),在茎顶端分生组织、14cm长幼穗及叶片中次之。我们进一步分析了OsDTH10在同一株水稻上不同叶龄叶片中的表达。结果显示,OsDTH10在最上部还未完全展开叶片和倒2叶中的表达量较高,但在较老的倒3叶和倒4叶中表达量比较低(图3-B)。
2.4 OsDTH10基因在不同光周期条件下昼夜节律性表达

A-过表达转基因植株的表型;WT为空载体转基因植株对照,OsDTH10-OV为过表达转基因植株;B-转基因植株的表达分析;1~5分别为5个独立的转基因株系;C-转基因株系的抽穗期(平均值±标准差,n≥15)。A,Phenotype of transgenic plants;WT,Transgenic plants with empty vector;OsDTH10-OV,OsDTH10-Overexpressing transgenic plants.B,qRT-PCR and RT-PCR analysis of OsDTH10 expression level in different OsDTH10-OVlines;1to 5are five independent OsDTH10-OV lines;C,Heading date of OsDTH10-OVline(mean±SD,n≥15).图2 过表达转基因植株的表型及表达分析Fig.2.Phenotype and expression analysis of OsDTH10-overexpressing transgenic lines(OsDTH10-OV).
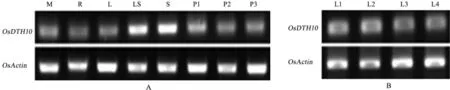
A-OsDTH10组织特异性表达;M-茎顶端分生组织;R-根;L-叶片;LS-叶鞘;S-茎;P1-14cm长幼穗;P2-4cm长幼穗;P3-25 cm长幼穗。B-OsDTH10在不同叶龄叶片中的表达。L1-未完全展开的剑叶叶片,L2-倒2叶,L3-倒3叶,L4-倒4叶。A,Tissue-specific expression of OsDTH10;M,Shoot apical meristem;R,Root;L,Leaf;LS,Leaf sheath;S,Stem;P1,14cm panicle;P2,4cm panicle;P3,25cm panicle.B,Expression of OsDTH10in leaves at various leaf-ages.L1,The unexpanded flag leaf;L2,The second leaf from the top;L3,The third leaf from the top;L4,The fourth leaf from the top.图3 OsDTH10的组织特异性表达分析Fig.3.Tissue-specific expression of OsDTH10.

A-短日照条件下OsDTH10的表达水平;B-长日照条件下OsDTH10的表达水平。A,Expression level of OsDTH10in short-day conditions;B,Expression level of OsDTH10in long-day conditions.图4 OsDTH10基因在不同光周期条件下的昼夜节律性表达Fig.4.Circadian rhythm expression of OsDTH10 under different photoperiods.
由于过表达转基因植株表现延迟抽穗,我们推测OsDTH10基因可能与光周期途径相关,为此我们进一步分析了目的基因在不同光周期下的表达。结果表明,无论在长日照还是短日照条件下,Os-DTH10在白天(光期)的表达量较高,而在晚上(暗期)表达量较低(图4-A,B)。短日照条件下,Os-DTH10 mRNA水平从早上6:30开始积累到下午15:30达到峰值,在凌晨降到最低(图4-A)。长日照条件下,OsDTH10 mRNA积累表现为双峰,分别在9:30和15:30达到峰值,在凌晨3:30降到最低(图4-B)。
3 讨论
植物开花是环境因素与控制植物开花的基因网络相互作用的结果,目前已明确水稻的成花主要是由成花素负责[6-7]。短日照条件下起主要作用的成花素是Hd3a,在长日照条件下起主要作用的成花素是RFT1[11]。这两个成花素都是FT-like家族基因[16]。模式植物拟南芥中FT和TFL1是FT-like家族中的两个成员,但它们的功能完全相反[15],FT促进拟南芥开花,而TFL1则抑制拟南芥开花,维持其营养生长,充当反成花素的作用[19-20]。水稻中FT-like家族共有13个成员[16],除了已经报道的RFT1和Hd3a分别履行水稻长日照及短日照成花素的功能外,其他成员的功能仍不清楚。本研究揭示了该家族另一成员OsDTH10在水稻中的功能,即过量表达OsDTH10推迟了水稻的抽穗开花时间。这与Hd3a及RFT1有所不同,Hd3a及RFT1是水稻开花的激活子,过量表达Hd3a及RFT1导致水稻提早抽穗。OsDTH10是水稻开花的抑制子,这与拟南芥TFL1基因的功能相似,是水稻中的反成花素。这一结果充分说明了水稻FT-like家族的成员在进化中功能有了很大的分化,因此弄清该家族中每个成员的功能,揭示哪些成员在功能上是冗余的,哪些成员在功能上已有分化,对于揭示水稻成花素基因的进化及功能具有重要的意义。
实时定量PCR结果表明,OsDTH10具有昼夜节律性表达,在白天表达量较高,而在夜晚表达量较低,这与Hd3a及RFT1表达模式一致。但Os-DTH10无论短日照还是长日照条件下都有较高的表达,这与Hd3a在短日照条件下表达量低有所不同。另外,现已明确成花素基因Hd3a及RFT1首先在叶片接受光的信号,然后运输到茎顶端分生组织中起作用。分子机理方面,Hd3a或RFT1首先在细胞质中与水稻14-3-3基因(OsGF14b,OsGF14c)互作形成复合体,然后进入细胞核中与水稻OsFD1结合形成复合体,最终调控开花身份基因OsMADS14和OsMADS15的表达水平来调控水稻抽穗的早晚[21-22]。OsDTH10是否有这样的功能,能否和这些已知的基因相互调控还不清楚,因此,后期创建OsDTH10 RNAi转基因植株,利用RNAi及过表达转基因植株,用不同的光周期条件处理,研究光周期调控通路中其他已知基因的表达,揭示目的基因与光周期通路中其他关键基因的调控关系,从而揭示OsDTH10在水稻抽穗开花时间上的功能。
参考文献:
[1] 宋远丽,栾维江.水稻开花的光温调控分子机理.中国水稻科学,2012,26(4):383-392.Song Y L,Luan W J.The molecular mechanism of light and temperature control the flowering in rice.Chin J Rice Sci,2012,26(4):383-392.(in Chinese with English abstract)
[2] Song Y L,Gao Z,Luan W S.Interaction between temperature and photoperiod in regulation of flowering time in rice.Sci China Life Sci,2012,55(3):241-249.
[3] Izawa T.Daylength measurements by rice plants in photoperiodic short-day flowering.Int Rev Cytol,2007,256:191-222.
[4] Garner W W,Allard H A.Effect of the relative length of day and night and other factors of the environment on growth and reproduction in plants.J Agric Res,1920,18:553-606.
[5] Itoh H,Nonoue Y,Yano M,et al.A pair of floral regulators sets critical day length for Hd3aflorigen expression in rice.Nat Genet,2010,42:635-638.
[6] Song Y L,Luan W J.Molecular regulatory network of flowering by photoperiod and temperature in rice.Rice Sci,2012,19 (3):169-176.
[7] Tsuji H,Taoka K,Shimamoto K.Regulation of flowering in rice:Two florigen genes,a complex gene network,and natural variation.Curr Opin Plant Biol,2011,14(1):45-52.
[8] Luan W,Chen H,Fu Y,et al.The effect of the crosstalk between photoperiod and temperature on the heading-date in rice.PLoS ONE,2009,4:e5891.
[9] Yano M,Katayose Y,Ashikari M,et al.Hd1,a major photoperiod sensitivity quantitative trait locus in rice,is closely related to the Arabidopsis flowering time gene CONSTANS.Plant Cell,2000,12:2473-2484.
[10]Tamaki S,Matsuo S,Wong H L,et al.Hd3aprotein is a mobile flowering signal in rice.Science,2007,316:1033-1036.
[11]Komiya R,Yokoi R,Shimamoto K.A gene network for longday flowering activates RFT1 encoding a mobile flowering signal in rice.Development,2009,136:3443-3450.
[12]Doi K,Izawa T,Fuse T,et al.Ehd1,a B-type response regulator in rice,confers short-day promotion of flowering and controls FT-like gene expression independently of Hd1.Genes Dev,2004,18:926-936.
[13]Xue W,Xing Y,Weng X,et al.Natural variation in Ghd7 is an important regulator of heading date and yield potential in rice.Nat Genet,2008,40:761-767.
[14]Chardon F,Damerval C.Phylogenomic analysis of the PEBP gene family in cereals.J Mol Evol,2005,61(5):579-590.
[15]Kobayashi Y,Kaya H,Goto K,et al.A pair of related genes with antagonistic roles in mediating flowering signals.Science,1999,286(5446):1960-1962.
[16]Ogiso-Tanaka E,Matsubara K,Yamamoto S,et al.Natural variation of the RICE FLOWERING LOCUS T 1 contributes to flowering time divergence in rice.PLoS One,2013,8(10):e75959.
[17]Hiei Y,Ohta S,Komari T,et al.Efficient transformation of rice(Oryza sativa L.)mediated by Agrobacterium and sequence analysis of the boundaries of the T-DNA.Plant J,1994,6(2):271-282.
[18]卢扬江,郑康乐.提取水稻DNA的一种简易方法.中国水稻科学,1992,6(1):47-48.Lu Y J,Zheng K L.A simple extraction methods of rice DNA.Chin J Rice Sci,1992,6(1):47-48.(in Chinese with English abstract)
[19]Kardailsky I,Shukla V K,Ahn J H,et al.Activation tagging of the floral inducer FT.Science,1999,286(5446):1962-1965.
[20]Abe M,Kobayashi Y,Yamamoto S,et al.FD,a bZIP protein mediating signals from the floral pathway integrator FT at the shoot apex.Science,2005,309(5737):1052-1056.
[21]Taoka K,Ohki I,Tsuji H,et al.Structure and function of florigen and the receptor complex.Trends Plant Sci,2013,18 (5):287-294.
[22]Tsuji H,Taoka K,Shimamoto K.Florigen in rice:Complex gene network for florigen transcription,florigen activation complex,and multiple functions.Curr Opin Plant Biol,2013,16(2):228-235.
Overexpressing Analysis of the Flowering Time Gene OsDTH 10in Rice
LI Ying-ying,XIONG Wei,SONG Ting,CAOZhen-hua,LUAN Wei-jiang*
(College of Life Science,Tianjin Normal University/Tianjin Key Laboratory of Animal and Plant Resistance,Tianjin 300387,China;*Corresponding author,E-mail:lwjzsq@163.com)
LI Yingying,XIONG Wei,SONG Ting,et al.Overexpressing analysis of the flowering time gene OsDTH10 in rice.Chin J Rice Sci,2016,30(2):121-126.
Abstract:Flowering time(heading date)is an important agronomic trait of crops,and it is closely linked with the yield of crops.To reveal the function of rice flowering time gene OsDTH10in rice,we constructed an overexpressing vector of OsDTH10 and analyzed the function of OsDTH10 by the reverse genetics.The results showed that the OsDTH10-overexpressing(OsDTH10-OV)transgenic plants displayed a late heading phenotype,suggesting that the overexpression of OsDTH10delayed the heading-date of rice.RT-PCR expression analysis showed that the expression level of OsDTH10was increased obviously in OsDTH10-OVlines with phenotype compared with the transgenic lines without phenotype and wild type plants,indicating that late heading phenotype in OsDTH10-OVlines was caused by the overexpression of OsDTH10.The tissue-specific expression showed that OsDTH10 was expressed in different organs,with the higher expression levels in stem and leaf sheath.Also,we further analyzed the expression of OsDTH10 in leaves at various leaf-ages,and the result showed that OsDTH10 exhibited higher expression in unexpanded flag leaf and the second leaf from the top than these in the third and the fourth leaf from the top.In addition,we also analyzed the expression of OsDTH10 in different photoperiods,and the result indicated that OsDTH10displayed a higher expression during the day time(light stage)and a lower expression at night(dark stage)whatever short-day and long-day conditions,suggesting that OsDTH10 might be involved in the photoperiodic pathways to regulate the heading-date in rice.
Key words:rice;OsDTH10;overexpression;expression analysis
中图分类号:Q786;S511.01
文献标识码:A
文章编号:1001-7216(2016)02-0121-06
基金项目:国家自然科学基金资助项目(31171515);天津市高校中青年骨干创新人才培养计划资助项目(ZX110GG017)。
收稿日期:2015-09-21;修改稿收到日期:2015-12-17。

