高通量测序分析冷鲜滩羊肉储藏过程中的细菌群落多样性
张赫宇,杨 波,罗瑞明,吴亮亮,苏春霞
(宁夏大学农学院,宁夏银川 750021)
高通量测序分析冷鲜滩羊肉储藏过程中的细菌群落多样性
张赫宇,杨波,罗瑞明*,吴亮亮,苏春霞
(宁夏大学农学院,宁夏银川 750021)
通过Illuminu测序技术对滩羊肉贮藏过程中菌群的16S rRNA基因V3区进行测序,探讨冷鲜滩羊肉在贮藏过程中细菌的菌群组成,以及随着储藏时间延长其优势菌群的变化。结果表明:滩羊肉在贮藏过程中其微生物菌群变化具有多样性;三个不同贮藏时期滩羊肉的微生物都是由11个细菌门、23个纲、35个属组成,但是不同时期的细菌的丰富度不同,随着储存时间延长,到第8 d时,冷鲜滩羊肉表面的主要类群为变形菌门(Proteobacteria)占71.59%,厚壁菌门(Firmicutes)占23.15%。其中Proteobacteria类群随着储藏时间的增加呈现减少的趋势,而Firmicutes菌群变化趋势与之相反。随着储藏时间变化,滩羊肉贮藏中主要腐败菌为假单胞菌属(Pseudomonasspp.)、不动杆菌属(Acinetobacterspp.)、嗜冷杆菌属(Psychrobacterspp.)、肠杆菌属(Enterobacteriaceaespp.)、热死环丝菌(Brochothrixspp.)、乳酸菌属(Lactobacillusspp.)。其中假单胞菌属(Pseudomonasspp.)是造成冷鲜滩羊肉腐败变质的主要优势菌。
冷鲜羊肉,高通量测序,贮藏过程,群落多样性
滩羊为宁夏优势特色畜种,肉质细嫩,脂肪分布均匀,膻味很淡,加工后风味独特[1]。清真冷鲜滩羊肉是出口中东地区的首选产品。然而,滩羊冷鲜肉加工理论滞后,制约了加工技术发展。所以研究优势腐败菌的生长变化和深入了解肉品表面微生物群落特征对于控制冷鲜滩羊肉的品质具有十分重要的意义。目前,国内外冷鲜肉微生物研究着眼于生物多样性分析,确定优势微生物,通过选择性培养法研究优势微生物生长规律,结合肉品质感官与理化指标,建立货架期预测一级、二级模型,在冷鲜肉微生物生态学领域却未做深入研究,模型以具体实验值拟合,局限性强。近年来随着分子生物学方法的兴起,通过高通量测序法[2-3]研究微生物多样性已受到越来越广泛的关注。高通量测序技术在分析微生物的群落结构时,有着独特的优势,能够产生测序覆盖深度非常高的16S rRNA的测序数据,通过BLAST比对或者是将16S rRNA序列聚类成分类操作逻辑单元(OTU),根据OTU的数目和序列分析得出微生物多样性和物种的丰度[4]。基于16S rRNA基因的高通量测序技术因消除了克隆问题,并且可以综合研究各个可变区,同时分析方法也相对成熟,已成为微生物群落研究中非常重要的工具[5],并已广泛用于猪屠宰及猪肉贮藏过程中微生物多样性的研究[6]、分析冷藏过程中真空包装猪肉的细菌群落变化[7]、运用实时PCR技术建立分子预测模型,预测真空包装冷鲜猪肉中单增李斯特菌的生长情况[8]等,但目前利用16S rRNA基因的高通量测序技术研究冷鲜滩羊肉贮藏过程中细菌群落演替规律鲜有报道。
本研究主要借助于宏基因组测序技术,分析微生物群体基因组成及功能,以0 d作为对照,研究了贮藏4、8 d时的冷鲜滩羊肉微生物群体基因组成及功能,多样性与丰度,探求微生物与环境,微生物与宿主之间的关系。研究冷鲜滩羊肉微生物群落多样性及演替规律。宏基因组测序研究避开了微生物分离培养的过程,为研究微生物相互作用提供了有效工具。第二代高通量测序技术能够对环境微生物进行深度测序,灵敏地探测出环境微生物群落结构随外界环境的改变而发生的极其微弱的变化。
1 材料与方法
1.1材料与仪器
新鲜滩羊后腿肉(当天屠宰)取自宁夏盐池大夏牧场,后立刻放入干冰中储存,返回实验室后迅速放入-20 ℃冰箱1 h,使其中心温度降至4 ℃,将其取出置于4 ℃冰箱保藏。
Axygen细菌基因组DNA提取试剂盒江苏省康宁生命科学(吴江)有限公司;Miseq高通量测序仪美国Illumina公司;ScanSpeed MiniVac Alpha冷冻离心机美国Scan speed公司;SPECTRAMAX PLUS384MD酶标仪美国MD公司;EPS 300电泳仪中国天能公司;Tanon-2500凝胶成像系统中国天能公司;Concentrator plus浓缩仪美国Thermo公司;Sorvall Legend Micro 17常温离心机美国Thermo公司;G-560E漩涡混合仪vortex-genie2美国SI公司;GeneAmp PCR system 9700普通PCR仪美国Life公司;JT-C均质器漯河市金田实验设备研究所;Scan Drop 200微量紫外光分光光度计德国耶拿公司。
1.2实验方法
1.2.1样品采集新鲜采购的滩羊后腿肉用灭菌刀去除筋膜后分为45 份,每份10 g左右,放入无菌托盘,用无菌保鲜膜包好,置于4 ℃冰箱。每15份为一组,分别在0、4和8 d[9]时提取45个样品的DNA。
1.2.2细菌总DNA的提取采用Axygen细菌基因组DNA提取试剂盒,严格按照操作步骤进行,步骤见文献[10]。
1.2.3细菌16S rDNA V3区高通量测序将在0、4、8 d所提取DNA样品分别作为PCR反应的模板,对16S rRNA基因的V3区进行扩增,上下游引物分别是27F:aatgatacggcg Accaccgagatctacactcttt ccctacacg acgctcttccgatctNNNNCCTACGGGAGGCAGCAG;1492R:caagcagaagacggcatacgagatCGTGATgtgactggagttcagacgtgtg ctcttccgatctATTACCGGCTGCTGG[11]。PCR反应采用25 μL体系:10 μmol·L-1的上下游引物各1.5 μL,Taq酶(Q5Master Mix)12.5 μL,DNA模板和dd H2O共9. 5 μL,个样品的DNA总量约为5 ng。PCR反应程序为:98 ℃预变性30 s,98 ℃ 10 s,50 ℃ 30 s,72 ℃ 30 s扩增25个循环,72 ℃延伸7 min。1%琼脂糖凝胶检测,切胶回收。采用AxyPrep DNA Gel Extraction Kit胶回收试剂盒对PCR产物进行胶回收。采用Quantity-iT PicoGreen定量试剂盒对胶回收PCR产物进行定量。将通过PCR扩增得到的3组样品(0、4、8 d)的目的DNA采用16S rDNA V3区扩增,采用Illumina的MiSeq测序平台的双端251bp测序模式对3个环境样本进行高通量测序,根据MiSeq测序数据的低质量分数集中于末端的分布特点,首先对原始数据进行质量控制,利用Trimmomatic软件进行去杂,具体标准如下:去除尾部质量值低于25的碱基;同时设置50 bp滑动窗口,1 bp步移,窗口内碱基平均质量不低于25;最后去除长度低于50 bp的序列。其次利用flash软件将高质量双端序列进行连接,最小overlap区10 bp,错配率0.2,去除含模糊碱基的序列。根据barcode将序列按样品拆分,并去除barcode,获得最终用于分析的优化序列。将序列上传至MG-RAST(Metagenomics analysis server http://metagenomics.anl. gov/)服务器[12]。
1.2.4软件数据分析为了更好的验证测序量是否能够真实的反映原始样品的微生物群落多样性,对不同储藏时期冷鲜滩羊肉菌群变化通过绘制稀释性曲线(rarefaction curve)评价所测序量是否覆盖全部类群。使用QIIME(Version1.50)软件[13]对测序数据进行总体分析,然后利用Greengenes数据库进行16S rRNA基因序列比对,确定序列对应微生物的分类学地位。通过软件将相似性大于97%的序列定位为一个OTU(operational taxonomic units),通过Alpha多样性指数分析比较三组样品的微生物多样性;最后通过 Mothur(http://www.mothur.org)中的sum-mary. single命令,计算常用的α生物多样性指数[14](beta diversity):Chao、ACE、Simpson、Shannon.、和coverage指数,分析样品之间群落组成的相异性。细菌群落丰富度用Chao指数表示,其值越高表明群落物种的丰富度越高,其计算方法见文献[15];Shannon指数反映样品的多样性程度,其值越高表明群落物种的多样性越高;Simpson指数反映了物种的优势度,计算方法见文献[16];Shannon指数越大,而Simpson指数越小,说明群落多样性越高,均衡化程度越高。Coverage是指各样品文库的覆盖率,其数值越高,则样本中序列没有被测出的概率越低。该指数实际反映了本次测序结果是否代表样本的真实情况[17]。选取分析数据中每个OTU中一个有代表性的序列,通过进行16S r RNA基因序列的比对,然后利用Mega 4.0软件建立系统发育树。
2 结果与分析
2.1滩羊肉宏基因组DNA提取
分别提取贮藏0、4、8 d滩羊肉的宏基因组DNA,用1%的琼脂糖凝胶进电泳检测,如图1所示,3个样本基因组条带清洗,无降解,进一步利用Scan Drop 200微量紫外光分光光度计测得三个样本的DNA浓度都≥200 ng/μL,OD260/280值为1.8~2.0。没有蛋白及试剂污染,满足后续实验要求。

表3 OTU与样本的丰度矩阵
图1 滩羊肉宏基因组DNA提取Fig.1 Macro genomic DNA extraction detection of Tan-sheep
2.216S rDNA V3区测序分析
处理获得的序列信息,去除低质量片段、检测错误序列、连接后引物序列以及无法与数据库中序列信息比对一致的序列,获得的序列为有效信息见表1,结果显示有效长度在 453.87 bp左右,符合预期的片段大小[20]。

表1 预处理过程统计
2.3测序数据的合理性分析
为了验证测序量是否能够真实的反映原始样品的微生物群落的多样性,对0、4、8 d的多样性利用rarefaction curve进行评估。样品的稀释性曲线相对趋于平稳(图2),说明取样合理,测序深度已基本覆盖样品中所有微生物,同时也表明了2个样本丰富的多样性。

表2 连接后的序列长度统计

图2 序列相似度97%的丰富度稀疏分析图Fig.2 Sparse abundance analysischart of 97% sequence similarity
2.4群落结构分析
2.4.1基于OTU对样品进行初步分析3个样品共获得了1168个OUT,其中ZHY0 d样品225个OUT,ZHY4 d样品661个OUT,ZHY8 d样品328个OUT这些样品全部属于细菌界,部分属于变形菌、厚壁菌门,还有部分属于变形菌纲。进一步对各样品之间共享OTU进行分类统计,分类统计显示ZHY0 d占样品OUT的19.26%,ZHY4 d占样品OUT的56.59%,ZHY8 d占样品OUT的28.08%。可以看出,ZHY4 d样品的群落结构最为复杂。
2.4.2细菌群落多样性(ALPHA)和丰富度分析冷鲜滩羊肉在储藏过程中的Alpha多样性指数变化如表4所示,储存4 d的冷鲜滩羊肉Chao指数高于0 d和8 d,其Shannon指数显著高于0 d和8 d,其Simpson指数低于其他两组,说明储存4 d的冷鲜滩羊肉细菌群落丰富度和均衡化程度高于储存0 d和8 d的冷鲜滩羊肉。这两种指数能够对群落物种组成的丰富度及均匀度进行综合评价。同时三组样品的Coverage指数均在0.99以上,说明本次实验的测序量已经达到饱和,测序结果基本能够反映滩羊肉微生物菌群多样性组成。
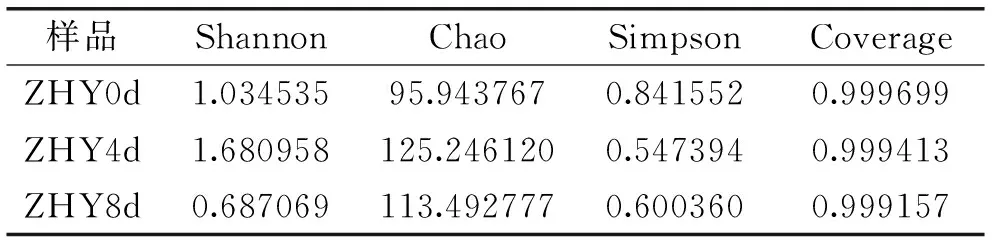
表4 序列相似度97%的Alpha多样性指数
2.4.3基于Weighted UniFrac PCoA的群落结构分析如图3所示,利用各样品序列间的进化信息计算样品距离,观察3种不同滩羊肉样品多样性差异。其第一主成分和第二主成分的贡献率分别是80.94%和9.98%,随着储存时间的延长,0 d和4 d两组样品呈现出一定的交叠现象,这说明0 d和4 d两组样品的细菌群落结构呈现一定的相似性,而8 d的样品呈现一定的聚类趋势,说明8 d样品相对于0 d和4 d两组样品来讲具有自己特殊的细菌群落结构。
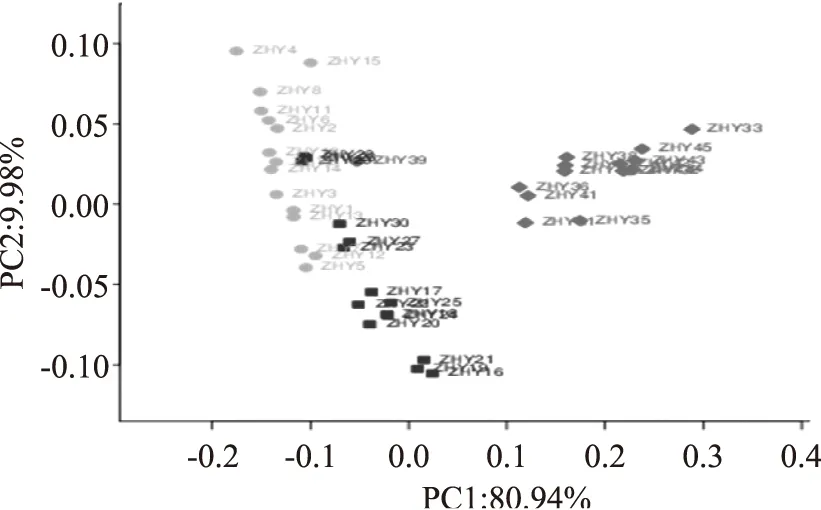
图3 种滩羊肉样品细菌菌群结构Unifrac加权PCoA分析分布图Fig.3 Unweighted phylogenetic distanceanalysis of three differents
2.5不同储藏时间冷鲜滩羊肉细菌的组成
对每个样本和每个物种单元分类进行序列丰度计算,构建物种分类单元和样本丰度矩阵文件。计算三种储藏时间下冷鲜滩羊肉在同一门分类下OUT分布及相对丰度,物种分类单元丰度比例图是分别根据物种分类单元的界、门、纲、目、科、属、种层对每个样本进行计数,从而计算相对比例。以门和纲的分类单元比例丰度图见图4。

图4 三种样本中细菌在不同分类水平上的分布Fig.4 Relative abundance statistical figure of phylum and class classificatio
通过序列比对和注释,根据各样品在门水平分布的分析可以看出,随着储藏时间的延长,滩羊肉样品中细菌在门水平上的分类是相同的,滩羊肉样品的序列共分属于11个门,而且类群的丰度很相似,但丰度比例不同。其中11个门都属于细菌门。分别是:变形菌门Proteobacteria,厚壁菌门Firmicutes、非典型细菌门unclassified_Bacteria、拟杆菌门Bacteroidetes、放线菌门Actinobacteria、绿弯菌门Armatimonadetes、酸杆菌门Acidobacteria、糖化细菌门Candidatus_Saccharibacteria、蓝藻菌门Cyanobacteria/Chloroplast、浮霉菌门Planctomycetes、互养菌门Synergistetes。变形菌门、厚壁杆菌门、拟杆菌门、放线菌门、蓝藻门、酸杆菌门、互养菌门,共同构成了滩羊肉菌群的主要结构。除了主要菌群外,其它门分类的菌群丰度都较低,甚至有的门水平出现了仅有为数不多的几条序列,占有百分比不足 0.01%。这些结果表明菌群具有多样性丰富,丰度集中的特点。

表5 储藏期间冷鲜滩羊肉表面优势菌门组成比例
注:-未检出。
分析冷鲜滩羊肉在储存期间各种细菌占总菌数的百分比,通过表3分析测序结果可以看出,冷鲜滩羊肉在储藏期间,变形菌门(Proteobacteria)广泛存在于3个单样品中,并且占据绝对优势,其含量都是最多,达到70%以上,且ZHY0 d样品中的变形菌门(Proteobacteria)的含量达到89.63%以上。变形菌门中的γ-变形菌是主要优势菌,在储藏到第8 d时,占比虽有所下降,但仍是优势菌。很多重要的病原菌都属于这个纲,如假单胞菌和肠杆菌。随着贮藏时间的延长,厚壁杆菌门的含量逐渐增多,逐渐成为优势菌。贮藏到第8 d,变形菌门依然是优势菌,占送检结果70%以上,是造成真空包装冷鲜滩羊肉腐败的主要微生物。随着储藏时间延长,芽孢杆菌所占比例成倍增加,这种趋势与芽孢杆菌的特性有关,芽孢杆菌能够产生内生孢子,对外部环境因素抵抗力强,生命力强,代谢快,繁殖快,它的占比比一般病原菌分子大四倍,占据空间优势,可以抑制有害菌的生长繁殖。

表6 储藏期间冷鲜滩羊肉表面优势菌纲组成比例
由此可以看出在储藏至第8 d时,冷鲜滩滩羊肉的主要优势菌为变形菌、芽孢杆菌、非典型细菌、梭状芽孢杆菌和鞘脂杆菌等。变形菌中的假单胞菌和肠杆菌是造成冷鲜肉腐败变质的主要微生物,这与已有研究指出冷鲜羊肉表面的优势腐败菌为假单胞菌的结论一致[14]。
2.6细菌菌群系统发育树的构建
细菌菌群系统发育树结果见图5。

图5 细菌菌群系统发育树 Fig.5 Phylogenetic tree of bacterial flora
3 结论
2009年,诸永志等采用选择性培养基分析了江苏徐州地区冷却羊肉的菌相,其中假单胞菌属、乳酸菌、葡萄球菌和微球菌、肠杆菌科、热死环丝菌是构成该品种冷却羊肉的主要微生物[18]。2013年,刘莹莹等采用选择性培养基分析了宁夏吴忠地区冷却羊肉的初始菌相:主要腐败菌为假单胞菌、肠杆菌、乳酸菌、葡萄球菌[9]。本实验通过MiSeq测序平台对16S rRNA基因分析滩羊肉贮藏过程中微生物多样性以及群落结构。
通过多样性分析发现,随着测序量的增加,各多样性曲线逐渐趋于平行,体现测序达到饱和,测序结果基本能够反映滩羊肉微生物菌群多样性组成,说明滩羊肉贮藏过程中其微生物组成具有丰富的多样性。通过群落结构分析发现,其细菌主要分布于变形菌门、厚壁菌门、拟杆菌门、非典型细菌门、放线菌门等11个门,23个纲,35个属,变形菌门为优势菌门,其中γ—变形菌纲中的假单胞菌属、不动杆菌属、肠杆菌属占一定优势。厚壁菌门随着贮存时间的延长也逐渐增多,其中芽孢杆菌纲中的热死环丝菌、乳酸菌、梭菌属占一定优势。相比其他研究方法[19-20]对于滩羊肉细菌群落分析更加全面,信息量更大。并且利用高通量测序技术能够快速了解冷鲜滩羊肉在储存过程中的重要菌株,并且得到主要腐败菌为假单胞菌属、不动杆菌属、嗜冷菌属、肠杆菌属、热死环丝菌、乳酸菌。其中假单胞杆菌为优势菌,很多文献都有研究。同时还能发现储藏过程中的candidate division TM7、bacterium PE03-7A6 等未知微生物,这对冷鲜滩羊肉的贮藏和保鲜和研究优势腐败菌的生长变化,并且深入了解肉品表面微生物群落特征对于控制冷鲜滩羊肉的品质具有十分重要的意义。
[1]李伟,罗瑞明,李亚蕾,等. 宁夏滩羊肉的特征香气成分分析[J].现代食品科技,2013,29(5):1173-1177.
[2]Amanda Gribble,Gale Brightwell. Spoilage characteristics of Brochothrix thermosphacta and campestris in chilled vacuum packaged lamb and their detection and identification by real time PCR[J]. Meat Science,2013,94(3):361-368.
[3]Rutvisuttinunt W,Chinaawirotpisanp,Simasa S,et al. Simultaneous and complete genome sequencing of influenza A and B with high coverage by Illumina Mi Seq Platform[J]. Virol Methods,2013,193(2):394-404.
[4]南春燕,马雅军,徐建农,等. 中华按蚊幼虫肠道细菌宏基因组的组成研究[J]. 中国寄生虫学与寄生虫病,2013,31(2):114-119.
[5]Williams S,Foster P,Littlewood D. The complete mitochon-drial genome of a turbinid vetigastropod from Mi Seq Illumina sequencing of genomic DNA and steps towards a resolved gastropod phylogeny[J]. Gene,2014,533(1):38-47.
[6]周琰冰,艾启俊,张德权. 应用PCR-DGGE技术研究贮藏期内冷鲜羊肉表面的优势菌[J]. 食品科学,2015,36(16):237-240.
[7]Y. Jiang F,Gao XL,Xu. Changes in the bacterial communities of vacuum-packaged pork during chilled storage analyzed by PCR-DGGE[J]. Meat Science,2010,86(4):889-895.
[8]何帆,徐幸莲,周光宏. 不同包装条件下冷却猪肉品质特征指标及动态模型研究[J]. 食品科学,2010,31(24):473-478.
[9]刘莹莹,罗瑞明,卢君逸,等. 冷却羊肉贮藏中品质变化及假单胞菌生长预测模型的建立[J]. 肉类研究,2013,27(5):5-9.
[10]高秋月,景奉香,李海燕. 基于磁珠的细菌基因组DNA提取方法[J]. 安徽农业科学,2010,38(21):11071-11074.
[11]Chun-Lan Xu,Rui Sun,Xiang-Jin Qiao,et al. Protective effect of glutamine on intestinal injury and bacterial community in rats exposed to hypobaric hypoxia environment[J]. World Journal of Gastroenterology,2014,20(16):4662-4674.
[12]Wang S,Hou W,Dong H,et al. Control of temperature on microbial community structure in hot springs of the Tibetan Plateau[J]. PLoS One,2013,8(5):1-14.
[13]F Dussault,RA Holley. Evaluation of the hygienic performances of the processes for cleaning,dressing and cooling pig carcasses at eight packing plants[J]. International Journal of Food Microbiology,2000,58(12):65-72.
[14]向文良,罗海,梁华忠,等. 基于16S-23S rRNA ITS AFLP 对米酒发酵过程中原核微生物的演替分析[J]. 酿酒科技,2010,188(2):43-46.
[15]Chao A N. Non-parametric estimation of the number of classes in a population[J]. Scandinavian Journal of Statistics,1984,11(4):265-270.
[16]张薇,胡跃高,黄国和,等. 西北黄土高原柠条种植区土壤微生物多样性分析[J]. 微生物学报,2007,47(5):751-756.
[17]Buee M,Reich M,Murat C,et al.Pyrosequencing analyses of forest soils reveal an unexpectedly high fungal diversity[J]. NewPjytologist,2009,184(2):449-456.
[18]诸永志,姚丽娅,王道营,等. 冷却羊肉微生物菌群分析与鉴定[J]. 江西农业学报,2009,21(3):124-126.
[19]LUO Hui-bo,HUANG Zhi-guo,LI Hao,et al. PCR-SSCP Analysis of the Pronucleus Microbial Communities in Luzhou-flavor Daqu[J]. Microbiology China,2009,36(9):1363-1367.
[20]叶光斌,罗惠波,杨晓东,等.基于免培养法研究泸州地区浓香型白酒窖泥原核微生物群落结构[J].食品科学,2013,34(17):176-181.
Analysis of bacterial community diversity in cold fresh Tan lamp during different storage time using pyrosequencing
ZHANG He-yu,YANG Bo,LUO Rui-ming*,WU Liang-liang,SU Chun-xia
(College of Agricultural,Ningxia University,Yinchuan 750021,China)
The Bacteria composition of Tan lamb and predominant flora changes with storage time extension were discussed by sequencing the microbial community of Tan lamb during the storage with Illuminu sequencing technology. The results indicated that the microbial flora showed diversity during the storage of Tan lamb. The microorganisms in the Tan lamb were constituted of 11 phylum of bacteria,23 classes and 35 genuses during its different storage periods,but it has showed different richness of bacteria in the different periods. With the extension of storage time,in the 8th day,the main bacterial populations on the surface of the cold fresh Tan lamb were:71.59% Proteobacteria;23.15% Firmicutes. The proteobacteria showed a decreasing trend with increase of storage time,but the Firmicutes changed in contrary. As the storage time increased,thePseudomonasinProteobacteriaaccounting for the most of the bacteria,which was the Specific Spoilage Organisms that caused clod fresh Tan lamb’s corruption.
cold fresh Tan lamp;barcoded pyrosequencing;storage process;community diversity
2016-01-14
张赫宇(1989-),女,硕士研究生,研究方向:畜产品贮藏与加工,E-mail:zhangheyu410@163.com。
罗瑞明(1964-),男,博士,教授,研究方向:畜产品贮藏与加工,E-mail:ruimingluo.nx@163.com。
国家自然科学基金项目(31460431)。
TS251
A
1002-0306(2016)13-0177-06
10.13386/j.issn1002-0306.2016.13.027

