擎天凤梨苞片叶绿素代谢关键基因的分离及褪绿的分子机理
刘建新,丁华侨,田丹青,王炜勇,刘慧春
擎天凤梨苞片叶绿素代谢关键基因的分离及褪绿的分子机理
刘建新,丁华侨,田丹青,王炜勇,刘慧春
(浙江省萧山棉麻研究所,杭州 311202)
【目的】擎天凤梨是一种新潮花卉,其苞片在成花变色过程中常常伴随着绿色的逐渐消退。分离叶绿素生物合成与降解途径的关键基因,探索在苞片变色过程中叶绿素代谢的分子基础和机理,有助于解析呈色苞片中绿色退化的原因。【方法】通过cDNA文库构建及EST批量测序获得叶绿素合成关键酶:谷氨酰tRNA合成酶基因(glutamyl-tRNA synthetase,)和尿卟啉原-Ⅲ合酶基因(uroporphyrinogen-Ⅲsynthase,);通过同源克隆技术获得叶绿素降解关键酶:脱镁叶绿素酶(pheophytin pheophorbide hydrolase/pheophytinase,)基因;通过采用透光系数法测量叶绿素含量、HPLC法测定类黄酮含量以及采用实时定量PCR法测定叶绿素代谢关键基因的基因表达模式,探索呈色苞片中绿色部分消失的分子机理。【结果】获得的(GenBank:KP144289),序列长为1 140 bp,编译171AA的蛋白氨基酸序列,并存在GlnRS-cataytic core和Nt-trans superfamily保守区;获得的(GenBank:KP144288)cDNA序列长为613 bp,编译188AA的蛋白氨基酸序列,并存在HemD和HemD Superfamily保守区;获得的(GenBank:KP723523)cDNA序列长为266 bp,编译88AA的蛋白氨基酸序列,并存在Abhydrolase-6保守区。通过对不同变色阶段苞片的叶绿素、类黄酮含量测定以及叶绿素代谢相关基因的表达水平分析,可知苞片变色过程伴随着叶绿素含量的显著降低和类黄酮含量的明显增加,同时叶绿素合成相关的和表达水平也明显降低,而叶绿素降解关键酶基因的表达水平在苞片变色初期显著上升,在变色完成后则降低到接近绿叶状态的最低水平。而作为对照材料的绿叶,其叶绿素含量是最高的,而类黄酮含量是最低的,同时叶绿素的合成相关酶基因表达量最高,而降解相关酶基因表达量最低,这与彩叶植物中叶绿素和类黄酮色素的表现较为一致。【结论】获得了擎天凤梨的叶绿素合成相关酶基因和以及降解关键酶基因。由于叶绿素合成量减少,降解量显著增加引起叶绿素含量的降低,从而影响苞片变色过程中绿色的消退,并伴随类黄酮含量的增加。在叶绿素的降解过程中起关键性作用。
擎天凤梨;叶绿素;谷氨酰tRNA合成酶基因;尿卟啉原-Ⅲ合酶基因;脱镁叶绿素酶
0 引言
【研究意义】擎天凤梨()是一种新潮花卉,属于凤梨科(Bromeliaceae Juss.)植物。原产于美洲的热带和亚热带地区,多年生常绿草本,其主要观赏部位是呈色苞片[1]。擎天凤梨主流品种‘Ostara’的呈色苞片为红色,苞片红色的形成常常伴随着绿色部分的消退,最终红色部分向苞片尖端扩散而导致整个苞片全部呈现出红色(图1)。研究呈色苞片中绿色物质的消退原因对探索叶绿素在变色苞片中合成和降解的代谢机制以及观赏凤梨的催花研究有重要意义。【前人研究进展】作为一种变态叶的苞片,一般认为其呈色物质与彩叶植物叶片色素的形成类似。与成色相关的色素包括叶绿素、类黄酮、类胡萝卜素、水溶性生物碱及其衍生物等[2-4],并受叶片的PH值以及光温等环境因子的影响[5]。Kate等[6]对擎天凤梨在高强度光照下叶绿体发生适应性变化进行了研究。通过对一些彩色植物进行研究,发现叶色的呈色程度常常是由花青素苷/叶绿素的比值决定[7-9]。Nakayama等[10]对姜荷花与苞片颜色形成相关的锦葵花素3-芸香糖苷(Malvidin 3-rutinoside)进行了研究。王长泉等[11]进行了一品红苞片花色素的分离。GTS和UROS都是叶绿素合成途径中起关键性作用的酶。脱镁叶绿素酶(pheophytin pheophorbide hydrolase/pheophytinase,PPH)是一个最新发现的叶绿素降解代谢关键酶[12-14],普遍存在于高等植物基因组,目前已在拟南芥(AT5G13800)、黄瓜[15]、花椰菜、西兰花(OL386)、大白菜(AC189212.2)等植物中被克隆获得。【本研究切入点】前人对擎天凤梨叶绿素合成与降解的研究主要集中在各类彩叶植物上,是从生理生化层面对叶色的成因以及影响因素进行研究,但关于苞片颜色的研究很少。UROS编码基因在细菌和哺乳动物(主要是人类和小鼠)中研究较为清楚[16],而在植物中研究较少。【拟解决的关键问题】本研究通过分离擎天凤梨叶绿素生物合成和降解途径的关键基因,研究其表达模式,探索呈色苞片中绿色消退的分子机理,为研究观苞类植物的苞片色素变化提供依据。
1 材料与方法
1.1 试验材料
擎天凤梨主流品种‘Ostara’的开花早期植株(图1-A)来源于浙江省萧山棉麻研究所(即浙江省农业科学院花卉研究开发中心)的凤梨种质资源圃。由于苞片是一种来源于叶片的变态器官,因而取用绿色叶片作为试验的基础对照材料。由凤梨观赏部位中心开始计算,由内而外选取第三轮苞片作为试验对象,此轮苞片形状较完整,变色效果最好。根据不同的变色阶段,分别选取绿色阶段、半红阶段以及全红阶段作为检测苞片变色动态的试验材料(图1-B),重复3次。试验于2015年和2016年在浙江省萧山棉麻研究所实验室实施完成。

A:取样部位;B:取样的具体材料;1:绿色苞片;2:半红苞片;3:全红苞片
1.2 叶绿素及类黄酮色素含量的测定
采用SPAD-502叶绿素测定仪(Konica,日本)对擎天凤梨‘Ostara’开花植株的绿色、半红以及全红苞片(图1-B)进行叶绿素含量的测定,同时测量绿色叶片部分以作为对照。每个材料随机取5个点测定(排除边缘、色斑等异常之处),取平均值,然后重复平行取样检测2次,最后进行统计分析。
采用HPLC法,以儿茶酚(catechin)作为标准参照,对类黄酮含量进行测定。使用仪器为waters二元泵1525,自动进样器2707,PAD检测器2998,温控箱1500,检测柱Sun FireTM C18 column(4.6 mm×250 mm,5 μm)。流动相组成:A-0.1%三氟乙酸(水相)和B-乙腈。梯度洗脱程序如下:0—3 min,5%B;3—20 min,5%—25%B;20—42 min,25%—95%B;42—45 min,95%B;45—50 min,95%—5%B;50—60 min,5%B。流速为1.0 mL·min-1,注入样品体积为20 μL。
1.3 叶绿素合成相关酶基因和的分离
以刘建新等[17]构建的擎天凤梨属栽培品种‘Ostara’早期花器官cDNA 全长文库以及随机挑取2 004个单克隆测序信息为基础,经过多序列比对筛选,获得2条与叶绿素合成相关的基因片段,一条是来源于谷氨酰tRNA合成酶(glutamyl-tRNA synthetase)编码基因的cDNA序列片段(单克隆ppfca0_001562.z1.scf);另一条是来源于尿卟啉原-Ⅲ合酶(uroporphyrinogen-Ⅲ synthase)编码基因的cDNA序列片段(单克隆ppfca0_002194.z1.scf),并进行重测序验证。
1.4 叶绿解降解基因的分离
参考王伟等[15]在黄瓜中克隆的过程设计引物:PPH-F:5′-CATCGAACAGGTCATTGGTG-3′和PPH-R:5′-ACTTTCGGGGTCGCTTATTT-3′。以红色苞片为材料提取RNA,并反转录成cDNA作为模板。按2×Taq PCR MasterMix(Tiangen,KT201)方法进行PCR扩增。PCR程序为95℃ 3 min;95℃ 30 s,48℃ 30 s,72℃ 30 s,35个循环;72℃ 15 min。产物进行切胶回收,再进行TA克隆测序。
1.5 开花期叶绿素合成、降解代谢相关基因的表达分析
以分离得到的叶绿素合成关键基因和及降解关键基因的表达变化来代表叶绿素合成和降解的代谢变化。以绿叶作为对照,采用实时荧光定量PCR检测目标基因在不同变色阶段中(绿色、半红和全红)的变化趋势,以确定相关代谢途径的变化情况以看家基因为内参[18],设计引物(表1)。

表1 实时定量PCR反应中各基因的引物序列
对各组织材料提取总RNA。并检测其浓度、纯度及完整性。使用TOYOBO反转录试剂盒合成cDNA。以cDNA为模板,进行扩增,扩增产物按梯度稀释作为模板再进行PCR扩增,制作、、及的标准曲线。然后进行实时荧光定量PCR检测(表2)。

表2 定量PCR循环程序
1.6 数据分析
相关数据以Excel 2007软件分析并生成图片。基因表达分析以作为内参进行校正并获得目标基因的相对表达量。
2 结果
2.1 不同变色时期苞片叶绿素及类黄酮色素含量的比较
采用SPAD-502叶绿素测定仪对绿色、半红、全红苞片以及绿色叶片的叶绿素含量进行测定。结果如图2-A所示,在苞片变色过程中,绿色苞片的叶绿素含量最高,其次是半红苞片,最后是红色苞片。而作为对照的绿叶,其叶绿素含量比绿色苞片更高。以儿茶酚(catechin)作为对照,采用HPLC法检测类黄酮含量(图2-B),红色苞片中类黄酮最高,其次是半红苞片,再者是绿色苞片,而绿叶中类黄酮含量最少。

图2 不同变色阶段中的叶绿素及类黄酮含量变化
2.2 叶绿素合成关键基因和的分离
根据获得的2 004个单克隆测序数据进行多序列比对筛选,有一条cDNA序列片段(单克隆ppfca0_001562.z1.scf)来源于谷氨酰tRNA合成酶编码基因。经重测序确定该序列全长1 140 bp,可编码171个氨基酸序列,GenBank登录号为KP144289。将获得的cDNA序列进行核苷酸同源性(BLAST N)比对,发现与玉米()、蓖麻()、可可树()、拟南芥()等植物有较高同源性。其中与玉米的|XM_008660333.1|和|NM_001153978.1|同源性最高,为82%。将编码的171个蛋白质氨基酸序列进行氨基酸序列同源性(BLASTP)比对,结果表明,与甘蔗()、玉米()、蓖麻()、可可树()、粗山羊草()、拟南芥()的谷氨酰tRNA合成酶的蛋白氨基酸有较高的同源性,其中与甘蔗的|AGT17076.1|同源性最高,为86%,其次为玉米的XP_008658555.1、AFW89634.1和NP_001147450.1,同源性为83%。同时该蛋白氨基酸序列存在GlnRS-cataytic core和Nt-trans superfamily保守区[19-22](图3)。GlnRS-core是Glutaminyl-tRNA synthetase(GlnRS)的催化核心区,吸附Gln到合适的tRNA。Nt-trans superfamily是核苷酸转移酶超家族,有一个保守的双核苷酸结合域,该超家族包括Ⅰ类氨基酰-tRNA合成酶、泛酸合成酶、ATP硫酸化酶和胞苷酰转移酶。

图3 GTS推定氨基酸的保守区
另一条cDNA序列片段(单克隆ppfca0_002194. z1.scf)是来源于尿卟啉原-Ⅲ合酶的编码基因。经重测序确定该序列长613 bp,可编码188个氨基酸序列,GenBank登录号为KP144288。将获得的cDNA序列进行核苷酸同源性比对,发现与海枣()、油棕()、大麦()、小麦()、二穗短柄草()、中国莲()、小米()、短花药野生稻()、玉米()等有较高的同源性。其中与海枣的XM_008796935.1、XM_ 008796934.1、XM_008796933.1和XM_008796932.1一致性最高,为76%。将编码的188个氨基酸序列与蛋白质数据库进行氨基酸序列同源性比对,结果表明,与海枣的XP_008795157.1、XP_008795156.1、XP_008795155.1和XP_008795154.1的一致性最高,为68%。同时该蛋白氨基酸序列存在HemD和HemD Superfamily保守区[19-22](图4),HemD结构域是所有已知UROS特有的一个功能结构域,与UROS的催化活性相关。
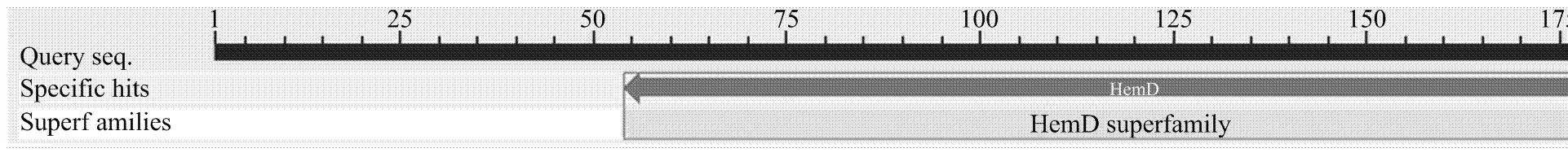
图4 UROS推定氨基酸的保守区
2.3 叶绿解降解基因的分离
以来源于红色苞片的合成cDNA作为模板,以PPH-F和PPH-R为引物进行PCR扩增(图5),发现250、300、450和650 bp 4条带,对预期的250 bp片段进行回收,测序,获得266 bp的cDNA序列(GenBank登录号为KP723523),该序列编码88个氨基酸序列。通过BLASTN比对,与油棕()、海枣()、小果野蕉()、葡萄()、荷花高的()、二穗短柄草()、大麦()等有较高同源性。其中,与油棕的XM_010923945.1一致性最高,为83%。将编码的88AA氨基酸序列进行BLASTP比对,与黄瓜()、油棕()、海枣()、梅花()、二穗短柄草()、甜瓜()等的脱镁叶绿素酶的氨基酸序列有较高同源性。其中与油棕的XP_010931560.1同源性最高,为89%;其次为黄瓜的|BAH84863.1|,为85%。此外,该蛋白氨基酸序列存在Abhydrolase-6保守区[19-22](图6)。
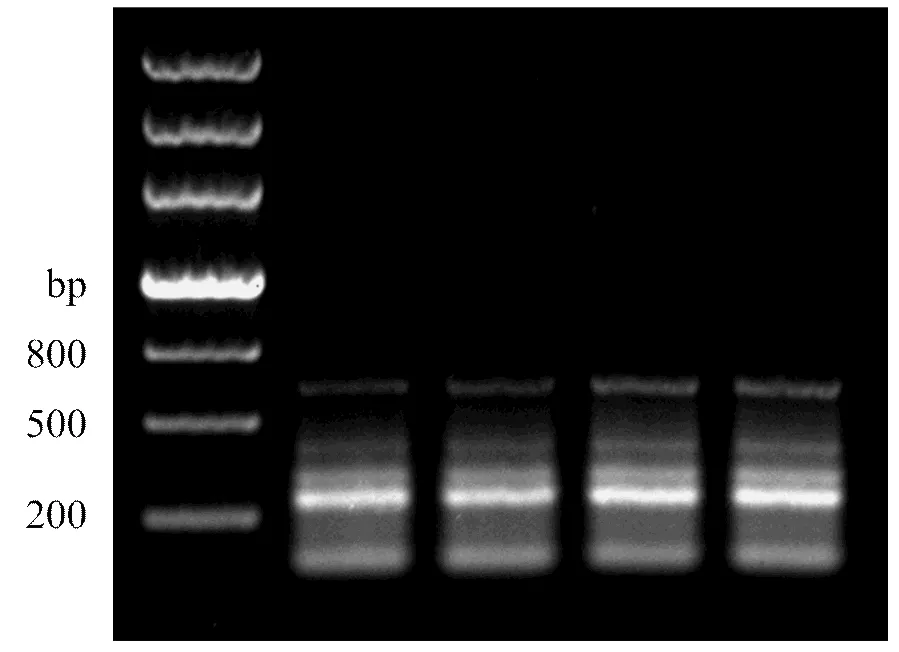
图5 PPH的PCR扩增电泳图

图6 PPH推定氨基酸的保守区
2.4 苞片变色过程中叶绿素合成关键基因、和降解关键基因的表达分析
通过对和在苞片变色3个阶段(绿色、半红及全红)及绿叶中的相对表达量进行检测分析(图7和图8)。叶绿素合成关键酶基因和的表达量在苞片的不同变色阶段都表现为绿色苞片>半红苞片>全红苞片。而作为对照的绿色叶片,和的表达量超过所有的苞片组织。叶绿素降解关键酶基因,在半红苞片部分含量最高,其次是绿苞,再者为全红苞片,而绿叶和红色苞片中的表达量大致相近,处于最低水平。
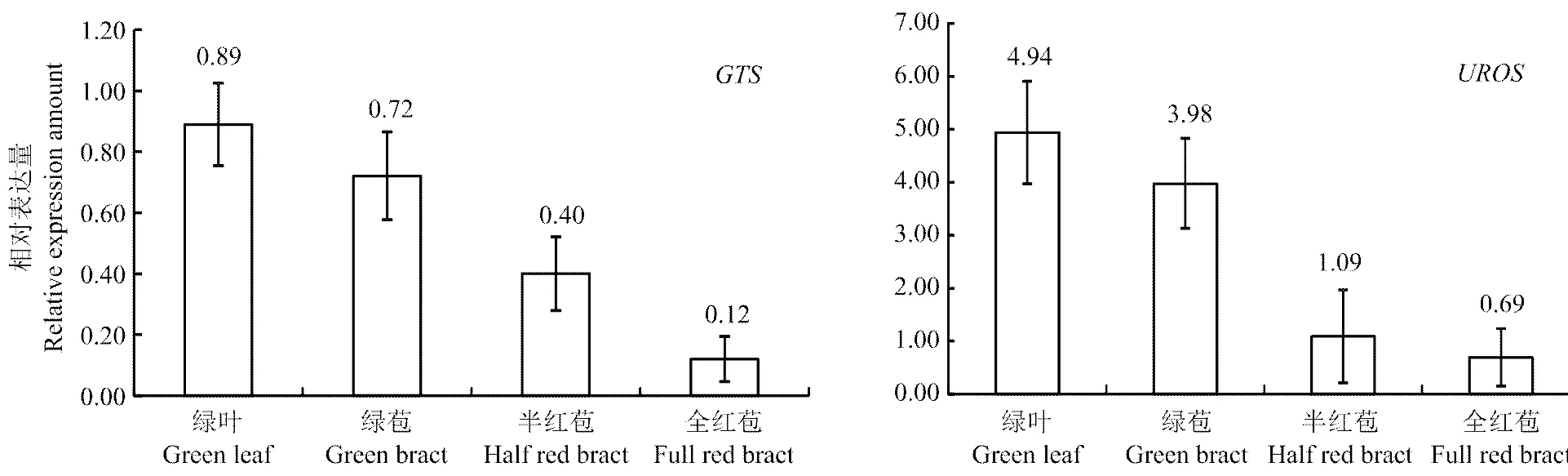
图7 叶绿素合成关键酶基因GTS和UROS的表达分析

图8 叶绿素降解关键酶基因PPH的表达分析
3 讨论
高等植物的绿色来源于叶绿素,其生物合成降解途径如图9[13,28-30]所示。GTS和UROS均是叶绿素合成途径中的关键酶。BLAST比对可知,获得的擎天凤梨与其他植物存在同源性,尤其是单子叶植物,其中与海枣同源性最高,核苷酸及氨基酸同源性分别为76%和68%。与动物及微生物之间无同源性,但都存在共同的Hem D结构域。Hem D结构域是所有已知的特有的一个功能结构域,与的催化活性相关。这与曹凤[16]的研究结论“该基因种间差异较大,保守性相对比较低”基本一致。脱镁叶绿素酶(PPH)是最新发现的叶绿素降解代谢关键酶[9-11],可以专一地裂解脱镁叶绿素a的植醇,促使脱镁叶绿素a脱去植醇而变成脱镁叶绿酸a[23-25]。同源基因普遍存在于高等植物基因组中,但有关该基因的研究非常之少,目前,国内仅在黄瓜[17]及小麦[26]中有相关报道,本研究获得的片段存在Abhydrolase-6保守区(pfam12697),它是一个Alpha/beta水解酶折叠,这与前人的研究类似,所有的PPH蛋白质都存在这一特征[17],具有高度的保守性。此外前人在花椰菜表达中的研究表明,乙烯处理能促进表达,加快植物的衰老[27]。生产上擎天凤梨的催花常常需要采用乙烯来处理植株,处理过的植株然后进入开花阶段,其本质也是步入成熟衰老阶段。本研究也表明,擎天凤梨的开花阶段即伴随着苞片绿色的消退和红色的形成,的表达量也越来越大,推动植株向成熟方向前进。

A:叶绿素合成示意图[28];B:叶绿素降解示意图[28-30]
在苞片变色过程中,绿色苞片中的叶绿素含量最高,其次是半红苞片,全红苞片的叶绿素含量最少,几乎消失不见。而类黄酮色素的含量刚好与之相反,表明苞片变色过程中绿色的消退伴随着叶绿素含量的降低以及类黄酮色素含量的增加。根据前人在各类彩叶植物上获得的结论:叶色的呈色程度常常是由花青素苷/叶绿素的比值决定的[3-5],通过将本研究获得的类黄酮色素与叶绿素的相对比值进行计算,绿苞为4.53;半红苞为18.53;全红苞为235.79。绿叶的类黄酮色素与叶绿素的相对比值为1.29,与绿苞较为接近。随着比值越来越大,绿色逐渐退去,红色却越来越浓,叶绿素和类黄酮色素之间呈现了此消彼长的现象,这与前人在彩叶植物中获得的结论相一致。类黄酮色素含量的增加起到了掩盖叶绿素的作用,但叶绿素自身含量的降低应是苞片绿色退去的根本原因。
为明确叶绿素含量降低的原因,文中对叶绿素的合成及降解相关基因进行了分析,叶绿素合成途径中的和都表现出了相同的趋势,即绿苞中含量最高,半红苞片次之,红色苞片最少。而叶绿素降解的关键基因在绿色苞片和半红苞片中的含量很高,其中在半红苞片是最高的,最低的是红色苞片,而绿叶和红色苞片含量大致相近。绿色苞片和半红苞片中表达量显著增加说明苞片在呈色过程中,对于叶绿素的降解起了非常重要的作用。红色苞片中含量很低,说明此时叶绿素的降解已基本完成,无需大量表达。绿叶中含量很低,而此时叶中的叶绿素含量最高,说明此时叶片需要大量的叶绿素进行光合作用来维持植物的生长,如果大量表达则会通过降解叶绿素而对植物的正常生长会产生破坏。因此,从绿色苞片再到全红苞片的褪绿变红过程中,叶绿素的含量逐渐降低的原因可能是伴随着叶绿素的合成水平的逐渐降低,叶绿素的降解反应显著加强引起的,在此过程中起关键性作用。而在已经变色的红色苞片中,叶绿素的降解反应达到接近绿叶状态的最低水平。
4 结论
获得了擎天凤梨的叶绿素合成相关酶基因和以及降解关键酶基因的cDNA序列。苞片变色过程中绿色的消退的关键在于叶绿素含量的降低,并同时伴随类黄酮含量的增加;而叶绿素含量的降低是由于叶绿素合成量减少,叶绿素的降解量显著增加引起的。在叶绿素的降解过程中起关键性作用。
References:
[1] 沈晓岚, 王炜勇, 毛碧增, 俞少华, 田丹青, 俞信英. 基因枪介导擎天凤梨遗传转化体系的建立. 分子植物育种, 2013, 11(1): 77-84.
Shen X L, Wang W Y, Mao B Z, Yu S H, Tian D Q, Yu X Y. Establishment of particle bombardment-mediated genetic transformation system of guzmania bromeliad., 2013, 11(1): 77-84. (in Chinese)
[2] Tanaka Y, Sasaki N, Ohmiya A. Plant pigments for coloration: Anthocyanins, betalains and carotenoids., 2008, 54: 733-749.
[3] Tanaka Y, Brugliera F, Chandler S. Recent progress of flower colour modification by biotechnology., 2009,10: 5350-5369.
[4] 赵云鹏, 陈发棣, 郭维明. 观赏植物花色基因工程研究进展. 植物学通报, 2003, 20(1): 51-58.
Zhao Y P, Chen F D, Guo W M. Advances in genetic engineering of flower color of ornamental plants., 2003, 20(1): 51-58. (in Chinese)
[5] 赵昶灵, 郭维明, 陈俊愉. 植物花色形成及其调控机理. 植物学通报, 2005, 22(1): 70-81.
Zhao C L, Guo W M, Chen J Y. Formation and regulation of flower color in higher plants., 2005, 22(1): 70-81. (in Chinese)
[6] Kate M, Joanne L M, Rachel M L, Howard G, Peter H. Chloroplast acclimation in leaves ofin response to high light., 1999, 121(1): 89-96.
[7] 祝钦泷. 彩叶草()叶色形成相关的花色素苷生物合成途径的分子调控研究[D]. 重庆: 西南大学, 2007.
Zhu Q L. Studies on the molecular regulation of the anthocyanin biosynthesis related to leaf color of[D]. Chongqing: Southwest University, 2007. (in Chinese)
[8] 季鹏章, 梁名志, 宋维希, 蒋会兵, 马琳, 王丽, 矣兵. 茶树珍稀品种“紫娟”的叶片色素含量与叶色变化的关系研究. 西南农业学报, 2010, 23(6): 1860-1862.
Ji P Z, Liang M Z, Song W X, Jiang H B, Ma L,Wang L,Yi B. Relationship between changes of pigments content and leaf color changing in ‘Zijuan’ (var.)., 2010, 23(6): 1860-1862. (in Chinese)
[9] 胡敬志, 田旗, 鲁心安. 枫香叶片色素含量变化及其与叶色变化的关系. 西北农林科技大学学报(自然科学版), 2007, 35(10): 219-223.
Hu J Z, Tian Q, Lu X A. Relationship between changes of the pigments content and leaf color changing in Liquidambar formosana., 2007, 35(10): 219-223. (in Chinese)
[10] Nakayama M, Roh M S, Uchida K, Yamaguchi Y, Takano K, Koshioka M. Malvidin 3-rutinoside as the pigment responsible for bract color in., 2000, 64(5): 1093-1095.
[11] 王长泉, 魏小兵. 一品红苞片花色素的分离及初步鉴定. 植物学通报, 2006, 23(4): 356-362.
Wang C Q, Wei X B. The extraction and primary identification of anthocyanidin in euphorbia pulcherrima., 2006, 23(4): 356-362. (in Chinese)
[12] Nancy A. A new chlorophyll degradation pathway., 2009, 21: 700.
[13] Schelbert S, Aubry S, Burla B, Agne B, Kessler F, Krupinska K, Hörtensteiner S. Pheophytin pheophorbide hydrolase (Pheophytinase) is involved in chlorophyll breakdown during leaf senescencein., 2009, 21: 767-785.
[14] Asumi F, Yasuo S, Hirofumi T, Naoki Y. Effects of postharvest ethanol vapor treatment on activities and gene expression of chlorophyll catabolic enzymes in broccoli florets., 2010, 55: 97-102.
[15] 王伟. 黄瓜衰老过程中叶绿素降解相关酶基因cDNA片段的克隆与表达初步研究[D]. 武汉: 华中农业大学, 2011.
Wang W. The preliminary study on cloning and expression of cdna fragments of related enzymes gene of chlorophyll degradation of cucumber during senescence[D]. Wuhan: Huazhong Agricultural University, 2011. (in chinese)
[16] 曹凤. 小麦尿卟啉原III合酶基因克隆、序列分析及其在大肠杆菌中的表达[D]. 杨凌: 西北农林科技大学, 2007.
Cao F. Cloning, sequence analysis and expression of wheat uroporphybilinogen III synthase in[D]. Yangling: Northwest A&F University, 2007. (in Chinese)
[17] 刘建新, 沈福泉, 田丹青, 张智, 葛亚英, 沈晓岚, 俞信英, 王炜勇. 擎天凤梨花器官全长cDNA文库的构建及EST分析. 分子植物育种, 2009, 7(6): 1137-1143.
Liu J X, Shen F Q, Tian D Q, Zhang Z, Ge Y Y, Shen X L, Yu X Y, Wang W Y. Construction of full-length cDNA library from floral organ and EST analysis in guzmania., 2009, 7(6): 1137-1143. (in Chinese)
[18] Liu J X, Ge Y Y, Tian D Q, Zhang Z, Ding H Q, Shen F Q, Wang W Y. Cloning and sequence analysis of actin gene from., 2012, 3(1): 1-5.
[19] Marchler-Bauer A, Derbyshire M K, Gonzales N R, Lu S, Chitsaz F, Geer L Y, Geer R C, He J, Gwadz M, Hurwitz D I, Lanczycki C J, Lu F, Marchler G H, Song J S, Thanki N, Wang Z, Yamashita R A, Zhang D, Zheng C, Bryant S H. CDD: NCBI's conserved domain database., 2015, 43: 222-226.
[20] Marchler-Bauer A, Lu S, Anderson J B, Chitsaz F, Derbyshire M K, DeWeese-Scott C, Fong J H, Geer L Y, Geer R C, Gonzales N R, Gwadz M, Hurwitz D I, Jackson J D, Ke Z, Lanczycki C J, Lu F, Marchler G H, Mullokandov M, Omelchenko M V, Robertson C L, Song J S, Thanki N, Yamashita R A, Zhang D, Zhang N, Zheng C, Bryant S H. CDD: A conserved domain database for the functional annotation of proteins., 2011, 39: 225-229.
[21] Marchler-Bauer A, Anderson J B, Chitsaz F, Derbyshire M K, DeWeese-Scott C, Fong J H, Geer L Y, Geer R C, Gonzales N R, Gwadz M, He S, Hurwitz D I, Jackson J D, Ke Z, Lanczycki C J, Liebert C A, Liu C, Lu F, Lu S, Marchler G H, Mullokandov M, Song J S, Tasneem A, Thanki N, Yamashita R A, Zhang D, Zhang N, Bryant S H. CDD: Specific functional annotation with the Conserved Domain Database., 2009, 37(D): 205-210.
[22] Marchler-Bauer A, Bryant S H. CD-Search: protein domain annotations on the fly., 2004, 32(W): 327-331.
[23] 田风霞, 惠振, 王国坤, 范振宇, 王玮. 植物体内的叶绿素降解与滞绿突变体. 植物生理学通讯, 2010, 46(5): 505-511.
Tian F X, Hui Z, Wang G K, Fan Z Y, Wang W. Chlorophyll degradation and stay-green mutant in plant., 2010, 46(5): 505-511. (in Chinese)
[24] 唐蕾, 毛忠贵. 植物叶绿素降解途径及其分子调控. 植物生理学报, 2011, 47(10): 936-942.
Tang L, Mao Z G. Degradation pathway of plant chlorophyll and its molecular regulation., 2011, 47(10): 936-942. (in Chinese)
[25] Eckardt N A. A new chlorophyll degradation pathway., 2009, 21: 700.
[26] 王绘艳. 小麦叶绿素和脱镁叶绿素酶基因的作用及表达分析[D]. 太谷: 山西农业大学, 2015.
Wang H Y. The Function and gene expression analysis of chlorophyllase and pheophytinase in wheat[D]. Taigu: Shanxi Agricultural University, 2015. (in Chinese)
[27] Agustin M B, Pedro M C, Gustavo A M. Chlorophyllase versus pheophytinase as candidates for chlorophyll dephytilation during senescene of broccoli., 2010, 7: 1-7.
[28] Asam F, Bob I, Peter M. Purification and properties of uroporphyrinogen iii synthase (co-synthase) from an overproducing recombinant strain ofK-12., 1989, 264: 397-402.
[29] Barry C S, McQuinn R P, Chung M Y, Besudent A, Giovannoni J J. Amino acid substitutios in homologs of the STAY- GREEN protein are responsible for the greefles hand chlorophyll retainer mutations of tomato and pepper., 2008, 147(l): 179-187.
[30] Suzuki T, Kunieda T, Murai F, Morioka S, Shioi Y. Mgdechelation activity in radish cotyledons with artificial and native substrates, Mg-chlorophyllin a and chlorophyllidea., 2005,43: 459-464.
(责任编辑 李莉)
Isolation of Chlorophyll Metabolism Key Genes and Molecular Mechanism of Green Fade in Guzmania Bracts Discoloration Process
LIU Jian-xin, DING Hua-qiao, TIAN Dan-qing, WANG Wei-yong, LIU Hui-chun
(Xiaoshan Cotton and Bast Fiber Crops Research Institute of Zhejiang Province, Hangzhou 311202)
【Objective】Guzmania is a trendy flower. Its flower formation and bracts discoloration process are often accompanied by green fade. To analyze the reason which bract’s green fade, it is necessary to isolate key genes and explore molecular mechanism of chlorophyll biosynthesis and degradation metabolism in the process of bracts discoloration. 【Method】By cDNA library construction and EST sequencing batch, two key enzyme genes in chlorophyll biosynthesis pathway, including glutamyl-tRNA synthetase () and Uroporphyrinogen-Ⅲ synthase (), were obtained. By homology cloning technology, a key enzyme gene in chlorophyll degradation pathway, pheophytin pheophorbide hydrolase gene (), was obtained. Through measuring chlorophyll content using light transmittance measurement method,flavonoid content using HPLC method and analyzing gene expression pattern of key genes in chlorophyll metabolism using real-time quantitative PCR method, the molecular mechanism which green fade in discoloration bracts was studied. 【Result】obtained has 1 140 bp (GenBank: KP144289) in cDNA sequence, which compiles a 171 amino acid protein sequence, and has GlnRS-cataytic core and Nt-trans superfamily conservative regions;obtained has 613 bp (GenBank: KP144288) in cDNA sequence, which compiles a 188 amino acid protein sequence, and has HemD and HemD Superfamily conservative regions;obtained has 266 bp (GenBank: KP723523) in cDNA sequence, which compiles a 88 amino acid protein sequence, and has Abhydrolase-6 conservative region. Through measuring chlorophyll,flavonoid content and studying the expression level of key genes in chlorophyll metabolism, it could know that bract discoloration process accompanied by significant reduction of chlorophyll content and significant increase of flavonoid content. Furthermore, the expression level of chlorophyll biosynthesis key enzyme’s encoding genes,and, were also decreased obviously. As for the chlorophyll degradation key enzyme’s encoding gene,, its expression level increased significantly at the beginning of bracts discoloration, then reduced to close to the lowest level, which in accord with green leaves, after finishing of discoloration. As for the control material, green leaf had the highest chlorophyll content and the lowest flavonoid content. Furthermore, the expression amount of chlorophyll biosynthesis related genes were the highest, while that of degrade related genes was the lowest. This was consistent with colorful plants in chlorophyll and flavonoid pigment change trend. 【Conclusion】We obtained biosynthesis key enzymes genes,,, and degradation key enzyme gene,, in chlorophyll metabolism pathway from Guzmania. The reason that bracts’ green fade was due to reduction of chlorophyll content, and accompanied by increase of flavonoid content. The reduction of chlorophyll content was due to chlorophyll’s biosynthesis reduced and degradation increased significantly.played a key role in chlorophyll degradation process. The results will provide a basis for researching pigment change of view bract plants.
; chlorophyll; glutamyl-tRNA synthetase; uroporphyrinogen-III synthase; pheophytin pheophorbide hydrolase/pheophytinase
2015-11-16;接受日期:2016-04-21
浙江省自然科学基金(LQ13C150002)、浙江省公益性技术应用研究计划(2012C22085)
刘建新,Tel:0571-83713882;E-mail:liujianxin2000@aliyun.com。通信作者田丹青,E-mail:tdq0123@163.com

