7种芸香科植物的RAPD分析
桂 茜, 蒋 昊, 阮 颖 (湖南农业大学,湖南长沙 410128)
7种芸香科植物的RAPD分析
桂 茜, 蒋 昊, 阮 颖*(湖南农业大学,湖南长沙 410128)
[目的]构建芸香科植物7个种12个材料的聚类分析树状图,为芸香科植物系统学的研究提供分子水平上的证据。[方法]以芸香科植物7个种12个材料为研究对象,利用RAPD技术对其亲缘关系进行分析。[结果]在建立适合芸香科植物PCR-RAPD反应体系的基础上,从21条随机引物(10 mer)中筛选出10个,对所有供试材料进行扩增,共获得10张DNA指纹图谱,85条DNA谱带,其中84条为多态带,多态性谱带比例为98.82%。[结论]建立了基于RAPD的芸香科植物亲缘关系树状图,该结果与经典的芸香科植物的起源、分类基本一致。
芸香科;RAPD;亲缘关系分析
芸香科是双子叶植物中一个较大的科,全世界共有7个亚科,约150属1 700种,分布于全球热带和亚热带地区,少数分布在温带。其中,我国野生及引种栽培的共有4个亚科29属151种28变种,分布于全国各地,主产于西南和南部[1-2]。由于芸香科植物的异花授粉特性,以及长期的引种选择,其变异较大,中间类型多,种的界限不清楚[3-4]。加之以往对芸香科植物亲缘关系的确定主要根据形态标记、生殖特性及染色体核型,结果不尽一致,这给芸香科植物种质资源的筛选、鉴定和利用带来了困难[5-7]。笔者以芸香科植物7个种12个材料为研究对象,采用RAPD (Random Amplified Polymorphic DNA)技术对其亲缘关系进行分析,旨在构建12个材料的聚类分析树状图,为芸香科植物系统学研究提供分子水平上的证据,同时也为芸香科植物种质资源保护和品种鉴定提供理论依据。
1 材料与方法
1.1 试验材料供试材料为芸香科植物中的7个种12个材料,分别为蜜柚(Honeypomelo)(A)、甜柚[Citrusmaxima(Burm) Merr.](B)、土柚[Citrusmaxima(Burm) Merr.](C)、年橘(Citrusreticulatacv. NianJu)(D)、纽荷尔脐橙(Newhall navel orange)(E)、矮果香橼(Citrusmedica)(F)、黔阳无核椪柑(CitrusreticulataBlanco cv.Ponkan)(G)、柠檬(Citruslimon(L.) Burm.f.)(H)、大分四号柑橘(CitrusreticulataBlanco)(I)、琯溪蜜柚(Citrusmaxima(Burm.) Merr.cv.Guanxiyou)(J)、枳(Poncirustrifoliata(L.) Raf)(K)、江永香柚(Citrusmaxima(Burm.) Merr.cv.Jiangyong Yu)(L)。其中,A~D取自湖南永州,E~L取自湖南农业大学柑橘中心。随机引物购自上海生工生物工程有限公司,Mix购自近岸蛋白质科技有限公司。
1.2 试验方法基因组DNA的提取和质量检测参照文献[8-9]的方法,并根据芸香科植物的特征进行修改。用氯仿∶异戊醇(24∶1)抽提2次,以提高DNA的浓度。DNA的质量和浓度检测在Themo核酸蛋白质分析仪上进行。根据所测得的DNA浓度,统一配制成100 ng/μL的模板贮液。取样品DNA在2.0%的琼脂糖凝胶上(溴化乙锭浓度为0.5 μg/mL)电泳,用Gel DOC-IT凝胶分析系统观察并照相,以100 bp plus Ⅱ为Marker,推算DNA分子的大小。
以E、K 基因组DNA为模板,利用RAPD随机引物进行PCR扩增。从21条随机引物(10 mer)中筛选出3次PCR重复能出现稳定条带的引物。利用筛选出的引物对12份芸香科植物基因组DNA进行PCR扩增。琼脂糖凝胶电泳,观察并照相。
PCR反应程序:94 ℃预变性4 min;94 ℃变性40 s,35 ℃ 退火40 s,72 ℃延伸100 s,39个循环;72 ℃延伸7 min,16 ℃保存。
根据凝胶电泳图显示的结果,在同一位置上有扩增条带的记为“1”,无扩增条带的记为“0”,所有样品未共有的条带记为多态性位点。将所有引物的扩增结果进行记录,编辑成“0”和“1”数据阵。利用UPGMA软件分析,采用聚类分析的方法构建聚类图。
2 结果与分析
2.1 引物筛选结果从21条随机引物(10 mer)1~21中筛选到3次PCR重复能出现稳定条带的为引物,有多态性扩增条带的引物为2、4、5、7、10、12、16、19、20、21,共10条(图1)。
2.2 基因组DNA的扩增结果对12个样品扩增产物电泳结果进行统计分析,结果见表1。由表1可知,10条随机引物共扩增出85条DNA谱带,其中,多态性DNA 片段(多态带)有84条,占扩增总带数的98.82%。每个引物扩增的带数在4~12条,平均为9条;其中扩增带数最多的为引物10,具12条扩增带;最少的为引物7,仅具4条带。扩增出的DNA 片段大小在150~2 000 bp。在扩增出的85条谱带中, 有1条为全部供试材料所共有(共有带),这表明各材料间的同源性。不同引物扩增出的带数不同;同一引物,不同供试材料间的扩增带数也不同;这表明芸香科植物遗传背景的复杂性和DNA 的多态性。引物5 和10 的扩增结果见图2。

图1 以E、K基因组DNA为模板对引物1~21的筛选结果
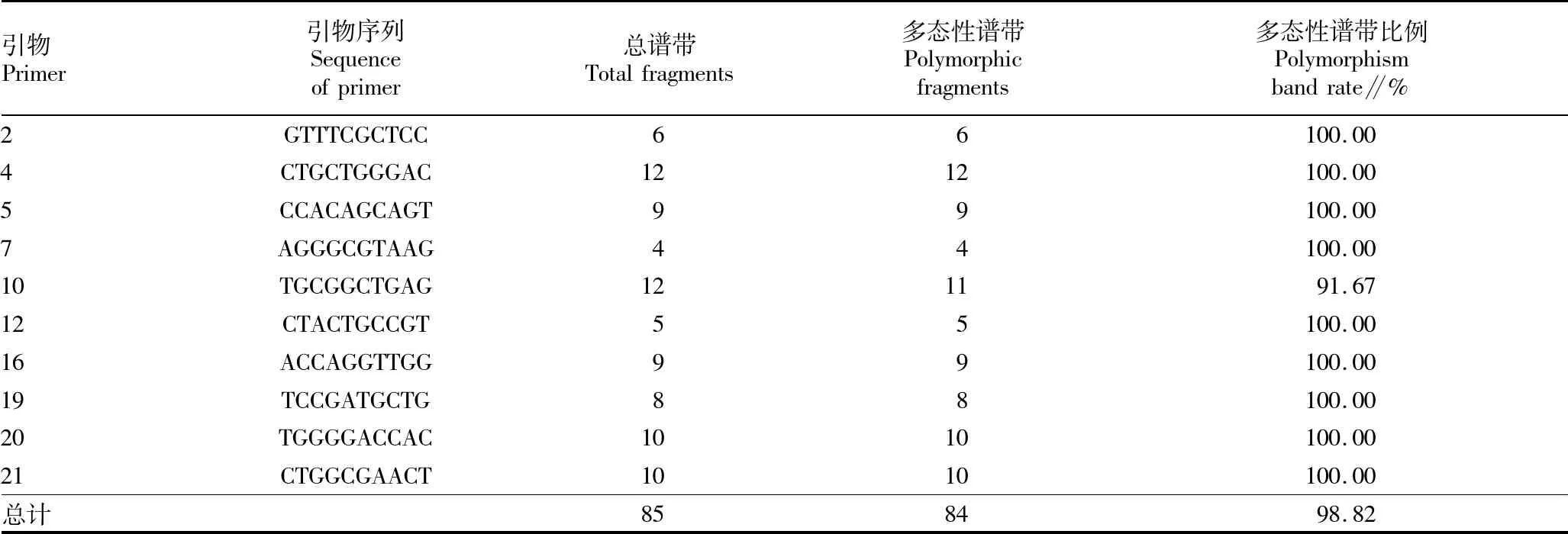
表1 10条随机引物对12个供试材料的扩增结果
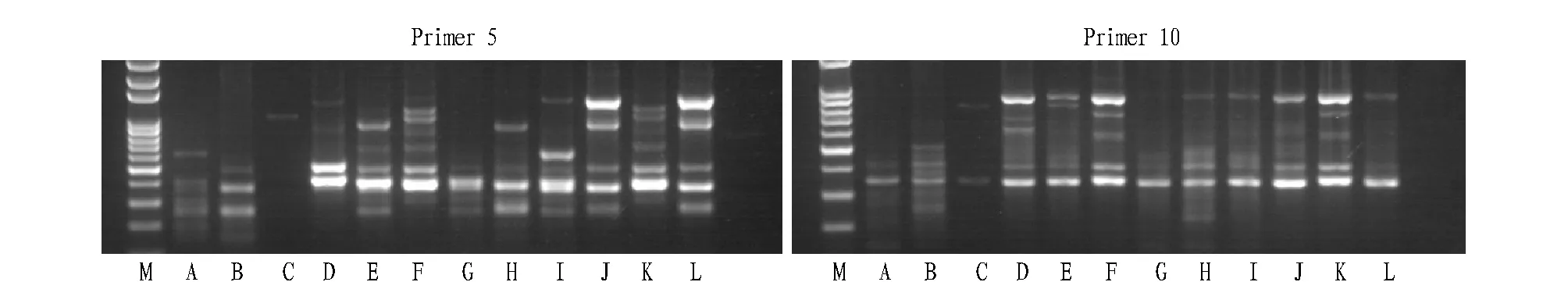
图2 引物5和10对12份芸香科材料的扩增结果

图3 芸香科植物聚类分析
2.3 聚类分析基于RAPD的扩增结果,用UPGAM法进行聚类分析,结果见图3[10]。由图3可知,当遗传距离在0.301 5~0.441 0时,12个样品可分成3类:第1类为蜜柚(A)、甜柚(B)、黔阳无核椪柑(G)和柠檬(H);第2类为纽荷尔脐橙(E)、大分四号柑橘(I)、琯溪蜜柚(J)、江永香柚(L)、年橘(D)、矮果香橼(F)和枳(K);第3类为土柚(C)。从这3个类群看,不同种的芸香科植物亲缘关系错综复杂,但总体来看,与传统的芸香科植物起源完全吻合。在传统的芸香科植物起源中,葡萄柚、椪柑和柠檬由橙分别与柚、桔、青柠杂交而来,聚为第1类;橙由橘和柚杂交而来,聚为第2类。当遗传距离在0.133 7~0.159 0时,芸香科植物可分为9类,第1类为蜜柚(A),第2类为甜柚(B),第3类为黔阳无核椪柑(G),第4类为柠檬(H),第5类为纽荷尔脐橙(E)、大分四号柑橘(I),第6类为琯溪蜜柚(J)、江永香柚(L),第7类为年橘(D),第8类为矮果香橼(F)和枳(K),第9类为土柚(C)。纽荷尔脐橙、大分四号柑橘与琯溪蜜柚、江永香柚在遗传距离较小时不能区分,说明其亲缘关系较近,而与其他芸香科植物亲缘关系较远。其中,第8类矮果香缘与我国特有的枳亲缘关系更近,遗传距离在0.061 5时,才能将其区分开。
3 讨论
从基于RAPD标记建立的树状图看,所有供试材料的遗传距离都大于0,又都能够聚类在一起,表明芸香科植物一方面具有相同遗传背景,另一方面相互之间又存在一定差异。当遗传距离在0.133 7~0.159 0时,供试芸香科植物可明显地分为Ⅰ(A)、Ⅱ(B)、Ⅲ(G)、Ⅳ(H)、Ⅴ(E、I)、Ⅵ(J、L)、Ⅶ(D)、Ⅷ(F、K)、Ⅸ(C)。其中,V为橙类,Ⅵ为柚类。从聚类树状图可以看出,我国特有的枳与矮果香橼遗传距离非常小,甚至比2种柚(J.琯溪蜜柚、L.江永香柚)之间的遗传距离还小,表明枳与矮果香橼的亲缘关系非常近。
[1] 罗焜,陈士林,陈科力,等.基于芸香科的植物通用DNA条形码研究[J].中国科学:生命科学,2010(4):342-351.
[2] 王贱荣,董美玲,郭跃伟.芸香科柠檬苦素类化合物及其生物活性研究进展[J].国际药学研究杂志,2009,36(5):321-331,365.
[3] GREGORY T R.DNA barcoding does not compete with taxonomy[J].Nature, 2005, 434(7037): 1067.
[4] MILLER S E.DNA barcoding and the renaissance of taxonomy[J].Proc Natl Acad Sci USA, 2007, 104(12): 4775-4776.
[5] 刘文哲,胡正海.中国芸香科植物叶分泌囊比较解剖学研究[J].植物分类学报,1998,36(2):119-127.
[6] 邵邻相,范晓萍.几种芸香科植物花粉形态观察[J].果树学报,2003,20(2):146-148.
[7] WILLIAMS J G K,KUBELIK A R,LIVAK K J,et al.DNA polymorphisms amplified by arbitrary primers are useful as genetic markers[J].Nucleic Acid Res,1990,18(22):6531-6535.
[8] 刘春林,阮颖,官春云,等.油菜 RAPD 反应体系的优化研究[J].湖南农业大学学报(自然科学版),2001,27(6):432-433.
[9] 阮颖,周朴华,刘春林.九种李属植物的 RAPD 亲缘关系分析[J].园艺学报,2002,29(3):218-223.
[10] 虢婷婷.蓖麻遗传多样性、脂肪酸组分分析及种子发育过程观察[D].长沙:湖南农业大学,2012.
RAPD Analysis of Seven Species of Rutaceae Plants
GUI Xi, JIANG Hao, RUAN Ying*(Hunan Agricultural University, Changsha, Hunan 410128)
[Objective] To establish cluster analysis dendrogram of 12 Rutaceae materials in seven species, and to provide evidence for the phylogenetic research on Rutaceae plants at molecular level.[Method] With 12 Rutaceae materials in seven species as the research objects, RAPD technology was used for genetic relationship analysis.[Result]Based on establishing PCR-RAPD reaction system of Rutaceae plants, ten primers were screened from 21 random primers.All the tested materials were used for amplification.A total of ten DNA fingerprint and 85 DNA bands were obtained.Among them, 84 bands were polymorphic, with the polymorphic band ratio being 98.82%.[Conclusion] Genetic relationship dendrogram of Rutaceae plants based on RAPD was established.Cluster results of dendrogram had basically the same origin and classification with classic Rutaceae plants.
Rutaceae; RAPD; Genetic relationship analysis
湖南省教育厅科学研究项目“多梳蛋白抑制植物体细胞胚特性发生的分子机制”(13B057)。
桂茜(1991- ),女,湖南沅江人,硕士研究生,研究方向:植物防御与植物抗性。*通讯作者,教授,博士,博士生导师,从事发育生物学研究。
2016-09-05
S 188
A
0517-6611(2016)30-0101-03

