人工合成六倍体小麦SHW—L1紫色花药的遗传定位研究
耿小红++武艳芍++余马
摘要:以138份重组自交系群体为材料,并利用已构建的高密度遗传图谱对人工合成六倍体小麦SHW-L1的紫色花药目标性状进行遗传定位。结果表明,该目标性状为单基因遗传,且被定位于染色体3D,该基因与R基因、Pal基因及Dfr等基因毗邻或具有等位性。
关键词:人工合成小麦;紫色花药;遗传定位
中图分类号:S326 文献标识码:A 文章编号:0439-8114(2016)22-5748-03
DOI:10.14088/j.cnki.issn0439-8114.2016.22.005
Genetic Mapping of Purple Anthers in Synthetic Hexaploid Wheat of SHW-L1
GENG Xiao-hong1, WU Yan-shao1, YU Ma2
(1.Shanxi Yuncheng Agriculture Vocational and Technical College, Yuncheng 044000,Shanxi,China;
2.College of Life Science and Engineering,Southwest University of Science and Technology,Mianyang 621010,Sichuan,China)
Abstract: A population contained 138 recombinant inbred lines and a high density genetic mapping were used to precisely detect genetic allele controled purple anther. The genetic analysis results indicated that the purple anther was a monogenic trait in SHW-L1, and this single gene was located on chromosome 3D, which might be the same alleles or at a short distance from R, Pal, and Dfr genes on 3D.
Key words: synthetic hexaploid wheat; purple anthers; genetic mapping
花青苷、原花色素及鞣酐等多酚色素皆通过类黄酮生物合成途径所得,是植物色素的主要成员[1]。对小麦、水稻等风传授粉禾谷类作物而言,各组织色素不只是感官性状,其在禾谷类植物中因抗氧化活性而具有很多生物学功能[2]。1883年,达尔文在其著作中记载有色的烟叶、葡萄、洋葱较无色种质资源更具抗病性。红皮小麦在北方气候中较白皮小麦更易于生存。此后,色素累积度(或类黄酮含量及类黄酮结构基因转录水平)与植物在不同胁迫下的抗性水平相关性开始被大量报道[3,4]。类黄酮对植物生长、发育、细胞膜通透性、种子萌发及穗发芽抗性都有显著影响。其中花青苷色素对植物抵御紫外线、干旱、盐胁迫、重金属、辐射、营养缺乏及真菌性病害都起到非常重要的作用[5]。因此研究花青苷色素的遗传机理对植物抗逆性提升意义重大。
普通小麦是由三个二倍体物种经种间杂交所得,其A、B和D基因组供体祖先种分别为乌拉尔圖小麦(Triticum urartu),斯卑尔脱小麦(Aegilops speltoides)及节节麦(A. tauschii)。节节麦分为ssp. tauschii亚种及ssp. strangulate亚种,其中,ssp. strangulate亚种被认为是普通小麦的D基因组供体,而ssp. tauschii亚种并未涉入普通小麦的遗传进化史,且亚种中的该亚种具有优异的农艺性状尚未被引入普通小麦种。本研究以ssp. tauschii亚种为亲本创制的人工合成六倍体小麦SHW-L1为材料。对SHW-L1进行紫色花药颜色的遗传定位分析,以发掘小麦花药中花青素色素沉积的关键位点。
1 材料与方法
1.1 供试材料及田间设计
本研究采用的试验材料为小麦SC重组自交系群体(SHW-L1×川麦32,简称SC群体),该群体为171份重组自交系。群体亲本SHW-L1系人工合成六倍体小麦,其A、B、D基因组供体情况参考文献[6]。川麦32为四川省主要审定推广品种,本研究所有材料皆由四川农业大学小麦研究所提供。
171份重组自交系群体、亲本SHW-L1及川麦32,以及SHW-L1合成亲本AS60和AS2255于2012~2013年种植于四川农业大学雅安及温江校区田间实验农场。田间设计采用单粒播种,种植行长1.5 m,单株间距0.1 m,行间距0.3 m,每株系种植3行,常规肥水管理及病虫害防治。
1.2 表型鉴定
待植株花药从颖壳内伸出,记录花药颜色。表型与SHW-L1一致的株系记为A,与川麦32一致的株系记为B。
1.3 标记-性状连锁分析
方差分析采用SPSS16.0分析软件。SC群体遗传连锁图谱数据及各株系基因型数据来源于参考文献[7]。SC群体遗传图谱全长3 766.9 cM,共1 862个标记位点,平均标记密度为2 cM每个位点。因MapMaker/EXP软件不能处理高通量的高密度图谱数据,故目标性状基因染色体定位采用JoinMap分析软件对图谱标记位点及目标性状进行群体划分,以识别到与目标性状连锁紧密的标记位点连锁群。目标性状在该连锁群的遗传定位采用MapMaker/EXP 3.0软件分析。
2 结果与分析
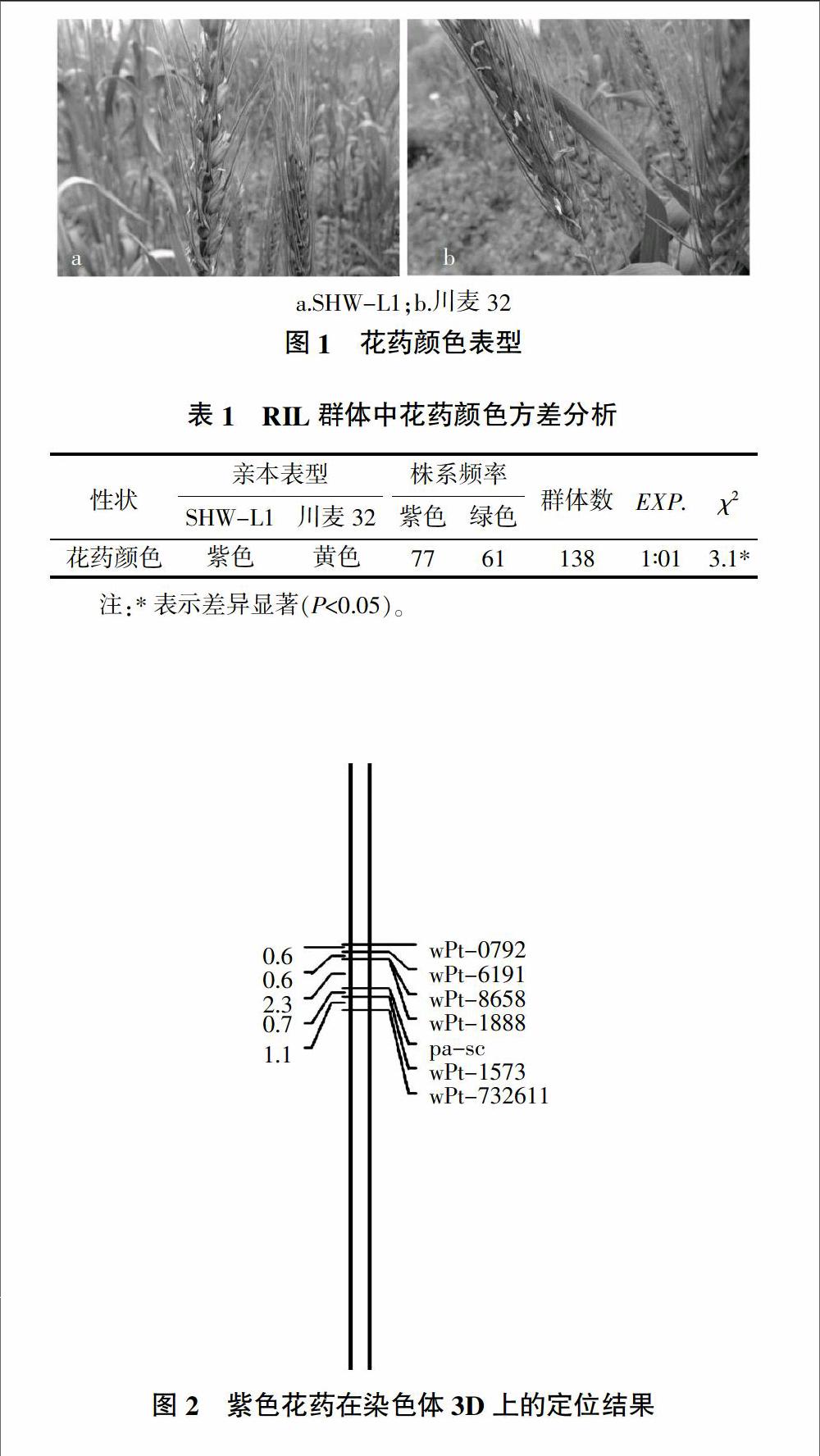
人工合成小麦SHW-L1抽穗后,开花较晚,当年6月初开花,其花药露出颖壳时,花药颜色为紫色(图1a)。川麦32花期较早,当年5月初即开花,花药颜色为黄色(图1b)。SHW-L1的A、B基因组供体AS2255亲本花药颜色为黄色,与川麦32表型一致;D基因组供体亲本AS60花药颜色为紫色,与SHW-L1花药颜色一致。171份重组自交系中,扬花期从4月中旬持续到7月初。其中77个株系为紫色花药,61个株系为黄色花药,剩余33个株系因极端天气原因未记录到花药颜色,因此被记为缺失。所有138份株系、群体亲本SHW-L1、川麦32、AS60及AS2255在雅安和温江都表现出稳定的表型一致性,这表明花药颜色性状的环境独立性。卡方检验结果表明,花药颜色在SC重组自交系群体中1∶1分离显著性不明显,但其卡方值仅为3.1,因此认为这是群体样本较小造成的取样误差,该目标性状为单基因遗传分离模式的概率极大(表1)。
性状-标记关联分析中,花药颜色被定位于3D染色体上(图2)。该染色体包含247个标记位点,染色体总长250.1 cM,平均标记密度1.01 cM每个标记。目标性状相关位点与3D染色体上217.1~220.1 cM区段内6个标记位点连锁最为紧密(遗传距离<10 cM)(图2)。利用这6个位点对SHW-L1及其A、B、D基因组供体亲本进行基因型鉴定,结果表明,wPt-8658、wPt-1888和wPt-1573组成的遗传区段在AS2255(AABB)中存在,但在SHW-L1中缺失。其中wPt-8658和wPt-1888为共分离位点。其余3个位点在SHW-L1的人工合成过程中符合孟德尔遗传规律进行传递。
3 讨论
小麦紫色花药与腥黑穗病抗性显著相关[8]。本研究发现人工合成六倍体小麦SHW-L1中紫色花药颜色受单基因控制,且将该基因位点定位于3D染色体上。目前对小麦紫色花药相关基因研究主要定位于染色体7A(Pan-A1)短臂和7D(Pan-D1)短臂上[9]。而Pan-A1和Pan-D1分别与紫色胚芽鞘相关基因的Rc-A1和Rc-D1毗邻。在3D染色体上有控制子粒颜色性状的R基因报道[10]。R基因与C1基因互作可以上调控制颜色的花青苷色素合成所需的所有结构基因表达[11]。此外,两个控制花青苷色素合成的苯丙氨酸氨裂合酶基因(Pal)及二氢黄酮醇还原酶基因(Dfr)结构基因也被定位于3D染色体上。本研究发掘到的紫色花药相关基因极有可能与3D上花青苷色素合成结构基因或调控基因具有等位性。
普通小麦的D基因组供体物种节节麦分为tauschii和strangulata两个亚种,其中strangulata亚种被认为是普通小麥的供体物种。tauschii亚种未参与普通小麦的遗传进化,较strangulata亚种早抽穗,其穗部性状也与strangulata亚种差异显著[12,13]。本研究中SHW-L1的D基因组供体亲本AS60系tauschii亚种。目前已利用SC群体进行了36个产量及抗性相关性状遗传定位,共发掘到136个遗传位点[14-16],这些遗传位点对小麦抗性即产量提升都具有重要意义。普通小麦的A、B、D基因组高度相似,对大多数基因来说都存在至少三个拷贝的同源基因。较二倍体物种而言,这些同源基因在异源多倍化过程中都经历了序列及表达模式改变。本研究所发掘到的紫色花药相关基因来源于A. tauschii,深入研究该基因在A. tauschii和SHW-L1中的序列及表达差异,对普通小麦的适应性进化研究有重要意义。
参考文献:
[1] WINKEL-SHIRLEY B. Flavonoid biosynthesis:A colourful model for genetics,biochemistry,cell biology,and biotechnology[J].Plant Physiol,2001,126(2):485-493.
[2] KHLESTKINA E K,(TERESHCHENKO) SHOEVA O Y,SALINA E. Flavonoid biosynthesis genes in wheat and wheat-alien hybrids: Studies into gene regulation in plants with complex genomes[A].MOTHERSILL C E,KOROGODINA V,SEYMOUR C B. Radiobiology and Environmental Security[C].Spriger:NATO Science for Peace and Security,2011.31-41.
[3] GIOVANINI M P,PUTHOFF D,NEMACHECK J A,et al. Gene-for-gene defense of wheat against the Hessian fiy lacks a classical oxidative burst[J].Mol Plant Microbe Interact,2006, 19(9):1023-1033.
[4] HOCH W A,SINGSAAS E L,MC COWN B H. Resorption protection. Anthocyanins facilitate nutrient recovery in autumn by shielding leaves from potentially damaging light levels[J].Plant Physiol,2003,133:1296-1305.
[5] PLAZA B M,JIMENEZ S,SEGURA M L,et al. Physiological stress caused by salinity in cordyline fruticosa and its indicators[J].Commun Soil Sci Plant Anal,2009,40:473-484.
[6] ZHANG L Q,LIU D C,YAN Z H,et al. Rapid changes of microsatellite flanking sequence in the allopolyploidization of new synthesized hexaploid wheat[J].Science in China Series C: Life Sciences,2004,47(6):553-561.
[7] YU M,MAO S L,CHEN G Y,et al. QTLs for uppermost internode and spike length in two wheat RIL populations and their affect upon plant height at an individual QTL level[J].Euphytica,2014,200(1):95-108.
[8] BOGDANOVA E D,SARBAEV A T,MAKHMUDOVA K K. Resistance of Common Wheat to Bunt. In: Proceedings of the Research Conference on Genetics[M].Moscow,2002.
[9] KHLESTKINA E K,PSHENICHNIKOVA T A,R?魻DER M S,et al. Clustering anthocyanin pigmentation genes in wheat group 7 chromosomes[J].Cereal Res Commun,2009,37:391-398.
[10] MCINTOSH R A,YAMAZAKI Y,DUBCOVSKY J,et al. Catalogue of gene symbols for wheat[R].2013-09-13.
[11] HIMI E,AHMED N,NODA K. Colour genes(R and Rc) for grain and coleoptile upregulate flavonoid biosynthesis genes in wheat[J].Genome,2005,48(4):747-754.
[12] HUANG L,WANG Q,ZHANG L Q,et al. Haplotype variations of gene Ppd-D1 in Aegilops tauschii and their implications on wheat origin[J].Genetic Resources and Crop Evolution,2011, 59(6):1027-1032.
[13] DUDNIKOV JU A,KAWAHARA T. Aegilops tauschii: Genetic variation in Iran[J].Genetic Resources and Crop Evolution,2006,53(3):579-586.
[14] YU M,CHEN G Y,ZHANG L Q,et al. QTL mapping for important agronomic traits in synthetic hexaploid wheat derived from Aegiliops tauschii ssp. tauschii[J].Journal of Integrative Agriculture,2014,13(9):1835-1844.
[15] YU M,CHEN G Y,PU Z E,et al. Quantitative trait locus mapping for growth duration and its timing components in wheat[J].Molecular Breeding,2015,35(1):1-11.
[16] PU Z E,YU M,HE Q Y,et al. Quantitative trait loci associated with micronutrient concentrations in two recombinant inbred wheat lines[J].Journal of Integrative Agriculture,2014, 13(11):2322-2329.

