利用高通量测序技术分析川中黑山羊瘤胃纤毛虫种群结构
刘 旗 陈 芸 邓俊良 任志华 杨颜铱 高 爽 陈 憧(四川农业大学动物医学院,动物疫病与人类健康四川省重点实验室,环境公害与动物疾病四川省高校重点实验室,成都611130)
利用高通量测序技术分析川中黑山羊瘤胃
纤毛虫种群结构
刘 旗 陈 芸 邓俊良*任志华 杨颜铱 高 爽 陈 憧
(四川农业大学动物医学院,动物疫病与人类健康四川省重点实验室,环境公害与
动物疾病四川省高校重点实验室,成都611130)
本试验旨在应用高通量测序技术研究川中黑山羊瘤胃纤毛虫种群结构。选取3只4月龄川中黑山羊[体重(15.53±0.21) kg],正常饲喂20 d后采集瘤胃液(A)样品,间隔40 d后再次采集瘤胃液(F)样品,提取样品总DNA后,扩增真核生物18S rRNA V4区,扩增产物使用Illumina MiSeq平台测序。结果表明:1)共获得高质量有效序列242 321条,聚类后得1 650个操作分类单位(OTU)。2)A样品、F样品在alpha多样性Chao、ACE、Shannon和Simpson指数上,差异不显著(P>0.05)。3)在纲水平分类上,2个时间点样品相对丰度最高的均为纤毛门,侧口纲(A样品为46.0%、F样品为44.7%),2个样品的相对丰度差异不显著(P>0.05)。4)在科水平分类上,A样品优势科为头毛科(31.8%),其次为均毛科(14.2%);F样品优势科为头毛科(42.8%);并且F样品头毛科相对丰度显著高于A样品(P<0.05),A样品均毛科相对丰度显著高于F样品(P<0.05)。5)在属水平分类上,A样品与F样品相对丰度最高的属均为多加多泡双毛属(A样品为20.9%、F样品为25.4%),无显著差异(P>0.05);存在显著性差异的属是均毛属、头毛属、腹甲双毛属、刺甲双毛属相对丰度,其中A样品均毛属(14.1% vs. 1.9%)、腹甲双毛属相对丰度(2.8% vs. 1.5%)显著高于F样品(P<0.05),而头毛属(6.7% vs. 12.5%)、刺甲双毛属相对丰度(0.3% vs. 2.5%)显著低于F样品(P<0.05)。本试验结果表明,幼龄川中黑山羊瘤胃液纤毛虫相对丰度最高的种属为多加多泡双毛属,瘤胃中还有许多未被分类鉴定且相对丰度较高的真核生物,需要进一步研究。
川中黑山羊;瘤胃纤毛虫;种群结构;多样性;MiSeq测序
反刍动物瘤胃中栖息着大量细菌、原虫、真菌和古菌,其中原虫主要指纤毛虫,数量为1×104~1×106个/mL[1-2]。纤毛虫数量虽然比细菌少,但体积大,占整个瘤胃微生物生物量的30%~80%[3],在瘤胃生态系统中具有重要生物学意义,包括稳定瘤胃内环境(pH、发酵类型、甲烷生成、氨浓度),降解纤维素、蛋白质及防止宿主中毒(硝酸盐、亚硝酸盐、酸)等[4-9]。纤毛虫种群结构受年龄、饲粮结构和地域等因素的影响[1,10-11]。有研究表明,在新生幼畜的瘤胃内没有纤毛虫,随着年龄的增长幼畜瘤胃内纤毛虫种群结构发生着剧烈的变化[12-14]。随着反刍家畜营养与疾病研究的不断深入,反刍家畜瘤胃内纤毛虫种群构成、种群关系变化及其与宿主之间复杂的关系成为瘤胃营养代谢研究的热点。
目前山羊瘤胃微生物的研究对象主要是波尔山羊等,而我国特有小品种羊研究甚少。川中黑山羊原产于四川省金堂县、乐至县一带,具有个体大、生长快、肉质鲜美、繁殖率高、适应性强、耐粗饲等优点,是我国优秀的黑山羊品种,于2010年1月15日,正式列入《中国国家畜禽遗传资源目录》。但目前对川中黑山羊瘤胃微生物的研究尚未见报道。
高通量测序(high-throughput sequencing)技术是近年来发展起来的一种研究微生物生态学的全新技术手段,能全面地反映样品微生物的结构与组成。应用高通量测序技术研究羊瘤胃微生物的报道主要有关于细菌,对瘤胃纤毛虫的研究却鲜有报道。因此,本试验应用高通量测序技术研究川中黑山羊瘤胃原生动物纤毛虫结构,旨在为我国黑山羊种质资源的开发利用奠定基础,也为川中黑山羊微生态营养的深入研究提供参考。
1 材料与方法
1.1 试验动物与饲粮
选取3头健康雄性4月龄川中黑山羊,平均体重为(15.53±0.21) kg,供瘤胃液采集。试验动物单栏饲喂(饲喂精料给足青草)、自由饮水。精料组成及营养水平见表1,饲喂量按照体重2%饲喂,粗料为新鲜青草,每天08:00和17:00饲喂。

表1 精料组成及营养水平(风干基础)
1)预混料为每千克饲粮提供Premix provided the following per kg of the diet:Fe (as ferrous sulfate) 30 mg,Cu (as copper sulfate) 10 mg,Zn (as zinc sulfate) 50 mg,Mn (as manganese sulfate) 60 mg,VA 2 937 IU,VD 343 IU,VE 30 IU。
2)营养水平均为计算值。Nutrient levels were calculated values.
1.2 样品采集与试验设计
预饲10 d后开始正式试验,正常饲喂20 d后,于第21天晨饲前采集瘤胃液(A)样品,间隔40 d后再次采集瘤胃液(F)样品,每个样品3个重复,参照王继文等[15]的方法(胃管真空泵抽吸样品法)采集样品。
1.3 18S rDNA的扩增及高通量测序
瘤胃微生物基因组DNA的提取,参照试剂盒[天根生化科技(北京)有限公司]说明书进行,采用紫外可见分光光度计(NanoDrop-ND1000)测定提取的DNA浓度,并利用1%的琼脂糖凝胶电泳检测DNA提取质量,提取的DNA于-20 ℃保存备用。提取质量合格的DNA样品送至上海派森诺生物科技有限公司扩增真核生物18S rDNA V4区(420 bp),通用引物为V547F:5'-CCAGCASCYGCGGTAATTCC-3';V4R:5'-ACTTTCGTTCTTGATYRA-3'。PCR产物经2%琼脂糖凝胶电泳鉴定,并用Axygen凝胶回收试剂盒回收。对文库质检和定量,将合格的文库利用MiSeq Reagent Kit V3(600cycles)进行双端测序。
1.4 数据分析
测序原始数据以Fastq格式保存,利用Flash 1.2.7软件筛选(按照引物和Index信息,识别分配入对应样本,并去除嵌合体等疑问序列)获取每个样本的有效序列,再运用QIIME 1.8.0识别疑问序列(要求序列长度≥150 bp,且不允许存在模糊碱基N之外,剔除5'端引物错配碱基数>1的序列以及含有连续相同碱基数>8的序列),筛选得到高质量序列,调用Uclust这一序列比对工具[16],对前述获得的高质量序列按97%的序列相似度进行归并和可操作分类单元(operational taxonomic unit,OTU)划分[17],并选取每个OTU中丰度最高的序列作为该OTU的代表序列,根据每个OTU在每个样本中所包含的序列数,构建OTU在各样本中丰度的矩阵文件。获得的OTU与NCBI数据库比对,通过GenBank鉴定OTU代表性序列的微生物分类地位。alpha多样性指数(Chao、ACE、Shannon、Simpson指数)的计算利用Mothur 1.30.1软件完成[18]。2个时间点样品菌群相对丰度差异显著性分析采用SPSS 18.0软件进行配对t检验,结果以平均值±标准差表示。
2 结 果
2.1 样品测序深度和多样性分析
本次试验6个样品共获得242 321条高质量序列,每个样品含有(40 386±4 082)条序列。基于相似度大于97%的原则,将获得的高质量序列进行OTU聚类,共获得1 650个OTU,剔除稀有OTU后,其中A样品获得175个,F样品获得197个,样品间差异不显著(P>0.05),2个样品间共享OTU为143个(图1)。本试验瘤胃液样品的稀释曲线见图2,如图所示,在本试验的测序深度下,除了A1样品外,其余各样品稀释曲线最终均趋于平缓,表明本试验的测序深度可以覆盖各样品中大多数微生物。
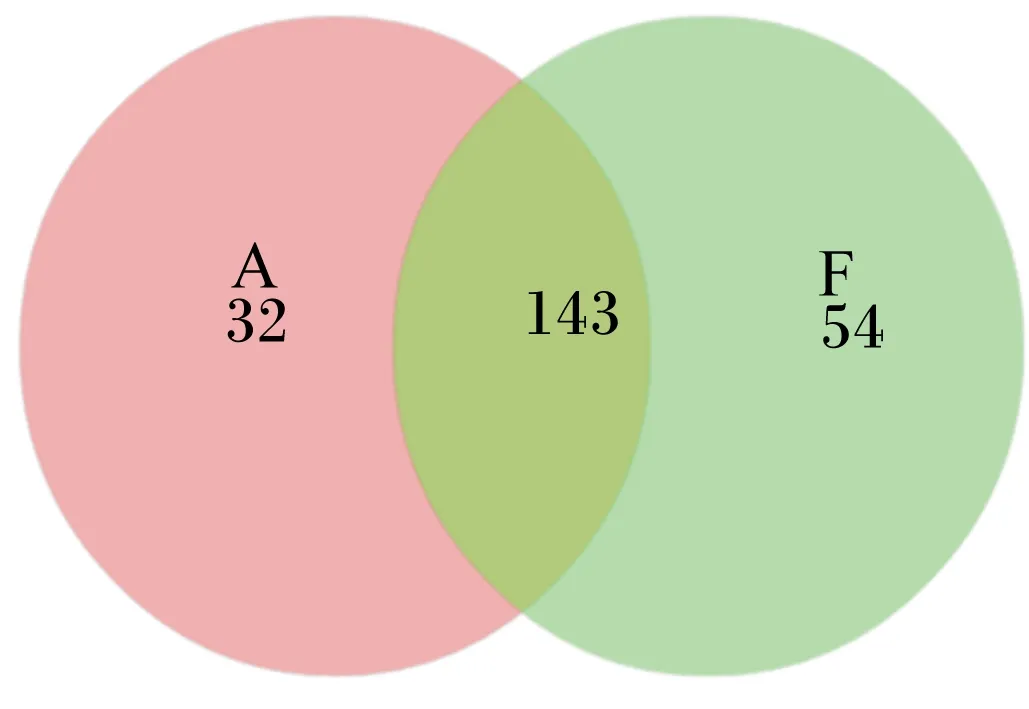
图1 OTU维恩图
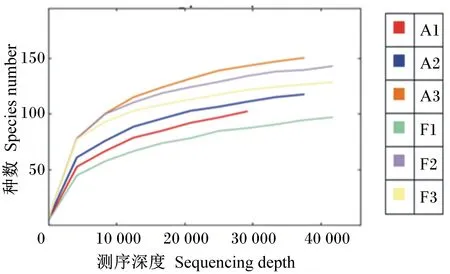
图2 样品稀释曲线
主成分分析结果见图3,如图所示,2个样品聚类在一起。主成分分析获得主成分1(PC1)的贡献率为70.84%,主成分2(PC2)的贡献率为23.80%。
alpha多样性常用于反映微生物群落的丰富度和均匀度,常用的度量指数主要包括侧重于体现群落丰富度Chao和ACE指数,以及兼顾群落均匀度的Shannon和Simpson指数。2个时间点样品的alpha多样性结果见表2,与A样品相比,F样品群落丰富度指数(Chao和ACE指数)稍低,但差异不显著(P>0.05),而群落均匀度及多样性指数(Shannon和Simpson指数)稍高,差异亦不显著(P>0.05)。

图3 主成分分析结果
2.2 瘤胃原生动物纤毛虫种群结构
表3中列出了纤毛虫在纲、科、属上的种群分布,各分类水平中不属于纤毛虫的均未列出,包括新丽鞭毛菌门、链形门和微孢子门等,双毛属(Diplodinium)含量过低而未列出。纲分类水平上,2个时间点样品相对丰度差异不显著(P>0.05),且相对丰度最高的均为纤毛门,侧口纲。科水平分类上,A样品中均毛科相对丰度显著高于F样品(P<0.05,14.2% vs. 1.9%),F样品中头毛科相对丰度显著高于A样品(P<0.05,42.8% vs. 31.8%)。属分类水平上,所有样品中共有9个属被鉴定出,各样品中纤毛虫属水平上种群组成见图4;已知属中,多甲多泡双毛属相对丰度最高(A样品为20.9%,F样品为25.4%),且2个样品间差异不显著(P>0.05);内毛属、腹甲双毛属、头毛属与刺甲双毛属在2个样品中存在显著差异(P<0.05),A样品中内毛属、腹甲双毛属显著高于F样品(P<0.05),而头毛属、刺甲双毛属显著低于F样品(P<0.05);2个时间点样品中均含有为未知属(A样品为35.4%,F样品为35.6%,未在表中列出)。因此,2个样品中纤毛虫种群结构存在较大差异。
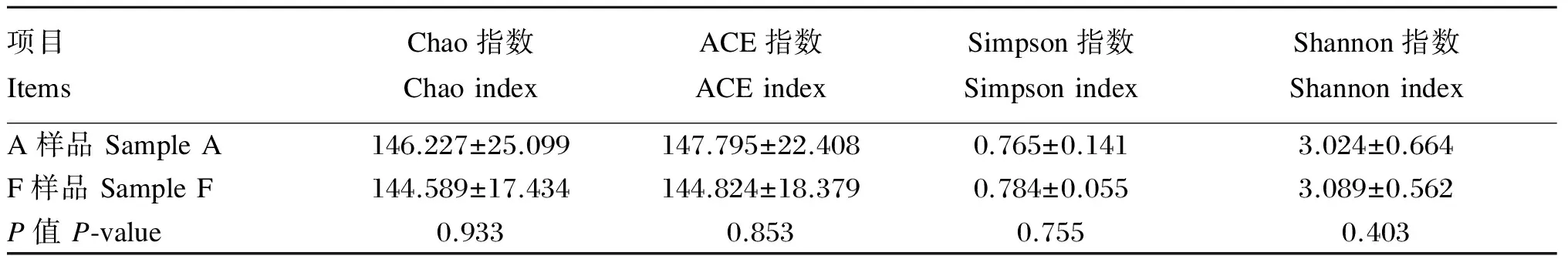
表2 取样深度为33 019时的瘤胃微生物alpha多样性指数对比

表3 瘤胃液中纤毛虫种群分布
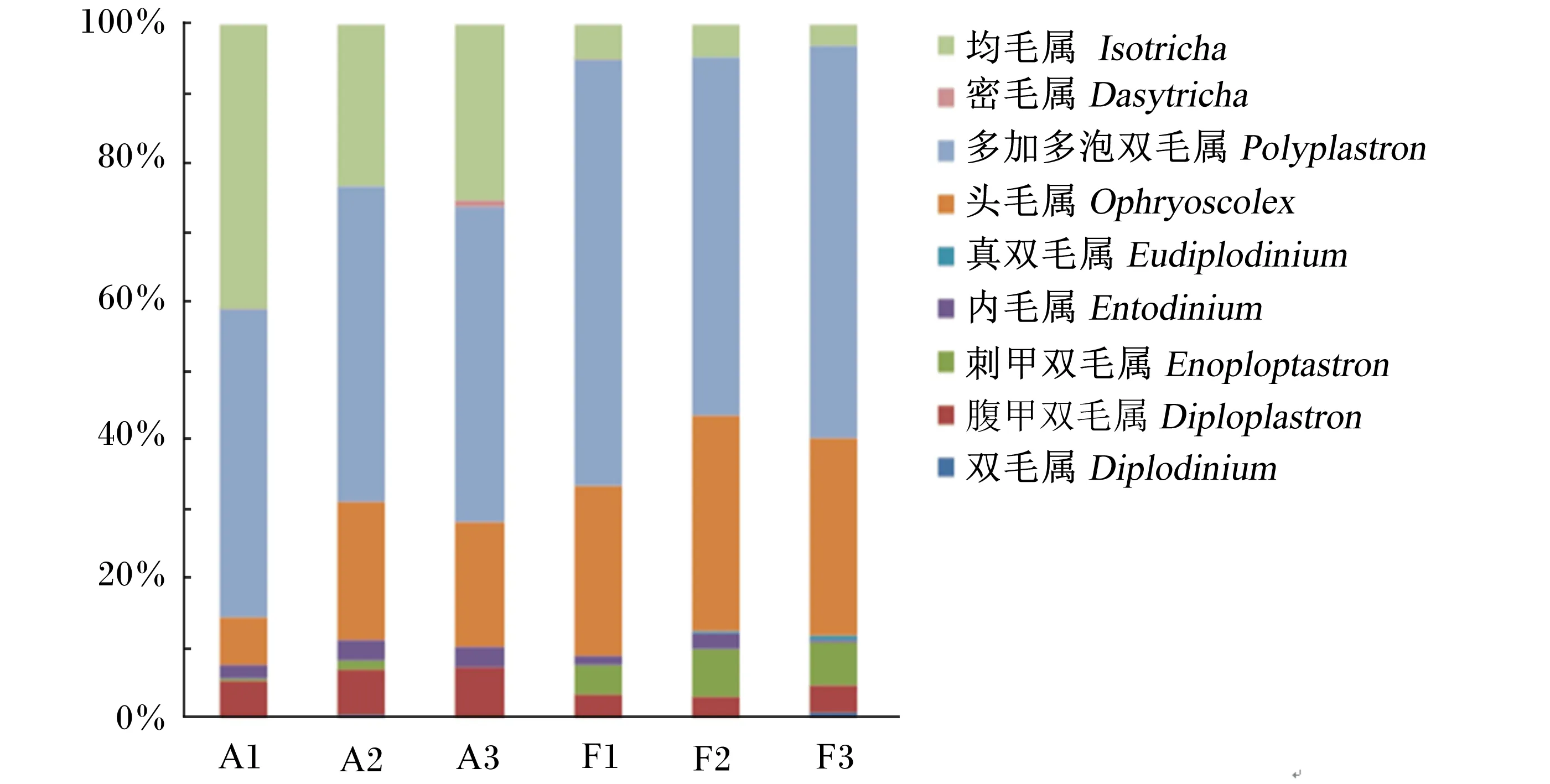
图4 属水平上瘤胃纤毛虫种群组成
3 讨 论
3.1 川中黑山羊瘤胃真核生物多样性分析
通过单样品的OTU和alpha多样性分析来反映微生物群落的丰富度和多样性。本试验通过MiSeq测序平台,平均每个样品获得275个OTU,远远低于应用该技术研究瘤胃细菌所得OTU数(962~2 499)[15,19-20],并且alpha多样性指数也远远低于应用该技术研究瘤胃细菌所获结果[Chao指数(836~2 687)[15,18,21]、ACE指数(330~841)[18-19]、Shannon指数(3.85~8.28)][19-21],由此可见瘤胃内细菌丰富度及多样性远远高于瘤胃内真核生物。此外本次所获Shannon指数(3.024、3.089)相对王新峰等[22]应用PCR-变性梯度凝胶电泳(DGGE)技术研究山羊瘤胃纤毛虫获得的Shannon指数(0.824)较高,可见针对群落多样性的研究,高通量测序技术明显优于PCR-DGGE技术。
Kittelmann等[23]通过454焦磷酸测序平台研究家养绵羊瘤胃真核生物组成,其结果中Simpson指数浮动较大(0.004~0.767),紫花苜蓿牧场放牧(胃管插管采样)组Simpson指数仅为0.004,饲喂谷粒圈养组(屠宰法采样)组Simpson指数为0.096,而夏季放牧和冬季放牧2组(永久瘘管采样)Simpson指数相差不大(0.711和0.731)。本研究结果(Simpson指数0.765、0.784)与Kittelmann等[23]研究结果(0.004、0.096)相差较大,这可能是受到了饲粮结构的影响。瘤胃真核生物作为瘤胃内饲粮消化吸收的重要组成成分,饲粮结构变化是影响群落多样性的关键因素[21,24]。Kobayashi[25]认为饲粮结构的单一性可能是影响反刍动物瘤胃微生物多样性的原因。此外,也可能是测序平台、品种与饲养方式不同引起的。
另外,2个时间点样品对比下,alpha多样性指数无显著差异。由此可见,短时间内瘤胃真核生物多样性不会发生显著变化。
3.2 川中黑山羊瘤胃纤毛虫种群结构分析
本次试验在2个时间点样品中均含有大量未知属,所占的比例高达35.4%与35.6%,这充分显示出瘤胃内还有很多有价值的真核生物种群信息需要深入挖掘。所有样品中共发现9个属(均毛属、密毛属、头毛属、多加多泡双毛属、腹甲双毛属、刺甲双毛属、真双毛属、双毛属、内毛属),其中多加多泡双毛属为最优势种属,这与以往大部分研究结果并不一致。大部分研究结果表明,内毛属为山羊瘤胃内最优势种属,包括徐淮白山羊(81.3%)[26]、内蒙古山羊(74.25%)[27]、呼伦贝尔草原绵羊(77.1%)[28],西班牙山羊(74%~85%)[29]和家养绵羊(40%)[23]等。而本试验结果中内毛属仅占总纤毛虫的2.6%、1.3%,引起这种差异可能由于饲粮结构的不同。本试验饲粮以牧草为主,牧草为南方牧草,南方牧草与其他地区牧草相比,粗蛋白质和粗脂肪含量低,但粗纤维含量高[30],而多甲多泡双毛属木聚糖酶和葡聚糖酶活力旺盛,内毛属只有微弱的作用[31],因此高纤维牧草可能是导致此结果的诱因。此外,也有可能是因为饲养方式的因素,Mishima等[32]研究称放养坦桑福尼亚瘤牛短角牛瘤胃纤毛虫内毛属含量(7.0%~25%)显著低于大部分家养反刍动物(80%~99%);Coleman[14]称放牧有利于反刍动物瘤胃原虫的定植,放牧的羔羊在正常饲养条件下3~6个月即可获得与成年羊类似的原虫种群;Franzolin等[33]研究2种饲养方式下印度水牛瘤胃微生物的组成,结果表明放牧组瘤胃纤毛虫总数及组成明显优于饲喂组。除了饲粮结构与饲养方式的影响外,日龄等也会影响瘤胃微生物;Hungate[11]称幼龄反刍动物瘤胃内容物呈酸性,妨碍瘤胃原生动物的定值;另外,高通量测序技术也会对真实种群结构有所影响,Kittelmann等[34]称高通量测序技术结果使体积较小的纤毛虫属(内毛属、端毛属和双毛属等)含量被低估,而体积较大的种属(后毛属、前毛属、真双毛属、硬甲双毛属和多甲多泡双毛属等)含量被高估。
2个时间点样品相对比下,相对丰度最高的均为多加多泡双毛属,并无显著差异。而均毛属、腹甲双毛属、头毛属和刺甲双毛属存在显著差异。这种差异可能是饲粮结构影响的结果,在长期高纤维牧草饲喂下,利于消化纤维的头毛属等逐渐增多,而利于淀粉多糖消化的均毛属逐渐减少[35-36]。因此饲粮可能是引起这种变化的主要因素,此外,样本数量少也可能影响本研究反映纤毛虫种群结构真实的变化。
4 结 论
① 本研究通过高通量测序技术分析了2个时间点川中黑山羊瘤胃纤毛虫种群结构。结果证明,幼龄川中黑山羊瘤胃纤毛虫最优势种属为多甲多泡双毛属,且饲粮显著影响瘤胃纤毛虫属结构的变化。
② 瘤胃真核生物群落丰富度与多样性在短时间内无显著差异。
③ 瘤胃中还有许多未被分类鉴定且相对丰度较高的真核生物,其结构和功能有待深入研究。
[1] IMAI S,OKU Y,MORITA T,et al.Rumen ciliate protozoal fauna of reindeer in Inner Mongolia,China[J].Journal of Veterinary Medical Science,2004,66(2):209-212.
[2] DE MATOS D S,GUIM A,BATISTAM V,et al.Rumen ciliate protozoa in sheep raising in the caatinga region of Pernambuco State,Brazil[J].Revista Brasileira de Saúde e Produção Animal,2008,9(2):270-279.
[3] SYLVESTER J T,KARNATI S K R,YU Z T,et al.Development of an assay to quantify rumen ciliate protozoal biomass in cows using real-time PCR[J].The Journal of Nutrition,2004,134(12):3378-3384.
[4] CLARKE R T J.Role of the Rumen ciliates in bloat in cattle[J].Nature,1965,205(4966):95-96.
[5] 周亚文,张玉杰,林波,等.瘤胃甲烷生成过程中微生物之间的相互关系[J].动物营养学报,2011,23(4):556-562.
[6] NEWBOLD C J,DE LA FUENTE G,BELANCHE A,et al.The role of ciliate protozoa in the Rumen[J].Frontiers in Microbiology,2015,6:1313.
[7] ONODERA R,TSUTSUMI W,KANDATSU M.Formation of δ-aminovaleric acid from proline,ornithine and arginine by rumen ciliate protozoa[J].Agricultural and Biological Chemistry,1977,41(11):2169-2175.
[8] TOKURA M,USHIDA K,MIYAZAKI K,et al.Methanogens associated with rumen ciliates[J].FEMS Microbiology Ecology,2006,22(2):137-143.
[10] IVAN M,KOENIG K M,TEFEREDEGNE B,et al.Effects of the dietaryEnterolobiumcyclocarpumfoliage on the population dynamics of rumen ciliate protozoa in sheep[J].Small Ruminant Research,2004,52(1/2):81-91.
[11] HUNGATE R E.The rumen and its microbes[M].New York:Academic Press,1966:466-525.
[12] 刘景喜,韩静,潘振亮,等.不同月龄荷斯坦犊牛瘤胃纤毛虫计数及形态观察[J].饲料研究,2013(2):38-41.
[14] COLEMAN G S.Rumen ciliate protozoa[J].Advances in Parasitology,1980,18:121-173.
[15] 王继文,王立志,闫天海,等.山羊瘤胃与粪便微生物多样性[J].动物营养学报,2015,27(8):2559-2571.
[16] EDGAR R C.Search and clustering orders of magnitude faster than BLAST[J].Bioinformatics,2010,26(19):2460-2461.
[17] BLAXTER M,MANN J,CHAPMAN T,et al.Defining operational taxonomic units using DNA barcode data[J].Philosophical Transactions of the Royal Society B:Biological Sciences,2005,360(1462):1935-1943.
[18] 高雨飞,欧阳克蕙,瞿明仁,等.利用MiSeq测序技术分析锦江牛瘤胃细菌多样性[J].动物营养学报,2016,28(1):244-248.
[19] 曾燕,简平,倪学勤,等.Illumina MiSeq测序平台测定蒙古羊瘤胃液相和固相菌群多样性[J].动物营养学报,2015,27(10):3256-3262.
[20] GUO W,LI Y,WANG L Z,et al.Evaluation of composition and individual variability of rumen microbiota in yaks by 16S rRNA high-throughput sequencing technology[J].Anaerobe,2015,34:74-79.
[21] PITTA D W,PINCHAK W E,DOWD S E,et al.Rumen bacterial diversity dynamics associated with changing from bermudagrass hay to grazed winter wheat diets[J].Microbial Ecology,2010,59(3):511-522.
[22] 王新峰,苏勇,毛胜勇,等.基于18S rRNA基因的PCR/DGGE技术研究山羊瘤胃原虫动态变化[J].生物技术通报,2008(4):141-144.
[23] KITTELMANN S,SEEDORF H,WALTERS W A,et al.Simultaneous amplicon sequencing to explore Co-occurrence patterns of bacterial,archaeal and eukaryotic microorganisms in rumen microbial communities[J].PLoS One,2013,8(2):e47879.
[24] BELANCHE A,DOREAU M,EDWARDS J E,et al.Shifts in the rumen microbiota due to the type of carbohydrate and level of protein ingested by dairy cattle are associated with changes in rumen fermentation[J].The Journal of Nutrition,2012,142(9):1684-1692.
[25] KOBAYASHI Y.Inclusion of novel bacteria in rumen microbiology:need for basic and applied science[J].Animal Science Journal,2006,77(4):375-385.
[26] 王梦芝,王洪荣,徐爱秋,等.徐淮白山羊瘤胃细菌和原虫的类群结构研究[J].中国农业科学,2009,42(8):2915-2922.
[27] 桂荣,今井壮一.中国内蒙古山羊瘤胃纤毛虫种类构成的研究[J].动物营养学报,1990,2(1):64.
[28] 桂荣.呼伦贝尔草原绵羊瘤胃内纤毛虫种类及其构成比的研究[J].中国兽医科学,1988(6):8-11.
[29] DE LA FUENTE G ,BELANCHE A,ABECIA L,et al.Rumen protozoal diversity in the Spanish ibex (Caprapyrenaicahispanica) as compared with domestic goats (Caprahircus)[J].European Journal of Protistology,2009,45(2):112-120.
[30] 杜占池,杜菁昀.我国不同自然区域天然牧地和人工牧地营养价值的比较研究[J].中国草地,2003,25(1):22-26.
[31] WILLIAMS A G,COLEMAN G S.Role of protozoa in the Rumen[M].New York:Springer,1992.
[32] MISHIMA T,KATAMOTO H,HORII Y,et al.Rumen ciliates from Tanzanian short horn zebu cattle,Bostaurusindicus,and the infraciliature ofEntodiniumpalmaren. sp. andEnoploplastronstokyi(Buisson,1924)[J].European Journal of Protistology,2009,45(2):77-86.
[33] FRANZOLIN R,WRIGHT A D G.Microorganisms in the rumen and reticulum of buffalo (Bubalusbubalis) fed two different feeding systems[J].BMC Research Notes,2016,9(1):243.
[34] KITTELMANN S,DEVENTE S R,KIRK M R,et al.Phylogeny of intestinal ciliates,includingCharoninaventriculi,and comparison of microscopy and 18S rRNA gene pyrosequencing for rumen ciliate community structure analysis.[J].Applied and Environmental Microbiology,2015,81(7):2433-2444.
[35] 桂荣.放牧绵羊瘤胃纤毛虫种类分布及补饲对它的影响[J].畜牧兽医学报,1992,23(4):372-378.
[36] BELANCHE A,RAMOS-MORALES E,NEWBOLD C J.Invitroscreening of natural feed additives from crustaceans,diatoms,seaweeds and plant extracts to manipulate rumen fermentation[J].Journal of the Science of Food and Agriculture,2016,96(9):3069-3078.
*Corresponding author, professor, E-mail: dengjl213@126.com
(责任编辑 王智航)
Ruminal Ciliate Community Structure ofChuanzhongBlack Goats: An Analysis Using High-Throughput Sequencing Technology
LIU Qi CHEN Yun DENG Junliang*REN Zhihua YANG Yanyi GAO Shuang CHEN Chong
(KeyLaboratoryofAnimalDiseaseandHumanHealthofSichuanProvince,KeyLaboratoryofEnvironmentalHazardandAnimalDiseaseofSichuanProvince,CollegeofVeterinaryMedicine,SichuangAgriculturalUniversity,Chengdu611130,China)
This study was designed to reveal the ruminal ciliate community structure ofChuanzhongblack goats using MiSeq sequencing technology. Three 4-month-oldChuanzhongblack goats weighted (15.53±0.21) kg were used to collect rumen fluid, and the samples were collected after normal feeding of 20 (sample A) and 60 d (sample F), respectively. Total DNA was extracted for amplification V4 area of 18S rRNA, and the products were sequenced by Illumina MiSeq sequencing system. The results showed as follows: 1) a total of 242 321 high quality valid sequences and 1 650 operational taxonomic units were obtained. 2) There were no significant difference on alpha diversity indexes (Chao, ACE, Shannon and Simpson) between samples A and F (P>0.05). 3) At class level, the most abundant class in samples A and F were Ciliophora, Litostomatea (sample A was 46.0%, sample F was 44.7%), and there was no significant difference between samples A and F (P>0.05). 4) At family level, the most abundant family in sample A was Ophryoscolecidae (31.8%) followed by Isotrichidae (14.2%); the most abundant family in sample F was Ophryoscolecidae (42.8%); the relative abundance of Ophryoscolecidae in sample F was significantly higher than that in sample A (P<0.05), and the relative abundance of Isotrichidae in sample A was significantly higher than that in sample F (P<0.05). 5) At genus level, the most abundant ciliate genus wasPolyplastron(sample A was 20.9%, sample F was 25.4%), and there are no significant differences between samples A and F (P>0.05); there were significant differences of the relative abundances ofIsotricha,Ophryoscolex,DiploplastronandEnoploplastron, the relative abundances ofIsotricha(14.1% vs. 1.9%) andDiploplastron(2.8% vs. 1.5%) in sample A were significantly higher than those in sample F (P<0.05), and relative abundances ofOphryoscolex(6.7% vs. 12.5%) andEnoploplastron(0.3% vs. 2.5%) in sample A were significantly lower than those in sample F (P<0.05). In conclusion,Polyplastronis the most abundant genus in the rumen ciliate communities of youthChuanzhongblack goats, and there are still a lot of unclassified and unidentified eukaryotic microorganisms with high abundance worth further study in rumen.[ChineseJournalofAnimalNutrition, 2017, 29(5):1574-1581]
Chuanzhongblack goat; ruminal ciliate; community structure; diversity; MiSeq sequencing
10.3969/j.issn.1006-267x.2017.05.016
2016-11-04
“长江学者和创新团队发展计划”创新团队项目(IRT0848);四川农业大学双支计划(03572070)
刘 旗(1992—),男,河南漯河人,硕士研究生,研究方向为中西兽医与临床。E-mail: 398772948@qq.com
*通信作者:邓俊良,教授,博士生导师,E-mail: dengjl213@126. com
S826
A
1006-267X(2017)05-1574-08

