HBV基因突变及其临床意义研究进展
刘灿,欧启水
(福建医科大学附属第一医院检验科,福州 350005)
·综述·
HBV基因突变及其临床意义研究进展
刘灿,欧启水
(福建医科大学附属第一医院检验科,福州 350005)
HBV以准种形式存在患者体内,不同基因区的准种变化具有明显的异质性,其对疾病的诊断、治疗及转归均有一定影响。理解HBV突变及其改变的分子及病理特征,有助于实现不同乙肝感染患者的分层管理,促进更精准的个体化治疗。该文就HBV基因突变及其临床意义研究进展作一综述。
HBV;基因;突变;临床意义
HBV为部分双链环状嗜肝DNA病毒,基因组含4个部分重叠的开放读码框(open reading frame,ORF),即前-S/S区、前-C/C区、P区和X区。HBV侵入机体后脱衣壳形成松弛环状DNA(relaxed circular DNA,rcDNA),进入靶细胞核内,在DNA聚合酶作用下合成共价闭合环状DNA(covalently closed circular DNA,cccDNA),再经过RNA聚合酶转录成各种大小不同的mRNA,其中前基因组RNA(pregenomic RNA,pgRNA)在HBV逆转录酶作用下合成完整的HBV DNA,其他mRNA则翻译成病毒的各种蛋白质成份(见图1)[1]。根据HBV基因组序列差异性>8%和S基因序列差异性>4%,可将HBV区分为至少10个基因型(A~J型)及若干个基因亚型,我国主要以B、C基因型感染为主[2]。然而,由于HBV逆转录过程缺乏严格的校正机制,每104~105核苷酸就会出现1个突变。因此,每一个患者体内的病毒构成并不均一,而是由HBV基因序列存在微小差异的多种病毒株组成,并始终处于动态变化过程中,即以准种(quasispecies)形式存在[3]。HBV准种的异质性变化包括复杂性和多样性改变[4],不同基因型HBV的准种异质性变化对疾病的诊断、治疗及转归均有一定的影响。本文就HBV不同基因区的突变、抗病毒疗效及临床转归的关系等作一综述。
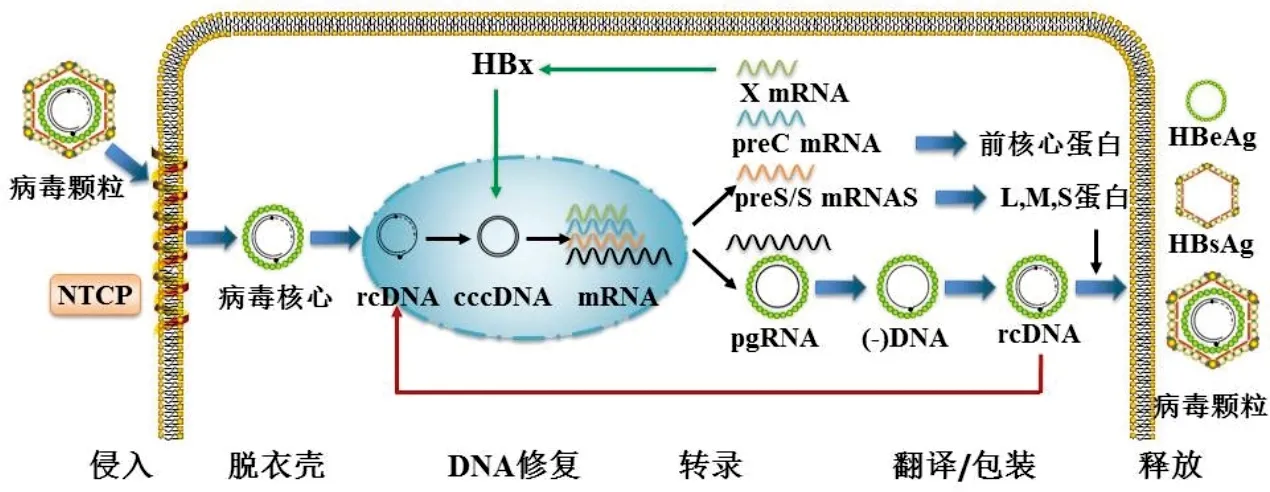
注:本图在文献[1]基础上修改而成;NTCP,钠离子/牛磺胆酸共转运多肽;rcDNA,松弛环状DNA;cccDNA,共价闭合环状DNA;pgRNA,前基因组RNA。
图1 HBV感染流程
1 前-S/S区基因突变及其临床意义
HBV前-S/S区基因包括preS1区、preS2区和S区,分别有各自的起始密码子,但共用一个终止密码子[5](见图2A)。前-S/S区基因主要编码3种包膜蛋白:大蛋白、中蛋白和主蛋白,分别又称L、M和S蛋白。preS1因基因亚型的不同而造成编码氨基酸长度不固定,由108至126个氨基酸构成,此区域包含与肝细胞结合受体钠离子/牛磺胆酸共转运多肽(Na+/taurocholate cotransporting polypeptide, NTCP)的结构域,在病毒感染进入细胞中起重要作用[6]。preS2由55个氨基酸组成,与T、B淋巴细胞免疫表位识别有关[7]。S区基因由226个氨基酸组成,包括N端区(aa1~98)、主要亲水区(major hydrophilic region,MHR,aa99~169)和C端区(aa170~226),负责编码HBsAg,是病毒包膜蛋白的主要抗原区域[7]。
前-S序列是整个HBV基因组中异质性最高的区域,突变类型包括替换、缺失和插入等多种形式,见表1。相对于B基因型,preS1/preS2区突变更常发生于HBV C型感染患者[5]。S区基因突变主要集中在MHR,发生在“a”决定区(aa124~147,能诱导机体针对所有HBV亚型产生保护性应答的区域)的突变,可改变HBsAg的空间构象引起抗原性质改变,无法被抗HBs抗体中和,成为免疫逃逸突变;发生在“a”决定区以外的突变,除了发生免疫逃逸外,还常常影响肝脏疾病进展及核苷(酸)类似物(NAs)耐药的发生,见表1。前-S/S区基因突变也会对临床HBsAg检测造成影响[8]。如产生的HBsAg无法被商品化试剂检出,容易出现HBsAg/抗HBs抗体双阳性的结果,甚至造成隐匿性肝炎(occult hepatitis B virus infection,OBI)的发生[9]。另一方面,不同的检测系统对突变株的检测能力不一[10]。Servant-Delmas等[11]比较了13种商品化试剂检测HBsAg的结果,发现对不同类型突变株不同试剂检测结果间存在差异,其总体检出率从62.9%~97.9%不等。因此,当临床对HBsAg检测结果持有疑议时,应尽可能采用另一种方法进行复检。
S区基因与HBV逆转录酶区基因重叠,有研究利用二代测序平台(next generation sequencing,NGS)如超深焦磷酸测序(ultra-deep pyrosequencing, UDPS)等在NAs治疗患者中发现了一些新的低比例(0.13%~0.17%)突变位点,如sW156stop、sW163stop、sW165stop和sW191stop等[12-13],会导致HBsAg合成缺陷。然而,这些低比例突变的确定临床意义目前尚不明了。

注:A,前-S/S区基因结构; B,前-C/C区基因结构及编码产物; C,HBV P区基因结构;D,HBV X区基因结构;nt,核苷酸;rt,逆转录酶;aa,氨基酸;MHR区,主要亲水区;NRD负向调控原件。

图2 HBV各基因区结构
注:*,终止密码;突变碱基前未加“nt”均代表氨基酸位点(全文同)。
2 前-C/C区基因突变及其临床影响
HBV前-C/C区基因依据各自的起始密码子,在基本核心启动子(basal core promoter,BCP)引导下,转录成preC mRNA和pgRNA,前者翻译成前核心蛋白和核心蛋白,进而剪切成HBeAg和HBcAg[5](图2B)。文献报道HBV前-C/C区基因的异质性常发生在BCP和前-C区,见表2。BCP和前-C区突变会促进HBeAg血清学转换,因此早期检出BCP和前-C区突变可以预测HBeAg的血清学转换[24]。与HBeAg阳性患者相比,HBeAg阴性患者更易发生前-C区突变,而且这些突变并不引起DNA拷贝数的变化,但与肝脏疾病进展密切相关[25]。有研究显示,BCP/前-C区突变可能对抗病毒药物具有更高的敏感性[26-27],但如果BCP/前-C突变联合前-S1/S2缺失,往往会导致更严重的肝脏疾病如暴发性肝炎和HCC的发生[28]。而且,BCP/前-C区突变与基因型相关,好发于HBV基因型B和D感染的患者[29]。

表2 常见BCP、前-C/C区突变及临床影响
最近有研究利用高灵敏UDPS技术,分析BCP、前-C/C区基因的异质性,发现在基线水平即存有少量G1896A、G1899A和前-C区起始密码子突变,并随疾病进展不断变化[26];Bayliss等[32]利用NGS检测HBeAg阳性患者发现,基线水平存在少量可检测的BCP/前-C突变,会显著降低NAs治疗后HBsAg的清除率。
3 P区基因突变及其临床意义
P区是最长的HBV基因区,包含4个功能结构域:末端蛋白(terminal protain,TP)、间隔区(spacer region, Spc)、逆转录酶(RT)区和RNA酶H区(RNH),其中RT区又可分为7个结构域(图2C)。P区编码DNA聚合酶,参与HBV DNA的合成及pgRNA的逆转录。TP位于P区N端,作为DNA负链合成的初始引物;Spc区作为聚合酶功能非必要的补充,更能耐受突变,由于间隔区与前-S区部分交叉,因此该区域变异亦会引起相应的HBsAg氨基酸位点的改变;RT区具备DNA聚合酶活性和逆转录活性,其催化中心位于nt736~747(即YMDD区),该区在所有基因型中高度保守,在HBV复制中发挥着重要作用;RNA酶H区位于P区C端,参与RNA的衣壳包装和负链DNA的合成[34]。
抗病毒治疗药物NAs主要通过竞争结合HBV DNA聚合酶尤其是RT区,发挥强效抑制病毒复制的作用,一旦该区基因发生变异,就会导致NAs结合力下降,从而失去对突变株的抑制能力,产生耐药[35]。因此,以往研究最多的HBV P区异质性主要集中在RT区,见表3。与干扰素治疗相比,A、B基因型较C、D基因型具有更好的应答效果,然而基因型差异对NAs的疗效应答情况目前仍知之甚少[2]。

表3 常见RT区突变及临床影响
注:LMV,拉米夫定;ADF,阿德福韦酯;ETV,恩替卡韦;TDF,富马酸替诺福韦酯;LdT,替比夫定;ETV耐药相关变异是在rtM204V/I+rtLl80M变异基础上,再联合其他至少一个位点的氨基酸替代变异;*,仅在体外实验发现,在临床实践中并未完全确认TDF的耐药位点。
本课题组曾构建高灵敏RT-AS-LNA-qPCR[44]和RT-ARMS-qPCR[45]技术,分别用于HBV YIDD(rtM204I)和YVDD(rtM204V)突变株的定量检测,初步揭示了早期检出低比例突变DNA对NAs的疗效预测价值。近来有研究利用NGS平台显示,NAs治疗的CHB患者其HBV RT区准种异质性呈现多样性及低比例变化的趋势[5,46-48]。进一步研究发现,选择LAM或LdT治疗4周时,应答组RT区准种复杂性和多样性显著低于无应答组[46],采用ETV时,完全应答组较部分应答组准种复杂性降低,而准种多样性增加[5,48],提示不同准种异质性对药物的动力学和药效学变化有影响;而且高基线水平RT 2区(与S区基因重叠的B、C结构域)的准种异质性可预测病毒学应答[47]。尽管如此,这些在基线水平及NAs治疗过程中出现的低比例耐药突变,其确切的临床意义仍未完全阐明。
4 X区基因突变及其临床意义
X区是HBV最小的基因区,N端含负向调控域(negative regulatory domain,NRD),具有抗凋亡基因;C端为转录因子结合区(transcription factor binding),含促凋亡基因[49]。其编码产物HBx蛋白不与宿主及病毒DNA直接结合,主要通过激活或抑制转录因子对HBV DNA病毒转录、复制及宿主细胞基因起调控作用,是导致肝癌发生的关键分子[50]。X区基因突变主要发生在肝癌患者,有研究显示,当存在K130M+V131I双联突变及V5M/L+K130M+V131I三联突变时,HCC的风险分别提高3.75倍和5.34倍[51]。鉴于X区基因突变与HCC密切相关,有研究利用测序技术,发现当8个X区基因位点(包括ntG1613A,ntC1653T,ntT1753V,ntA1762T,ntG1764A,ntA1846T,ntG1896A和ntG1899A)出现≥6个突变位点阳性作为预测HCC的指标,其敏感性为44.0%,特异性可达99.7%[52]。
目前,单独针对X区基因的二代测序研究较少。研究显示与前-C区重叠的X区基因(nt 1 814~1 838)中,当存有少量(0.16%)ntT1836C突变时,会导致原有的终止密码子TAA突变为编码谷氨酰胺的密码子CAA,从而产生的HBx蛋白的羧基末端比正常HBx多51个氨基酸,致使HBx蛋白发生潜在的功能性改变[26]。
5 小结与展望
HBV基因组具有明显的准种异质性,有些与病毒治疗反应有关,有些与免疫逃逸相关,有些可加速肝脏疾病进展,有些会引起检测结果不一致。因此,借助于分子诊断技术的发展及临床研究的深入,能进一步了解不同HBV基因突变的临床意义,尤其是证实一些低比例突变的时空特性,更好地针对不同的乙肝感染个体做出精准医疗决策。
[1]Nassal M. HBV cccDNA: viral persistence reservoir and key obstacle for a cure of chronic hepatitis B[J]. Gut, 2015, 64(12): 1972-1984.
[2]Lin CL, Kao JH. Hepatitis B virus genotypes and variants[J]. Cold Spring Harb Perspect Med, 2015, 5(5): a021436.
[3]Locarnini S, Zoulim F. Molecular genetics of HBV infection[J]. Antivir Ther, 2010, 15(Suppl 3): 3-14.
[4]傅亚, 欧启水. 乙型肝炎病毒准种的研究进展[J]. 中华检验医学杂志, 2016, 39(8):1-4.
[5]Gao S, Duan ZP, Coffin CS. Clinical relevance of hepatitis B virus variants[J]. World J Hepatol, 2015, 7(8): 1086-1096.
[6]Yan H, Zhong G, Xu G,etal. Sodium taurocholate cotransporting polypeptide is a functional receptor for human hepatitis B and D virus[J]. Elife, 2012,1: e00049.
[7]Pollicino T, Cacciola I, Saffioti F,etal. Hepatitis B virus PreS/S gene variants: pathobiology and clinical implications[J]. J Hepatol, 2014, 61(2): 408-417.
[8]傅晓春, 刘灿, 欧启水. 慢性乙型肝炎患者HBsAg和抗HBs共存的发生机制及其临床意义[J]. 临床检验杂志, 2016, 34(8): 566-570.
[9]Raimondo G, Pollicino T, Romanò L,etal. A 2010 update on occult hepatitis B infection[J]. Pathol Biol(Paris), 2010, 58(4): 254-257.
[10]Osiowy C, Kowalec K, Giles E. Discordant diagnostic results due to a hepatitis B virus T123A HBsAg mutant[J]. Diagn Microbiol Infect Dis, 2016, 85(3): 328-333.
[11]Servant-Delmas A, Mercier-Darty M, Ly TD,etal. Variable capacity of 13 hepatitis B virus surface antigen assays for the detection of HBsAg mutants in blood samples[J]. J Clin Virol, 2012, 53(4): 338-345.
[12]Rodriguez-Frías F, Tabernero D, Quer J,etal. Ultra-deep pyrosequencing detects conserved genomic sites and quantifies linkage of drug-resistant amino acid changes in the hepatitis B virus genome[J]. PLoS One, 2012, 7(5): e37874.
[13]Solmone M, Vincenti D, Prosperi MC,etal. Use of massively parallel ultradeep pyrosequencing to characterize the genetic diversity of hepatitis B virus in drug-resistant and drug-naive patients and to detect minor variants in reverse transcriptase and hepatitis B S antigen[J]. J Virol, 2009, 83(4): 1718-1726.
[14]Olinger CM, Weber B, Otegbayo JA,etal. Hepatitis B virus genotype E surface antigen detection with different immunoassays and diagnostic impact of mutations in the preS/S gene[J]. Med Microbiol Immunol, 2007, 196(4): 247-252.
[15]Datta S, Ghosh A, Dasgupta D,etal. Novel point and combo-mutations in the genome of hepatitis B virus-genotype D:characterization and impact on liver disease progression to hepatocellular carcinoma[J]. PLoS One, 2014, 9(10): e110012.
[16]Kim H, Lee SA, Kim DW,etal. Naturally occurring mutations in large surface genes related to occult infection of hepatitis B virus genotype C[J]. PLoS One, 2013, 8(1): e54486.
[17]Liu W, Hu T, Wang X,etal. Coexistence of hepatitis B surface antigen and anti-HBs in Chinese chronic hepatitis B virus patients relating to genotype C and mutations in the S and P gene reverse transcriptase region [J]. Arch Virol, 2012, 157(4): 627-634.
[18]Abdelnabi Z, Saleh N, Baraghithi S,etal. Subgenotypes and mutations in the s and polymerase genes of hepatitis B virus carriers in the West Bank, palestine[J]. PLoS One, 2014, 9(12): e113821.
[19]Araujo NM, Vianna CO, Moraes MT,etal. Expression of Hepatitis B virus surface antigen(HBsAg) from genotypes A, D and F and influence of amino acid variations related or not to genotypes on HBsAg detection[J]. Braz J Infect Dis, 2009, 13(4): 266-271.
[20]Salpini R, Colagrossi L, Bellocchi MC,etal. Hepatitis B surface antigen genetic elements critical for immune escape correlate with hepatitis B virus reactivation upon immunosuppression [J]. Hepatology, 2015, 61(3): 823-833.
[21]Pollicino T, Raffa G, Costantino L,etal. Molecular and functional analysis of occult hepatitis B virus isolates from patients with hepatocellular carcinoma[J]. Hepatology, 2007, 45(2): 277-285.
[22]Torresi J, Earnest-Silveira L, Deliyannis G,etal. Reduced antigenicity of the hepatitis B virus HBsAg protein arising as a consequence of sequence changes in the overlapping polymerase gene that are selected by lamivudine therapy[J]. Virology, 2002, 293(2): 305-313.
[23]Mirabelli C, Surdo M, Van Hemert F,etal. Specific mutations in the C-terminus domain of HBV surface antigen significantly correlate with low level of serum HBV-DNA in patients with chronic HBV infection[J]. J Infect, 2015, 70(3): 288-298.
[24]Chu CJ, Hussain M, Lok AS. Hepatitis B virus genotype B is associated with earlier HBeAg seroconversion compared with hepatitis B virus genotype C[J]. Gastroenterology, 2002, 122(7): 1756-1762.
[25]Wang XL, Ren JP, Wang XQ,etal. Mutations in pre-core and basic core promoter regions of hepatitis B virus in chronic hepatitis B patients[J]. World J Gastroenterol, 2016, 22(11): 3268-3274.
[26]Homs M, Buti M, Quer J,etal. Ultradeep pyrosequencing analysis of the hepatitis B virus preCore region and main catalytic motif of the viral polymerase in the same viral genome[J]. Nucleic Acids Res, 2011, 39(19): 8457-8471.
[27]Homs M, Buti M, Tabernero D,etal. Quasispecies dynamics in main core epitopes of hepatitis B virus by ultra-deep-pyrosequencing[J]. World J Gastroenterol, 2012, 18(42): 6096-6105.
[28]Yin J, Xie J, Liu S,etal. Association between the various mutations in viral core promoter region to different stages of hepatitis B, ranging of asymptomatic carrier state to hepatocellular carcinoma[J]. Am J Gastroenterol, 2011, 106(1): 81-92.
[29]Rodriguez-Frias F, Buti M, Tabernero D,etal. Quasispecies structure, cornerstone of hepatitis B virus infection: mass sequencing approach[J]. World J Gastroenterol, 2013, 19(41): 6995-7023.
[30]Liao Y, Hu X, Chen J,etal. Precore mutation of hepatitis B virus may contribute to hepatocellular carcinoma risk: evidence from an updated meta-analysis[J]. PLoS One, 2012, 7(6): e38394.
[31]Tu WH, Lv Y, Zhang YM,etal. Precore/basal core promoter mutants quantification throughout phases of hepatitis B virus infection by Simpleprobe[J]. World J Gastroenterol, 2015, 21(21): 6639-6648.
[32]Bayliss J, Yuen L, Rosenberg G,etal. Deep sequencing shows that HBV basal core promoter and precore variants reduce the likelihood of HBsAg loss following tenofovir disoproxil fumarate therapy in HBeAg-positive chronic hepatitis B[J]. Gut, 2016, pii: gutjnl-2015-309300.
[33]Homs M, Jardi R, Buti M,etal. HBV core region variability: effect of antiviral treatments on main epitopic regions[J]. Antivir Ther, 2011, 16(1): 37-49.
[34]Ko C, Shin YC, Park WJ,etal. Residues Arg703, Asp777, and Arg781 of the RNase H domain of hepatitis B virus polymerase are critical for viral DNA synthesis[J]. J Virol, 2014, 88(1): 154-163.
[35]Sharon A, Chu CK .Understanding the molecular basis of HBV drug resistance by molecular modeling[J]. Antiviral Res, 2008, 80(3): 339-353.
[36]Chen J, Yan L, Zhu FC,etal. Amino acid polymorphism in the reverse transcriptase region of hepatitis B virus and the relationship with nucleos(t)ide analogues treatment for preventing mother-to-infant transmission[J]. J Med Virol, 2014, 86(8): 1288-1295.
[37]Cento V, Van Hemert F, Neumann-Fraune M,etal. Anti-HBV treatment induces novel reverse transcriptase mutations with reflective effect on HBV S antigen[J]. J Infect, 2013, 67(4): 303-312.
[38]Bartholomeusz A, Locarnini SA. Antiviral drug resistance: clinical consequences and molecular aspects[J]. Semin Liver Dis, 2006, 26(2): 162-170.
[39]Sarin SK, Kumar M, Lau GK,etal. Asian-Pacific clinical practice guidelines on the management of hepatitis B: a 2015 update[J]. Hepatol Int, 2016, 10(1): 1-98.
[40]Qin B, Budeus B, Cao L,etal. The amino acid substitutions rtP177G and rtF249A in the reverse transcriptase domain of hepatitis B virus polymerase reduce the susceptibility to tenofovir[J]. Antiviral Res, 2013, 97(2): 93-100.
[41]Amini-Bavil-Olyaee S, Herbers U, Sheldon J,etal. The rtA194T polymerase mutation impacts viral replication and susceptibility to tenofovir in hepatitis B e antigen-positive and hepatitis B e antigen-negative hepatitis B virus strains[J]. Hepatology, 2009, 49(4): 1158-1165.
[42]Amini-Bavil-Olyaee S, Herbers U, Mohebbi SR,etal. Prevalence, viral replication efficiency and antiviral drug susceptibility of rtQ215 polymerase mutations within the hepatitis B virus genome[J]. J Hepatol, 2009, 51(4): 647-654.
[43]Karatayli E, Karatayli SC, Cinar K,etal. Molecular characterization of a novel entecavir mutation pattern isolated from a multi-drug refractory patient with chronic hepatitis B infection[J]. J Clin Virol, 2012, 53(2): 130-134.
[44]Zeng Y, Li D, Wang W,etal. Establishment of real time allele specific locked nucleic acid quantitative PCR for detection of HBV YIDD(ATT) mutation and evaluation of its application[J]. PLoS One, 2014, 9(2): e90029.
[45]Zeng Y, Yang B, Wu Y,etal. Clinical significance of periodic detection of hepatitis B virus YVDD mutation by ultrasensitive real-time amplification refractory mutation system quantitative PCR during lamivudine treatment in patients with chronic hepatitis B[J]. J Med Microbiol, 2015, 64(Pt 3): 237-242.
[46]Peveling-Oberhag J, Herrmann E, Kronenberger B,etal. Dynamics of hepatitis B virus quasispecies heterogeneity and virologic response in patients receiving low-to-moderate genetic barrier nucleoside analogs[J]. J Viral Hepat, 2013, 20(4): 234-239.
[47]Han Y, Gong L, Sheng J,etal. Prediction of virological response by pretreatment hepatitis B virus reverse transcriptase quasispecies heterogeneity: the advantage of using next-generation sequencing[J]. Clin Microbiol Infect, 2015, 21(8): 797.e1-797. e8.
[48]Liu F, Chen L, Yu DM,etal. Evolutionary patterns of hepatitis B virus quasispecies under different selective pressures: correlation with antiviral efficacy[J]. Gut, 2011, 60(9): 1269-1277.
[49]Slagle BL, Andrisani OM, Bouchard MJ,etal. Technical standards for hepatitis B virus X protein(HBx) research[J]. Hepatology, 2015, 61(4): 1416-1424.
[50]Ming Geng, Xuan Xin, Li-Quan Bi,etal. Molecular mechanism of hepatitis B virus X protein function in hepatocarcinogenesis[J]. World J Gastroenterol, 2015, 21(38): 10732-10738.
[51]Lee JH, Han KH, Lee JM,etal. Impact of hepatitis B virus(HBV) x gene mutations on hepatocellular carcinoma development in chronic HBV infection[J]. Clin Vaccine Immunol, 2011, 18(6): 914-921.
[52]Jang JW, Chun JY, Park YM,etal. Mutational complex genotype of the hepatitis B virus X /precore regions as a novel predictive marker for hepatocellular carcinoma[J]. Cancer Sci, 2012, 103(2): 296-304.
(本文编辑:周万青,刘群)
10.13602/j.cnki.jcls.2017.07.11
国家自然科学基金(81572067,81672101);福建省自然科学基金(2015J01385)。
刘灿,1979 年生,男,博士研究生,主要事乙型肝炎病毒诊断与疗效监测研究。
欧启水,主任技师,教授,博士研究生导师,E-mail:ouqishui@163.com。
R446.6
A
2016-12-20)

