玉米不同组织器官谷氨酰胺合成酶同工酶表达差异及聚合方式
王小纯张浩然韦一昊贾喜婷谷明鑫马新明,*
1河南农业大学河南粮食作物协同创新中心, 河南郑州 450002;2河南农业大学省部共建小麦玉米作物学国家重点实验室, 河南郑州450002;3河南农业大学生命科学学院生物化学系, 河南郑州 450002
玉米不同组织器官谷氨酰胺合成酶同工酶表达差异及聚合方式
王小纯1,2,3张浩然3韦一昊1贾喜婷3谷明鑫3马新明1,*
1河南农业大学河南粮食作物协同创新中心, 河南郑州 450002;2河南农业大学省部共建小麦玉米作物学国家重点实验室, 河南郑州450002;3河南农业大学生命科学学院生物化学系, 河南郑州 450002
谷氨酰胺合成酶(GS)是作物氮同化及转移利用的关键酶, 本试验研究了玉米灌浆期不同组织器官的GS同工酶表达特性, 鉴定了玉米GS同工酶的聚合方式。Western blot结果表明, 玉米不同组织器官的GS同工酶亚基表达存在明显差异, 分子量约40 kD的GS1亚基在所有组织中均大量表达, 39 kD的GS1亚基仅在穗位节及穗柄中大量表达, 分子量约44 kD的GS2亚基在叶片等光合组织中微量表达。通过改进BNE技术, 结合胶内转移酶活性的测定, 分析了玉米GS同工酶全酶的大小; 利用2-D胶结合Western blot鉴定了GS同工酶相应的亚基组成。结果表明, 在玉米组织鉴定出3种分子量不同的GS同工酶, GS2全酶分子量约460 kD, 为十聚体; GS1全酶有2种聚合状态, 一种是分子量约410 kD的十聚体, 另一种是分子量约240 kD的五聚体形式, 可见玉米GS同工酶表达存在多种方式。
玉米; 谷氨酰胺合成酶(GS); 表达; BNE; 聚合方式
氮是玉米生长发育必须的大量矿质营养元素, 也是玉米产量的一个主要限制因素。在高等植物中, 谷氨酰胺合成酶(GS)/谷氨酸合酶(GOGAT)循环是氮素同化的主要途径, 是无机氮转化为有机氮的枢纽[1], GS是GS/GOGAT循环中的关键酶, 因此, GS同工酶表达成为提高作物氮素利用率的一个研究热点。
高等植物中有2种GS同工酶, 定位于细胞液的GS1和质体的GS2。研究表明GS同工酶的表达受组织器官、生长发育、新陈代谢及环境因素等的调控[2-4]。GS1主要参与蛋白质等含氮有机化合物降解产生氨的再同化及转移利用[1], GS2主要参与光呼吸和硝酸盐还原产生的氨的同化[5]。高等植物GS2亚基较大(42~45 kD), 由单一核基因编码, 而GS1亚基较小(38~40 kD), 由2~5个核基因编码[6-12]。玉米GS2亚基约44 kD, 由单一核基因编码, GS1亚基39~40 kD, 由5个核基因编码[13]。
植物GS必须组装成聚合体才具有催化活性, 早期电子显微镜研究表明大豆GS1全酶是八聚体, 由2个平面环组成, 每个平面环由 4个亚基组成[14]。Llorca等[10]利用X-ray晶体技术研究菜豆重组 GS1结构, 发现其与大豆GS1全酶结构相同, 即由两个4元环聚合体组成的八聚体;用分析离心机测定菜豆GS1全酶相对分子质量为344 kD,与晶体研究结果一致。X-ray晶体分析表明玉米GS1、苜蓿 GS1全酶是由 2个五元环聚合体组成的十聚体[15-17],但没有关于GS2全酶结构的报道。
利用凝胶过滤及分析离心机测定纯化GS全酶的分子量, 也可以初步判断 GS的聚合状态, 但是所用材料多,费时长, 仪器设备昂贵, 而且分辨率低。近年来 Blue native PAGE (BNE)快速发展[18-19], 利用凝胶电泳对蛋白质复合体依据其大小进行分离, 具有样品需求少、分辨率高、简便快捷且保持蛋白质聚合状态等特点, 越来越多地应用于活性蛋白的低聚物状态研究[20-22]。本研究通过改良BNE, 结合蛋白质免疫印迹等技术, 分析了玉米不同组织部位GS的表达特性, 并快速鉴定了GS同工酶的聚合状态, 与玉米GS1晶体研究结果一致, 为简便快捷及时研究GS同工酶表达调控方式及其与玉米氮素利用的关系提供了技术保障。
1 材料与方法
1.1 材料
将玉米品种豫单916种植于河南农业大学农场(郑州),常规田间管理, 于灌浆期选取根(地下节根和地上气生根)及不同叶位的叶片、叶脉、叶鞘、节、节间、穗柄、苞叶和籽粒, 快速清洗、剪碎, 于液氮中速冻, 置-80℃保存备用。
1.2 粗酶液的制备
称取0.5 g不同组织部位的样品, 加液氮研磨, 再加3倍体积的提取缓冲液(100 mmol L–1Tris-HCl, pH 7.6, 1 mmol L–1EDTA, 1 mmol L–1MgCl2和10 mmol L–1β-巯基乙醇[13,22])混成匀浆, 冰浴静置30 min后, 4℃、13 000×g离心30 min, 上清液即粗酶提取液。
1.3 电泳分离鉴定GS同工酶
利用3种凝胶电泳系统分离鉴定GS同工酶。
1.3.1 不连续活性聚丙烯酰胺凝胶电泳系统(Native-PAGE) 由3%的浓缩胶(pH 6.7)和5%的分离胶(pH 8.7)组成, 用于玉米不同组织器官的蛋白提取液中GS同工酶亚型的分离和酶活性检测[22]。4℃预冷电极缓冲液(25 mmol L–1Tris, 192 mmol L–1甘氨酸), 粗酶液与5×上样缓冲液[25 mmol L–1Tris-HCl, pH 7.6, 5% (w/v) β-巯基乙醇, 0.05% (w/v)溴酚蓝, 50% (v/v)甘油]混匀后上样, 于4°C条件下电泳, 浓缩胶稳压80V, 分离胶稳压120 V。
1.3.2 Blue native PAGE (BNE) 依据Wittig等[18]改良BNE电泳方案, 根据GS同工酶全酶大小进行分离并鉴定其相对分子质量。凝胶由3%的浓缩胶和4%~13%的梯度分离胶组成, 样品处理同 Native-PAGE, 加入预冷阳极缓冲液(25 mmol L–1咪唑-HCl, pH 7.0), 首先使用阴极电泳缓冲液 A (0.02% Coomassie Blue G250, 50 mmol L–1Tricine-HCl, 5 mmol L–1咪唑, pH 7.0), 于4°C、100 V电泳20min后, 更换为阴极电泳缓冲液 B (50 mmol L–1Tricine-HCl, 5 mmol L–1咪唑, pH 7.0), 继续电泳至样品进入 4%~13%的梯度分离胶, 然后进行稳流(15 mA)电泳至蓝色指示剂出胶[22]。
1.3.3 Clear native PAGE (CNE) 参考Wittig等[19], 凝胶体系和 BNE一样, 但只使用阴极电泳缓冲液 B, 于100V电泳至样品进入分离胶, 然后进行稳流(15 mA)电泳至蓝色指示剂出胶。
胶内GS活性依据Wang等[22]方法检测, 活性染色结束后扫描结果, 然后再用考马斯亮蓝R250染色。BNE胶中, 以高分子蛋白 Marker (Amersham HMW Calibration Kit For Native Electrophoresis)为标准, 利用Gel-Pro analyzer计算GS同工酶全酶的相对分子质量。
1.4 Western blot检测GS同工酶亚基
取适量粗酶提取液与等体积 2×的上样缓冲液[100 mmol L–1Tris-HCl, pH 6.8, 4% (w/v) SDS, 10% (v/v) β-巯基乙醇, 0.2% (w/v)溴酚蓝, 20% (v/v)甘油]混合, 沸水浴5min变性处理, 室温条件下利用SDS-PAGE (5%浓缩胶, 12%分离胶)分离蛋白质, 将蛋白转移至 PVDF膜上进行Western-blot检测。使用本实验室制备的小麦 GS多克隆抗体检测玉米GS亚基, 使用Bio-Rad Clarity Western ECL试剂盒显色, 以蛋白Marker (Thermo Scientific PageRuler Prestained Protein Ladder)为标准, 利用 Gel-Pro analyzer计算GS同工酶亚基分子质量。
1.5 GS同工酶亚基组成的鉴定
切取BNE胶条, 浸泡于含1% SDS和1% β-巯基乙醇的变性液中, 37℃变性处理 2 h[18], 然后用去离子水冲洗凝胶 3~5次, 置 SDS-PAGE浓缩胶顶部, 室温条件下电泳。电泳结束后, 将蛋白质转移至PVDF膜上进行Western blot分析。
2 结果与分析
2.1 玉米GS同工酶表达特点
Western blot结果显示玉米中有3种GS亚基, 即40 kD和 39 kD的胞液型 GS (GS1)及 44 kD的质体型 GS (GS2), 且不同组织器官中 GS同工酶表达差异较大(图1-A)。40 kD的GS1在玉米中起主导作用, 不同组织器官均有较大的表达量(籽粒除外); 39 kD的GS1仅在穗位节间、节和果穗柄中表达, 且后2个部位表达量较高, 可能与玉米灌浆期氮素营养的运输有关; GS2只在功能叶的叶片、叶脉中少量表达, 可能与玉米是C4植物, 光呼吸强度低有关。Native-PAGE结合胶内 GS酶活性分析显示(图 1-B), 玉米根系、叶片、茎节、穗柄等组织中均检测到GS活性带, 叶片中最高, 籽粒中最低; 但叶片提取液中仅检测到 1条 GS活性带, 可能因 GS2活性太低或不连续Native-PAGE分离蛋白质的特点导致的2种GS同工酶没有被分离开; 此外与 Western blot结果一致, 在含两种GS1亚基的穗位节和果穗柄中检测到2个GS同工酶。
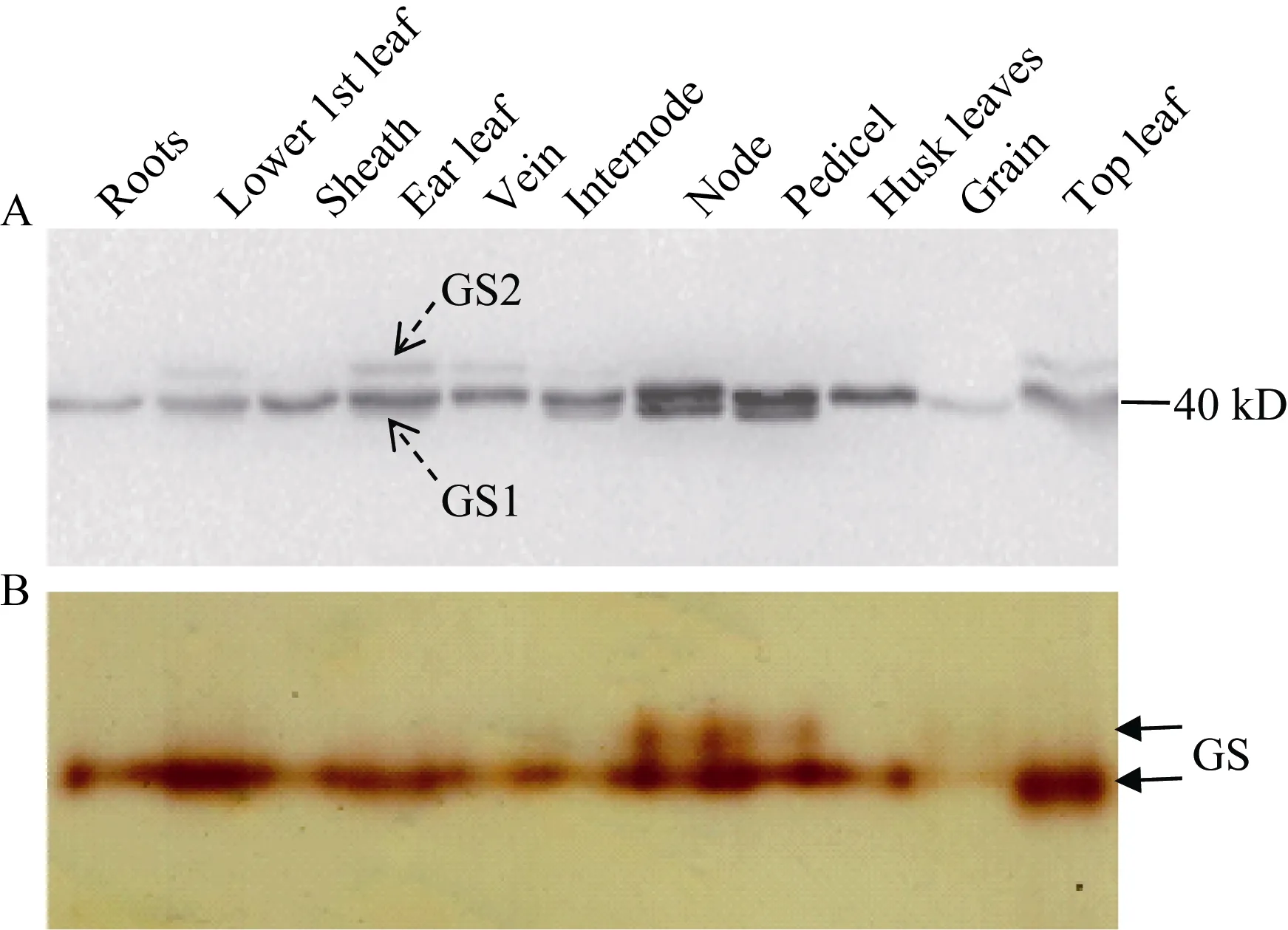
图1 玉米不同组织GS同工酶表达和活性分析Fig. 1 Analysis of the expression and activity of GS isoforms from different tissues of maize
2.2 玉米GS同工酶大小鉴定
通过改良 BNE方法, 在玉米叶片和穗位节间、节和果穗柄中检测到2个大小不同的GS活性带, 其他组织中则只检测到一条GS带(图2-A)。其中, 迁移率较大的GS同工酶全酶分子量约 240 kD, 活性较低; 迁移率较小的GS同工酶分子量约410 kD, 活性非常高。利用BNE分离玉米叶片总蛋白后, 进行 Western blot, 检测到一个相对分子质量约460 kD的GS同工酶(图2-C), 可能因其活性低, 且分子量接近RuBP羧化酶, 其活性被羧化酶的蓝色条带遮盖[22]。
2.3 GS同工酶及聚合状态鉴定
第一相胶为 BNE, 用以分离玉米组织蛋白复合体,切取相应组织的泳道进行蛋白质变性处理; 第二相胶为SDS-PAGE, 对 BNE上的蛋白质复合体的亚基进行电泳分离; 然后转膜, 利用Western blot鉴定玉米组织中GS同工酶的亚基组成。玉米叶片中分子量约460 kD的GS同工酶(图2-A)由44 kD的亚基组成且信号非常弱(图3-A),为GS2; 分子量约410 kD和240 kD的GS同工酶由39~40 kD的亚基组成且信号非常强, 为GS1, 表明胞液型GS存在两种不同的聚合方式。玉米穗柄中只有分子量约410 kD和240 kD的GS同工酶, 也是由39~40 kD的亚基组成, 说明穗柄中GS1全酶也存在两种不同的聚合方式。
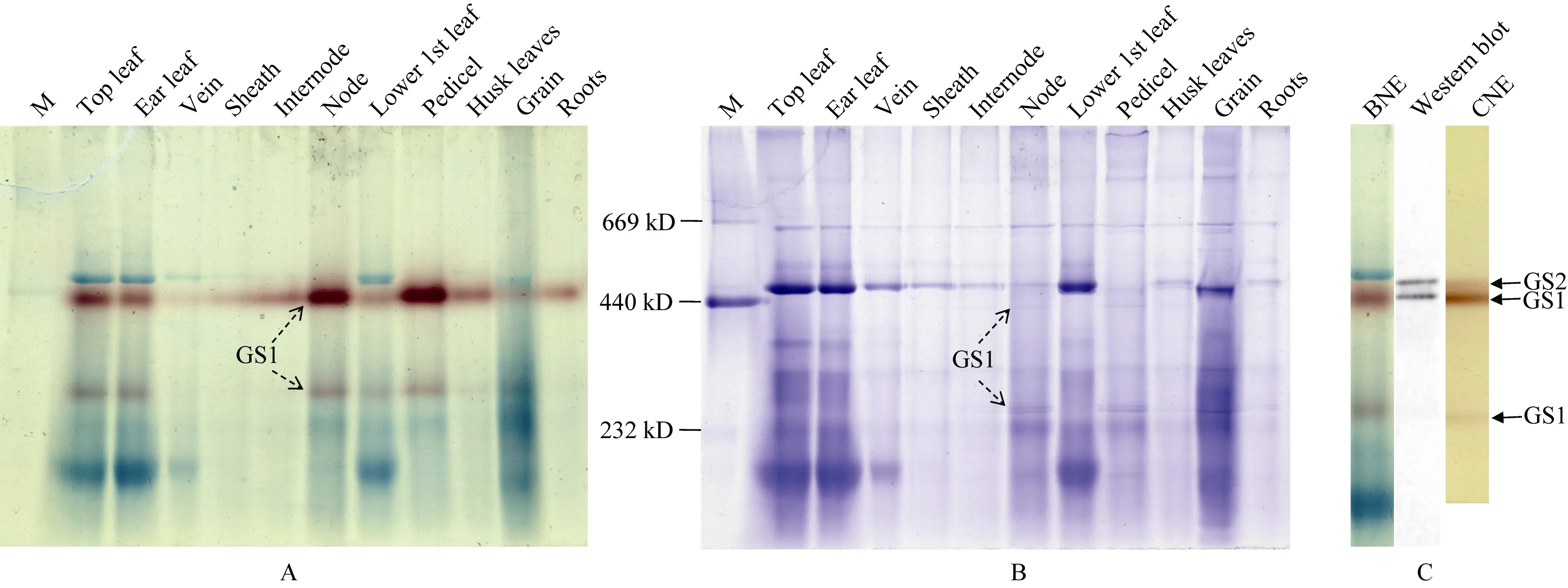
图2 玉米GS同工酶全酶大小鉴定Fig. 2 Identification of the molecular weight of GS holoenzymes
利用BNE计算GS同工酶的全酶大小(图2), 利用BNE与SDS-PAGE结合进行两相电泳分离GS同工酶亚基, 利用Western blot检测鉴定GS同工酶亚基组成(图3)。利用 GS同工酶分子量除以相应亚基的分子量, 在此基础上计算 GS同工酶聚合状态, GS2为十聚体, 胞液型GS1有2种聚合方式, 一种为十聚体, 和前人结果一致[15]; 此外, GS1同工酶还存在另外一种聚合状态,即五聚体。

图3 玉米GS同工酶亚基组成的鉴定Fig. 3 Identification of the subunit composition of GS isoenzymes in maize
3 讨论
高等植物2种GS同工酶分别定位于细胞液(GS1)和质体(GS2)[2,23]。GS2缺失在正常条件下是致死性突变; 但通过抑制光呼吸, GS2突变体能够正常生长发育, 证明GS2参与光呼吸过程中的氨同化[2]。C3植物和 C4植物光呼吸强度差异巨大[24], 小麦、水稻等 C3植物光呼吸强度很高, GS2表达量更丰富[25], 而玉米等C4植物光呼吸强度要小得多, 本研究结果显示仅在玉米叶片中检测到 GS2少量表达, 远远低于GS1表达量, 与 C4植物光呼吸弱的生理现象一致。
玉米GS1基因由5个核基因编码, 其表达因组织、叶龄及氮量而异, Gln1-3和 Gln1-4在叶片表达量高[26], Gln1-3决定穗粒数, Gln1-4调控粒重[13]。本研究发现玉米的穗位节、节间和穗柄组织中有2种高表达GS1亚基, 其中39 kD的GS1亚基是组织特异表达。推测39 kD的GS1可能在玉米灌浆期氮素营养转运方面起着重要作用。
由于从植物组织分离纯化足量蛋白的难度非常大, 因而,通常采用异源重组植物 GS研究其结构和聚合方式[10,15-17]。BNE/CNE电泳技术用于分离膜蛋白等蛋白质复合物、测定天然蛋白复合体的相对分子质量, 且具有极高的分辨率[18-19]。近年来广泛运用于植物可溶性蛋白的分离鉴定[20-22]。本研究通过改良BNE电泳技术分离玉米可溶性蛋白, 结合胶内活性测定、2-D胶和Western blot技术, 首次发现玉米叶片有3种分子量不同的GS同工酶, GS2为十聚体, GS1主要是十聚体, 与Unno等[15]利用晶体学X-ray分析的玉米GS1a是由2个五聚环组成的十聚体的结果一致;此外, GS1还存在少量的五聚体, 推测玉米 GS1的聚合状态也是一种在氮素代谢中广泛存在的 GS同工酶调控方式。
[1] Bernard S M, Habash D Z. The importance of cytosolic glutamine synthetase in nitrogen assimilation and recycling. New Phytol, 2009, 182: 608–620
[2] McNally S F, Hirel B, Gadal P, Mann A F, Stewart G R. Glutamine synthetases of higher plants: evidence for a specific isoform content related to their possible physiological role and their compartmentation within the leaf. Plant Physiol, 1983, 72: 22–25
[3] Hirel B, Gadal P. Glutamine synthetase isoforms in pea leaves: intracellular localization. Zeitschrift Fü Pflanzenphysiologie, 1981, 102: 315–319
[4] Hirel B, Lea P J. Ammonia Assimilation. Plant Nitrogen. Springer Berlin Heidelberg, 2001. pp 79–99
[5] Zozaya-Hinchliffe M, Potenza C, Ortega J L, Sengupta-Gopalan C. Nitrogen and metabolic regulation of the expression of plastidic glutamine synthetase in alfalfa (Medicago sativa). Plant Sci, 2005, 168: 1041–1052
[6] Cren M, Hirel B. Glutamine synthetase in higher plants regulation of gene and protein expression from the organ to the cell. Plant Cell Physiol, 1999, 40: 1187–1193
[7] Robert F M, Wong P P. Isozymes of glutamine synthetase in Phaseolus vulgaris L. and Phaseolus lunatus L. root nodules. Plant Physiol, 1986, 81: 142–148
[8] Carvalho H, Pereira S, Sunkel C, Salema R. Detection of a cytosolic glutamine synthetase in leaves of Nicotiana tabacum L. by immunocytochemical methods. Plant Physiol, 1992, 100: 1591–1594
[9] Ishiyama K, Inoue E, Watanabe-Takahashi A, Obara M, Yamaya T, Takahashi H. Kinetic properties and ammonium-dependent regulation of cytosolic isoenzymes of glutamine synthetase in arabidopsis. J Biol Chem, 2004, 279: 16598–16605
[10] Llorca O, Betti M, González J M, Valencia A, Márquez A J, Valpuesta J M. The three-dimensional structure of an eukaryoticglutamine synthetase: functional implications of its oligomeric structure. J Structural Biol, 2006, 156: 469–479
[11] Seabra A R, Vieira C P, Cullimore J V, Carvalho H G. Medicago truncatula contains a second gene encoding a plastid located glutamine synthetase exclusively expressed in developing seeds. BMC Plant Biol, 2010, 10: 183
[12] Singh K K, Ghosh S. Regulation of glutamine synthetase isoforms in two differentially drought-tolerant rice (Oryza sativa L.) cultivars under water deficit conditions. Plant Cell Rep, 2013, 32: 183–193
[13] Martin A, Lee J, Kichey T, Gerentes D, Zivy M, Tatout C, Dubois F, Balliau T, Valot B, Davanture M, Tercé-Laforgue T, Quilleré I, Coque M, Gallais A, Gonzalez-Moro M B, Bethencourt L, Habash D Z, Lea P J, Charcosset A, Perez P, Murigneux A, Sakakibara H, Edwards K J, Hirel B. Two cytosolic glutamine synthetase isoforms of maize are specifically involved in the control of grain production. Plant Cell, 2006, 18: 3252–3274
[14] McParland R H, Guevara J G, Becker R R, Evans H J. The purification and properties of the glutamine synthetase from the cytosol of soya-bean root nodules. Biochem J, 1976, 153: 597–606
[15] Unno H, Uchida T, Sugawara H, Kurisu G, Sugiyama T, Yamaya T, Sakakibara H, Hase T, Kusunoki M. Atomic structure of plant glutamine synthetase: a key enzyme for plant productivity. J Biol Chem, 2006, 281: 29287–29296
[16] Seabra A R, Carvalho H, Pereira P J. Crystallization and preliminary crystallographic characterization of glutamine synthetase from Medicago truncatula. Acta Crystallographica, 2009, 65: 1309–1312
[17] Torreira E, Seabra A R, Marriott H, Zhou M, Llorca Ó, Robinson C V, Carvalho H G, Fernández-Tornero C, Pereira P J. The structures of cytosolic and plastid-located glutamine synthetases from Medicago truncatula reveal a common and dynamic architecture. Acta Crystallographica, 2014, 70: 981–993
[18] Wittig I, Braun H P, Schägger H. Blue native PAGE. Nat Protocols, 2006, 1: 418–428
[19] Wittig I, Karas M, Schägger H. High resolution clear native electrophoresis for in-gel functional assays and fluorescence studies of membrane protein complexes. Mol Cell Proeom, 2007, 6: 1215–1225
[20] Kimata-Ariga Y, Hase T. Multiple complexes of nitrogen assimilatory enzymes in spinach chloroplasts: possible mechanisms for the regulation of enzyme function. PLoS One, 2014, 9(10): e108965
[21] Seabra A R, Silva L S, Carvalho H G. Novel aspects of glutamine synthetase (GS) regulation revealed by a detailed expression analysis of the entire GS gene family of Medicago truncatula under different physiological conditions. BMC Plant Biol, 2013, 13: 137–142
[22] Wang X C, Wei Y H, Shi L X, Ma X M, Theg S M. New isoforms and assembly of glutamine synthetase in the leaf of wheat (Triticum aestivum L.). J Exp Bot, 2015, 66: 6827–6834
[23] Mann A F, Fentem P A, Stewart G R. Identification of two forms of glutamine synthetase in barley (Hordeum vulgare). Biochem Biophys Res Commun, 1979, 88: 515–521
[24] 郭玉朋. 植物光呼吸途径研究进展. 草业学报, 2014, 23: 322–329
Guo Y P. A study on advances in plant photorespiration. Acta Pratac Sin, 2014, 23: 322–329 (in Chinese with English abstract)
[25] Bernard S M, Møller A L, Dionisio G, Kichey T, Jahn T P, Dubois F, Baudo M, Lopes M S, Tercé-Laforgue T, Foyer C H, Parry M A, Forde B G, Araus J L, Hirel B, Schjoerring J K, Habash D Z. Gene expression, cellular localisation and function of glutamine synthetase isozymes in wheat (Triticum aestivum L.). Plant Mol Biol, 2008, 67: 89–105
[26] Hirel B, Andrieu B, Valadier M H, Renarda S, Quilleré I, Chelleb M, Pommel B, Fournier C, Drouetb J L. Physiology of maize: II. identification of physiological markers representative of the nitrogen status of maize (Zea mays) leaves during grain filling. Physiol Plant, 2005, 124: 178–188
Differential Expression and Assembly Mode of Glutamine Synthetase Isoenzymes in Different Tissues and Organs of Maize
WANG Xiao-Chun1,2,3, ZHANG Hao-Ran3, WEI Yi-Hao1, JIA Xi-Ting3, GU Ming-Xin3, and MA Xin-Ming1,*
1Collaborative Innovation Center of Henan Grain Crops / Henan Agricultural University, Zhengzhou 450002, China;2State Key Laboratory of Wheat and Maize Crop Science in China / Henan Agricultural University, Zhengzhou 450002, China;3Department of Biochemistry, College of Life Science / Henan Agricultural University, Zhengzhou 450002, China
Glutamine synthetase (GS) is a key enzyme in nitrogen assimilation and recycling in cereals. In this study, the expression characteristics of GS isoenzymes in different tissues and organs of maize in grain-filling period were analyzed, and the assembly of GS isoenzymes were indentified. The GS isoforms expressed differentially in different organs were shown by Western-blot obviously; GS1 with a molecular weight of about 40 kD expressed highly in all tissues, and GS1 with a molecular weight of about 39 kD was merely expressed in the node of ear position and pedical, and GS2 with a molecular weight of about 44 kD was weakly expressed in the photosynthtic tissue such as leaf. With a modified blue naive PAGE (BNE) technique and in-gel activity analysis, the size of GS holoenzyme was calibrated; combined the 2-D gel with western-blot analysis, the subunits composition of GS isoenzymes were identified. Three GS isoenzymes with different sizes were identified in maize. GS2 holoenzyme was about 460 kD and likely a decamer, GS1 holoenzyme existed two kinds of assembly state, one was about 410 kD and likely a decamer, another was about 240 kD and more likely a pentamer; therefore, the expression of GS isoenzymes exists diversity in maize.
Maize; Glutamine synthetase (GS); Expression; Blue native PAGE (BNE); Assembly
(
): 2016-12-21; Accepted(接受日期): 2017-04-20; Published online(网络出版日期): 2017-05-08.
10.3724/SP.J.1006.2017.01410
本研究由国家重点研发计划项目(2016YFD0300205)和小麦玉米作物学国家重点实验室(39990047)资助。
The study was supported by the National Key Research and Development Program of China (2016YFD0300205) and State Key Laboratory of Wheat and Maize Crop Science (39990047).
*通讯作者(Corresponding author): 马新明, E-mail: xinmingma@126.com, Tel: 13937100780
联系方式: E-mail: xiaochun.w@163.com, Tel: 13783586761
URL: http://kns.cnki.net/kcms/detail/11.1809.S.20170508.1007.010.html

