肾透明细胞癌与表观遗传学
郭倚天,陈明
(1.东南大学 医学院,江苏 南京 210009; 2.东南大学 附属中大医院,江苏 南京 210009)
肾透明细胞癌与表观遗传学
郭倚天1,陈明2
(1.东南大学 医学院,江苏 南京 210009; 2.东南大学 附属中大医院,江苏 南京 210009)
肾透明细胞癌是常见的具有基因及表观遗传学变异的泌尿系统恶性肿瘤。本文作者综述了肾透明细胞癌表观遗传学特征包括DNA甲基化、组蛋白修饰、微小RNA和最近发现的长链非编码RNA的研究成果。这些表观遗传学特点可以用于肾透明细胞癌的早期诊断、患者预后评估及靶向治疗。随着高通量测序的广泛应用,肾透明细胞癌的表观遗传学变化可以获得更精准的确定,并为其研究与治疗提供新的导向。
DNA甲基化; 组蛋白修饰; 非编码RNA; 肾透明细胞癌; 综述
美国2016年预计约有62 700例肾癌患者,且约有14 240例肾癌患者死亡[1]。肾癌最常见的类型是肾细胞癌(renal cell carcinoma, RCC),70%~80%RCC诊断为肾透明细胞癌(clear cell renal cell carcinoma, ccRCC)。RCC近年来在中国的发病率也呈现明显上升趋势[2]。
1 DNA甲基化和ccRCC
1.1 DNA甲基化和ccRCC的发生
DNA甲基化包括启动子区内CpG岛的超甲基化及基因主体的甲基化。这些因素可导致抑癌基因失活及原癌基因激活。家族性和近70%的散发性ccRCC是由体细胞突变、杂合性缺失或启动子超甲基化导致的Von Hippel- Lindau(VHL)基因表达异常导致的[4]。VHL异常可导致缺氧诱导因子(hypoxia- inducible factors, HIFs)的积累并促进ccRCC的血管生成及发生[5- 6]。ccRCC中抑癌基因的失活主要是因启动子甲基化而非基因突变,即使VHL的失活只表现在约15%的散发性ccRCC中。研究发现RASSF1、PITX2、CDH13、hs3st2、TWIST1、TAL1、TUSC3位点在VHL野生型散发ccRCC中比家族性ccRCC表现出更频繁的甲基化[7],可能表明DNA甲基化与ccRCC的发生相关。
许多基因包括CDH1、APAF1、COL1A1、DKK2、DKK3、SFRP1、SFRP4、SFRP5、WIF、PCDH17、TCF21在ccRCC中常出现甲基化(50%),而在配对的正常肾组织中呈无甲基化或较少的甲基化(<10%)[8- 10],这些基因参与了肿瘤的信号转导、凋亡、血管生成、黏附和侵袭。最近一项比较38例RCC和9例正常肾组织中CpG岛甲基化的研究显示,55个基因仅在ccRCC呈甲基化。进一步的功能研究揭示了8个新的潜在抑癌基因,包括OVOL1、DLEC1、BMP4、SST、TMPRSS2、TM6SF1、SLC34A2和COL1A2。其中,OVOL1的表观遗传沉默可促进原癌基因c- myc的表达[11]。
对5- 氮- 2- 脱氧胞苷处理过的ccRCC进行高密度微阵列基因表达分析显示,一些原发肿瘤中的基因在去甲基化后重新表达,BNC1、COL14A1、CST6、Pdlim4、SFRP1基因都表现出频繁(>30%)的启动子区的甲基化并与转录沉默相关,体外试验[10]证明了其抑癌作用。通过甲基化DNA免疫共沉淀结合全基因组表达谱芯片分析ccRCC基因组甲基化模式[12]显示,ATP5G2、PCDH8、CORO6、KLHL35、QPCT、SCUBE3、ZSCAN18、CCDC8和FBN2基因在ccRCC中表现频繁的基因甲基化和启动子甲基化,导致它们的表达水平明显降低。
一些异常的超甲基化的区域包括AP2a、AHR、HAIRY、ARNT和HIF1转录因子结合位点与RCC组织缺氧信号通路相关[13]。
1.2 DNA甲基化与ccRCC的预后
具有阳性GpG岛甲基子表型的FAM150A、GRM6、ZNF540、ZFP42、ZNF154、RIMS4、PCDHAC1、KHDRBS2、ASCL2、KCNQ1、PRAC、WNT3A、TRH、FAM78A、ZNF671、SLC13A5和NKX6- 2基因的ccRCC,呈现更差的预后及更强的肿瘤侵袭性[14]。抑癌/癌基因增强子的甲基化对患者的生存也具有较好的预测作用[13]。
一般来说,村民由于环境和有关条件所限,在眼界、市场意识、环境意识等方面存在一定局限性,在机遇面前容易怀疑和观望,容易被动接受和跟风,在村民眼里,能够获得利益、自己能够得到好处才是他们是否去做一件事情的判断标准。闲置农宅旅游开发对村民来说是新事物,开发过程中会存在困难和风险,村干部则是这个过程中的标杆和核心人物,起带头作用,村干部说的话做的事对村民更有说服力,村干部做好了,村干部受益了,才会对村民产生示范效应,村民才会更多地相信合作社、加入农宅合作社。
1.3 DNA甲基化与ccRCC的分子诊断
一些甲基化的DNA启动子存在于血清、外周血和尿中,提示了血液及体液中甲基化DNA在早期无创诊断ccRCC的可能性[8,15]。例如,RASSF1A(RAS相关区域家族成员1A)甲基化的启动子在ccRCC患者血清中的表达[16]以及甲基化的KILLIN(一种新的p53调控的靠近PTEN的抑癌基因)和LINE- 1在外周血中的含量[17- 18],明显高于良性肿瘤患者和健康对照组。
2 ccRCC中的组蛋白修饰
染色质是DNA、组蛋白和非组蛋白凝聚成的一种高度复杂的核蛋白。N- 末端特定残端的组蛋白修饰包括甲基化、乙酰化、磷酸化、泛素化和类泛素化。一般乙酰化导致转录激活,并与更开放的染色质构象有关,而其甲基化的转录作用取决于受影响的残端以及甲基化程度。
组蛋白脱乙酰酶(histone deacetylase, HDACs)、组蛋白乙酰转移酶(histone acetyltransferases, HATs)、组蛋白甲基化酶(histone methyltransferases, HMTs)和最近发现的组蛋白去甲基化酶(histone demethylases, HDMTs)可调控细胞增殖、血管生成、缺氧诱导和细胞周期。这些酶在ccRCC中显著降低。逆转这些酶的表观遗传修饰作用来重新活化抑癌基因或抑制癌基因可抑制肿瘤生长、进展。
2.1 组蛋白修饰和ccRCC的发生
HIF亚型与组蛋白修饰有关酶相关。HDAC4和HIF- 1α之间的相互作用可以防止HIF- α被蛋白酶体降解[19]。JARID1C是一种编码靶向HIF的组蛋白H3K4去甲基化酶,在RCC中存在突变(3%),被认为是非VHL突变肿瘤的抑癌基因[20]。JMJD1A是缺氧情况下由HIF2α所诱导的一种HMT且在RCC中呈过表达。SETD2是ccRCC的抑癌基因,编码位于靠近VHL基因的广泛缺失(约90%)的3p位点的组蛋白H3K36甲基转移酶[21]。SETD2基因截断突变(3%~8%)与RCC肿瘤样本中VHL基因突变[22]、缺氧因子诱导和染色质重塑蛋白PBRM1表达下降以及更高的核分级有关[23]。这些结果表明,组蛋白修饰酶在VHL基因突变或缺失的RCC中发挥重要作用。
上皮间质转化(epithelial- to- mesenchymal transition, EMT)在肿瘤进展中发挥重要作用。抑制HDACs可以抑制TGF- β诱导的EMT[24],HDACs通过调节pRB和p53影响细胞周期和细胞凋亡。
果蝇zeste基因增强子同源物2(enhancer of zeste homolog 2, EZH2)可通过向组蛋白H3赖氨酸27添加3个甲基团发挥HMT作用。H3K27的三甲基化导致染色质浓缩并介导抑癌基因的表观沉默。EZH2基因可能通过抑制E- cadherin促进RCC细胞的迁移和侵袭[25]。miR- 101是EZH2基因表达的负调控因子[26]。
2.2 组蛋白修饰和ccRCC的预后
组蛋白H4乙酰化水平与ccRCC的核分级和病理分期呈负相关,而低组蛋白H3乙酰化水平与全身转移和肿瘤进展相关。此外,低水平组蛋白H3的18位赖氨酸乙酰化与RCC进展密切相关,且可独立地预测肿瘤进展并与RCC术后较差的预后相关[27]。
RCC肿瘤标本中较低水平的组蛋白H3赖氨酸4甲基化与更高的病理分期和较高的Fuhrman核分级以及更低的无进展生存期和肿瘤特异性生存率相关,因此可用于预测RCC的不良预后[28- 29]。组蛋白H3赖氨酸9甲基化水平在RCC患者中也有一定预后意义,它的甲基化水平与RCC核分级、肿瘤位置和Ki67及p53表达水平有关,是RCC预后较差的重要预测因子[27]。组蛋白H3赖氨酸27较低的甲基化水平与更高的病理分期、较高的Fuhrman分级、远处转移和血管浸润有关。此外,一个单因素分析提示无进展生存期短的患者具有较低的组蛋白H3赖氨酸27甲基化水平[30]。
3 miRNAs和ccRCC
miRNAs通过表观遗传机制调节基因表达[31]。miRNAs的5’端6至8个核苷酸是可决定3’端非翻译区与靶mRNA相互作用特异性的区域,从而导致mRNA的翻译和(或)降解的抑制[32- 33]。肿瘤中高表达的miRNAs可通过下调抑癌基因发挥癌基因的作用。
3.1 miRNAs与肿瘤的发生
miR- 17- 5p、miR- 224、miR- 200家族、miR106a/b、miR- 21、miR- 221、miR- 199a和miR- 214已明确在ccRCC的形成中起重要作用。其靶点包括VHL- HIF通路、HIF/HIF转录因子、血管内皮生长因子、血小板源性生长因子、转化生长因子、PI3K/Akt下游、Ras/Raf/MEK/ERK通路和mTOR通路。同一个miRNA可沿相同的路径控制多个靶点,多个miRNAs可有相同的靶点,即使细微的变化也可能对这些miRNAs的调控途径造成重大影响。
3.2 miRNAs与ccRCC的分子诊断
miRNAs在体液中稳定且高度丰富,从坏死或凋亡的细胞释放后进入循环。一些miRNAs包裹在外泌体内,可抵抗RNA酶并在血液中持续存在,因此血miRNAs检测可能是一种简单快速的预测肿瘤的方式[34- 35]。miR- 210是早期诊断ccRCC的潜在生物标志物,由于VHL- HIF通路的激活,同肿瘤组织中表达类似,血清miR- 210水平在RCC患者中显著升高[36]。另一项研究发现RCC患者血清中miR- 378水平增加,miR- 451水平降低。两种miRNAs综合用于RCC诊断的特异性为81%,敏感性为83%[37]。Wang等[38]发现,血清中3个显著升高的miRNAs(mir- 193a- 3p,mir- 362和miR- 572)和2个明显下降的miRNAs(mir- 28- 5p和miR- 378)可以区别Ⅰ期RCC组织及其对照组,表明这些miRNAs可被用于RCC的早期诊断。
尿miRNAs分析是泌尿系肿瘤的早期诊断的方法之一[39],但有关研究仍旧有限,尿miRNAs水平可维持稳定[40]。miR- 15a在其他肿瘤中表达量低,在ccRCC组织及患者尿液中表达量高,这种现象在其他泌尿系统肿瘤和尿路炎症中并无发现,表明miR- 15a可能是一个潜在ccRCC标志物[41]。一些miRNAs如miR- 425、miR- 136、miR- 335、miR- 340和mir- 320可能是新的标志物[42]。
3.3 miRNAs与ccRCC的预后
miRNAs的异常表达常与较差的预后相关,miR- 21高表达在多种癌症包括RCC中是较差预后的标志[43]。此外,miR- 21/10b比率是无转移ccRCC预后的一个独立指标[44]。抑癌miRNAs研究表明,miR- 497、miR- 23b- 27b集群与RCC更高的分级及更短的总生存期有关[45- 46]。miR- 210的高表达也是预后差的标志之一[47],而另一项研究却呈现相反的结果[48]。
mir- 501在RCC中呈差异表达,它的高表达预示更差的预后[49]。同样,miR- 126也呈差异表达,但其低表达与预后差相关,高表达提示RCC患者具有显著较长的无病生存期[50]。
miR- 122在ccRCC组织原发灶高表达,在转移灶低表达。mir- 514在原发灶低表达,并在转移灶进一步低表达。这些miRNAs的改变与复发风险显著相关,且mir- 514是ccRCC患者预后的独立预测因素[51]。
4 长链非编码RNA和ccRCC的发生发展
长链非编码RNA(long non- coding RNAs, lncRNAs)是长度大于200个核苷酸的非编码RNA。lncRNAs的差异表达具有组织特异性,并在细胞增殖、凋亡中发挥作用[52]。见表1。
表1 ccRCC中异常表达的lncRNAs及其靶点、预后
Tab 1 lncRNAs abnormally expressed in ccRCC and their targets and prognosis
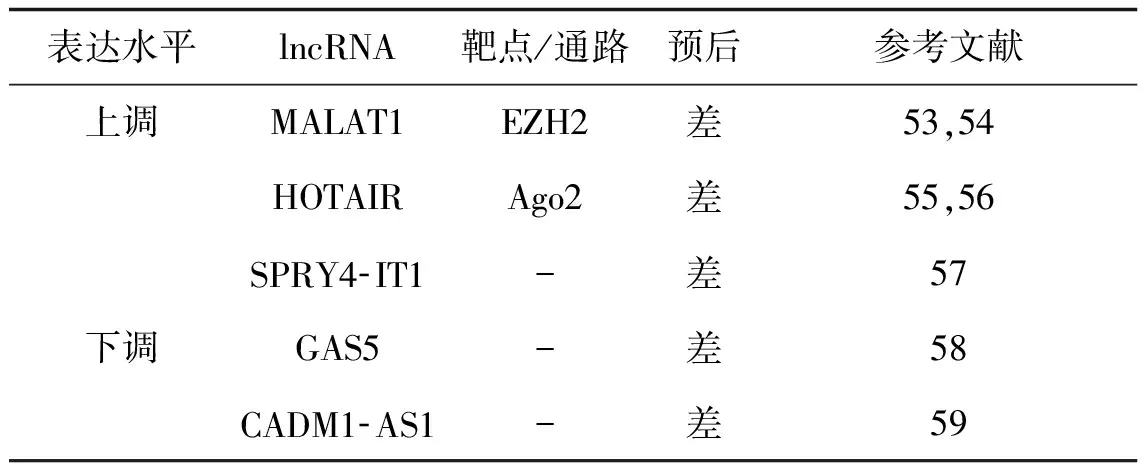
表达水平lncRNA靶点/通路预后参考文献上调MALAT1EZH2差53,54HOTAIRAgo2差55,56SPRY4⁃IT1-差57下调GAS5-差58CADM1⁃AS1-差59
5 ccRCC的表观遗传学与靶向治疗
ccRCC中靶向VEGF的药物包括贝伐单抗联合干扰素、索拉非尼、舒尼替尼、帕唑帕尼和阿西替尼。另一个ccRCC治疗靶点是mTOR通路,如应用替西罗莫司和依维莫司[60]。抑制HDACs和逆转抑癌基因启动子甲基化是一种新的治疗方法,一些新的药物可作为单药或与硼替佐米、西罗莫司、干扰素和IL- 2等联用发挥作用。此外,去甲基化药物如阿扎胞苷和地西他滨在治疗ccRCC中也有一定效果。
总之,表观遗传改变调控RCC的形成和进展的研究仍处于起步阶段。对ccRCC表观遗传学调控改变的基因及通路的进一步研究,可能会促进新的ccRCC的诊断和预后工具的发现与发展。长远来看,表观遗传学可能为RCC提供除标准治疗方案以外的全新疗法。
[1] SIEGEL R L,MILLER K D,JEMAL A.Cancer statistics,2016[J].CA Cancer J Clin,2016,66(1):7- 30.
[2] CHEN W,ZHENG R,BAADE P D,et al.Cancer statistics in China,2015[J].CA Cancer J Clin,2016,66(2):115- 132.
[3] BIRD A.DNA methylation patterns and epigenetic memory[J].Genes Dev,2002,16(1):6- 21.
[4] BAUSCH B,JILG C,GLASKER S,et al.Renal cancer in von Hippel- Lindau disease and related syndromes[J].Nat Rev Nephrol,2013,9(9):529- 538.
[5] KIM W,KAELIN W G Jr.The von Hippel- Lindau tumor suppressor protein:new insights into oxygen sensing and cancer[J].Curr Opin Genet Dev,2003,13(1):55- 60.
[6] LINEHAN W M,RUBIN J S,BOTTARO D P.VHL loss of function and its impact on oncogenic signaling networks in clear cell renal cell carcinoma[J].Int J Biochem Cell Biol,2009,41(4):753- 756.
[7] MCRONALD F E,MORRIS M R,GENTLE D,et al.CpG methylation profiling in VHL related and VHL unrelated renal cell carcinoma[J].Mol Cancer,2009,8:31.
[8] RYDZANICZ M,WRZESINSKI T,BLUYSSEN H A,et al.Genomics and epigenomics of clear cell renal cell carcinoma:recent developments and potential applications[J].Cancer Lett,2013,341(2):111- 126.
[9] COSTA V L,HENRIQUE R,DANIELSEN S A,et al.TCF21 and PCDH17 methylation:An innovative panel of biomarkers for a simultaneous detection of urological cancers[J].Epigenetics,2011,6(9):1120- 1130.
[10] MORRIS M R,MAHER E R.Epigenetics of renal cell carcinoma:the path towards new diagnostics and therapeutics[J].Genome Med,2010,2(9):59.
[11] RICKETTS C J,MORRIS M R,GENTLE D,et al.Genome- wide CpG island methylation analysis implicates novel genes in the pathogenesis of renal cell carcinoma[J].Epigenetics,2012,7(3):278- 290.
[12] MORRIS M R,RICKETTS C J,GENTLE D,et al.Genome- wide methylation analysis identifies epigenetically inactivated candidate tumour suppressor genes in renal cell carcinoma[J].Oncogene,2011,30(12):1390- 1401.
[13] HU C Y,MOHTAT D,YU Y,et al.Kidney cancer is characterized by aberrant methylation of tissue- specific enhancers that are prognostic for overall survival[J].Clin Cancer Res,2014,20(16):4349- 4360.
[14] ARAI E,CHIKU S,MORI T,et al.Single- CpG- resolution methylome analysis identifies clinicopathologically aggressive CpG island methylator phenotype clear cell renal cell carcinomas[J].Carcinogenesis,2012,33(8):1487- 1493.
[15] BALDEWIJNS M M,van VLODROP I J,SCHOUTEN L J,et al.Genetics and epigenetics of renal cell cancer[J].Biochim et Biophys Acta,2008,1785(2):133- 155.
[16] de MARTINO M,KLATTE T,HAITEL A,et al.Serum cell- free DNA in renal cell carcinoma:a diagnostic and prognostic marker[J].Cancer,2012,118(1):82- 90.
[17] BENNETT K L,CAMPBELL R,GANAPATHI S,et al.Germline and somatic DNA methylation and epigenetic regulation of KILLIN in renal cell carcinoma[J].Genes Chromosomes Cancer,2011,50(8):654- 661.
[18] LIAO L M,BRENNAN P,VAN BEMMEL D M,et al.LINE- 1 methylation levels in leukocyte DNA and risk of renal cell cancer[J].PLoS One,2011,6(11):e27361.
[19] QIAN D Z,KACHHAP S K,COLLIS S J,et al.Class II histone deacetylases are associated with VHL- independent regulation of hypoxia- inducible factor 1 alpha[J].Cancer Res,2006,66(17):8814- 8821.
[20] NIU X,ZHANG T,LIAO L,et al.The von Hippel- Lindau tumor suppressor protein regulates gene expression and tumor growth through histone demethylase JARID1C[J].Oncogene,2012,31(6):776- 786.
[21] DUNS G,van den BERG E,van DUIVENBODE I,et al.Histone methyltransferase gene SETD2 is a novel tumor suppressor gene in clear cell renal cell carcinoma[J].Cancer Res,2010,70(11):4287- 4291.
[22] DALGLIESH G L,FURGE K,GREENMAN C,et al.Systematic sequencing of renal carcinoma reveals inactivation of histone modifying genes[J].Nature,2010,463(7279):360- 363.
[23] HAKIMI A A,CHEN Y B,WREN J,et al.Clinical and pathologic impact of select chromatin- modulating tumor suppressors in clear cell renal cell carcinoma[J].Eur Urol,2013,63(5):848- 854.
[24] YOSHIKAWA M,HISHIKAWA K,MARUMO T,et al.Inhibition of histone deacetylase activity suppresses epithelial- to- mesenchymal transition induced by TGF- beta1 in human renal epithelial cells[J].J Am Soc Nephrol,2007,18(1):58- 65.
[25] LIU L,XU Z,ZHONG L,et al.Enhancer of zeste homolog 2 (EZH2) promotes tumour cell migration and invasion via epigenetic repression of E- cadherin in renal cell carcinoma[J].BJU Int,2016,117(2):351- 362.
[26] SAKURAI T,BILIM V N,UGOLKOV A V,et al.The enhancer of zeste homolog 2 (EZH2),a potential therapeutic target,is regulated by miR- 101 in renal cancer cells[J].Biochem Biophys Res Commun,2012,422(4):607- 614.
[27] SELIGSON D B,HORVATH S,MCBRIAN M A,et al.Global levels of histone modifications predict prognosis in different cancers[J].Am J Pathol,2009,174(5):1619- 1628.
[28] MOSASHVILLI D,KAHL P,MERTENS C,et al.Global histone acetylation levels:prognostic relevance in patients with renal cell carcinoma[J].Cancer Sci,2010,101(12):2664- 2669.
[29] ELLINGER J,KAHL P,MERTENS C,et al.Prognostic relevance of global histone H3 lysine 4 (H3K4) methylation in renal cell carcinoma[J].Int J Cancer,2010,127(10):2360- 2366.
[30] ROGENHOFER S,KAHL P,MERTENS C,et al.Global histone H3 lysine 27 (H3K27) methylation levels and their prognostic relevance in renal cell carcinoma[J].BJU Int,2012,109(3):459- 465.
[31] HAUSSER J,ZAVOLAN M.Identification and consequences of miRNA- target interactions- - beyond repression of gene expression[J].Nat Rev Genet,2014,15(9):599- 612.
[32] LIM L P,LAU N C,GARRETT- ENGELE P,et al.Microarray analysis shows that some microRNAs downregulate large numbers of target mRNAs[J].Nature,2005,433(7027):769- 773.
[33] LEWIS B P,SHIH I H,JONES- RHOADES M W,et al.Prediction of mammalian microRNA targets[J].Cell,2003,115(7):787- 798.
[34] ZEN K,ZHANG C Y.Circulating microRNAs:a novel class of biomarkers to diagnose and monitor human cancers[J].Med Res Rev,2012,32(2):326- 348.
[35] MITCHELL P S,PARKIN R K,KROH E M,et al.Circulating microRNAs as stable blood- based markers for cancer detection[J].Proc Natl Acad Sci U S A,2008,105(30):10513- 10518.
[36] IWAMOTO H,KANDA Y,SEJIMA T,et al.Serum miR- 210 as a potential biomarker of early clear cell renal cell carcinoma[J].Int J Oncol,2014,44(1):53- 58.
[37] REDOVA M,POPRACH A,NEKVINDOVA J,et al.Circulating miR- 378 and miR- 451 in serum are potential biomarkers for renal cell carcinoma[J].J Transl Med,2012,10:55.
[38] WANG C,HU J,LU M,et al.A panel of five serum miRNAs as a potential diagnostic tool for early- stage renal cell carcinoma[J].Sci Rep,2015,5:7610.
[39] MLCOCHOVA H,HEZOVA R,STANIK M,et al.Urine microRNAs as potential noninvasive biomarkers in urologic cancers[J].Urol Oncol,2014,32(1):41.e41- 49.
[40] YUN S J,JEONG P,KIM W T,et al.Cell- free microRNAs in urine as diagnostic and prognostic biomarkers of bladder cancer[J].Int J Oncol,2012,41(5):1871- 1878.
[41] von BRANDENSTEIN M,PANDARAKALAM J J,KROON L,et al.MicroRNA 15a,inversely correlated to PKCalpha,is a potential marker to differentiate between benign and malignant renal tumors in biopsy and urine samples[J].Am J Pathol,2012,180(5):1787- 1797.
[42] ZAMAN M S,SHAHRYARI V,DENG G,et al.Up- regulation of microRNA- 21 correlates with lower kidney cancer survival[J].PLoS One,2012,7(2):e31060.
[43] FRITZ H K,LINDGREN D,LJUNGBERG B,et al.The miR(21/10b) ratio as a prognostic marker in clear cell renal cell carcinoma[J].Eur J Cancer,2014,50(10):1758- 1765.
[44] ZHAO X,ZHAO Z,XU W,et al.Down- regulation of miR- 497 is associated with poor prognosis in renal cancer[J].Int J Clin Exp Pathol,2015,8(1):758- 764.
[45] ISHIHARA T,SEKI N,INOGUCHI S,et al.Expression of the tumor suppressive miRNA- 23b/27b cluster is a good prognostic marker in clear cell renal cell carcinoma[J].J Urol,2014,192(6):1822- 1830.
[46] SAMAAN S,KHELLA H W,GIRGIS A,et al.miR- 210 is a prognostic marker in clear cell renal cell carcinoma[J].J Mol Diagn,2015,17(2):136- 144.
[47] MANGOLINI A,BONON A,VOLINIA S,et al.Differential expression of microRNA501- 5p affects the aggressiveness of clear cell renal carcinoma[J].FEBS Open Bio,2014,4:952- 965.
[48] MCCORMICK R I,BLICK C,RAGOUSSIS J,et al.miR- 210 is a target of hypoxia- inducible factors 1 and 2 in renal cancer,regulates ISCU and correlates with good prognosis[J].Br J Cancer,2013,108(5):1133- 1142.
[49] KHELLA H W,SCORILAS A,MOZES R,et al.Low expression of miR- 126 is a prognostic marker for metastatic clear cell renal cell carcinoma[J].Am J Pathol,2015,185(3):693- 703.
[50] WOTSCHOFSKY Z,BUSCH J,JUNG M,et al.Diagnostic and prognostic potential of differentially expressed miRNAs between metastatic and non- metastatic renal cell carcinoma at the time of nephrectomy[J].Clin Chim Acta,2013,416:5- 10.
[51] MERCER T R,DINGER M E,MATTICK J S.Long non- coding RNAs:insights into functions[J].Nat Rev Genet,2009,10(3):155- 159.
[52] 郭倚天,许斌,陈明.长链非编码RNA在前列腺癌中的作用及研究进展[J].医学研究生学报,2017,30(2):199- 203.
[53] ZHANG H M,YANG F Q,CHEN S J,et al.Upregulation of long non- coding RNA MALAT1 correlates with tumor progression and poor prognosis in clear cell renal cell carcinoma[J].Tumour Biol,2015,36(4):2947- 2955.
[54] HIRATA H,HINODA Y,SHAHRYARI V,et al.Long Noncoding RNA MALAT1 Promotes Aggressive Renal Cell Carcinoma through Ezh2 and Interacts with miR- 205[J].Cancer Res,2015,75(7):1322- 1331.
[55] WU Y,LIU J,ZHENG Y,et al.Suppressed expression of long non- coding RNA HOTAIR inhibits proliferation and tumourigenicity of renal carcinoma cells[J].Tumour Biol,2014,35(12):11887- 11894.
[56] CHIYOMARU T,FUKUHARA S,SAINI S,et al.Long non- coding RNA HOTAIR is targeted and regulated by miR- 141 in human cancer cells[J].J Biol Chem,2014,289(18):12550- 12565.
[57] ZHANG H M,YANG F Q,YAN Y,et al.High expression of long non- coding RNA SPRY4- IT1 predicts poor prognosis of clear cell renal cell carcinoma[J].Int J Clin Exp Pathol,2014,7(9):5801- 5809.
[58] QIAO H P,GAO W S,HUO J X,et al.Long non- coding RNA GAS5 functions as a tumor suppressor in renal cell carcinoma[J].Asian Pac J Cancer Prev,2013,14(2):1077- 1082.
[59] YAO J,CHEN Y,WANG Y,et al.Decreased expression of a novel lncRNA CADM1- AS1 is associated with poor prognosis in patients with clear cell renal cell carcinomas[J].Int J Clin Exp Pathol,2014,7(6):2758- 2767.
[60] MATTEI J,da SILVA R D,SEHRT D,et al.Targeted therapy in metastatic renal carcinoma[J].Cancer Lett,2014,343(2):156- 160.
2016- 12- 08
2017- 04- 28
国家自然科学基金资助项目(81370849)
郭倚天(1992-),男,陕西西安人,医学硕士。E- mail:guotian.3366@163.com
陈明 E- mail:mingchenseu@126.com
郭倚天,陈明.肾透明细胞癌与表观遗传学[J].东南大学学报:医学版,2017,36(4):651- 656.
R737.11
A
1671- 6264(2017)04- 0651- 06
10.3969/j.issn.1671- 6264.2017.04.032

