荧光定量PCR解析酸性条件下丙酸氧化菌的演替
班巧英,张 瑞,张立国,李建政,梅 芝
荧光定量PCR解析酸性条件下丙酸氧化菌的演替
班巧英1*,张 瑞1,张立国1,李建政2,梅 芝1
(1.山西大学环境与资源学院,山西太原 030006;2.哈尔滨工业大学市政与环境工程学院,黑龙江哈尔滨 150090)
采用实时荧光定量PCR技术考察了UASB酸性条件下丙酸氧化菌的演替规律.结果表明,至少有3种已鉴定的丙酸氧化菌(,和)存在于UASB反应器中.在pH值为7.5~7.1时,是主要的丙酸氧化菌群,其数量为(5.0~5.8)×10316S rRNA 基因拷贝数/ng DNA,约占检测到丙酸氧化菌总数的90.6%以上. pH从7.1降到6.8导致数量显著增加,和成为优势菌群,其数量占丙酸氧化菌总数的88.9%.在pH6.5条件下,(74.8%)成为优势丙酸氧化菌群.当pH£6.0时,.和演替为优势丙酸氧化菌,表明这两种丙酸氧化菌具有较强的耐酸性.当pH£6.0时,丙酸氧化菌总数随pH降低而明显减少,这可能是厌氧反应器在酸性条件下发生丙酸积累的根本原因之一.
UASB;丙酸氧化菌;酸性条件;荧光定量PCR
厌氧生物处理技术广泛用于处理各种有机废弃物,同时回收甲烷作为清洁能源[1-3].有机物厌氧发酵可产生大量的中间代谢产物,包括丙酸[4].从热力学的角度看,丙酸降解是一个高度吸能的过程,很难自发进行.所以,当系统遭受温度、有机负荷率、毒性物质等冲击时,丙酸降解受到抑制,并在厌氧消化系统内过量积累,进而导致反应器“酸化”、运行失败[5-6].因此,强化厌氧消化系统中丙酸氧化菌群的代谢活性对于提高厌氧反应器运行效能及运行稳定性具有重要意义.
在厌氧生境中,丙酸的降解是由丙酸氧化菌来完成的,其在有机物厌氧降解过程中起着不可替代的作用[7].由于丙酸氧化菌的生长需要耗氢菌的协同作用而导致其分离培养十分困难.迄今为止,只有一些生长缓慢的丙酸氧化菌被分离和鉴定[5].已鉴定的丙酸氧化菌在分类学上属于属,属,属和属[5].它们广泛分布于厌氧颗粒污泥、沼泽、污泥消化器等厌氧生境中[8-10].厌氧反应器运行的效能与微生物群落结构密切相关.DGGE(变性梯度凝胶电泳)、FISH(荧光原位杂交)、qPCR(定量PCR)等分子生物学技术常用于解析有机物厌氧降解过程中的微生物群落结构[11].丙酸氧化菌的组成和分布受到诸多因素的影响,例如:温度、水力停留时间、pH、氢分压等[5].尽管关于中性条件下丙酸氧化菌定性和定量分析的研究已有报道[12-13].然而,酸性环境中丙酸氧化菌定量分析的相关报道仍然较少.前期研究发现和是UASB(升流式厌氧污泥床)反应器中主要的丙酸氧化菌[14].因此,本研究针对这两个属中已鉴定的丙酸氧化菌,采用实时荧光定量PCR技术分析其在不同pH值条件下的数量差异,为优化厌氧反应器运行参数、强化反应器运行效能及运行稳定性提供理论基础.
1 材料与方法
1.1 反应器运行控制
试验装置为UASB反应器,其有效容积为4L.试验废水为合成废水,丙酸为唯一碳源,并补充其他微生物生长所必须的大量、微量元素及维生素等[14].在温度(35±1)℃,水力停留时间6h,进水丙酸浓度为2000mg/L,污泥床pH7.5条件下启动UASB反应器,待运行稳定后,将UASB反应器的污泥床pH逐渐降低至7.1,6.8,6.5,6.0,5.5,5.0, 4.5,4.0.每次改变pH值均在前一次运行达到稳定时进行.每次运行达到稳定后从污泥床取样,保存至-20℃备用.
1.2 分析项目及方法
pH值和生物量采用标准方法测定[15],产气量通过湿式气体流量计(LML-1,长春汽车滤清器有限责任公司)测定,气体组成和挥发酸组成采用气相色谱仪测定(SP-6800A和SP6890,山东鲁南瑞虹化工仪器有限公司)[14].
1.3 DNA提取及qPCR

表1 qPCR中所用引物序列
称取0.15g污泥(湿重),采用DNA提取试剂盒(MO Bio Laboratories,Inc,Carlsbad,CA,USA)提取厌氧活性污泥总DNA.DNA浓度用分光光度计测定(Thermo Fisher Scientific Inc.USA).
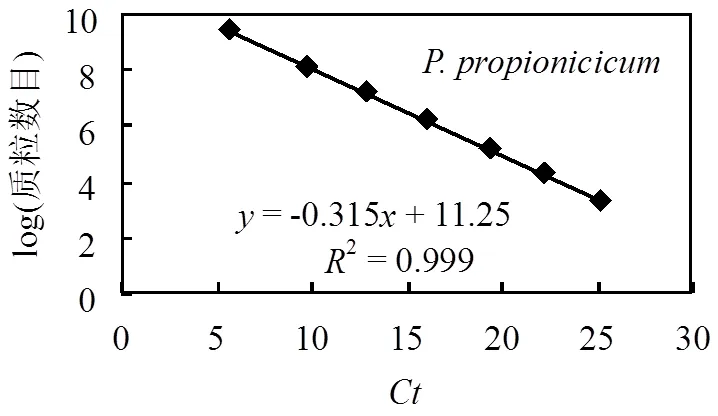
根据和中已鉴定丙酸氧化菌的16S rRNA基因序列设计特异引物,引物序列见表1[16].本研究中实时荧光定量PCR分析采用绝对定量方式.标准样品为包含目的基因片段的融合质粒,用超纯水进行10倍梯度稀释(101~107).标准样品和待测样品同时进行荧光定量PCR扩增.qPCR在荧光定量PCR系统(Model 7500,ABI,USA)中完成.所用荧光染料为SYBR green I.反应体系为:SYBR Green PCR Master Mix (Toyobo Co.,LTD.,Japan)10μL,DNA样品3μL,引物各1μL,ROX 0.04μL,4.96μL H2O.反应程序: 94℃预变性1min,94℃ 15s,58℃ 30s,72℃ 35s,40循环.每个样品设置3个重复.以达到对数增长时的循环数(值)为横坐标,标准质粒数目的对数值为纵坐标绘制标准曲线(图1).
2 结果与分析
2.1 UASB运行效能
在35℃、有机负荷率12kg COD/(m3·d)条件下,考察了pH值降低对UASB中丙酸降解的影响.结果表明,当污泥床pH值为7.5~6.8时,丙酸去除率高达93.6%以上,且大部分丙酸(81.5%~90%)是在污泥床去除的(表2).当污泥床pH值分阶段从6.8逐渐降低至4.5时,污泥床对丙酸的去除率减少了17.1%~84.9%.当pH值降低至4.0时,丙酸几乎不能分解.在整个运行期间,氢气含量低于色谱检测限,出水乙酸浓度总是低于70mg/L,比产甲烷速率为190.7~346.9LCH4/(kg CODremoved·d).
2.2 丙酸氧化菌的qPCR分析
厌氧生物处理反应器的运行效能与系统中的微生物群落结构密切相关.前期研究发现该UASB中的主要丙酸氧化菌在分类学上属于属和属[14].因此,本研究针对上述两个属中已鉴定的丙酸氧化菌,通过qPCR检测技术探讨了UASB反应器污泥床在pH7.5、pH7.1、pH6.8、pH6.5、pH6.0、pH5.5、pH5.0、pH4.5、pH4.0条件下丙酸氧化菌的数量.结果表明,有3种已鉴定丙酸氧化菌存在于所有检测的样品中,分别是、和.其中,和是两个专性互营菌,只有与产甲烷菌共培养时才能生长[5].而.除了能与产甲烷菌共培养外,还能在一些简单底物上进行纯培养[5].spp.通过甲基丙二酰-辅酶A途径(MMC)降解丙酸,而.先将两分子丙酸整合转化为1分子乙酸和1分子丁酸,然后丁酸再通过-氧化分解成2分子乙酸[5].和生长的pH 范围分别是6.5~7.5和6.3~7.8,而生长的pH范围仍然是未知的[17-19].
丙酸氧化菌的qPCR分析表明,在中性条件下(pH 7.5和pH 7.1),是UASB反应器中优势丙酸氧化菌,其含量为5.0~5.8×10316S rRNA基因拷贝数/ng DNA,约占检测到丙酸氧化菌总数的90.6%以上(图2和图3).这是由于有更高的比生长速率和丙酸利用率,在适宜的条件下其生长和代谢更快[19].类似地,在一个处理生物垃圾-丙酸合物的中温厌氧消化器中(pH7.0),spp.是优势丙酸氧化菌[20].Shigematsu等[21]的研究也发现,在一个以丙酸为唯一碳源的恒化器中,高稀释率促进spp.生长,而低稀释率有利于spp.的生长.与此相反,Worm等发现中性条件下UASB处理丙酸合成废水时,spp.长期占据优势地位.但是当反应器中长期缺乏钼、钨、硒时,spp.取代了spp.[13].另外,也有研究发现,在处理全脂奶粉,蔗糖,混合酸(丁酸、丙酸、乙酸)废水时,或是优势丙酸氧化菌[12,22].这些研究暗示了底物组成对丙酸氧化菌的组成也有一定影响.从UASB反应器中丙酸氧化菌数量上看,尽管丙酸氧化菌的数量在pH7.5~7.1时较低,但由于具有较高的代谢活性,使得丙酸去除率高达95%以上(图4和表2).
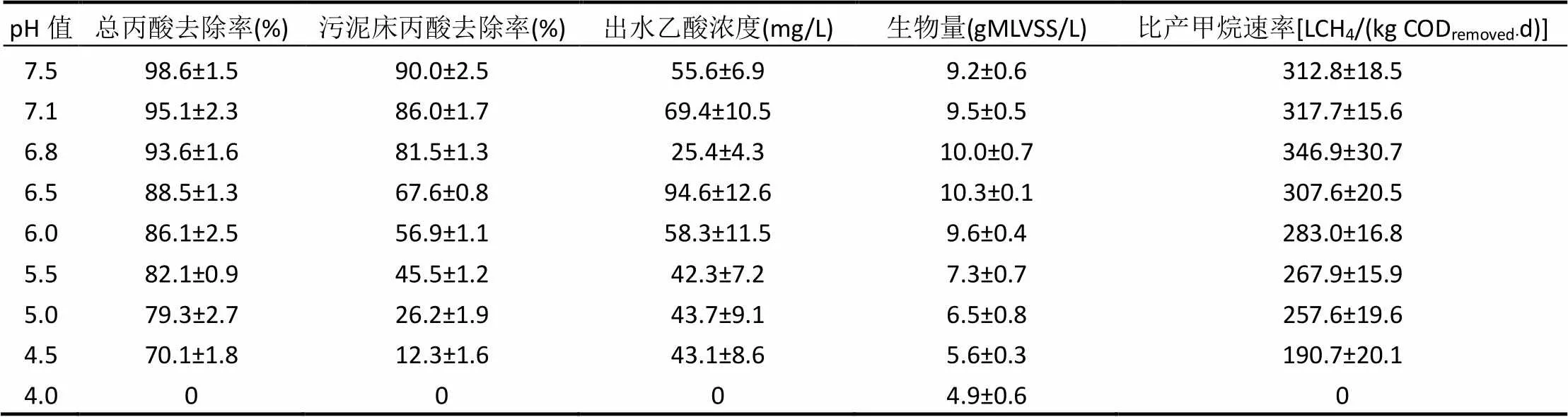
表2 不同pH条件下UASB反应器运行特征
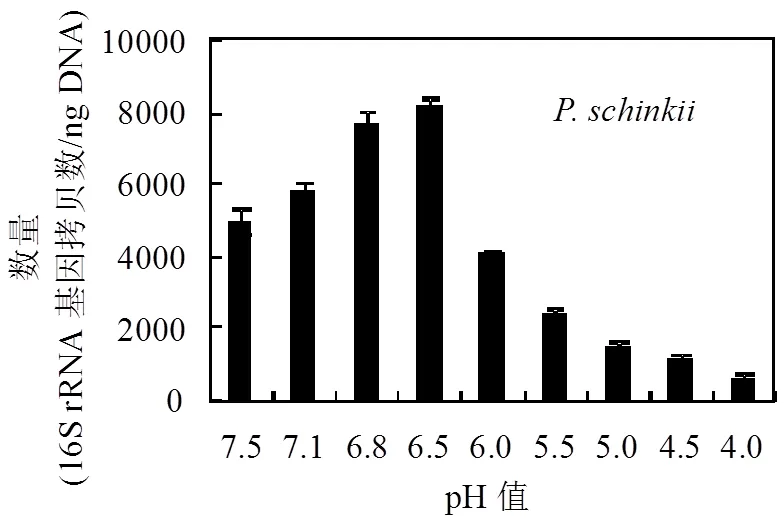
pH从7.1降低至6.8导致的数量增加了2.1倍,但是它的相对丰度却降低至38.2%.相反,的含量显著增加,占丙酸氧化菌总数的50.6%.和成为pH6.8条件下的主要丙酸降解菌群,相对丰度为88.9%.同时,UASB的生物量较之前增加了16.0%增加量,使得污泥床的丙酸去除率达到81.5%(表2).
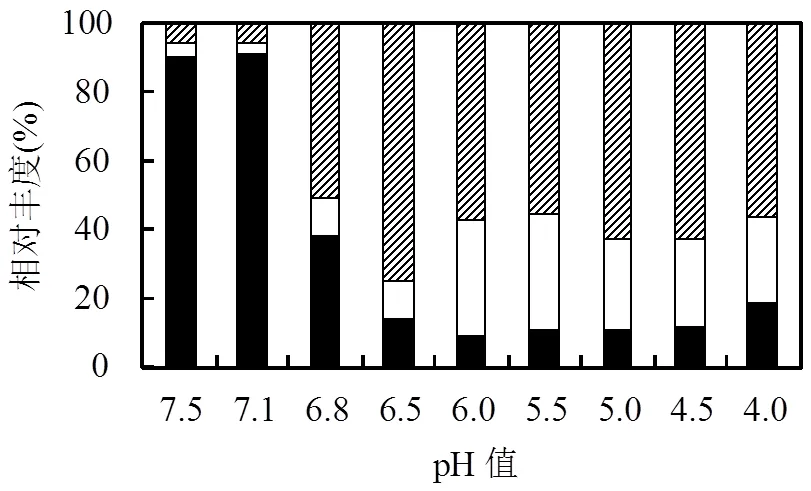
图3 不同pH条件下丙酸氧化菌的相对丰度

当pH由6.8降低至6.5时,S.propionica的数量达到最大值(4.6×10416S rRNA基因拷贝数/ng DNA),成为优势丙酸氧化菌群(图2).当pH从6.5降低至6.0,导致P.propionicum的数量增加了1倍达到最大值.相反,S.propionica的数量减少了43.9%.二者总数为4.1×104 16S rRNA基因拷贝数/ ng DNA,占丙酸氧化菌总数的90.9%,共同成为优势丙酸氧化菌群.当pH 进一步降低至5.5,5.0,4.5以及 4.0时,所有检测到的丙酸氧化菌逐渐减少(图4).由图3可知,在pH6.0~4.5条件下,P.propionicum和S.propionica依然是UASB反应器中优势丙酸氧化菌群,占丙酸氧化菌总数的81.4%~89.5%.分析认为, S.propionica能在低pH条件下生存可能与其代谢途径有关[17]. S.propionica降解丙酸时可产生中间代谢产物丁酸,丁酸的降解在热动力学上比丙酸更容易,以至于产物抑制容易解除,可促进丙酸降解.事实上,前期研究的确发现该UASB系统中存在降解丁酸的微生物—Syntrophomonas spp.[14]. S.propionica独特的代谢途径使其能在酸性环境下生长.然而,P.propionicum能在低pH 条件下生存的原因有待进一步研究.
另外,尽管丙酸是该UASB反应器中唯一的碳源,但是检测到的丙酸氧化菌仅占细菌总数的0.55%~3.65%,说明该UASB反应器中可能存在着未知的丙酸氧化菌.
以上结果表明,pH降低使得丙酸氧化菌群发生了明显的演替.在中性条件下,是UASB反应器中的优势丙酸氧化菌,随着pH降低逐渐演替为和(pH6.8)、(pH6.5)、和(pH6.0~4.0).这可能是弱酸性环境中(pH6.5~5.0),丙酸去除率得到一定程度恢复的主要原因.
3 结论
3.1 pH降低对丙酸氧化菌的生长有明显的抑制作用.在pH6.5条件下达到最大值6.2×10416S rRNA基因拷贝数/ngDNA.当pH由6.5分阶段降低至4.0时,丙酸氧化菌总数逐渐减少,这可能就是厌氧生物处理反应器在酸性条件下发生丙酸积累的根本原因.
3.2 随着pH逐渐降低,丙酸氧化菌群发生了明显的生态演替.在pH7.5~7.1时,是优势丙酸氧化菌;然后逐渐演替为和(pH6.8)、(pH6.5),和(pH6.0~4.0).
[1] 何 强,孙兴福,艾海男,等.两相一体式污泥浓缩消化反应器运行效能及其微生物特性[J]. 中国环境科学, 2012,32(11): 2039-2046.
[2] 陈广银,郑 正,常志州,等.不同氮源对麦秆厌氧消化过程的影响[J]. 中国环境科学, 2011,31(1):73-77.
[3] 宋佳秀,任南琪,钱东旭,等.醌呼吸影响厌氧消化产CO2/CH4及转化有毒物质的研究[J]. 中国环境科学, 2014,34(5):1236- 1241.
[4] Meng X, Zhang Y, Li Q, et al. Adding Fe0powder to enhance the anaerobic conversion of propionate to acetate [J]. Biochemical Engineering Journal, 2013,73:80–85.
[5] Li J, Ban Q, Zhang L, et al. Syntrophic propionate degradation in anaerobic digestion: A review [J]. International Journal of Agriculture and Biology, 2012,5:843–850.
[6] Narihiro T, Terada T, Ohashi A, et al. Quantitative detection of previously characterized syntrophic bacteria in anaerobic wastewater treatment systems by sequence-specific rRNA cleavage method [J]. Water Research, 2012,46:2167–2175.
[7] Boone D R, Bryant M P. Propionate-degrading bacterium, Syntrophobacter wolinii sp. nov. gen. nov., from methanogenic ecosystems [J]. Applied and Environmental Microbiology, 1980, 40:626–632.
[8] Sekiguchi Y, Kamagata Y, Nakamura K, et al. Fluorescence in situ hybridization using 16S rRNA- targeted oligonucleotides reveals localization of methanogens and selected uncultured bacteria in mesophilic and thermophilic sludge granules [J]. Applied and Environmental Microbiology, 1999,65:1280–1288.
[9] Lueders T, Pommerenke B, Friedrich M W. Stable-isotope probing of microorganisms thriving at thermodynamic limits: syntrophic propionate oxidation in flooded soil [J]. Applied and Environmental Microbiology, 2004,70:5778–5786.
[10] Harmsen H J M, Kengen H M P, Akkermans A D L, et al. Detection and localization of syntrophic propionate-oxidizing bacteria in granular sludge by in situ hybridization using 16S rRNA-based oligonucleotide probes [J]. Applied and Environmental Microbiology, 1996,62:1656–1663.
[11] Kundu K, Bergmann I, Hahnke S, et al. Carbon source-a strong determinant of microbial community structure and performance of an anaerobic reactor [J]. Journal of Biotechnology, 2013,168: 616–624.
[12] Ariesyady H D, Ito T, Okabe S. Functional bacterial and archaeal community structures of major trophic groups in a full-scale anaerobic sludge [J]. Water Research, 2007,41:1554–1568.
[13] Worm P, Fermoso F G, Lens P N L, et al. Decreased activity of a propionate degrading community in a UASB reactor fed with synthetic medium without molybdenum, tungsten and selenium [J]. Enzyme and Microbiology Technology, 2009,45:139–145.
[14] Zhang L, Ban Q, Li J, et al. Response of syntrophic propionate degradation to pH decrease and microbial community shifts in an UASB reactor [J]. Journal of Microbiology and Biotechnology, 2016,26:1409–1419.
[15] 国家环保局.水和废水监测分析方法[M]. 北京:中国环境科学出版社, 2002.
[16] Ban Q, Zhang L, Li J. Shift ofpropionate-oxidizing bacteria with HRT decrease in an UASB reactor containing propionate as a sole carbon source [J]. Applied Biochemistry and Biotechnology, 2015,175:274–286.
[17] Liu Y, Balkwill D L, Aldrich H C, et al. Characterization of the anaerobic propionate-degrading syntrophs Smithella propionica gen. nov., sp. nov. and Syntrophobacter wolinii [J]. International Journal Systematic Bacteriology, 1999,49:545–556.
[18] Imachi H S, Sakai Ohashi A, Harada H, et al.sp. nov., an, mesophilic, obligately syntrophic, propionate- oxidizing bacterium [J]. International Journal of Systematic and Evolutionary Microbiology, 2007,57:1487–1492.
[19] de Bok F A M, Harmsen H J M, Plugge C M, et al. The first true obligately syntrophic propionate-oxidizing bacterium,sp. nov., co-cultured with, and emended description of the genus Pelotomaculum [J]. International Journal of Systematic and Evolutionary Microbiology, 2005,55:1697–1703.
[20] Li C, Moertelmaier C, Winter J, et al. Microbial community shifts during biogas production from biowaste and/or propionate [J]. Bioengineering, 2015,2:35–53.
[21] Shigematsu T, Era S, Mizuno Y, et al. Microbial community of a mesophilic propionate-degrading methanogenic consortium in chemostat cultivation analyzed based on 16S rRNA and acetate kinase genes [J]. Applied and Environmental Microbiology, 2006,72:401–415.
[22] Harmsen H J M, Kengen H M P, Akkermans A D L, et al. Detection and localization of syntrophic propionate-oxidizing bacteria in granular sludge by in situ hybridization using 16S rRNA-based oligonucleotide probes [J]. Applied and Environmental Microbiology, 1996,62:1656–1663.
The succession of propionate-oxidizing bacteria at acidic conditions was analyzed byRT-PCR.
BAN Qiao-ying1*, ZHANG Rui1, ZHANG Li-guo1, LI Jian-zheng2, MEI Zhi
(1.College of Environment and Resource, Shanxi University, Taiyuan 030006, China;2.School of Municipal and Environmental Engineering, Harbin Institute of Technology, Harbin 150090, China)., 2017,37(10):3861~3866
Succession of propionate-oxidizing bacteria at acidic conditions was investigated in an upflow anaerobic sludge bed (UASB) reactor by quantitative real-time fluorescence polymerase chain reaction (qPCR). The results showed that at least three identified species of propionate-oxidizing bacteria (,, and) existed in the UASB system.was dominated at pH7.5~7.1 and its quantity was (5.0~5.8)×10316S rDNA copies per ng DNA, accounting for above 90.6% of the total detectable propionate-oxidizing bacteria. pH decreased from 7.1 to 6.8 resulted inwas significantly increased.andbecame dominant propionate-oxidizing bacteria and occupied 88.9%.accounted for 74.8% in total detectable propionate-oxidizing bacteria at pH 6.5, whereasandsucceed to dominant propionate-oxidizing bacteria at pH£6.0, indicating thatandwere more acid resistant. In addition, the total number of propionate-oxidizing bacteria was gradually reduced at pH£6.0. It might be one of the reasons for propionate accumulation at acidic conditions in anaerobic reactors.
UASB;propionate-oxidizing bacteria;acidic conditions;qPCR
X703.5
A
1000-6923(2017)10-3861-06
班巧英(1982-),女,山西忻州人,讲师,博士,主要从事废水厌氧生物处理研究.发表论文30余篇.
2017-03-13
国家自然科学基金资助项目(51708341);天津市水质科学与技术重点实验室开放研究基金资助项目 (TJKLAST-ZD- 2016-05);山西省基础研究项目(2015021136, 2015021134)
* 责任作者, 讲师, banqiaoying@163.com

