SarA调控金黄色葡萄球菌生物被膜形成的研究进展
郑艳阳,曹凤娇,明 迪,母 丹,向 华
(1.吉林农业大学动物科学技术学院,长春 130118;2.吉林大学动物科学学院,长春 130062)
SarA调控金黄色葡萄球菌生物被膜形成的研究进展
郑艳阳1,曹凤娇1,明 迪2,母 丹2,向 华1
(1.吉林农业大学动物科学技术学院,长春 130118;2.吉林大学动物科学学院,长春 130062)
金黄色葡萄球菌生物被膜的形成是一个多基因和多因子共同调控的过程。葡萄球菌属辅助调节子SarA是金黄色葡萄球菌主要的毒力基因表达调控系统,能直接或间接调控多种生物被膜形成相关基因的表达。本文对SarA的结构及其调控生物被膜形成的机制作一综述,以期为深入研究金黄色葡萄球菌的致病机理和开发新的抗生物膜感染策略提供参考。
金黄色葡萄球菌;SarA;生物被膜
金黄色葡萄球菌(Staphylococcusaureus,以下简称金葡菌)是引起人类和动物临床感染的重要病原体之一[1],感染金葡菌后会引发一系列感染,从轻度的皮肤感染到严重的全身感染[2]。金葡菌易形成生物被膜(biofilm,BF),这是金葡菌引发慢性感染的主要原因[3],它的形成与金葡菌细胞外基质包括多糖细胞间黏附素(extracellular polysaccharide intercellular adhesion,PIA),蛋白质和胞外DNA(extracellular DNA,eDNA)密切相关。葡萄球菌属辅助调节子SarA是金葡菌主要的毒力基因表达调控系统。研究表明,不论是在体内或是在体外条件下,SarA均能通过介导各种生物被膜形成相关基因的表达来调控金葡菌BF的形成[4]。本文将对SarA的结构以及SarA调控生物被膜形成相关因子表达的机制进行全面的综述,从而为寻找新型有效的抗菌膜剂提供新的思路和理论依据。
1 SarA的结构
SarA是由sarA基因座编码的大小为14.7 kDa的DNA结合蛋白,它能与靶基因启动子富含A/T保守序列的区域结合形成二聚体,也能通过下游基因作用于其他调节子(例如与agr启动子结合)直接或间接影响目的基因的转录[5-6]。sarA基因座由1.2 kb个碱基组成,包含三个重叠的转录单位,分别是sarA(0.58 kb)、sarC(0.84 kb)、sarB(1.15 kb),每个转录单位分别由P1、P2、P3启动子驱动[5]。

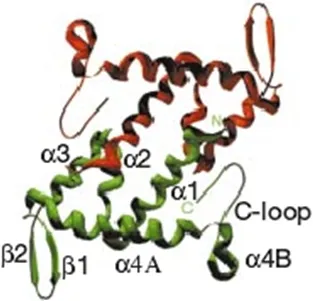
图1 Apo-SarA晶体结构[7]Fig 1 Crystal structures of Apo-SarA[7]

图2 Apo-SarA晶体结构[8]Fig 2 Crystal structures of Apo-SarA[8]
1.2 DNA-SarA的结构 翼状螺旋蛋白家族成员均有保守的DNA识别结合基序,SarA与DNA结合的结构域包括HTH基序(α3α4)和由β-发夹转角螺旋(β2β3)组成的翼状结构域(W1)[6]。Liu等研究提出SarA与DNA相互作用的方式与其翼状螺旋蛋白家族成员SarR相似,即HTH基序与DNA大沟相互作用,翼状结构域与DNA小沟相互作用[8](图3)。SarA突变蛋白表达SDS-PAGE分析图表明,翼状结构域氨基酸残基R84和R90对SarA与目标DNA(160-bp spa启动子片段)的相互作用至关重要,并且这两个氨基酸残基在SarA蛋白家族中高度保守[8](图4)。翼状结构域氨基酸残基D88和E89,金属结合口袋氨基酸残基D8和E11以及半胱氨酸残基C9均对SarA活性起到一定的作用,可见翼状结构域是SarA的关键活性部位[8]。

图3 SarA-DNA晶体结构Fig 3 Crystal structures of SarA-DNA complex

图4 SarA突变蛋白表达的SDS-PAGE分析Fig 4 SDS-PAGE analysis of mutant SarA proteins expression
2 SarA调控金葡菌生物被膜形成的机制
SarA是金葡菌的主要毒力因子表达调控系统,它能促进或抑制金葡菌许多毒力基因的表达,基因芯片分析发现SarA至少会影响120个基因的转录[9]。其中,SarA可以促进agr的表达,而agr会降低金葡菌形成BF的能力,推测若sarA突变,金葡菌BF形成能力会增强[9]。但Valle等研究发现,sarA突变却造成BF形成能力减弱,这可能与SarA调控多种生物被膜相关基因有关。例如SarA突变,会引起胞外蛋白酶和核酸酶的相关表达增加,它们会削弱BF形成的能力[10]。并且研究还发现agr突变株和agr野生株形成生物被膜的能力相同,说明SarA是以agr非依赖途径调控金葡菌BF的形成[10]。金葡菌BF形成过程中存在ica依赖途径和ica非依赖途径[11]。
2.1 ica依赖途径 PIA是金葡菌BF形成过程中的重要因子,主要在BF形成的黏附和繁殖阶段发挥作用。PIA是由胞间黏附素(intercellular adhesion,ica)操纵子icaABCD表达产物共同催化合成[9]。大部分的金葡菌都携带ica操纵子,但ica操纵子对金葡菌生物被膜形成所起的作用很复杂,并且存在菌株和环境依赖性[12]。
Valle等研究首次发现金葡菌sarA突变株生物被膜表型缺失的现象,他们指出sarA突变会显著减少ica和PIA的表达量,导致细菌无法形成生物被膜[10]。然而研究并未指出SarA可否与ica操纵子的启动子结合。而Tormo等研究证实了SarA能与ica操纵子的启动子结合,他们发现SarA与icaR-icaA启动子序列之间的亲和力高,启动子序列之间存在多个DNA结合位点,SarA易与这些DNA位点结合,从而促进ica操纵子的表达[13]。Cerca等研究还发现SarA可以上调icaR的表达,IcaR是位于ica操纵子上游的调控基因,IcaR对ica操纵子的表达有抑制作用。但研究表明,与表皮葡萄球菌不同,金葡菌中IcaR对ica操纵子的表达基本没有影响[14]。
除此之外,有研究表明磷酸化/去磷酸化与SarA活性和BF的形成密切相关。Didier等研究发现金葡菌丝氨酸激酶StK1/PknB和苏氨酸激酶SA0077对SarA有磷酸化作用。SarA经StK1/PknB磷酸化后,能增强与某些启动子序列的亲和力[15]。Tamber等的研究提出磷酸化后的SarA可能会显著影响ica操纵子的表达,但具体机制尚不清楚[16]。
2.2 ica非依赖途径 icaADBC操纵子和PIA固然对金葡菌BF的形成至关重要,但近来有研究提出了ica非依赖金葡菌BF形成机制[17]。Beenken等研究发现剔除人源金葡菌临床分离株UASM-1 的ica基因,对生物被膜的形成并没有影响,而突变sarA后则会削弱生物被膜的形成能力[17]。此外有研究发现sarA突变会消除耐甲氧西林金黄色葡萄球菌(Methicillin-resistantStaphylococcusaureus,MRSA)形成生物被膜的能力,而MRSA主要是以蛋白质介导的ica非依赖途径形成生物被膜[18-19]。因此,以上数据表明SarA也能调控ica非依赖生物被膜的形成。
2.2.1 蛋白质依赖途径 在ica非依赖金葡菌BF形成途径中,主要是蛋白质和eDNA等发挥作用,它们均是细菌BF基质的重要成分。在金葡菌中,BF的形成与某些细胞壁锚定蛋白有关,包括表面蛋白C和G(surface protein C and G,SasC和SasG)、凝集因子A/B(clumping factor A/B,ClfA/ClfB)、丝氨酸-天冬氨酸重复蛋白(serine aspartate repeat proteins,SdrC、SdrD和SdrE),生物膜相关蛋白(biofilm associated protein,Bap),表面蛋白A (surface protein A,SpA)和纤连蛋白结合蛋白(fibronectin-binding proteins ,FnBPA和FnBPB)[20]。研究表明这些葡萄球菌属表面蛋白可以在ica非依赖BF形成的初始黏附和聚集阶段中发挥作用[21]。J.Penades和I.Lasa研究团队最先阐明了ica非依赖的生物被膜形成机制,他们证实了Bap在奶牛乳房炎金葡菌分离株V329 BF形成的初始黏附和聚集阶段中发挥了关键作用[22]。研究发现突变V329的ica操纵子,并不会影响BF的形成。另外Totonda等研究分析了SarA对Bap表达的影响,发现SarA以agr非依赖途径上调了bap的表达,SarA可以与bap的启动子结合,SarA是bap表达的活化剂[23]。他们构建的sarA突变株JP62和JP65均不能形成生物被膜,而将携带sarA的重组质粒导入JP66和JP69中后,Bap重新表达,并且均能形成生物被膜,表明SarA可以恢复Bap依赖的生物被膜形成能力[23]。
研究还发现,胞外蛋白酶在蛋白质依赖的生物被膜形成中发挥关键作用[24]。SarA可以抑制胞外蛋白酶(extracellular protease)的活性。并且有研究表明,在SarA调控生物被膜形成的机制中,相比于PIA依赖机制,胞外蛋白酶介导机制更为重要,而且在此过程中,SarA不依赖agr途径[4]。Zielinska等研究发现在金葡菌sarA突变会导致胞外蛋白酶包括金属蛋白酶(aureolysin,Aur),丝氨酸蛋白酶(serine protease,SspA和SplA-F),半胱氨酸蛋白酶(cysteine protease ,ScpA/ SspB)分泌增加,这些金葡菌胞外蛋白酶均能限制BF形成,其中Aur影响最大[25-26]。sarA突变后,胞外蛋白酶分泌增加,这会导致超过250多种金葡菌蛋白质包括FnBPA,SpA,α-毒素以及酚可溶性调控蛋白的表达减少,这些蛋白与金葡菌的多种致病机理有关包括BF形成[25]。
FnBPA和FnBPB是金葡菌与纤维蛋白原结合的主要蛋白,它们能促进细菌定殖到宿主组织,完成BF形成的初始黏附过程。研究发现,MRSA菌株的生物被膜形成能力主要与表面蛋白,特别是FnBPA和FnBPB密切相关。Loughran等的研究发现aur可以抑制MRSA和MSSA形成生物被膜的能力[26]。他们还构建了MRSA sarA突变株,并且分别构建了同源的四株双重突变株sarA/aur、sarA/sspAB、sarA/sspB、sarA/scpA,发现SarA可以限制这些基因的转录,从而减少其对应编码的胞外蛋白酶的分泌,降低了生物被膜形成相关蛋白的解聚,继而促进了BF的形成[27]。他们还发现突变sspABC操纵子可以恢复sarA突变株FnBPA的表达,而突变编码aur的基因可以恢复SpA,α-毒素以及酚可溶性调控蛋白的表达[26]。但SspA需要SspB的激活,Aur需要SspA的激活,所以Aur可能是间接地影响这些毒力因子的表达[27]。然而研究发现,突变sarA突变株sspABC操纵子并没有恢复SpA,α-毒素以及酚可溶性调控蛋白的表达,说明aur自身会影响这些毒力因子的表达[26]。
除此之外有研究发现,在Newman菌株中,SarA可以通过与SaePQRS协同合作来抑制蛋白酶的合成,SarA能提高SaePQRS的活性,而SaePQRS能抑制胞外蛋白酶的分泌,使得生物被膜表面相关蛋白如FnBPA分泌量增加,从而促进了生物被膜的形成。但目前对于SaePQRS抑制蛋白酶分泌的机制还尚不清楚[28]。他们还发现往sarA突变株添加全局蛋白酶抑制剂后,FnBPs水平提高了6倍,而加入半胱氨酸蛋白酶抑制剂E64并没有影响FnBPs的水平,表明SarA不是通过影响SspB和ScpA的分泌来影响FnBPs的水平[29]。研究发现ssp或aur活性丧失,会导致10到20倍金葡菌sarA突变株SpA水平的增加[29]。
2.2.2 eDNA依赖途径 eDNA在ica非依赖金葡菌生物被膜形成过程中发挥重要作用,它主要影响生物被膜形成的早期黏附阶段和成熟阶段[29-31]。研究发现,用DNaseI 处理成熟的生物被膜,生物被膜会溶解[30]。此外,eDNA在生物被膜中的水平会受到耐热核酸酶(nuclease,nuc)的调控,而许多研究表明SarA 会下调nuc的表达[32-33]。
Tsang等研究构建了UAMS-1 nuc突变株和nuc/sarA双重突变株来分析它们对生物被膜形成的影响。研究发现sarA突变,nuc的表达水平提高,而突变sarA突变株的nuc后,便无法合成核酸酶[32]。他们还发现与sarA突变株相比,nuc/sarA双重突变株形成生物被膜的能力显著提高,但生物被膜数量并未恢复到野生水平,而往nuc/sarA双重突变株中添加携带sarA的重组质粒后,生物被膜数量恢复到正常水平[32]。这些数据表明UAMS-1 sarA突变株生物被膜表型缺失与核酸酶表达增强有关,但在此过程中,SarA也参与调控了其他生物被膜形成相关因子的表达[32]。
Dengler等对报道过能影响eDNA合成的一些基因进行了突变,包括自溶素基因(Alt、Aaa)、CidA、LrgAB以及SarA。其中,他们发现当sarA突变,eDNA的合成明显减少,同时生物被膜数量也显著减少[33]。除此之外,Atwood等研究发现在耐甲氧西林金葡菌LAC中sarA突变反而会抑制核酸酶的活性,但消除sarA突变株合成胞外蛋白酶能力后便能恢复其核酸酶的活性,说明SarA是通过降低胞外蛋白酶的分泌间接影响核酸酶的活性[34]。更重要的是,消除sarA突变株的蛋白酶后,尽管核酸酶活性增强,生物被膜数量仍会增加。这些数据表明,在耐甲氧西林金葡菌LAC 中,相比核酸酶,胞外蛋白酶对sarA突变株生物被膜缺失表型起到更重要的作用[34]。可见SarA调控eDNA依赖的生物被膜形成具有菌株特异性。
3 结 语
金葡菌生物被膜形成是一个多基因和多因子共同调控的过程。SarA作为金葡菌主要的毒力基因表达调控系统,参与调控了多种生物被膜形成相关因子的表达。PIA,蛋白质,eDNA作为金葡菌生物被膜的重要组成成分,自然是SarA调控金葡菌生物被膜形成的关键靶点。由于菌株特异性和环境依赖性,SarA参与调控不同菌株生物被膜形成的机制不尽相同。本文通过总结前人的研究结果,概述了SarA调控金葡菌生物被膜形成的三条途径,为日后以SarA作为有效靶点开发新型金葡菌BF感染抑制剂提供了重要的参考。但目前针对SarA靶向抑制剂的研究并不多,因此,医学工作者今后应将工作重点放在寻找出对SarA活性有明显抑制作用的药物。近来许多研究表明,中药单独或者联合其他传统抗生素能够有效破坏金葡菌成熟生物被膜结构,从而使得药物成功进入膜内杀灭细菌。生物被膜的形成是细菌对抗菌药物产生耐药性的主要原因之一,由于抗菌药物的不合理使用,日益加剧了细菌对抗菌药物的耐药性。中药不仅具有天然的活性成分,并且具备来源广泛,安全性能高,毒副作用小,作用靶点不单一,不易产生耐药性等多种优点,因此,筛选中药化合物作为SarA靶向抑制剂可行性很大,同时也符合绿色抗生素市场的需求。总而言之,随着金葡菌生物被膜形成机制研究的深入发展和抗菌膜剂的不断开发,人类将能更好地控制金葡菌感染。
[1] Verkade E, Kluytmans J. Livestock-associatedStaphylococcusaureusCC398: animal reservoirs and human infections[J]. Infections, Genetics and Evolution,2014, 21: 523-530.
[2] Becker K, Eliff C V.Staphylococcus,Micrococcus, and other catalase-positive cocci[J]. Manual of Clinical Microbiology,2011, 308-330.
[3] Gordon R J, Lowy F D. Pathogenesis of Methicillin-ResistantStaphylococcusaureusInfection[J]. Clinical Infectious Disease,2008, 46: S350-S359.
[4] Paharik A E, Horswill A R. TheStaphylococcusBiofilm: Adhesions, regulation, and host response[J]. Microbiology Spectrum,2016, 4(2): 22-215.
[5] Bayer M G, Heinrichs J H, Cheung A L. The molecular architecture of the sar locus inStaphylococcusaureus[J]. Journal of Bacteriology,1996, 178(15): 4563-4570.
[6] Cheung A L, Bayer A S, Zhang G,etal. Regulation of virulence determinantsinvitroandvivoinStaphylococcusaureus[J]. FEMS Immunology and Medical Microbiology,2004, 40(1): 1-9.
[7] Schumacher M A, Hurlburt B K, Brennan R G. Crystal structures of SarA, a pleiotropic regulator of virulence genes inS.aureus[J]. Nature,2001, 409(6817): 215-219.
[8] Liu Y, Manna A C, Pan C H,etal. Structure and function analyses of the global regulatory protein SarA fromStaphylococcusaureus[J]. PNAS,2006, 103(7): 2392-2397.
[9] Cue D, Lei M G, Lee C Y. Genetic regulation of the intercellular adhesion locus in staphylococci[J]. Frontiers in Cellular and Infection Microbiology,2012, 2: 38.
[10] Valle J, Toledo-Arana A, Berasain C,etal. SarA and not sigmaB is essential for biofilm development byStaphylococcusaureus[J]. Molecular Microbiology, 2003, 48(4): 1075-1087.
[11] Arciola C R, Campoccia D, Ravaioli S,etal. Polysaccharide intercellular adhesin in biofilm: structural regulatory aspects[J]. Frontiers in cellular and Infection microbiology, 2015, 5: 7.
[12] O'Gara J P. ica and beyond: biofilm mechanisms and regulation inStaphylococcusepidermidis andStaphylococcusaureus[J]. FEMS Microbiology Letters,2007, 270(2): 179-188.
[13] Tormo M A, Martí M, Valle J,etal. SarA is an essential positive regulator ofStaphylococcusepidermidis biofilm development[J]. Journal of Bacteriology,2005, 187(7): 2348-2356.
[14] Cerca N, Brooks J L, Jefferson K K. Regulation of the intercel ̄lular adhesin locus regulator (icaR) by SarA, sigmaB and IcaR inStaphylococcusaureus[J]. Journal of Bacteriology,2008, 190(19): 6530-6533.
[15] Didier J P, Cozzone A J, Duclos B. Phosphorylation of the virulence regulator SarA modulates its ability to bind DNA inStaphylococcusaureus[J]. FEMS Microbiology Letters,2010, 306(1): 30-36.
[16] Tamber S, Schwartzman J, Cheung A L. Role of Pkn Bkinase in antibiotic resistance andvirulence in community-acquired methicillin-resistantStaphylococcusaureusstrain USA300[J]. Infection and Immunity,2010, 78(8): 3637-3646.
[17] Beenken K E, Blevins J S, Smeltzer M S. Mutation of sarA inStaphylococcusaureuslimit biofilm formation[J]. Infection and Immunity,2003, 71(7): 4206-4211.
[18] O'Neil E, Pozzi C, Houston P,etal. Association between Methicillin Susceptibility and Biofilm Regualtion inStaphylococcusaureusIsolates from Device-Related Infections[J]. Journal of Clinical Microbiology,2007, 45(5): 1379-1388.
[19] O'Neil E, Pozzi C, Houston P,etal. A novelStaphylococcusaureusbiofilm phenotype mediated by the fibronectin-binding proteins, FnBPA and FnBPB[J]. Journal of Bacteriology,2008, 190(11): 3835-3850.
[20] Speziale P, Pietrocola G, Foster T J,etal. Protein-based biofilm matrices in Staphylococci[J]. Frontiers in Cellular and Infection Microbiology,2014, 4: 171.
[21] Foster T J, Geoghegan J A, Ganesh, V K,etal. Adhesion, invasion and evasion: the many fuctions of the surface proteins ofStaphylococcusaureus[J]. Nature Reviews.Microbiology,2014, 12(1): 49-62.
[22] Cucarella C, Solano C, Valle J,etal. Bap, aStaphlococcusaureussurface protein involved in biofilm formation[J]. Journal of Bacteriology,2001, 183(9): 2888-2896.
[23] Trotonda M P, Manna A C, Cheung A L,etal. SarA positively controlsbap-dependent biofilm formation inStaphylococcusaureus[J]. Journal of Bacteriology,2005,187(16): 5790-5798.
[24] Beenken K E, Mrak L N, Griffin L M,etal. Epistatic relationships between sarA and agr inStaphylococcusaureusbiofilm formation[J]. PLoS One,2010, 5(5): e10790.
[25] Zielinska A K, Beeken K E, Mrak L N,etal. sarA-mediated repression of protease production plays a key role in the pathogenesis ofStaphylococcusaureusUSA300 isolates[J]. Molecular microbiology,2012, 86(5): 1183-1196.
[26] Loughran A J, Atwood D N, Anthony A C,etal. Impact of individual extracellular proteases onStaphylococcusaureusbiofilm formation in diverse isolates and their isogenic sarA mutants[J]. Microbiology Open,2014, 3(6): 897-909.
[27] Shaw L, Golonka E, Potempa J. The role and regulation of the extracellular protease ofStaphylococcusaureus[J].Microbiology,2004, 150: 217-28.
[28] Mrak L N, Zielinska A K, Beenken K E,etal. saeRS and sarA Act Synergisticcally to Repress Protease Production and Promote Biofilm Formation inStaphylococcusaureus[J]. PLoS ONE,2012, 7(6): e38453.
[29] Houston P, Rowe S E, Pozzi C,etal. Essential role for the major autolysin in the fibronectin-binding protein-mediatedStaphylococcusaureusbiofilm phenotype[J]. Infection and Immunity,2011, 79(3): 1153-1165.
[30] Okshevsky M, Regina V R, Meyer R L. Extracellular DNA as a target for biofilm control[J]. Current Opinion in Biotechnology,2015, 33: 73-78.
[31] Kalan J B, LoVetri K, Cardona S T,etal. Recombinant human DNase I decrease biofilm and increase antimicrobial susceptibility in staphylococci[J]. Journal of Antibiotics,2012, 65(2):73-77.
[32] Tsang L H, Cassat J E, Shaw L N,etal. Factors contributing to the biofilm-deficiency phenotype ofStaphylococcusaureussarA mutants[J]. PLoS One,2008, 3(10): e3361.
[33] Dengler V, Foulston L, DeFrancesco A S,etal. An electrostatic net model for the role of extracellular DNA in biofilm formation byStaphylococcusaureus[J]. Journal of Bacteriology, 2015, 197(4): 3779-3787.
[34] Atwood D N, Loughran A J, Courtney A P,etal. Comparative impact of diverse regulatory loci onStaphylococcusaureusbiofilm formation[J]. MicrobiologyOpen,2015, 4(3): 436-451.
ProgressinStudyofImpactofSarAonBiofilmFormationbyStaphylococcusaureus
ZHENG Yan-yang1, CAO Feng-jiao1, MING Di2, MU Dan2, XIANG Hua1
(1.CollegeofAnimalScienceandTechnology,JilinAgriculturalUniversity,Changchun130118,China;2.CollegeofAnimalScience,JilinUniversity,Changchun130062,China)
Staphylococcusaureusbiofilm mode of growth is coherently regulated by multiple genes and factors. Staphylococcal accessory regulator SarA is one of the major regulatory systems which mediate virulence genes expression inS.aureus. Several biofilm-associated genes expression is controled by SarA either in a direct or indirect way. This article conducted a general review regarding the structures of SarA and its regulatory mechnisams on biofilm formation byStaphylococcusaureus. These findings provide an important reference for the pathogenesis ofS.aureusand the development of new strategies applied to eradicate biofilm-associated infections.
Staphylococcusaureus; SarA; biofilm
10.11751/ISSN.1002-1280.2017.12.12
2017-05-27
A
1002-1280 (2017) 12-0062-07
S852.61
吉林省教育厅"十三五"科学技术研究项目(吉教科合字[2016]第194号)
郑艳阳,硕士研究生,从事动物药理学研究。
向 华。E-mail:xianghua@126.com
(编辑:侯向辉)

