云南丘北仙女虾一物种的分子鉴定
李 蓉, 陈艳艳, 和忠廷, 杨洪福, 李春青, 曾本娟, 陈善元, 肖 蘅
(1. 云南大学 生命科学学院, 昆明 650091; 2. 云南省丘北县渔业站, 丘北 663200)
仙女虾(fairy shrimp)是无背甲目(Anostraca)所有成员的总称,包括弯头虫科(Streptocephalidae)、钗额虫科(Thamnocephalidae)、虾仙科(Branchinectidae)等8科26属300余种[1],均属于节肢动物门(Arthropoda),甲壳纲(Crustacea),鳃足亚纲(Branchiopoda)[2-4],其主要生活于临时水域或季节性淹没的洼地,生命周期较短,一般只能存活1~3个月,其卵粒耐冻耐旱,能在水体中存活数年甚至数十年。成年仙女虾含有丰富的蛋白质和必需氨基酸成分[5],具有较高的营养价值,幼体是鱼、虾及其他水生动物良好的天然饲料[6-8]。但是由于仙女虾物种的形态鉴定主要局限于成体,并且对于仙女虾物种的鉴定主要依据显著的几个形态特征来进行区分,如弯头虫科物种的鉴定主要依据雄性触角和四面体的卵进行区分[9-11],钗额虫科物种主要依据前附肢和雄性第一对胸足进行区分[12]。除了显著的几个特征之外,其余很多定性和定量的形态特征常常被忽略,对于形态相似的近缘种很难做到准确地鉴定,加之现在很难找到全球统一公认的物种分类相关资料,且随着仙女虾新种的不断出现,采用形态特征对物种进行鉴定存在一定困难。
近年来随着分子生物学技术的发展,基于PCR技术的分子鉴定可用于相同物种内和不同物种间细微差异的检测,为物种的鉴定提供了有效手段[13-14]。线粒体DNA具有分子结构简单、进化速率快、无重组、多拷贝及严格的母系遗传等特点[15-18],相比核DNA更适于进化关系的分析研究。其中线粒体细胞色素氧化酶亚基I(Cytochrome coxidase subunit I,COI)基因的特定区域可作为DNA条形码用于物种的鉴定[19]。例如,Vandergast等[20]运用两种多态聚合酶链式反应(PCRs)扩增mtDNA COI基因的特异性片段,很好地解决了分布于加利福尼亚州南部同一水域濒危仙女虾Branchinectasandiegonensis、Branchinectalynchi、Branchinectalindahli的分类鉴定难题。Daniels等[21]利用线粒体COI基因和其他基因片段对仙女虾Streptocephalus的物种进行系统发育分析显示,Streptocephalus的物种进化于岗得瓦纳,其分布格局是地理隔离和有限扩散共同作用的结果。Alonso等[22]对仙女虾Phallocryptustserensodnomi与近缘种P.spinosa进行COI基因序列及形态学分析一致显示,它们是两个不同的物种。
COI基因作为遗传标记能够很好地用于物种鉴定,然而某些因素的存在可能影响COI基因用于物种鉴定的有效性。由于COI是母系遗传,渐渗杂交或谱系分拣可能会导致近缘种之间单倍型共享[23-24]。此外,线粒体DNA的核拷贝(NUMTs)中存在的终止密码子、插入或缺失、非同义突变很容易被检出,会导致物种数量的高估[25]。因此,以防COI基因不能对物种进行有效鉴定,结合其他分子标记进行分析显得十分重要。18S rDNA基因常被用于系统发育研究和生物多样性分析的分子标记之一[26]。例如,Sanoamuang等[3]对泰国Streptocephalussirindhornae进行形态学鉴定发现与印度S.dichotomus形态特征极为相似,通过SSU(18S)rDNA基因序列的系统发育分析表明S.sirindhornae与S.dichotomus、S.simplex的亲缘关系较近。然而,近年来18S rDNA基因也被用于物种的分子鉴定,例如,Gebhardt等[27]利用18S rDNA基因和COI基因序列对来自北海和波罗的海Alloteuthis的物种进行聚类分析,结果显示Alloteuthis仅包含一个单物种。
云南省丘北县渔业站的杨洪福等在云南丘北进行鱼类资源调查时,在天星乡笼陶村水淹塘意外发现似虾非虾的物种,经形态特征初步鉴定为一种仙女虾“钗额虫科、枝额虫属、枝额虫”,后经国内外专家鉴定为“旋额虫科、旋额虫属、旋额虫”。云南丘北仙女虾究竟为钗额虫科枝额虫还是旋额虫科旋额虫,有待进一步明确。目前,国内对仙女虾物种进行研究报道的文献仅有一篇[28],并且对其种类鉴定尚未用到分子生物学手段。鉴于此,本研究以进化速度较快的线粒体COI基因和相对保守的18S rDNA基因序列片段作为分子标记,对云南丘北仙女虾样本进行测序,基于NCBI数据库BLAST进行相似性比对,并结合GenBank中已发表的相关同源序列,进行序列特征、遗传距离及系统发育分析,以期进一步明确云南丘北仙女虾物种的具体种类,为仙女虾物种的准确鉴定提供参考依据。
1材料与方法
1.1 材料
本研究的样本由云南丘北县渔业站赠送,总计30份(编号:YQ01~YQ30),经形态学特征初步鉴定为一种仙女虾物种,样本浸泡于95%的乙醇溶液中,-20℃保存备用。同时基于仙女虾COI基因和18S rDNA基因序列,从GenBank中下载了该物种的已知同源序列(表1)。

表1 GenBank序列及其登录号
1.2 方法
1.2.1 基因组DNA提取
取仙女虾泳足组织约20~30 mg,用蒸馏水缓冲液洗净泳足中的酒精,将其剪碎用超纯水处理为细胞悬液,采用血液/细胞/组织基因组提取试剂盒提取基因组总DNA。用1.5%琼脂糖凝胶电泳检测技术和Nanodrop 2000超微量分光光度计进行DNA浓度和纯度测定,将浓度大于60 ng/μL的DNA,稀释至40 ng/μL工作液浓度之后,存于-20℃冰箱,用于之后的PCR扩增实验。
1.2.2 PCR扩增及序列测定
线粒体COI基因与18S rDNA基因的扩增引物均为通用引物,线粒体COI基因的引物为LCO1490(5′-GGTCAACAAATCATAAAGATATTGG-3′);HCO2198(5′-TAAACTTCAGGGTGACCAAAAAATCA-3′)[29]。18S rDNA基因的引物为NSI(5′-GTAGTCATATGCTTGTCTC-3′);Fung(5′-ATTCCCCGTTACCCGTTG-3′)[30]。所有引物均由上海捷瑞生物工程技术有限公司合成。PCR扩增的反应体系为25 μL,包括10×Buffer 2.5 μL,线粒体COI基因与18S rDNA基因25 mmol/L的MgCl2分别为2.5 μL、3.5 μL,10 mmol/L的ddNTP Mix 2 μL,10 μmol/L的正反引物各0.5 μL,0.75 U ExTaq酶0.15 μL,DNA模板为2~3 μL,然后用超纯水定容至25 μL。PCR反应程序为:95℃预变性4 min;95℃变性1 min,线粒体COI基因与18S rDNA基因分别为40℃、45℃退火1 min,72℃延伸1 min,35个循环;循环结束后72℃延伸5 min。每次PCR扩增都设置阴性对照,以防外源DNA分子的污染。扩增产物进行1.5%琼脂糖凝胶电泳检测后,将胶置于UVP全自动凝胶成像系统上观察并拍照,选取条带亮且单一的PCR产物送往北京六合华大基因科技有限公司广州分公司进行双向测序。
1.2.3 数据分析
采用DNASTAR软件包中的SeqMan进行正反链校对和编辑,手动去除序列两端的引物区,获得COI基因与18S rDNA基因序列有效长度分别为658 bp和325 bp。采用NCBI在线BLAST(https://blast.ncbi.nlm.nih.gov/Blast.cgi)进行相似性比对分析,以确定扩增序列的种类。将实验所得序列与GenBank中下载的同源序列利用Clustal X软件进行排序。采用MEGA6.06软件将COI基因序列翻译成蛋白序列,检测是否有提前终止现象,计算碱基含量和遗传距离,并采用邻接法(Neighbor-joining,NJ)构建系统发育树,树上各分支的支持率由Bootstrap重复次数为1000次[31]。采用DnaSP v5软件计算简约信息位点、变异位点和单倍型数目。
2结果与分析
2.1 PCR扩增、电泳检测与相似性比对
对云南丘北仙女虾共30份样本的COI基因与18S rDNA基因的序列分析发现,所有实验样本均可成功进行PCR扩增与测序,PCR产物经凝胶电泳检测所得的电泳图效果良好,扩增条带清晰完整(图1)。将比对好的COI基因与18S rDNA基因片段序列经NCBI数据库的BLAST相似性比对发现与弯头虫科(Streptocephalidae)的Streptocephalussirindhornae相似度最高,分别为96%和100%,说明云南丘北仙女虾可初步确定为弯头虫科的Streptocephalussirindhornae。
2.2 COI基因与18S rDNA基因序列特征
对COI基因与18S rDNA基因序列分析显示所有序列都未发现碱基的插入或缺失。将COI基因序列翻译成蛋白序列,检测未包含终止密码子,说明未出现提前终止现象。在30个样本的COI基因序列中共检测到15个简约信息位点,22个变异位点,分别占序列有效长度(658 bp)的2.3%、3.3%,8个单倍型(YQH1~YQH8),碱基A、T、C、G的平均含量分别为23.2%、38.2%、20.7%、17.9%,其中A+T含量(61.4%)大于G+C含量(38.6%),说明COI基因的碱基组成具有明显的偏向性。而在30个样本的18S rDNA基因序列中只检测到1个简约信息位点,1个变异位点,均占序列有效长度(325 bp)的0.3%,2个单倍型(YQH1~YQH2),碱基A、T、C、G的平均含量分别为25.7%、27.2%、23.7%、23.4%,其中A+T含量(52.9%)与G+C含量(47.1%)相当,说明18S rDNA基因的碱基组成无偏向性。
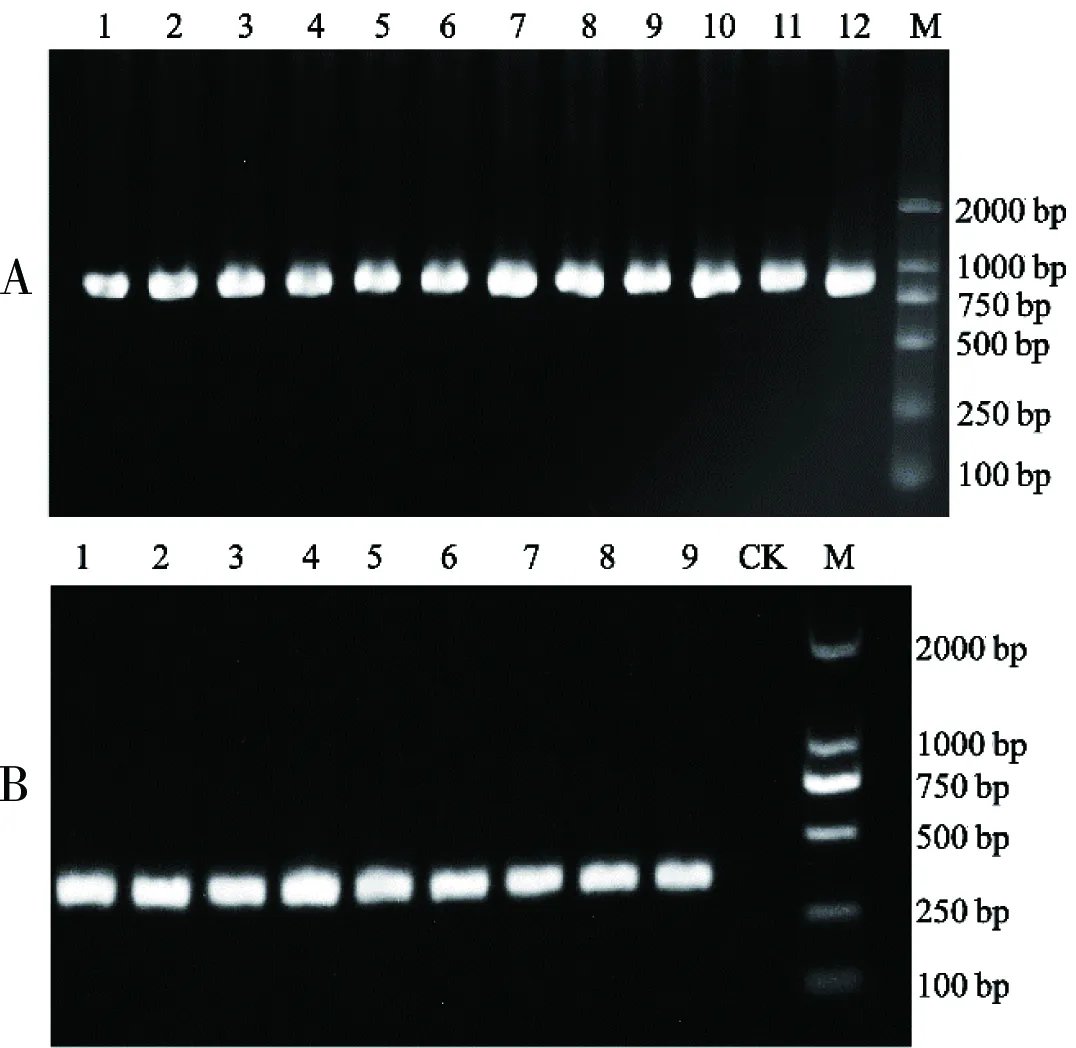
图1 COI基因与18S rDNA基因部分仙女虾的PCR扩增条带
A:COI基因PCR扩增条带;B:18S rDNA基因PCR扩增条带。CK:空白对照;M:分子标记 DL 2000
2.3 遗传距离与系统发育分析
利用MEGA6.06软件中的K2P(Kimura-2-parameter)模型计算不同仙女虾物种的遗传距离(表2)。基于COI基因序列的结果显示:云南丘北仙女虾种内平均遗传距离为0.011;丘北仙女虾与其他仙女虾的种间遗传距离范围为0.043~0.211,其中与S.sirindhornae的种间遗传距离最小,为0.043;与钗额虫科T.platyurus的种间遗传距离最大,为0.211。
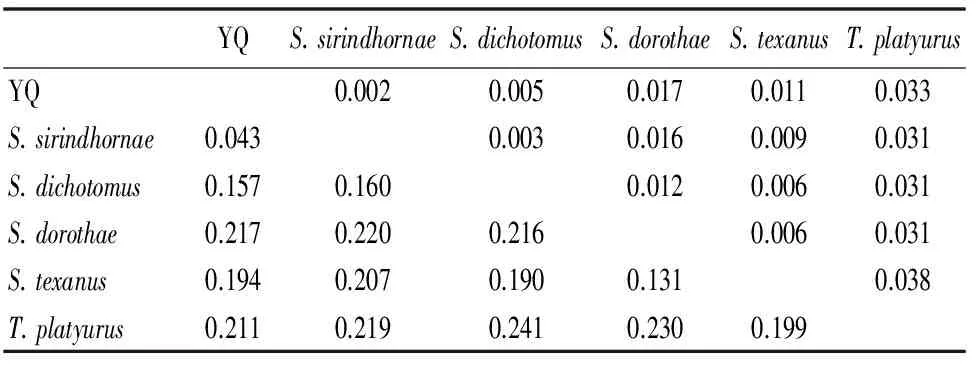
表2 基于COI基因与18S rDNA基因序列计算的不同仙女虾遗传距离
对角线上方为不同仙女虾18S rDNA基因序列的遗传距离;对角线下方为不同仙女虾COI基因序列的遗传距离基于18S rDNA基因序列的结果显示:云南丘北仙女虾种内平均遗传距离为0.003;丘北仙女虾与其他仙女虾的种间遗传距离范围为0.002~0.033,其中与S.sirindhornae的种间遗传距离最小,为0.002;与钗额虫科T.platyurus的种间遗传距离最大,为0.033。
选取钗额虫科的Thamnocephalusplatyurus为外类群,基于COI基因与18S rDNA基因序列采用邻接法构建系统发育树(图2和图3),结果发现云南丘北仙女虾的单倍型序列都与泰国S.sirindhornae序列聚在一起,形成单系枝,支持率分别为100%和72%。丘北仙女虾与S.sirindhornae聚为单系枝后同形态学上极为相似的印度S.dichotomus形成单系枝,支持率分别为97%和56%。

图2 基于COI基因序列构建的不同仙女虾物种的NJ树

图3 基于18S rDNA基因序列构建的不同仙女虾的NJ树
3讨论
COI基因是线粒体DNA上的一段蛋白质编码基因。近年来随着COI基因作为DNA条形码概念的提出,因其线粒体DNA具有拷贝数量大、独立于细胞核DNA等特点,被广泛用于物种的分子鉴定与系统发育的研究[19]。18S rDNA是真核生物染色体上编码核糖体小亚基RNA的基因,因其DNA序列较为保守,被普遍用于高级阶元的分子进化和系统学的研究。本研究中,COI基因序列的碱基A+T含量(61.4%)明显高于G+C含量(38.6%),碱基组成的偏向性是线粒体蛋白质编码基因的一个显著特征,COI基因序列不包含终止密码子,且未发现碱基的插入或缺失,说明本研究所测COI基因序列来源于线粒体,而非核基因序列。18S rDNA基因序列碱基组成同COI基因序列相比,G和C的含量明显高于前者,且G+C含量(47.1%)与Sanoamuang等[3]对泰国仙女虾18S rDNA基因序列研究得出的G+C含量(50.55%~50.72%)相当。从序列变异来看,18S rDNA基因序列的变异位点低于COI基因,说明18S rDNA基因的进化速率较COI基因慢,同18S rDNA基因更适合于种上阶元分子分类研究的结论相一致[32-33]。
遗传距离的大小是进行物种鉴别的主要标准。本研究中基于COI基因丘北仙女虾的种内平均遗传距离为1.1%,同Ratnasingham等[34]认为的DNA条形码的种内遗传距离<3%的一般规律相符合。此外,丘北仙女虾与S.sirindhornae的种间遗传距离(4.3%)相比甲壳纲同属物种间的平均遗传距离(15.4%)[35],属于种内差异;与弯头虫科S.dichotomus、S.dorothae、S.texanus及钗额虫科Thamnocephalusplatyurs的种间遗传距离均高于甲壳纲同属物种间的分化水平,属于种间差异。这一结论初步表明丘北仙女虾与S.sirindhornae属于同一物种。而基于18S rDNA基因序列的丘北仙女虾与S.sirindhornae的种间遗传距离(0.2%)小于丘北仙女虾的种内平均遗传距离(0.3%),且其他仙女虾物种的种间遗传距离较小(0.3%~3.8%),不符合Hebert等[35]计算的甲壳纲同属物种间的平均遗传距离(15.4%),未达到物种分化水平,因此无法对仙女虾各物种进行区分。构建的NJ系统发育树均显示丘北仙女虾的单倍型序列与泰国S.sirindhornae序列聚为独立的分枝,COI树的节点支持率较高(100%),但18S rDNA基因构建的系统发育树节点支持率低(72%),说明该节点为独立分支的结论不可靠,同理,S.texanus与S.dorothae为独立分支的节点支持率为58%,其结论也不可信。因此,利用18S rDNA基因序列对仙女虾物种进行系统发育分析时,节点的支持率低,若要获取准确可信的系统发育关系,还需考虑结合其他基因序列进行进一步分析。
仙女虾Streptocephalussirindhornae是在泰国首次被发现及描述的无甲目物种,广泛分布于泰国东北部的临时水域,如泰国的马哈沙拉堪养殖塘等[3];该物种也被发现于中国西南地区的干涸水塘,如广西壮族自治州崇左县白头叶猴生态公园的沼泽地、云南泸西老干塘的临时水塘等[28]。本研究首次应用COI与18S rDNA基因序列片段对云南丘北仙女虾进行相似性比对、遗传距离和系统发育分析,结果一致支持所测仙女虾样本为弯头虫科的Streptocephalussirindhornae。这一鉴定结果同泰国、云南泸西发现及描述的S.sirindhornae应属于同一物种。该结论说明了中国科学院昆明动物研究所舒树森等通过形态鉴定,该物种不属于钗额虫科、枝额虫属、枝额虫的报道相符合。同时也推测国外专家鉴定为旋额虫科、旋额虫属、旋额虫的相关报道是错误的。但由于本研究仅利用了COI与18S rDNA基因片段对仙女虾物种进行了分子鉴定,初步证实COI基因能对仙女虾物种进行有效鉴定,18S rDNA基因对其鉴定存在不足,且目前难以收集到近缘的仙女虾物种开展比较形态学研究。鉴于此,为获取更加准确可靠的结论,还需结合其他核基因或基因组标记,尽可能收集其他仙女虾物种,并与形态学数据相结合作进一步分析。
[1]BRENDONCK L, ROGERS D C, OLESEN J, et al. Global diversity of large branchiopods (Crustacea: Branchiopoda) in freshwater [J]. Hydrobiologia, 2008, 595 (1): 167-176.
[2]SANOAMUANG L O, SAENGPHAN N. A new species ofstreptocephalusfairy shrimp (Crustacea, Anostraca) with tetrahedral cysts from central Thailand [J]. International Review of Hydrobiology, 2006, 91(3): 250-256.
[3]SANOAMUANG L O, MURUGAN G, WEEKERS P H H, et al.Streptocephalussirindhornae, new species of freshwater fairy shrimp (Anostraca) from Thailand [J]. Journal of Crustacean Biology, 2000, 20(3): 559-565.
[4]SANOAMUANG L O, SAENGPHAN N, MURUGAN G. First record of the family Thamnocephalidae (Crustacea: Anostraca) from Southeast Asia and description of a new species ofBranchinella[J]. Hydrobiologia, 2002, 486(1): 63-69.
[5]DARARAT W, LOMTHAISONG K, SANOAMUANG L O. Biochemical composition of three species of fairy shrimp (Branchiopoda: Anostraca) from Thailand [J]. Journal of Crustacean Biology, 2012, 32(1): 81-87.
[6]SAEJUNG C, HATAI K, WADA S, et al. Clinical observations of black disease in fairy shrimps,StreptocephalussirindhornaeandBranchinellathailandensis, from Thailand and pathogen verification [J]. Journal of Fish Diseases, 2011, 34(12): 911-920.
[7]SRIPUTHORN K, SANOAMUANG L. Fairy shrimp (Streptocephalussirindhornae) as live feed improve growth and carotenoid contents of giant freshwater prawnMacrobrachiumrosenbergii[J]. International Journal of Zoological Research, 2011, 7(2): 138-146.
[8]SORNSUPHARP S, DAHMS H U, SANOAMUANG L. Nutrient composition of fairy shrimpStreptocephalussirindhornaenauplii as live food and growth performance of giant freshwater prawn postlarvae [J]. Aquaculture Nutrition, 2013, 19(3): 349-359.
[9]BELADJAL L, MERTENS J, DUMONT H J.Streptocephaluscaljonin.sp.(Crustacea: Anostraca) from Burundi and analysis of its limb structure [J]. Hydrobiologia, 1996, 319(2):149-157.
[10]LEWIS B. Molar surface morphology inStreptocephaluscoomansi(Crustacea: Branchiopoda: Anostraca) [J]. Journal of Morphology, 1994, 219(2): 165-172.
[11]HERBERT B, TIMMS B V. A new species ofStreptocephalus(Parastreptocephalus) (Crustacea: Anostraca: Streptocephalidae) from North Queensland, Australia [J]. Memoirs of the Queensland Museum, 2000, 45(2): 385-390.
[12]HIROSE G L, BARROS-ALVES S P, ALVES D F, et al. Morphological variation in males ofDendrocephalusorientalis(Anostraca: Thamnocephalidae): implications for species identification [J]. Zootaxa, 2015, 3915(1): 569-580.
[13]TAUTZ D, ARCTANDER P, MINELLI A, et al. DNA points the way ahead in taxonomy [J]. Nature, 2002, 418(6897): 479.
[14]肖金花, 肖 晖, 黄大卫. 生物分类学的新动向-DNA条形编码 [J]. 动物学报, 2004, 50(5): 852-855.
[15]BIBB M J, VAN ETTEN R A, WRIGHT C T, et al. Sequence and gene organization of mouse mitochondrial DNA[J]. Cell, 1981, 26(2): 167-180.
[16]VAWTER L, BROWN W M. Nuclear and mitochondrial DNA comparisons reveal extreme rate variation in the molecular clock [J]. Science, 1986, 234(4773): 194-196.
[17]GYLLENSTEN U, WHARTON D, WILSON A C. Maternal inheritance of mitochondrial DNA during backcrossing of two species of mice [J]. Journal of Heredity, 1985, 76(5): 321-324.
[18]PAKENDORF B, STONEKING M. Mitochondrial DNA and human evolution [J]. Annu Rev Genomics Hum Genet, 2005, 6 (6):165-183.
[19]RAMADAN H A I, BAESHEN N A. Biological identifications through DNA barcodes [J]. Proceedings Biological Sciences, 2003, 270 (1512): 313-321.
[20]VANDERGAST A G, WOOD D A, SIMOVICH M, et al. Identification of co-occurringBranchinectafairy shrimp species from encysted embryos using multiplex polymerase chain reaction [J]. Molecular Ecology Resources, 2009, 9(3): 767-770.
[21]DANIELS S R, HAMER M, ROGERS D C. Molecular evidence suggests an ancient radiation for the fairy shrimp genusStreptocephalus(Branchiopoda:Anostraca) [J]. Biological Journal of the Linnean Society, 2004, 82(3): 313-327.
[22]ALONSO M, VENTURA M. A new fairy shrimpPhallocryptustserensodnomi(Branchiopoda: Anostraca) from Mongolia [J]. Zootaxa, 2013, 3670 (3): 349-361.
[23]CONFLITTI I M, SHIELDS G F, CURRIE D C. A "complex" problem: delimiting sibling species boundaries in black flies (Diptera: Simuliidae) [J]. Canadian Entomologist, 2012, 144 (2): 323-336.
[24]FUNK D J, OMLAND K E. Species-level paraphyly and polyphyly: frequency, causes, and consequences, with insights from animal mitochondrial DNA [J]. Annual Review of Ecology Evolution & Systematics, 2003, 34(1): 397-423.
[25]SONG H, BUHAY J E, WHITING M F, et al. Many species in one: DNA barcoding overestimates the number of species when nuclear mitochondrial pseudogenes are coamplified [J]. Proceedings of the National Academy of Sciences of the United States of America, 2008, 105 (36): 13486-13491.
[26]MEYER A, TODT C, MIKKELSEN N T, et al. Fast evolving 18S rRNA sequences from Solenogastres (Mollusca) resist standard PCR amplification and give new insights into mollusk substitution rate heterogeneity [J]. BMC Evolutionary Biology, 2010, 10 (1): 1-12.
[27]GEBHARDT K, KNEBELSBERGER T. Identification of cephalopod species from the North and Baltic Seas using morphology, COI and 18S rDNA sequences [J]. Helgoland Marine Research, 2015, 69 (3): 259-271.
[28]SHU S, MAEDA-MARTINEZ A M, ROGERS D C, et al. Morphological characterization ofStreptocephalussirindhornae(Branchiopoda: Anostraca) from South East Asia: First record of the Streptocephalidae from China [J]. Zootaxa, 2015, 3911(3): 447-450.
[29]FOLMER O M, BLACK M B, HOEH W R, et al. DNA primers for amplification of mitochondrial cytochrome coxidase subunit I from diverse metazoan invertebrates [J]. Molecular Marine Biology & Biotechnology, 1994, 3(5): 294-299.
[30]WU J, YU M, XU J, et al. Impact of transgenic wheat with wheat yellow mosaic virus resistance on microbial community diversity and enzyme activity in rhizosphere soil [J]. Plos One, 2014, 9(6): e98394.
[31]FELSENSTEIN J. Confidence limits on phylogenies: an approach using the bootstrap [J]. Evolution, 1985, 39(4): 783-791.
[32]BORCHIELLINI C, CHOMBARD C, MANUEL M, et al. Molecular phylogeny of Demospongiae: implications for classification and scenarios of character evolution [J]. Molecular Phylogenetics & Evolution, 2004, 32(3): 823-837.
[33]MANUEL M, BORCHIELLINI C, ALIVON E, et al. Phylogeny and evolution of calcareous sponges: monophyly of calcinea and calcaronea, high level of morphological homoplasy, and the primitive nature of axial symmetry [J]. Systematic Biology, 2003, 52(3): 311-333.
[34]RATNASINGHAM S, HEBERT P D N. BOLD: The barcode of life data system (www.barcodinglife.org) [J]. Molecular Ecology Notes, 2007, 7 (3): 355-364.
[35]HEBERT P D N, RATNASINGHAM S, DEWAARD J R. Barcoding animal life: cytochrome c oxidase subunit 1 divergences among closely related species [J]. Proceedings of the Royal Society B Biological Sciences, 2003, 270 (Suppl 1): S96-S99.

