烟草Hsp70基因家族的鉴定及NtHsp70Chl基因的表达分析
陈二龙 范志勇 王松峰 王德勋 朱凯 孙军伟 苏家恩 宋朝鹏


摘 要:為探明烟草热激蛋白70(Hsp70)基因家族的特征及烟叶主脉中NtHsp70Chl基因在烘烤条件下的表达模式,对Hsp70基因家族进行了亚细胞定位、系统进化、基因结构、染色体定位,并对NtHsp70Chl基因进行了烘烤条件下实时荧光定量表达PCR分析。结果表明,烟草基因组中Hsp70基因家族共有61个成员,蛋白序列长度不等,等电点在4.52~9.75范围内,各成员蛋白分别定位于细胞质、内质网、细胞外基质、叶绿体和线粒体中;烟草Hsp70基因家族分为6组,定位于叶绿体的成员均存在Ⅵ-a亚组;61个NtHsp70基因分布于18条染色体上,经亚细胞定位于叶绿体的基因分别位于6、17和19号染色体上;烟草Hsp70家族中具有10个基因重复和5个旁系同源基因;经RT-qPCR检测分析,定位于烟草叶绿体中的NtHsp70Chl基因对高温诱导的敏感性较弱。本研究为深入研究烟草Hsp70的功能奠定了基础。
关键词:烟草;主脉组织;Hsp70基因家族;生物信息学;NtHsp70Chl;高温诱导
中图分类号:S572.03 文章编号:1007-5119(2018)02-0008-09 DOI:10.13496/j.issn.1007-5119.2018.02.002
Bioinformatics of Tobacco(Nicotiana tabacum)Hsp70 Gene Family and Expression Analysis of NtHsp70Chl in Midrib
CHEN Erlong1, FAN Zhiyong2, WANG Songfeng3, WANG Dexun2, ZHU Kai2,
SUN Junwei2, SU Jiaen2, SONG Zhaopeng1*
(1. College of Tobacco Science, Henan Agricultural University, Zhengzhou 450002, China 2; Dali Tobacco Company of Yunnan Province, Dali 671000, Yunnan, China; 3. Key Laboratory of Tobacco Biology and Processing, Ministry of Agriculture, Tobacco Research Institute of CAAS, Qingdao 266101, China)
Abstract: In order to study the relevant biological information of the tobacco heat shock protein 70 (Hsp70) gene family and the specific expression of the chloroplast-localized members NtHsp70Chl in tobacco midrib, subcellular location, system evolution, Gene structure, chromosome location, and real-time fluorescence quantitative expression PCR analysis were carried out. The results showed that 61 members of the tobacco Hsp70 gene family were identified. The numbers of amino acid contained in the protein sequence were different, and the isoelectric point was in the range of 4.52~9.75. And the protein members were predicted to be localized in the cytoplasm, endoplasmic Reticulum, extracellular matrix, chloroplast (4 members) and mitochondria respectively. The tobacco Hsp70 gene family was divided into 6 groups, and the NtHsp70Chl genes were all in the VI-a subgroup. 61 NtHsp70 genes are located on 18 chromosomes, and NtHsp70Chl genes were located on chromosomes 6, 17, and 19, respectively. In tobacco Hsp70 family, there are 10 duplications and 5 paralogous genes. According to RT-qPCR analysis, the NtHsp70Chl genes in tobacco midrib were weakly sensitive to high-temperature stress. This study laid the foundation for further exploration and application of tobacco Hsp70 functions.
Keywords: tobacco; midrib tissue; Hsp70 gene family; bioinformatics; NtHsp70Chl; high temperature induction
烟叶在烘烤过程中处于高温和水分胁迫的环境[1]。Hsp70基因在高温或干旱诱导的条件下表达量上调,Hsp70蛋白参与蛋白质的正确折叠,调节蛋白在细胞内积累,增加组织的耐热性[2-4]。叶绿体内Hsp70蛋白可协助基质中蛋白转运,是必要的ATP酶,与类囊体膜的形成和病原微生物入侵的嗜
基金项目:中国烟草总公司云南省公司项目“防治烘烤过程中烟叶腐烂霉变技术研究推广”(2016YN10)、“返青寡日照等条件下难落黄烟叶的烘
烤技术研究及推广”(2015YN20)
作者简介:陈二龙(1992-),男,硕士研究生,主要从事烟草调制加工研究。E-mail:celyctz@163.com。*通信作者,E-mail:ycszp@163.com
收稿日期:2017-07-22 修回日期:2017-11-17
好有一定关系[5-8]。
烟叶在烘烤过程中(逆境条件)进行代谢活动,
且在一定程度上促进病原微生物的入侵或扩散,此时,细胞内一些抗性基因表达或上调,如TLHS1、LFD、NPK1和Hsp70等[9-12]。Hsp70蛋白作为分子伴侣,其N端核酸结合域(NBD)与ATP/ADP相互作用,控制C端底物结构域(SBD)以催化剂的形式调节蛋白质的折叠和积累[13-14]。Hsp70基因家族成员的功能具有多样性,如拟南芥中的AtHsp70-15在其植株的耐热性方面发挥重要的作用[15],哈茨木霉菌的Hsp70基因通过转基因表达使拟南芥对高温以及非生物胁迫的抗性增加[16],斑茅在干旱胁迫的条件下,Hsp70基因表达量上调,对调节细胞膜热稳定性和水分相对含量具有积极作用[17],部分植物体中Hsp70参与病原微生物的蛋白质折叠等[18]。Hsp70的亚细胞位置可分为4类:内质网、细胞质、线粒体及叶绿体[19]。目前,对植物Hsp70基因家族数据研究对象主要包括拟南芥[20]、胡杨[21]、水稻[22]、大豆[23]等,关于烟草Hsp70基因家族成员的研究较少。
研究烟草Hsp70基因家族成员的特征、进化和表达模式,对于进一步探讨烟草中Hsp70基因家族的功能及指导烟叶的烘烤具有重要的作用。本研究在烟草全基因组的背景下,运用生物信息学的方法挖掘烟草Hsp70基因家族的潜在信息,并以烟叶主脉组织内NtHsp70Chl基因成员为研究对象,采用实时荧光定量表达探索其在热激条件下的反应。
1 材料与方法
1.1 材料
试验于2016年在云南省弥渡县红花大金元基地调制实验室进行。
烟草(Nicotiana tabacum L)Hsp70基因家族的相关序列,下载于NCBI(https://www.ncbi. nlm.nih.gov/)和Sol Genomics Network(https:// solgenomics.net/)数据库[24-25]。拟南芥的Hsp70蛋白序列来源于TAIR(http://www.arabidopsis.org/)和NCBI数据库[26-27]。
烟草品种为红花大金元,取样地理位置为凤仪镇。取样:(1)在常规烘烤(变黄前期干球/湿球温度为35 ℃/35 ℃,烘烤时长8 h,烟叶状态为叶尖变软;变黄中期38 ℃/37 ℃,32 h,叶片达到8黄;变黄后期42 ℃/36 ℃,16 h叶片9成黄)过程中,于烘烤0、12、24、36、48、60 h随机取顶层烟叶3片,从主脉端口向下除去1 cm,以新端口为参照,向下取2 cm,去除两侧叶耳,将3条主脉混合包裹于锡箔纸中,放于液氮中并进行标记;(2)将适熟烟叶随机装于3组ZLKX-Ⅱ型烟叶烘烤箱(±0.5 ℃,郑州智联自动化设备有限公司),第1组烘烤箱的温湿度设定为35 ℃/35 ℃, 第2组设定为38 ℃/38 ℃, 第3组为42 ℃/42 ℃,期间的3组烘烤箱温湿度设定均保持不变,在0、12和24 h进行取样,步骤同上。
1.2 烟草Hsp70基因家族的鉴定
利用已获得拟南芥Hsp70基因家族的蛋白序列为比对文件[20],从上述数据库中进行BLAST(1e-10),将重复序列去除,并构建本地數据库;从Pfam(http://pfam.xfam.org/)数据库获得具有Hsp70结构域的种子文件(PF00012)[28],通过HMMER3.0软件搜索本地数据库,确认获取的蛋白序列具有典型Hsp70结构域。采用Protparam(http://us.expasy.org/tools/protparam.html)在线工具,预测烟草Hsp70的等电点、序列长度及分子质量[29]。烟草Hsp70的亚细胞定位的预测运用ProtComp(http://linux1.softberry.com/berry.phtml? topic=protcomppl&group=programs&subgroup=proloc)工具进行分析[30]。
1.3 烟草Hsp70基因的系统进化分析
运用ClustalW2软件对烟草中61个Hsp70及拟南芥中已被确认功能的Hsp70蛋白序列进行比对[31];MEGA6软件将比对结果进一步分析转化,生成进化树,其构建方式使用邻接法(NJ)[32]。
1.4 烟草Hsp70基因的染色体定位和结构分析
依据烟草Hsp70基因定位于染色体中的信息,采用Mapinspector 软件呈现基因位于染色体中轮
廓图[33];依据两个基因的匹配长度以及相似性是否大于80%判定基因复制,而基因是否处于同一染色 体中判定串联复制或片段复制[34]。根据烟草Hsp70基因DNA和CDS序列的数据,采用GSDS2(http://gsds.cbi.pku.edu.cn/)在线工具绘制基因结构图[35]。
1.5 烟草Hsp70基因的荧光定量表达分析
在亚细胞定位的结果中选择定位于叶绿体的4个烟草Hsp70(NtHsp70Chl)基因,由于其相似性程度较高,特设计共用引物。将提取的RNA采用NanoDrop 2000分光光度计(Thermo Scientific,USA)(OD260/OD280)测定浓度,琼脂糖凝胶电泳检测RNA完整性,RNA逆转录试剂HiScript II Q RT SuperMix for qPCR由Vazyme公司生产(货号:R223-01)。依据基因序列信息,通过Roche LCPDS2软件设计特异性引物,并由上海捷瑞生物工程有限公司合成,采用烟草L25作为内参基因,荧光定量PCR引物和扩增产物如表1。PCR使用的试剂盒为QuantiFast? SYBR? Green PCR Kit (Qiagen,Germany),并在荧光定量LightCycler? 480 Ⅱ型PCR仪(Roche,Swiss)上进行反应。PCR反应程序:95 ℃ 5 min;95 ℃ 10 s,60 ℃ 30 s,40个循环。根据熔解曲线检测扩增产物特异性:从60 ℃缓慢升温至97 ℃,每1 ℃采集5次荧光信号。依据2–ΔΔCT对数据进行换算[36]。
2 结 果
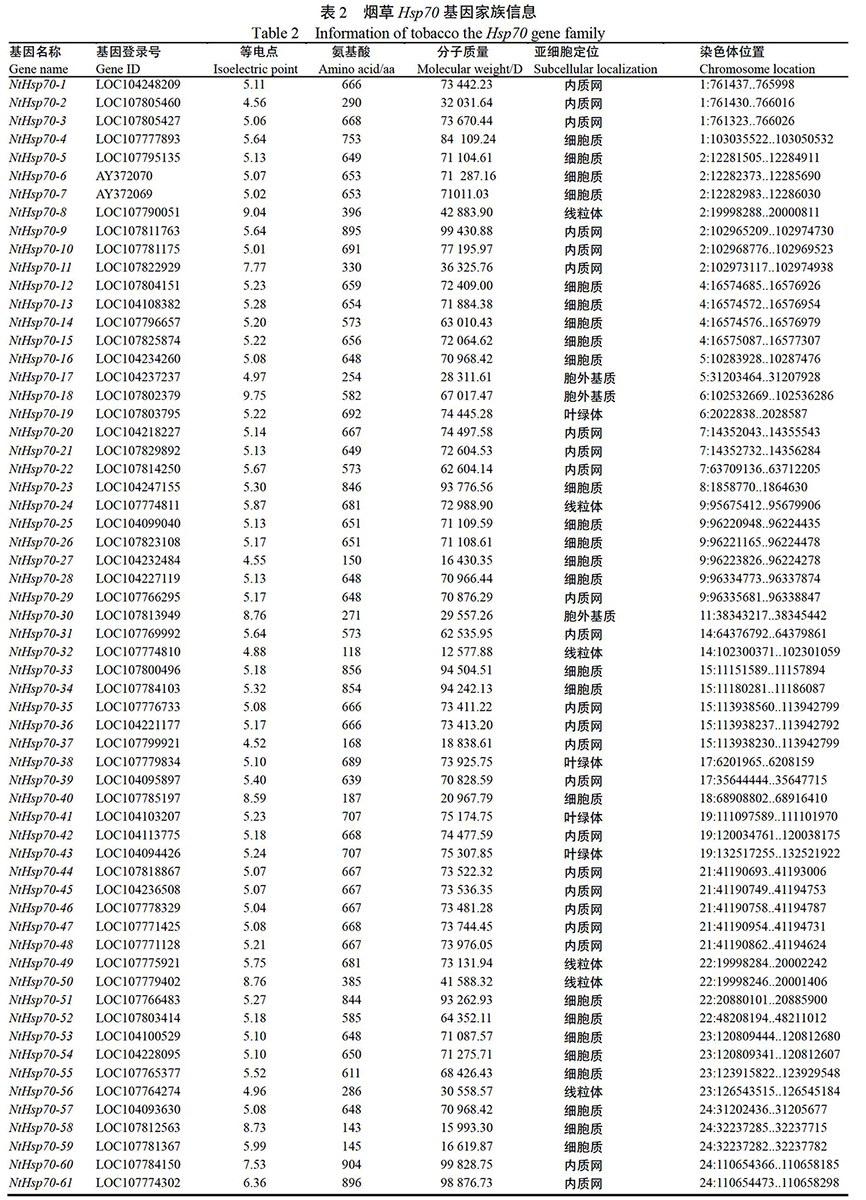
2.1 烟草Hsp70家族成员信息
在烟草全基因组数据中检索,分析获得61个烟草Hsp70家族成员,依据各成员在染色体中的定位结果,依次对其进行命名为NtHsp70-(1-61)。通过分析NtHsp70家族成员理化特性,等电点的范围为4.52(NtHsp70-37)至9.75(NtHsp70-18);Hsp70蛋白序列最长的为NtHsp70-60,由904个氨基酸组成,最短的序列为NtHsp70-32,由118个氨基酸组成。亚细胞定位结果表明,NtHsp70蛋白多定位于细胞质及内质网中,其家族成员数量分别为25和23个;6个家族成员定位于线粒体;4个家族成员(NtHsp70-19、NtHsp70-38、NtHsp70-41和NtHsp70-43)定位于叶绿体;仅3个家族成员定位于胞外基质。NtHsp70家族成员的详情信息见表2。
2.2 烟草Hsp70家族成员进化与基因结构分析
以烟草Hsp70基因家族的蛋白序列与拟南芥AtHsp70-1(CAB85987)、AtHsp70-2(CAB85986)、AtHsp70-3(AAF14038)、AtHsp70-4(BAB02269)、AtHsp70-5(AAF18501)、AtHsp70-6(CAB45063)、AtHsp70-7(BAA97012)、AtHsp70-8(AAD15393)、AtHsp70-9(CAB37531)、AtHsp70-10(CAB89371)、AtHsp70-11(AAF88019)、AtHsp70-12(BAB08435)、AtHsp70-13(AAB70400)、AtHsp70-14(AAG52240)、AtHsp70-15(AAG52244)、AtHsp70-16(AAD30257)、AtHsp70-17(CAB46039)和AtHsp70-18(AAG51503)的蛋白序列共同组建系统进化树(图1)。筛选进化树获得最终聚类结果,依据拓扑结构及拟南芥家族组别划分将其分为Ⅰ~Ⅵ组,其成员数分别为9、21、2、12、9、8个。Ⅰ、Ⅱ和Ⅲ组内的蛋白质主要定位于细胞质(Ⅰ组NtHsp70-17和NtHsp70-22、Ⅱ组NtHsp70-18、NtHsp70-29、NtHsp70-39、NtHsp70-60和NtHsp70-61除外),Ⅳ和Ⅴ组成员主要位于内质网(Ⅴ组NtHsp70-8、NtHsp70-27、NtHsp70-30和NtHsp70-32除外),Ⅵ组中的蛋白定位于能量供应细胞器中,其中Ⅵ-a亚组的NtHsp70成员定位于叶绿体,Ⅵ-b亚组成员位于线粒体;Ⅰ和Ⅴ组属于Hsp110/SSE亚家族,Ⅱ、Ⅲ、Ⅳ、Ⅵ组属于Dnak亚家族。NtHsp70具有5对旁系同源基因(NtHsp70-4/55、NtHsp70-19/38、NtHsp70-23/51、NtHsp70-33/34、NtHsp70-41/43)。Ⅰ组外显子主要集中在5'端,Ⅱ組外显子主要集中在3'端,Ⅲ组外显子无内含子或内含子序列较短,Ⅳ组外显子分散存在于3'端,Ⅴ和Ⅵ组的外显子分布无明显规律。
2.3 烟草Hsp70基因的染色体定位
为明确烟草Hsp70基因家族的组成与分布形式,依据其定位信息,制作染色体分布的信息图(图2)。烟草的24条染色体内分布61个NtHsp70基因,其在染色体中的分布位置有所不同,多数分布于染色体的端部;每条染色体中的NtHsp70基因个数具有差异,2号染色体含有7个基因家族成员,其成员数目最多,9号染色体含有6个家族成员,15、21和24号染色体均包含5个家族成员,1、4、22和23号染色体均具有4个家族成员,7和19号染色体均有3个家族成员,5、6、14和17号染色体中有2个家族成员,8、11和18号染色体成员数最少,均为1个基因家族成员,于叶绿体中表达的基因分别位于6、17和19号染色体,目前3、10、12、13、16和20号染色体无任何NtHsp70基因家族成员信息。通过分析基因之间的关系,发现NtHsp70基因家族中共存在10个基因复制,分别为1组(NtHsp70-44至NtHsp70-48)基因簇、5对串联重复(NtHsp70-10/11、NtHsp70-13/14、NtHsp70-36/37、NtHsp70-49/50、NtHsp70-58/59)和4对片段重复(NtHsp70-16/57、NtHsp70-20/42、NtHsp70-22/31、NtHsp70-28/52)。基因重复促进烟草Hsp70家族成员扩增。
2.4 NtHsp70Chl在烟草叶片主脉组织中的表达
[4]LI Z, LONG R, ZHANG T, et al. Molecular cloning and functional analysis of the drought tolerance gene MsHSP70 from alfalfa (Medicago sativa L.)[J]. Journal of Plant Research, 2017, 130(2): 387-396.
[5]SU P H, LI H M. Stromal Hsp70 is important for protein translocation into pea and Arabidopsis chloroplasts[J]. Plant Cell, 2010, 22(5): 1516-1531.
[6]LIU C, WILLMUND F, GOLECKI J R, et al. The chloroplast HSP70B-CDJ2-CGE1 chaperones catalyse assembly and disassembly of VIPP1 oligomers in Chlamydomonas[J]. Plant Journal, 2007, 50(2): 265-277.
[7]LATIJNHOUWERS M, XU X M, M?LLER S G. Arabidopsis stromal 70-kDa heat shock proteins are essential for chloroplast development[J]. Planta, 2010, 232(3): 567-578.
[8]HUANG Y W, HU C C, TSAI C H, et al. Chloroplast Hsp70 isoform is required for age-dependent tissue preference of bamboo mosaic virus in mature Nicotiana benthamiana leaves[J]. Molecular Plant-Microbe Interactions, 2017, 30(8): 631-645.
[9]PARK S M, HONG C B. Class I small heat-shock protein gives thermotolerance in tobacco[J]. Journal of Plant Physiology, 2002, 159(1): 25-30.
[10]TOGNETTI V B, PALATNIK J F, FILLAT M F, et al. Functional replacement of ferredoxin by a cyanobacterial flavodoxin in tobacco confers broad-range stress tolerance[J]. Plant Cell, 2006, 18(8): 2035-2050.
[11]SHOU H, BORDALLO P, WANG K. Expression of the Nicotiana protein kinase (NPK1) enhanced drought tolerance in transgenic maize[J]. Journal of Experimental Botany, 2004, 55(399): 1013-1019.
[12]SUNG D Y, KAPLAN F, Lee K J, et al. Acquired tolerance to temperature extremes[J]. Trends in Plant Science, 2003, 8(4):179-187.
[13]DRAGOVIC Z, BROADLEY S A, SHOMURA Y, et al. Molecular chaperones of the Hsp110 family act as nucleotide exchange factors of Hsp70s[J]. EMBO Journal, 2006, 25(11): 2519-2528.
[14]MAYER M P, BUKAU B. Hsp70 chaperones: cellular functions and molecular mechanism[J]. Cellular & Molecular Life Sciences, 2005, 62(6): 670-684.
[15]JUNGKUNZ I, LINK K, VOGEL F, et al. AtHsp70-15-deficient Arabidopsis plants are characterized by reduced growth, a constitutive cytosolic protein response and enhanced resistance to TuMV[J]. Plant Journal, 2011, 66(6): 983-995.
[16]MONTERO-BARRIENTOS M, HERMOSA R, CARDOZA R E, et al. Transgenic expression of the Trichoderma harzianum hsp70 gene increases Arabidopsis resistance to heat and other abiotic stresses[J]. Journal of Plant Physiology, 2010, 167(8): 659-665.
[17]AUGUSTINE S M, NARAYAN J A, SYAMALADEVI D P, et al. Erianthus arundinaceus HSP70 (EaHSP70) overexpression increases drought and salinity tolerance in sugarcane ( Saccharum, spp. hybrid)[J]. Plant Science, 2015, 232: 23-24.
[18]MATHIOUDAKIS M M, VEIGA R, GHITA M, et al. Pepino mosaic virus capsid protein interacts with a tomato heat shock protein cognate 70[J]. Virus Research, 2012, 163(1): 28-39.
[19]RENNER T, WATERS E R. Comparative genomic analysis of the Hsp70s from five diverse photosynthetic eukaryotes[J]. Cell Stress & Chaperones, 2007, 12(2): 172-185.
[20]LIN B L, WANG J S, LIU H C, et al. Genomic analysis of the Hsp70 superfamily in Arabidopsis thaliana[J]. Cell Stress & Chaperones, 2001, 6(3): 201-208.
[21]YER E N, BALOGLU M C, ZIPLAR U T, et al. Drought-responsive Hsp70 gene analysis in Populus at genome-wide level[J]. Plant Molecular Biology Reporter, 2016, 34(2): 483-500.
[22]SARKARE N K, KUNDNANI P, GROVER A. Functional analysis of Hsp70 superfamily proteins of rice (Oryza sativa)[J]. Cell Stress & Chaperones, 2013, 18(4): 427-437.
[23]ZHANG L, ZHAO H K, DONG Q L, et al. Genome-wide analysis and expression profiling under heat and drought treatments of HSP70 gene family in soybean (Glycine max L.)[J]. Frontiers in Plant Science, 2015, 6: 773.
[24]BARRETT T, WILHITE S E, LEDOUX P, et al. NCBI GEO: archive for functional genomics data sets-update[J]. Nucleic Acids Research, 2013, 41(1): 991-995.
[25]FERNANDEZ-POZO N, MENDA N, EDWARDS J D, et al. The Sol Genomics Network (SGN)-from genotype to phenotype to breeding[J]. Nucleic Acids Research, 2015, 43(1): 1036-1041.
[26]SWARBRECK D, WILKS C, LAMESCH P, et al. The Arabidopsis Information Resource (TAIR): gene structure and function annotation[J]. Nucleic Acids Research, 2008, 36(1): 1009-1014.
[27]LAMESCH P, BERARDINI T Z, LI D, et al. The Arabidopsis Information Resource (TAIR): improved gene annotation and new tools[J]. Nucleic Acids Research, 2012, 40(1): 1202-1210.
[28]ZHAO Q, GAO J, SUO J, et al. Cytological and proteomic analyses of horsetail (Equisetum arvense L.) spore germination[J]. Frontiers in Plant Science, 2015, 6: 441.
[29]VIZCA?NO J A, DEUTSCH E W, WANG R, et al. ProteomeXchange provides globally coordinated proteomics data submission and dissemination[J]. Nature Biotechnology, 2014, 32(3): 223-226.
[30]陳继圣,郑士琴,郑武,等. 全基因组预测稻瘟菌的分泌蛋白[J]. 中国农业科学,2006,39(12):2474-2482.
CHEN J S, ZHENG S Q, ZHENG W, et al. Prediction for secreted proteins from Magnaporthe grisea genome[J]. Scientia Agricultura Sinica, 2006, 39(12): 2474-2482.
[31]PATEL S, PANCHAL H, ANJARIA K. Phylogenetic analysis of some leguminous trees using CLUSTALW2 bioinformatics tool[C]//IEEE International Conference on Bioinformatics and Biomedicine Workshops. IEEE, 2013: 917-921.
[32]TAMURA K, STECHER G, PETERSON D, et al. MEGA6: Molecular Evolutionary Genetics Analysis version 6.0[J]. Molecular Biology & Evolution, 2013, 30(12): 2725-2729.
[33]周喆,張彩霞,张利义,等. 苹果LysM基因家族的生物信息学及表达分析[J]. 中国农业科学,2014,47(13):2602-2612.
ZHOU Z, ZHANG C X, ZHANG L Y, et al. Bioinformatics and expression analysis of the LysM gene family in apple[J]. Scientia Agricultura Sinica, 2014, 47(13): 2602-2612.
[34]YANG S, ZHANG X, YUE J X, et al. Recent duplications dominate NBS-encoding gene expansion in two woody species[J]. Molecular Genetics & Genomics, 2008, 280(3): 187-198.
[35]郭安源,朱其慧,陈新,等. GSDS:基因结构显示系统[J]. 遗传,2007(8):1023-1026.
GUO A Y, ZHU Q H, CHEN X, et al. GSDS: a gene structure display server[J]. Hereditas(Beijing), 2007, 29(8): 1023-1026.
[36]DING X, ZHANG X, YANG Y, et al. Polymorphism, expression of natural resistance-associated macrophage protein 1 encoding gene (NRAMP1) and its association with immune traits in pigs[J]. Asian-Australasian Journal of Animal Sciences, 2014, 27(8): 1189-1195.
[37]KOSE S, IMAMOTO N. Nucleocytoplasmic transport under stress conditions and its role in HSP70 chaperone systems[J]. Biochimica et Biophysica Aata, 2014, 1840(9): 2953-2960.
[38]WANG J, LEE J, LIEM D, et al. HSPA5 gene encoding Hsp70 chaperone BiP in the endoplasmic reticulum[J]. Gene, 2017, 618: 14-23.
[39]HAN S, LIU Y, CHANG A. Cytoplasmic Hsp70 promotes ubiquitination for endoplasmic reticulum- associated degradation of a misfolded mutant of the yeast plasma membrane ATPase, PMA1[J]. Journal of Biological Chemistry, 2007, 282(36): 26140-26149.
[40]SUNG D Y, VIERLING E, GUY C L. Comprehensive expression profile analysis of the Arabidopsis Hsp70 gene family[J]. Plant Physiology, 2001, 126(2): 789-800.
[41]LENG L, LIANG Q, JIANG J, et al. A subclass of HSP70s regulate development and abiotic stress responses in Arabidopsis thaliana[J]. Journal of Plant Research, 2016, 130(2): 349-363.
[42]SCHRODA M, VALLON O, WOLLMAN F A, et al. A chloroplast-targeted heat shock protein 70 (HSP70) contributes to the photoprotection and repair of photosystem II during and after photoinhibition[J]. Plant Cell, 1999, 11(6): 1165-1178.
[43]WANG Y, XU T, PATERSON A H. Different patterns of gene structure divergence following gene duplication in Arabidopsis[J]. BMC Genomics, 2013, 14(1): 652-660.
[44]王明强,张道远. 植物热激蛋白70基因家族及其生物学功能研究进展[J]. 基因组学与应用生物学,2015,34(2):421-428.
WANG M Q, ZHANG D Y. Research advance of heat shock protein 70 gene family and its biological functions in plant[J]. Genomics and Applied Biology, 2015, 34(2) : 421-428.

