基于Cytoscape的miRNA调控网络的构建与研究*
杨 淼 杜 菁* 李冬果 杨秋英 刘文艳 姚红串
近年来,非编码核糖核酸(ribonucleic acid,RNA),尤其是微小RNA(microRNA,miRNA)的研究越来越引人关注。miRNA是在真核生物中广泛存在的一类重要的非编码RNA,目前有超过2 500种miRNA已经在人类中注释[1]。成熟的miRNA是一类大小约21~23个核苷酸的非编码RNA,miRNA通过调节靶基因的表达水平影响到各种生物过程,包括发育、病毒防御、造血过程、器官形成、细胞增殖和凋亡以及脂肪代谢等。一个miRNA可以同时调控多个靶基因的表达,多个miRNA通过协同或组合关系调节靶基因[2]。因此miRNA与靶基因错综复杂的相互作用构成一种复杂的调控网络(regulatory network),称为MiRNA-Target调控网络。在这个调控网络中,通过有向边,描述miRNA与靶基因之间的调控与被调控关系。
有研究表明,miRNA在调节癌症基因表达程序中具有重要作用,一些miRNA起肿瘤抑制剂的作用,而另一些miRNA异常过表达可促进肿瘤的发生、生长或进展[3]。因此,miRNA在调控网络中的作用有助于理解高等真核生物的基因组的复杂性[4]。
随着miRNA调控基因表达研究的逐步深入,在MiRNA-Target调控网络中的节点数日趋庞大,要了解miRNA和靶基因之间复杂的“多对多”关系,最直观方式是使用基于网络的可视化方法,借助图论理论进行分析。
Cytoscape是一个基于Java技术的开放源代码的网络可视化软件平台,主要用于复杂生物网络分析研究设计,其可以绘制基因表达调控网络、蛋白互作网络等任何与网络结构、层级有关系的内容。Cytoscape软件为数据集成、分析和可视化提供了一套基本的功能,同时Cytoscape软件拥有大量的插件,这些插件提供了丰富的生物网络分析功能,目前Cytoscape软件的版本已经更新到3.6.1版。
1 材料与方法
1.1 miRNA数据获取
乳腺癌(breast cancer,BC)是迄今为止妇女中最常见的癌症。近年来,人们对在BC中表达异常的miRNA表达机制的研究已经取得了相当大的进展,并且发现了一些miRNA或miRNA家族作为BC标记的关键调控器[5-6]。尽管可以通过实验查找miRNA靶标基因,但是实验既耗时又昂贵,因此开发了大量的预测工具来预测miRNA的靶基因。
本研究选用BC与hsa-miR-125b-2-3p、hsamiR-125b-5p、hsa-miR-139-5p、hsa-miR-195-3p、hsa-miR-195-5p、hsa-miR-204-5p、hsa-miR-497-3p、hsa-miR-497-5p、hsa-miR-99a-3p和hsa-miR-99a-5p 10个最有差异性表达的下调miRNAs作为诊断BC的miRNA标志物,构建miRNA预测靶基因的调控网络[7]。采用生物网络分析软件Cytoscape3.6.1可视化乳腺癌的miRNA与预测靶基因之间的调控关系,并对靶基因进行基因本体(gene ontology,GO)富集分析和京都基因和基因组百科全书(kyoto encyclopedia of genes and genomes,KEGG)信号通路分析。
1.2 预测靶基因数据获取
miRWalk是迄今为止唯一自由开放,有关miRNA靶标相互作用的最大的数据库在线资源,目前miRWalk2.0提供了智人、小鼠和大鼠的miRNA靶基因信息。miRWalk数据内容分为预测模块和验证模块。预测模块提供了预测miRNA靶基因的功能,并与12个miRNA靶基因预测程序的预测结果进行比对。本研究使用miRWalk数据库获取miRNA的预测靶基因,采用TargetScan、Pictar2、miRanda以及RNA22预测工具[8]和miRWalk完成预测。为了降低假阳性率,同时被这5种方法预测标记的靶基因被选为调控网络中miRNA的预测靶基因。
1.3 miRNA-Target调控网络构建
Cytoscape软件支持多种格式输入,选择Cytoscape软件菜单“File|import|network|File”输入调控网络数据,并设置源点Source列和目标Target列及其属性列。生成初步的调控网络。
1.4 样式设置和网络布局
Cytoscape软件可用于设置节点(Node)、边(Edge)和网络(Network)的样式。选择Cytoscape软件“Control Panel”控制面板中的“Style”选项卡可对节点、边和网络进行样式设置。
网络布局主要设置节点和边在视图中的几何位置,根据网络的拓扑结构计算出各节点的几何位置,直观显示网络的拓扑结构。Cytoscape软件提供了多种布局算法,本研究选择力导向算法布局miRNA调控网络。力导向布局算法模拟力学物理,将节点建模为物理对象,将边作为将这些对象连接在一起的弹簧来布局整个网络。该算法可以快速的布局复杂网络中的节点,绘制一个清晰的网络布局图。选择Cytoscape软件菜单命令“Layout|Prefuse Force Directed Layout”,运行力导向布局,其中弹性系数为“1E-5”,弹性长度为“6”。
1.5 基因富集分析和KEGG通路分析
Cytoscape软件提供的插件生物网络基因本体论工具(biological networks gene ontology tool,BiNGO),将识别的靶基因映射到GO的注释库中,对预测靶基因集合进行GO富集分析,实现靶基因的生物学功能识别。BiNGO可以构建基于基因集合的生物过程(biological process,BP)、分子功能(molecular function,MF)、细胞组分(cell component,CC)的网络结构图,并找出富集的生物学注释。本研究对预测靶基因进行生物过程富集分析。在Cytoscape软件中,打开miRNA调控网络,选中需要映射的基因节点;选择Cytoscape菜单“Apps|BiNGO”,打开BiNGO对话框进行设置;选择统计学检验“Hypergenometric test”,以P<0.05为显著性阈值,分别选择GO的BP作为映射文件,选择物种“Homo Sapiens”后,点击“Start BiNGO”按钮,运行BiNGO。
KEGG通路分析可用于发现共享相同通路的共表达基因簇,通过miRWalk数据库完成KEGG通路分析,显著性阈值P<0.05。
2 结果
Cytoscape软件采用力导向布局绘制的MiRNATarget调控网络中大圆角矩形为miRNA,小圆形为预测的靶基因。调控网络中一共预测了791个靶基因,1062对调控关系。从miRNA调控网络可直观地看出,一个miRNA可调控多个靶基因,多个靶基因又同时被多个miRNA所调控(如图1所示)。
使用Cytoscape软件的插件BiNGO做GO富集分析,通过链接GO数据库得到靶基因的注释信息。结果显示,10个miRNA标志物中hsa-miR-195-3p、hsa-miR-139-5P、hsa-miR-195-5p和hsa-miR-497-5p在BiNGO富集分析中,有显著性差异。靶基因显著性富集的前5个生物学过程为生物调节,细胞过程调节,大分子代谢过程的调节和翻译后蛋白质的修饰。同时构建GO富集的生物学过程有向无环图,本研究仅列出hsa-miR-497-5p的靶基因的生物学过程有向无环图,其中有颜色的代表有显著性富集,颜色越深代表富集程度越高(如图2所示)。
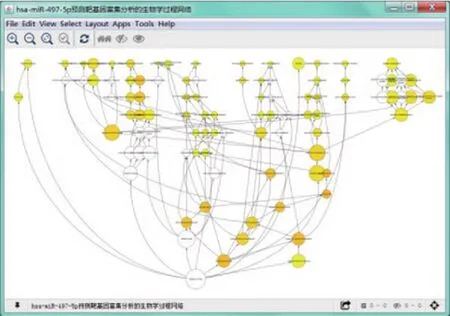
图2 hsa-miR-497-5p预测靶基因富集分析生物学过程层次网络图
对靶基因的KEGG通路进行分析,结果得到303个基因富集在85条通路上。其中,有7条通路与乳腺癌有关,分别是p53信号通路,Hedgehog信号通路,Wnt信号通路,Notch信号通路,Jak STAT信号通路,Toll样受体信号通路和ErbB信号通路[9-12]。
3 结论
本研究通过5种预测方法预测出791个靶基因,利用预测的调控数据,建立了乳腺癌的肿瘤标志物MiRNA-Target调控网络,该网络中包含801个节点,791个预测靶基因,1062对调控关系,并用可视化工具Cytoscape软件构建并布局MiRNA-Target调控网络,对miRNA与靶基因之间的调控关系有了初步的了解。分析结果显示,10个miRNA标志物中,预测的靶基因在生物学过程中主要富集在调节功能上。KEGG通路分析中,预测的靶基因主要富集在7种信号通路中,这些通路与乳腺癌存在一定关系。
从生物信息角度,预测miRNA靶基因的算法,BiNGO富集分析,KEGG通路分析有助于找出要研究的基因和通路,为乳腺癌靶基因的实验验证及生物学功能的研究提供了理论指导,有助于乳腺癌的靶向治疗。

