基于簇毛麦No.1026转录组的SSR序列分析及其PCR标记开发
陈竟男,马晓兰,王振,李仕金,谢皓,叶兴国,林志珊
基于簇毛麦No.1026转录组的SSR序列分析及其PCR标记开发
陈竟男1,2,马晓兰1,王振3,李仕金1,谢皓2,叶兴国1,林志珊1
(1中国农业科学院作物科学研究所/农作物基因资源与基因改良国家重大科学工程/农业部麦类生物学与遗传育种重点实验室,北京 100081;2北京农学院植物科学技术学院,北京 102206;3北方民族大学生物工程与科学学院,银川 750021)
【目的】探究从前苏联引进的簇毛麦No.1026(Dv#4)的EST-SSR序列特征及其在染色体的分布,分析它们在不同簇毛麦间及与小麦间的多态性,为其进一步的研究与利用提供依据。【方法】通过Illumina HiSeq测序获得No.1026植株的转录组序列,利用MISA软件分析转录组SSR序列及特征,采用Primer 3设计SSR引物,随机合成238对引物,对小麦中国春与簇毛麦Dv#4和引自英国剑桥的簇毛麦Dv#2的基因组DNA进行扩增,在琼脂糖凝胶上分离评价扩增产物的多态性,并利用一套小麦-簇毛麦异染色体系进行扩增分析。【结果】检测了No.1026总长62.76 Mb的转录组序列,发现10 497个SSR位点,它们分布于8 735条Unigene上。在1—6个核苷酸的重复单元中,单、双、三碱基的重复占95.85%,其中三核苷酸串联重复数量最多,占SSR总数的50.33%,而CCG/CGG基序的重复占三核苷酸串联重复的41.66%;单核苷酸重复是第二大类型,出现频率为27.13%,其中A/T重复占单核苷酸重复的74.58%。二核苷酸重复类型的数量位列第三,占SSR总数的18.39%。在238对EST-SSR引物中,88对在中国春与簇毛麦(包括Dv#2和Dv#4)之间的扩增产物显示多态性;8对只在簇毛麦和单一异染色体系扩增;4对可在簇毛麦和多个异染色体系中特异扩增;但多数可在簇毛麦中清晰扩增的引物不能在任何异染色体系中扩增,推测可能与簇毛麦基因组及染色体导入小麦过程中的变异有关;47对(19.74%)在2份不同来源的簇毛麦Dv#2和Dv#4之间的扩增产物呈现多态性。利用1对EST-SSR引物和1个EST-PCR标记检测48个簇毛麦植株,证明多态性的SSR引物可有效用于检测簇毛麦的异质性。【结论】簇毛麦No.1026(Dv#4)的转录组中存在丰富的SSR序列,其中CCG/CGG、A/T和AG/CT等三核苷酸、单核苷酸和二核苷酸是其最主要的串联重复基序。部分EST-SSR的侧翼保守序列与单一或若干外源染色体特异相关联,据此开发特异的分子标记,可用于跟踪检测小麦背景中特异的簇毛麦染色体或染色体片段。Dv#4与Dv#2间的部分EST-SSR具有多态性,提示2份不同来源簇毛麦的表达序列间存在一定程度的遗传差异,因此,簇毛麦Dv#4值得进一步的发掘研究。
簇毛麦;转录组;EST-SSR;异染色体系;染色体定位;遗传多样性
0 引言
【研究意义】簇毛麦((L.) P. Candargy, syn.Schur)是小麦的二倍体近源种属(2n=2x=14,VV),为一年生异花授粉植物,主要分布于地中海的东北部和高加索地区[1]。由于簇毛麦表现出许多生物胁迫的抗性(如抗条锈、秆锈、白粉、梭条花叶病毒病、瘿螨等多种小麦主要病虫害)和非生物胁迫抗性(如耐寒、耐盐、抗旱),同时拥有分蘖力强、生长繁茂、多小花、籽粒蛋白质含量高等重要农艺性状[1-3],因此,受到广泛关注,并被认为是小麦改良的潜在优异基因源。【前人研究进展】前人利用不同来源的簇毛麦开展了有益基因导入小麦的研究,培育了硬粒小麦-簇毛麦双二倍体[4-6]、普通小麦-簇毛麦异附加系、异代换系和易位系[6-10],鉴定了位于6V#2S和6#4S上的白粉病抗性基因[7-10],并克隆了主效抗病基因[11-12]。Zhang等[13]培育了抗小麦梭条花叶病毒病的T4VS.4DL易位系,将抗梭条花叶病毒病基因定位在4VS上。Zhao等[14]进一步利用CSph1b诱导获得4VS小片段易位,将定位在4VS染色体臂近端部FL为0.78—1.00区间。研究还表明,簇毛麦所特有的护颖颖脊刚毛的形态特征由2V染色体短臂上的基因所控制[15],而1V、5V染色体分别携带高分子量麦谷蛋白亚基(HMW-GS)基因和籽粒软质基因。此外,5V还具有成株期的白粉病抗性,它们已被导入普通小麦,分别育成T1DL·1VS、T1DS·1VL和T5VS·5DL易位系[16-18]。Qi等[19]利用一套源于簇毛麦Dv#3,并由Lukaszewski培育的小麦-簇毛麦二体异附加系进行鉴定,发现DA6V#3对锈病小种Ug99表现出较好的抗性,开发了7对6V#3特异的EST–STS标记用于从6D和6V#3染色体双单体的F2群体中筛选易位系,并在F3世代获得生长和育性正常的纯合易位系T6AS.6V#3L和T6AL.6V#3S,随之将抗锈病相关的基因定位于染色体长臂6V#3L,命名为。是一个温敏型基因,其作用在16℃最有效,而28℃失效。【本研究切入点】上述研究所涉及的簇毛麦大多来源于Dv#2以及Dv#3,而从前苏联引进的簇毛麦No.1026(Dv#4)除了6V#4染色体代换系及6V#4S.6DL易位系外,涉及其他染色体性状的研究鲜见报道。而且,尽管先前的研究表明,6V#4与6V#2间在病虫抗性[20]、染色体结构[21]、基因序列[22-26]间均存在多态性,但不同来源的簇毛麦基因组间的多态性还缺少深入研究。据报道,SSR标记的多态性明显高于其他类型的分子标记[27-28],但传统开发SSR标记引物过程繁琐,需要构建文库、筛选重复序列、测序等环节,成本高,时间长[29]。近年来,随着测序技术的发展及成本的下降,利用EST或转录组序列开发SSR标记变得更为简便快速[30-31]。因此,EST-SSR标记目前已被广泛应用于植物的遗传连锁图谱构建[32]、种质资源的遗传多样性分析[33-34]等研究。【拟解决的关键问题】本研究利用簇毛麦No.1026转录组测序数据分析表达序列中的SSR序列及特征;基于侧翼保守区序列设计引物,分析EST-SSR序列在不同簇毛麦间的多态性;利用一套小麦-簇毛麦附加系或代换系对这些标记进行染色体定位;筛选在小麦中国春与簇毛麦间具有多态性的标记,为No.1026的进一步研究和利用奠定基础。
1 材料与方法
1.1 植物材料
材料包括2份不同来源的簇毛麦:英国剑桥引进的簇毛麦和前苏联引进的簇毛麦No.1026,按照对不同来源簇毛麦的区分方法[35,10],分别用Dv#2和Dv#4表示。2份簇毛麦的种子均由中国农业科学院作物科学研究所陈孝研究员保存并提供,其中,源于英国剑桥的簇毛麦Dv#2原由南京农业大学陈佩度教授惠赠;小麦-簇毛麦异附加系DA2V#2、DA3V#2、DA4V#2、DA5V#2、DA6V#2和DA7V#2由南京农业大学王秀娥教授惠赠;江苏里下河地区农业科学研究所别同德博士提供了小麦-簇毛麦异代换系1V#2(1D)的DNA。为方便描述,本文将上述异附加系与异代换系统称为异染色体系,用于EST-SSR的染色体定位分析。中国农业科学院作物科学研究所小麦抗逆分子育种课题组收集保存的小麦品种中国春为对照。
1.2 转录组数据获得及EST-SSR引物设计
转录组数据来源于笔者所在实验室2016年对前苏联簇毛麦No.1026(Dv#4)进行高通量测序的结果[25-26]:取温室盆栽的植株成株期叶片,利用Trizol法提取RNA,委托北京诺禾致源生物信息科技有限公司完成cDNA文库构建和HiSeq测序。使用软件MISA(27/09/2010)http://pgrc.ipk-gatersleben.de/misa/misa.html进行SSR分析:设定各个重复单元的最少重复次数分别为1—10、2—6、3—5、4—5、5—5和6—5,即:以单核苷酸为重复单元时,其重复数至少为10次才可被检测到;以双核苷酸为重复单元时,其最少重复数为6次;以三、四、五、六核苷酸为重复单元的重复次数均需达到5次以上。找出SSR序列之后,采用Primer3进行SSR引物设计。
1.3 DNA提取及其引物扩增条件
采用DNA提取试剂盒(北京康为世纪生物科技有限公司)按说明书提取每份材料新鲜叶片的基因组DNA,并稀释成100 ng·μL-1,4℃保存备用。238对随机选取的EST-SSR引物由生工生物工程(上海)股份有限公司合成。先用中国春、DV#2、DV#4的DNA进行引物初筛选;对小麦与簇毛麦扩增产物间具有多态性的引物进一步用一套DV#2异附加系进行扩增,分析RST-SSR位点的染色体分布。PCR扩增体系为MIX 7.5 μL、上下游引物各0.5 μL、模板1 μL和dd H2O 5.5 μL,混合均匀后置于PCR仪(T100-Thermal Cycler, BIO-RAD)中扩增。扩增程序为95℃5 min;94℃30 s,54—60℃30 s(退火温度因不同引物而异),72℃1 min,34个循环;72℃8 min,4℃保存。
2 结果
2.1 源于簇毛麦Dv#4转录组数据的SSR序列构成特性分析
在总长62.76 Mb的转录组47 384条序列中,共检测到10 497个SSR位点,平均每5.98 kb有1个位点,它们分布于8 735条Unigene上。其中含有1个以上SSR位点的Unigene有1 442条。SSR有单一型和混合型2种类型,单一型重复基序的SSR位点数9 979个,混合型重复基序的位点数为518个。SSR包括单碱基重复2 848个、双碱基重复1 930个、三碱基重复5 283个、四碱基重复394个、五碱基重复36个和六碱基重复6个(表1)。可见,1—3个碱基重复是No.1026转录序列中主要的SSR重复类型,而三核苷酸串联重复数量最多,占SSR总数的50.33%,其构成基序主要有CCG/CGG、AGG/CCT、AGC/CTG、ACC/GGT、AAG/CTT、ACG/CGT等10种,其中CCG/CGG基序占三核苷酸串联重复的41.66%。单核苷酸重复类型排列第二位,其出现频率为27.13%,其中A/T重复明显多于G/C重复,占单核苷酸重复的74.58%。二核苷酸重复类型排列第三位,其出现频率为18.39%,以AG为主的重复基序占二核苷酸重复类型的54.61%,而且在拟南芥EST-SSR中极为罕见的GC基序在No.1026中拥有二核苷酸重复的8.13%。4—6核苷酸的重复基序仅为SSR总数的4.15%。
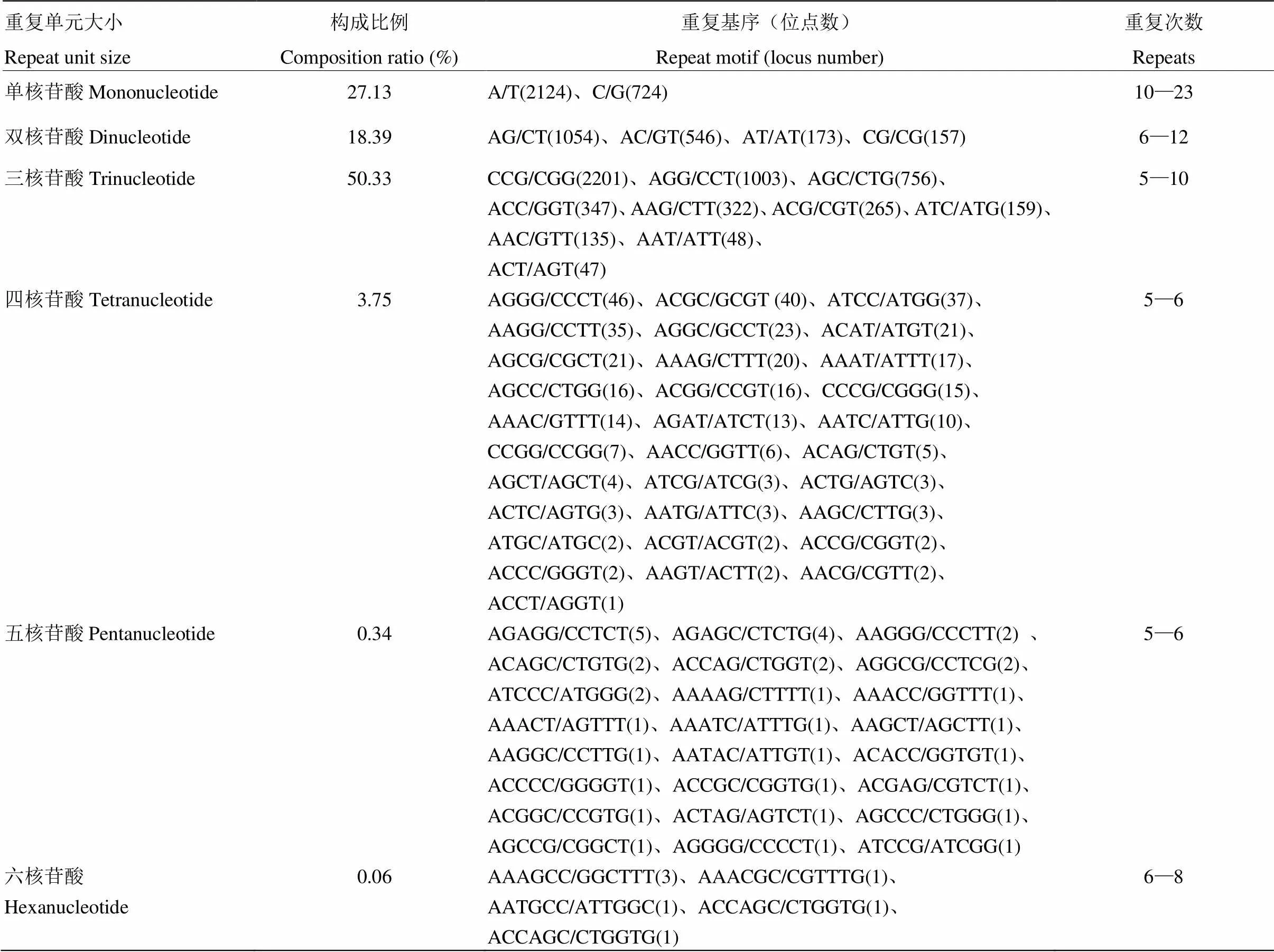
表1 Dv#4的EST-SSR构成
2.2 SSR引物在小麦与簇毛麦间扩增的多态性及在簇毛麦染色体的分布
随机合成238对引物,对包括单核苷酸、二核苷酸、三核苷酸、四核苷酸4种主要重复单元的SSR位点进行引物有效性验证,对CS、簇毛麦Dv#2和Dv#4进行扩增分析。结果显示,88对引物在CS与簇毛麦之间具有扩增产物的多态性。图1显示了部分引物的扩增结果。图中可见,多态性类型分为2种:类型1在簇毛麦中清晰扩增的引物在小麦中没有明显的扩增带,如图1中的5、6、8、24、44、51、54和65,表现为有、无的多态性;类型2则表现为在簇毛麦与小麦间扩增产物的长度多态性。将这些与小麦具有多态性的引物进一步对1V#2(1D)异代换系以及DA2V#2-7V#2异附加系进行扩增,8对引物只在簇毛麦和单一异染色体系中扩增出相同的目的条带(图2),表明这些引物位于簇毛麦单一染色体上,可作为这些染色体特异的EST-SSR标记。
本研究对每一个SSR位点都分别设计了3组引物,8584.0的3组引物p1、p2和p3都可在簇毛麦和4V异附加系扩增出特异带(图2),说明该SSR位点确实位于4V染色体上。引物3155.0p3和13764.0p1可在簇毛麦和二体异附加系DA2V#2特异扩增,而引物18808F3可在簇毛麦和二体异附加系DA5V#2特异扩增。引物1211F2和12613.0F3除了在簇毛麦扩增外,还分别在二体异附加系3V和6V扩增。此外,有4对引物可同时在簇毛麦和多个异染色体系中特异扩增(图3),表明这些SSR位点分布在簇毛麦多条染色体上。其中,引物18131.3751可在簇毛麦DV#2不同的异染色体系上扩增出分子量大小明显不同的产物。

M:Marker 2000的部分区段,条带对应于100、250和500 bp;1—65表示65对不同引物,其扩增产物显示小麦与簇毛麦或簇毛麦间的多态性M: Parts of Marker 2000, the bands are corresponding to 100 bp, 250 bp and 500 bp in size, respectively. 1-65 indicate 65 pairs of EST-SSR primers, the amplified products showing the PCR polymorphic fragments between wheat and D. villosum or betweentwo D. villosum accessions
2.3 特异SSR扩增产物在不同簇毛麦间的多态性
在238对SSR引物中,有47对引物(19.74%)在Dv#2和Dv#4之间显示出多态性。从图1可以看出,部分引物如2、3、4、32和44在Dv#4中具有预期扩增条带,而在Dv#2则没有明显的扩增带。而引物11、14、15、31、42、45、61、63、64及65则在2份簇毛麦间显示扩增片段长度多态性。由于这些SSR引物来自簇毛麦的转录组,说明这两份不同来源簇毛麦的表达序列存在着差异。
2.4 新开发分子标记对簇毛麦异质性的鉴定
曾经将簇毛麦Dv#2与簇毛麦Dv#4近距离种植,用开发的EST-SSR引物225(225F:5’-ACCAGTTGTT AAGGTGGGCC-3’,225R:5’-ACCGTTGTTACGCCG TACAA-3’)和1个定位于6VS(6V-12[26])的EST-PCR标记检测从簇毛麦Dv#2衍生的部分后代植株。6V-12可在原始的2个簇毛麦Dv#2和Dv#4扩增出多态性片段,对48个簇毛麦Dv#2衍生的植株无一例外都稳定扩增出来自6V#2S的特异带,没有6V#4S的特异带。但EST-SSR引物225则扩增出3种类型,不同植株在该SSR位点显示了分离(图4)。说明2份近距离种植的簇毛麦间可能曾经发生过天然的异交,也表明了SSR引物可有效用于簇毛麦异质性的鉴定。
3 讨论
3.1 EST-SSR序列在簇毛麦基因组的构成及其染色体的分布
本研究对来自前苏联簇毛麦No.1026转录组数据的分析结果表明,No.1026的表达序列中存在丰富的SSR序列。根据对拟南芥的研究[36-37],发现转录区的SSR分布呈现从5’端到3’端递减的特性,5’UTR的SSR分布频率显著高于其他转录区,且以二核苷酸AG和三核苷酸AAG重复为主,二核苷酸GC重复基序极其罕见。而基因编码区的SSR序列中,92.6%为三核苷酸及六核苷酸的串联重复序列,认为可能与三联体密码子所受的选择压有关。三核苷酸重复基序又以AAG的频率最高,CCG的基序最少。本研究的结果明显不同于拟南芥,整体上,簇毛麦No.1026转录组中三核苷酸的串联重复序列占SSR总数的50.33%。这与前人对水稻、小麦、大豆、玉米等作物[38]以及杉木[39]的研究结果相类似。而且,CCG/CGG是三核苷酸重复的主要基序,其出现频率占三核苷酸重复的41.66%,这一结果与水稻很相似。根据Gao等[38]的研究,水稻的EST中,平均每100 kb就有23.6个CCG基序的SSR重复,而小麦、玉米和大豆分别为10.3、3.5和0.9。Gao等[38]认为许多三核苷酸可能与重要的基因功能相关,如CCG重复可能涉及胁迫抗性、转录调控、代谢酶的合成、信号转导等基因功能。另外,单核苷酸作为簇毛麦转录组SSR的第二大主要的串联重复单元,A/T出现的频率远高于C/G,这一特征与小麦、水稻、玉米、大豆及拟南芥都是相同的[38,36]。
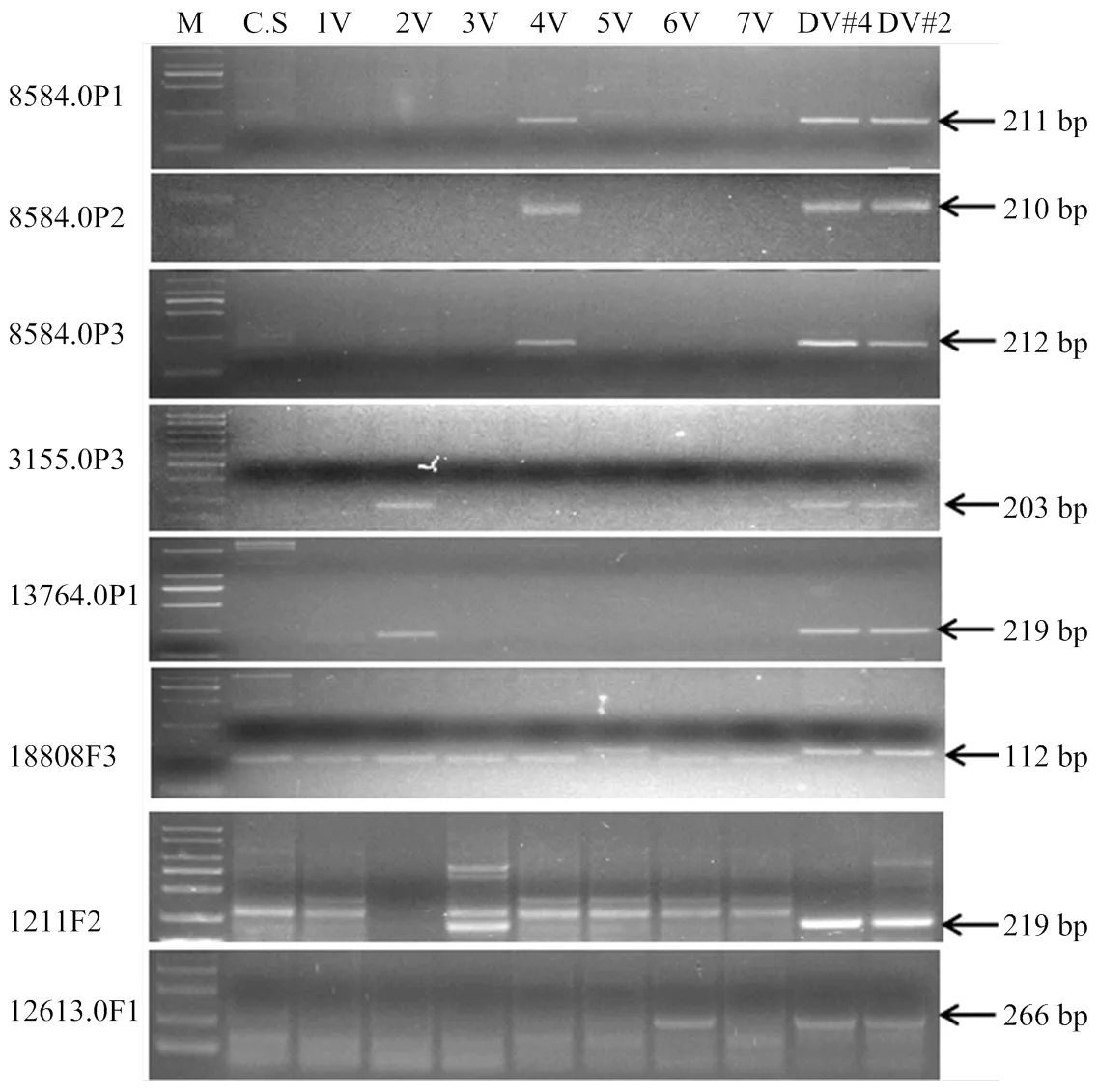
M:Marker 2000,左侧为引物名称,右侧箭头显示引物扩增的特异条带及片段大小。下同
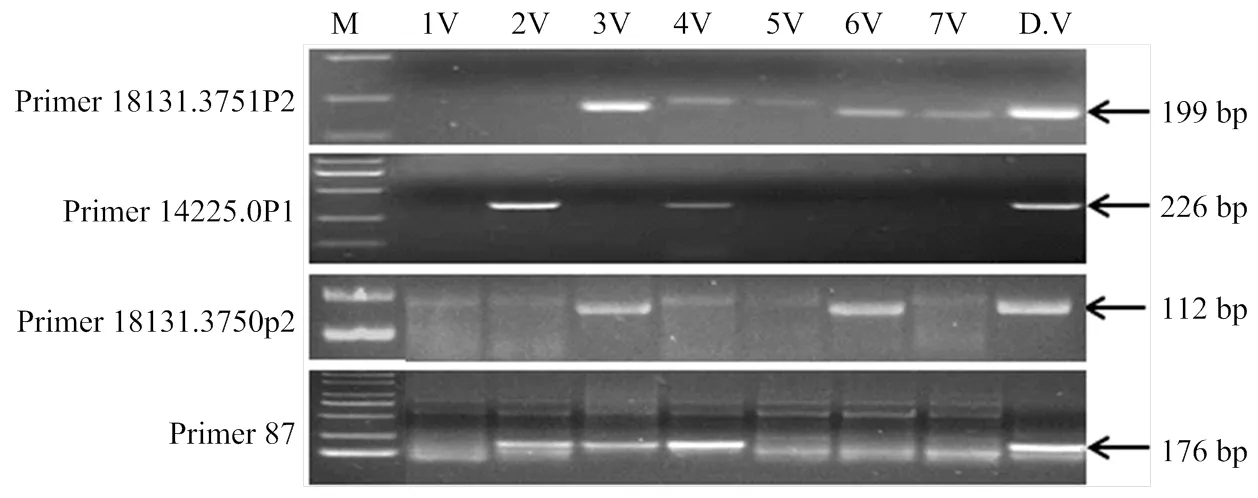
图3 4对引物对1V—7V异染色体系和Dv#4的扩增图谱
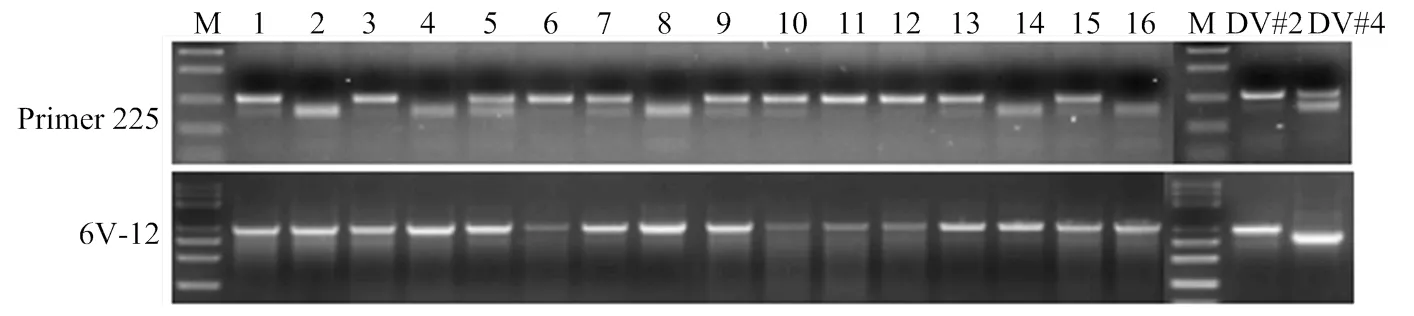
M:Marker 5000;1—16:曾经与簇毛麦Dv#4近距离种植的Dv#2衍生的部分植株,簇毛麦Dv#2和Dv#4为原始亲本
基于SSR开发的PCR标记具有操作简易的特点,尤其是,来自转录组的SSR直接与功能基因的表达相关,鉴定SSR位点与特定染色体的相关性,开发外源染色体特异的SSR分子标记,对于充分利用转录组的SSR序列资源,辅助筛选和鉴定向小麦基因组转移的簇毛麦染色体及其性状具有十分重要的意义。No.1026迄今还没有整套的附加或代换系,所以,本研究借助了来自英国剑桥的簇毛麦Dv#2的一套异染色体系(附加或代换系)对No.1026的SSR在染色体上的分布进行了调查。虽然结果不能完全代表No.1026,但依然具有一定的参考价值,因为作为基因的一部分,转录序列在种间相对保守,而且本研究选择用于染色体定位的引物都可在2份簇毛麦Dv#2和Dv#4中扩增出大小相似清晰的特异片段。
随机合成的238对引物扩增结果显示:簇毛麦基因组中SSR可能位于单条染色体,也可能分布于多条染色体。但是本研究有许多在簇毛麦可清晰扩增出目的条带的引物在任何一个异染色体系上都无扩增,这可能是由于:源于Dv#2的附加系中,外源染色体并没有完全保持簇毛麦原始亲本中染色体的完整性。因为从簇毛麦到附加系的培育过程中,经历过异源多倍体化(如硬-簇双二倍体的合成)及多次的回交和自交,在异源多倍体化过程中,基因组的重新整合可能出现基因的缺失、删除或突变[40]。事实上,SSR引物是基于SSR序列侧翼的保守序列设计的,这些保守序列的缺失或变异都可能造成扩增的失败。另外,这些来自转录组的SSR引物在基因组扩增时,可能由于内含子或其他插入序列的存在而比预期片段大。
3.2 不同簇毛麦间的遗传多样性
簇毛麦携带许多改良小麦潜在的优良性状,但对不同来源簇毛麦的遗传多样性的研究迄今为止多局限于6V,如研究表明,6V#2和6V#4染色体都携带白粉病抗性基因,而6V#1和6V#3的异染色体系则对白粉菌敏感。此外,不同来源的6VS对瘿螨及其所传播的病毒抗性也存在差异[20]。最近的研究中,至少有10个分子标记显示出6V#2和6V#4间的多态性[22-26]。然而,涉及其他染色体的此类研究还很缺乏。另外,除了白粉病抗性,簇毛麦中控制重要农艺性状的很多基因尚未鉴定和利用。本研究利用来自转录组的SSR序列,对2份不同簇毛麦进行扩增分析,揭示了它们之间存在丰富的多态性。而且,本研究是基于琼脂糖凝胶电泳技术区分样品的多态性的,其分辨率较低,一些原本存在的差异不足以在琼脂糖凝胶上区分。所以,推测实际上簇毛麦间的多态性可能更高。而遗传多样性是研究与利用种质资源的基础。因此,将不同来源簇毛麦导入普通小麦,培育附加、代换或易位系,对其有益基因的发掘、鉴定及在小麦改良中的利用具有重要的意义,而基于基因序列的差异开发的EST-SSR标记在上述过程中也可作为重要的辅助选择的工具。
4 结论
簇毛麦No.1026(Dv#4)的转录组存在大量的SSR序列,其中CCG/CGG、A/T和AG/CT等三核苷酸、单核苷酸和二核苷酸是其最主要的串联重复基序。部分EST-SSR侧翼保守序列与单一或多条外源染色体相关联,据此开发特异的分子标记,可用于跟踪检测小麦背景中特异的簇毛麦染色体或染色体片段。簇毛麦Dv#4与Dv#2间的部分EST-SSR具有多态性,提示2份不同来源的簇毛麦其表达序列间存在一定程度的遗传差异,因此,Dv#4值得进一步的发掘研究。
[1] Grądzielewska A. The genus Dasypyrum-part 2.- a wild species used in wheat improvement.,2006, 152: 441-454.
[2] 赵万春, 董剑, 陈其皎, 李晓燕, 高翔, 石引刚, 陈良国. 簇毛麦—用于小麦改良的一种野生植物. 草业科学, 2012, 29(10): 1613-1621.
ZHAO W C, DONG J, CHEN Q J, LI X Y, GAO X, SHI Y G, CHEN L G. Advance in-a valuable wild species used in wheat improvement., 2012, 29(10): 1613-1621. (in Chinese)
[3] CHEN Q, CONNER R L, LI H, LAROCHE A, GRAF R J, KUZYK A D. Expression of resistance to strip rust, powdery mildew and the wheat curl mite in×lines., 2002, 82(2): 451-456.
[4] 刘大钧, 陈佩度, 吴沛良, 王耀南, 邱伯行, 王苏玲. 硬粒小麦-簇毛麦双二倍体. 作物学报, 1986, 12(3): 155-162.
LIU D J, CHEN P D, WU P L, WANG Y N, QIU B X, WANG S L.amphidiploid., 1986, 12(3): 155-162. (in Chinese)
[5] 陈孝, 徐惠君, 杜丽璞, 尚立民, 韩彬, 施爱农, 肖世和. 利用组织培养技术向普通小麦导入簇毛麦抗白粉病基因的研究. 中国农业科学, 1996, 29(5): 1-8.
CHEN X, XU H J, DU L P, SHANG L M, HAN B, SHI A N, XIAO S H. Transfer of gene resistant to powdery mildew fromto common wheat by tissue culture.,1996, 29(5): 1-8. (in Chinese)
[6] 陈佩度, 周波, 齐莉莉, 刘大钧. 用分子原位杂交(GISH)鉴定小麦-簇毛麦双倍体、附加系、代换系和易位系. 遗传学报, 1995, 22(5): 380-386.
CHEN P D, ZHOU B, QI L L, LIU D J. Identification of wheat-amphiloid, addition, substitution and translocation lines byhybridization using biotin-labelled genomic DNA as a probe., 1995, 22(5): 380-386. (in Chinese)
[7] CHEN P D, QI L L, ZHOU B, ZHANG S Z, LIU D J. Development and molecular cytogenetic analysis of wheat-6VS/6AL translocation lines specifying resistance to powdery mildew., 1995, 91: 1125-1128.
[8] 齐莉莉, 陈佩度, 刘大钧, 周波, 张守中, 盛宝钦, 向齐君, 段霞渝, 周益林.小麦白粉病新抗源——基因. 作物学报, 1995, 21(3): 257-262.
QI L L, CHEN P D, LIU D J, ZHOU B, ZHANG S Z, SHEENG B Q, XIANG Q J, DUAN X Y, ZHOU Y L. The gene- a new source for resistance to wheat powdery mildew., 1995, 21(3): 257-262. (in Chinese)
[9] LI H, CHEN X, XIN Z Y, MA Y Z, XU H J, CHEN X Y, JIA X. Development and identification of wheat–T6DL.6VS chromosome translocation lines conferring resistance to powdery mildew., 2005, 124: 203-205.
[10] LIU C, QI L L, LIU W X, ZHAO W C, WILSON J, FRIEBE B, GILL B S. Development of a set of compensating-Robertsonian translocation lines., 2011, 54: 836-844.
[11] XING L, HU P, LIU J., WITEK K, ZHOU S, XU J, ZHOU W, GAO L, HUANG Z, ZHANG R, WANG X, CHEN P, WANG H, JONES J D G, KARAFIATOVA M, VRANA J, BARTOS J, DOLEZEL J, TIAN Y, WU Y, CAO A. Pm21 fromencodes a CC-NBS-LRR that confers powdery mildew resistance in wheat., 2018, doi: 10.1016/j.molp.2018.02.013.
[12] HE H, ZHU S, ZHAO R, JIANG Z, JI Y, JI J, QIU D, LI H, BIE T. Pm21, encoding a typical CC-NBS-LRR protein, confers broad- spectrum resistance to wheat powdery mildew disease., 2018, doi: 10.1016/j.molp.2018.03.004.
[13] ZHANG Q P, LI Q, WANG X E, WANG H Y, LANG S P, WANG Y N, WANG S L, CHEN P D, LIU D J. Development and characterization of a-translocation line T4VS·4DL conferring resistance to wheat spindle streak mosaic virus., 2005, 145: 317-320.
[14] ZHAO R, WANG H, XIAO J, BIE T, CHENG S, JIA Q, YUAN C, ZHANG R, CAO A, CHEN P, WANG X. Induction of 4VS chromosome recombinants using the CS ph1b mutant and mapping of the wheat yellow mosaic virus resistance gene from, 2013, 126: 2921-2930.
[15] 李淑梅, 徐川梅, 周波, 陈佩度. 普通小麦-簇毛麦2V染色体端体异附加系的选育与鉴定. 南京农业大学学报, 2009, 32(1): 1-5.
LI S M, XU C M, ZHOU B, CHEN P D. Development and identification ofditelesomic addition lines involving chromosome 2V of., 2009, 32(1): 1-5. (in Chinese)
[16] 董剑, 杨华, 赵万春, 李晓燕, 陈其皎, 高翔. 普通小麦中国春–簇毛麦易位系T1DL·1VS和T1DS·1VL的农艺和品质特性. 作物学报, 2013, 39(8): 1386-1390.
DONG J, YANG H, ZHAO W C, LI X Y, CHEN Q J, GAO X. Agronomic traits and grain quality of Chinese Spring-translocation lines T1DL∙1VS and T1DS∙1VL., 2013, 39(8): 1386-1390. (in Chinese)
[17] 张瑞奇, 冯祎高, 侯富, 陈树林, 别同德, 陈佩度. 普通小麦-簇毛麦T5VS.5DL易位染色体对小麦主要农艺性状、品质和白粉病抗性的遗传效应分析. 中国农业科学, 2015, 48(6): 1041-1051.
ZHANG R Q, FENG Y G, HOU F, CHEN S L, BIE T D, CHEN P D. The genetic effect of wheat-T5VS.5DL translocated chromosome on agronomic characteristics, quality, and powdery mildew resistance of common wheat., 2015, 48(6): 1041-1051. (in Chinese)
[18] ZHANG R, SUN B, CHEN J, CAO A, XING L, FENG Y, LAN C, CHEN P., a developmental-stage and tissue-specific powdery mildew resistance gene introgressed frominto common wheat., 2016, 129: 1975-1984.
[19] QI L L, PUMPHREY M O, FRIEBE B, ZHANG P, QIAN C, BOWDEN R L, ROUSE M N, JIN Y, GILL B S. A novel Robertsonian translocation event leads to transfer of a stem rust resistance gene () effective against race Ug99 frominto bread wheat., 2011, 123(1): 159-167.
[20] LI H J, CONNER R L, CHEN Q, JIA X, LI H, GRAF R J, LAROCHE A, KUZYK A D. Different reactions to the wheat curl mite and wheat streak mosaic virus in various wheat–6V and 6VS lines., 2002, 86(4): 423-428.
[21] LIU C, Ye X G, Wang M J, LI S J, LIN Z S. Genetic behavior oftranslocation chromosomes T6V#4S·6DL and T6V#2S·6AL., 2017, 16(10): 2136-2144.
[22] 张云龙, 王美蛟, 张悦, 褚翠萍, 林志珊, 徐琼芳, 叶兴国, 陈孝, 张宪省. 不同簇毛麦6VS 染色体臂的白粉病抗性特异功能标记的开发及应用. 作物学报, 2012, 38(10): 1827-1832.
ZHANG Y L, WANG M J, ZHANF Y, CHU C P, LIN Z S, XU Q F, YE X G, CHEN X, ZHANG X S. Development and application of functional markers specific to powdery mildew resistance on chromosome arm 6VS from different origins of, 2012, 38(10): 1827-1832. (in Chinese)
[23] LIN Z S, ZHANG Y L, WANG K, LI J R, XU Q F, CHEN X, ZHANG X S, YE X G. Isolation and molecular analysis of genesandhomologous to powdery mildew resistance genein aaccession and its derivatives., 2013, 54: 417-426.
[24] BIE T D, ZHAO R H, ZHU S Y, CHEN S L, HE H G. Development and characterization of marker MBH1 simultaneously tagging genesandconferring resistance to powdery mildew in wheat., 2015, 35: 189.
[25] 刘畅, 李仕金, 王轲, 叶兴国, 林志珊. 簇毛麦6VS特异转录序列P21461及P33259的获得及其分子标记在鉴定小麦–簇毛麦抗白粉病育种材料中的应用. 作物学报, 2017, 43(7): 983-992.
LIU C, LI S J, WANG K, YE X G, LIN Z S. Developing of specific transcription sequences P21461 and P33259 on6VS and application of molecular markers in identifying wheat-breeding materials with powdery mildew resistance., 2017, 43(7): 983-992. (in Chinese)
[26] LI S J, LIN Z S, LIU C, Wang K, Du L P, YE X G. Development and comparative genomic mapping of6V#4S- specific PCR markers using transcriptome data., 2017, 130(10): 2057-2068.
[27] PLASCHKE J, GANAL M E, RODER M S. Detection of genetic diversity in closely related bread wheat using microsatellite markers., 1995, 91: 1001-1007.
[28] BRYAN G J, COLLINS A J, STEPHENSON P, Orry A, Smith J B, Gale M D. Isolation and characterization of microsatellites from hexaploid bread wheat., 1997, 94(5): 557-563.
[29] 谢皓, 陈学珍, 杨柳, 王建立. EST-SSR标记的发展和在植物遗传研究中的应用. 北京农学院学报, 2005, 20(4): 73-76.
XIE H, CHEN X J, YANG L, WANG J L. Development and application of EST-SSR marker in plant genetic., 2005, 20(4): 73-76. (in Chinese)
[30] 曹亚萍, 曹爱忠, 王秀娥, 陈佩度. 基于EST-PCR的簇毛麦染色体特异分子标记筛选及应用. 作物学报, 2009, 35(1): 1-10.
CAO Y P, CAO A Z, WANG X E, CHEN P D. Screening and application of EST-based PCR markers specific to individual chromosomes of, 2009, 35(1): 1-10. (in Chinese)
[31] 李珊珊, 曾艳飞, 何彩云, 张建国. 基于沙棘转录组序列开发EST-SSR分子标记. 林业科学研究, 2017, 30(1): 69-74.
LI S S, ZENG Y F, HE C Y, ZHANG J G. Development of EST-SSR markers based ontranscriptomic sequences., 2017, 30(1): 69-74. (in Chinese)
[32] 陈岳, 张微微, 莫海波, 付乃峰, 田代科. EST-SSR标记构建莲()遗传连锁图谱. 分子植物育种, 2017, 15(6): 2265-2273.
CHEN Y, ZHANG W W, MO H B, FU N F, TIAN D K. Construction of a genetic linkage map of lotus () using EST-SSR markers., 2017, 15(6): 2265-2273. (in Chinese)
[33] 陈勋, 龚自明. 基于EST-SSR 标记的湖北茶树种质资源遗传多样性分析. 分子植物育种, 2017, 15(5): 1831-1838.
Chen X, Gong Z M. Analysis of genetic diversity with EST-SSR markers for tea germplasm in Hubei province., 2017, 15(5): 1831-1838. (in Chinese)
[34] 李翔, 张颖, 许俊强, 宋婷, 李文璐, 董玉梅, 张应华. 基于EST-SSR 标记的云南黄瓜种质资源遗传多样性分析. 分子植物育种, 2016, 14(5): 1179-1188.
LI X, ZHANG Y, XU J Q, SONG T, LI W L, DONG Y M, ZHANG Y H. Analysis on genetic diversity of local cucumber germplasm resources in Yunnan based on EST-SSR markers., 2016, 14(5): 1179-1188. (in Chinese)
[35] QI L L, WANG S L, CHEN P D, LIU D J, GILL B S. Identification and physical mapping of threechromosome-6V deletion lines., 1998, 97(7): 1042-1046.
[36] ZHANG L D, YUAN D J, YU S W, LI Z G, CAO Y F, MIAO Z Q, QIAN H M, TANG K X. Preference of simple sequence repeats in coding and non-coding regions of., 2004, 20(7): 1081-1086.
[37] 张利达, 唐克轩. 植物EST-SSR标记开发及其应用. 基因组学与应用生物学, 2010, 29(3): 534-541.
ZHANG L D, TANG K X. Development of plant EST-SSR markers and its application., 2010, 29(3): 534-541. (in Chinese)
[38] GAO L F, TANG J F, LI H W, JIA J Z. Analysis of microsatellites in major crops assessed by computational and experimental approaches., 2003, 12: 245-261.
[39] 文亚峰, 韩文军, 周宏, 徐刚标. 杉木转录组SSR挖掘及EST-SSR标记规模化开发. 林业科学, 2015, 51(11): 40-49.
WEN Y F, HAN W J, ZHOU H, XU G B. SSR mining and development of EST-SSR markers forbased on transcriptome sequences., 2015, 51(11): 40-49. (in Chinese)
[40] KASHKUSH K, FELDMAN M, LEVY A A. Silencing and activation in a newly synthesized wheat allotetraploid., 2002, 160(4): 1651-1659.
SSR sequences and development of PCR markers based on transcriptomeNo.1026
CHEN JingNan1,2, Ma XiaoLan1, WANG Zhen3, LI ShiJin1, Xie Hao2, YE XingGuo1, LIN ZhiShan1
(1Institute of Crop Science, Chinese Academy of Agricultural Sciences/National Key Facility of Crop Gene Resources and Genetic Improvement/Key Laboratory of Biology and Genetic Improvement of Triticeae Crops, Ministry of Agriculture, Beijing 100081;2School of Plant Science and Technology, Beijing university of agriculture, Beijing 102206;3College of bioscience & bioengineering, North Minzu university, Yinchuan 750021)
【Objective】The aim of this study is to explore the characteristics of the EST-SSR sequences of aaccession No.1026 (Dv#4) introduced from the former Soviet Union, and their distributions on chromosomes and polymorphism in differentaccessions and between Dv#4 and common wheat. 【Method】Transcriptome sequences of Dv#4 plants were obtained by Illumina HiSeq sequencing and used to search and analyze the SSR sequences using MISA software and design primers by Primer 3. In total, 238 pairs of primers were selected randomly for synthesis and used to amplify the genomic DNAs of wheat Chinese Spring (CS) and the two differentaccessions. The polymorphisms of the PCR products on agarose gel were evaluated. Further, the features of their chromosome distributions in Dv#4 were studied by using a set of wheat-alien chromosome lines (including disomic addition lines and disomic substitution lines). 【Result】 A total sequence length of 62.76 Mb was detected and 10 497 SSR loci were found in the transcriptome data. They are involved in 8 735 unigenes. Repeated unit of mono-, di-, tri- nucleotides are the main type, holding 95.85% of all loci among 1-6 nucleotides repeats, among which tri-nucleotide is the richest component that makes up of 50.33% and contains CCG/CGG motif by 41.66%. The next component is mono-nucleotide tandem repeat, and its occurrence frequency is 18.39%, and A/T repeats occupy 74.58% in this type. Di-nucleotide ranks the 3rd, and it holds 18.39% in the total SSR loci. Among 238 pairs of randomly synthetized EST-SSR primers, 88 pairs amplified polymorphic fragments between CS and(including Dv#2 and Dv#4); 8 pairs only had amplifications inand some single alien chromosome lines; 4 pairs could specifically amplify bands inand multiple alien chromosome lines. But, many primers which had amplification in bothaccessions had no amplification in any alien chromosome lines. Therefore, it can be inferred that variations on the flanking conserved sequences of SSR might occur during the transferring of the exogenous genome or chromosomes into wheat. Additionally, 47 pairs of primers (19.74%) showed polymorphisms between Dv#2 and Dv#4. By using a pair of EST-SSR primer and a PCR marker, respectively, polymorphic amplicons were detected in 48plants, indicating that polymorphic SSR primer can be used to detect the heterogeneity ofeffectively.【Conclusion】Dv#4 has abundant SSR sequences in its transcriptome, of which, CCG/CGG, A/T, AG/CT and other tri-, mono-, and di-nucleotide are the main tandem repeats.The flanking conserved sequences of partial EST-SSR in Dv#4 are associated with a single or several chromosomes specifically, which provides an ideal sequence resource for the development of specific molecular markers to track and detectchromosomes or chromosome fragments in wheat background. Some of the EST-SSR polymorphism between Dv#4 and Dv#2 indicates that there is a certain degree of genetic diversity in some of the expressed sequences between the two different originaccessions. Therefore, Dv#4 is valuable to be further investigated.
; transcriptome; EST-SSR; alien chromosome lines; chromosome localization; genetic diversity
10.3864/j.issn.0578-1752.2019.01.001
2018-07-01;
2018-09-19
国家重点研发计划(2016YFD0102002,2016YFD0102001)、中国农业科学院科技创新工程(2060302-2-17年)
陈竟男,E-mail:1293498949@qq.com。 通信作者谢皓,E-mail:xiehao126@126.com。通信作者林志珊,E-mail:linzhishan@caas.cn
(责任编辑 李莉)

