可见/近红外高光谱成像技术对冷藏过程中原料乳细菌总数的无损检测
□ 赵文静 内蒙古自治区产品质量检验研究院 崔腾飞 陈锦华 宁夏大学农学院
1 引言
在单一食品中,牛乳被称为为数不多的营养完全食品之一[1-2]。原料乳(生鲜牛奶)含有蛋白质、脂肪、乳糖和各种维生素以及矿物质等一百多种人体需要的营养元素,但也正是因为如此,原料乳中蛋白质等营养物质极易被微生物分解利用,在贮藏过程中生长、繁殖,导致原料乳品质变化。原料乳位于奶业产业链的最上游,所以其质量安全将直接影响到乳制品的质量与安全,而原料乳细菌总数是衡量生牛乳原料的重要指标,所以检测原料乳细菌总数对控制原料乳质量与安全至关重要。
传统检测原料乳中细菌总数指标按照GB478.2-2010方法进行的,此检测方法是基于平板培养和菌落计数,需耗时48 h,且检测结果也存在着严重的滞后性,并且容易导致在原料奶的储藏和运输环节造成二次污染,无法满足原料乳快速、安全等的验收要求。因此,实时、快速、准确地检测原料乳中细菌总数对加强原料乳质量控制具有重要的现实意义。现今用于原料乳快速检测方法有显微镜检测、流式细胞计数法45、还原实验法光电法、ATP荧光计数法、PCR技术等。各种方法各有优缺点,但没有一项技术同时具有快速、准确、无需样品预处理、实时、无损等优点,高光谱成像技术是基于非常多窄波段的影像数据技术,具有非常丰富的吸收谱带,能够识别样品的大量信息,并将图谱合二为一,是单一的光谱或者图谱无法比拟的,最大的特点是能够对几十个乃至几百个窄波段以进行连续的光谱覆盖,而且光谱分辨率高、操作方便等[2-3],用平均光谱反映样本,建立原料乳检测模型、相关性好,能够满足方便、快速、无损、准确等检测原料乳细菌总数的要求。
本文以原料乳为研究对象,利用可见/近红外高光谱成像技术采集样品400~1 000 nm的光谱数据,选取正交信号校正法(orthogonal signal correction,OSC)、多元散射校正(Multiple Scattering Correction,MSC)、 卷 积平 滑(Savitzky- Golay,SG) 3种预处理方法减少噪音干扰,结合PLSR建模方法优选最佳的预处理方法;使用连续投影算法(Successie Projection Algorithm,SPA)、 无 信息消除变量(Uninformative Variable Elimination,UVE)等2种数据降维方法,结合使用偏最小二乘回归(partial least squares regression,PLSR) 建模方法分别对全波段和特征波段建模;选取最优模型,为原料乳中细菌总数的快速检测奠定理论基础。
2 实验
2.1 材料与试剂
样品来源于宁夏银川市平吉堡牧场乳样,每份原料乳为100 mL。将获得的奶罐新鲜乳样快速置于灭菌取样瓶中,4 ℃恒温保温箱带回实验室,编号之后置于冰箱中4 ℃冷藏。每天测试一次(贮藏期1~7 d,共计七次),每次随机取20个乳样作为试验样本,用于高光谱数据采集和测定细菌总数。
2.2 仪器
实验过程中使用的仪器主要为:Hyper Spec VIS/NIR 高光谱成像系统(美国Headwall Photonics 公司),如图1所示。
2.3 高光谱信息采集
为避免图像采集时环境光的干扰,获得更加精确的数据,高光谱图像数据采集前,预先根据光源的照度设定好高光谱摄像头曝光时间以保证图像清晰,并调整好输送装置的速度以避免图像空间分辨率失真。采集光谱图像前需对高光谱进行预热[3]和参数设定,半小时后应对仪器进行校准[4]。通过多次预实验确定了最终光谱扫描的参数:物距为400 mm,输送装置的步距为180 μm/s,成像光谱仪的曝光时间为35 ms,扫描起始位置90 mm,扫描线实际长度为60 mm。
图像采集前,为减弱成像光谱仪暗电流和室内照明对图像的影响,应在暗室进行操作仪器,避免杂散光对采集样本的干扰。同时需对仪器进行黑白校正[5],如公式(1)所示。
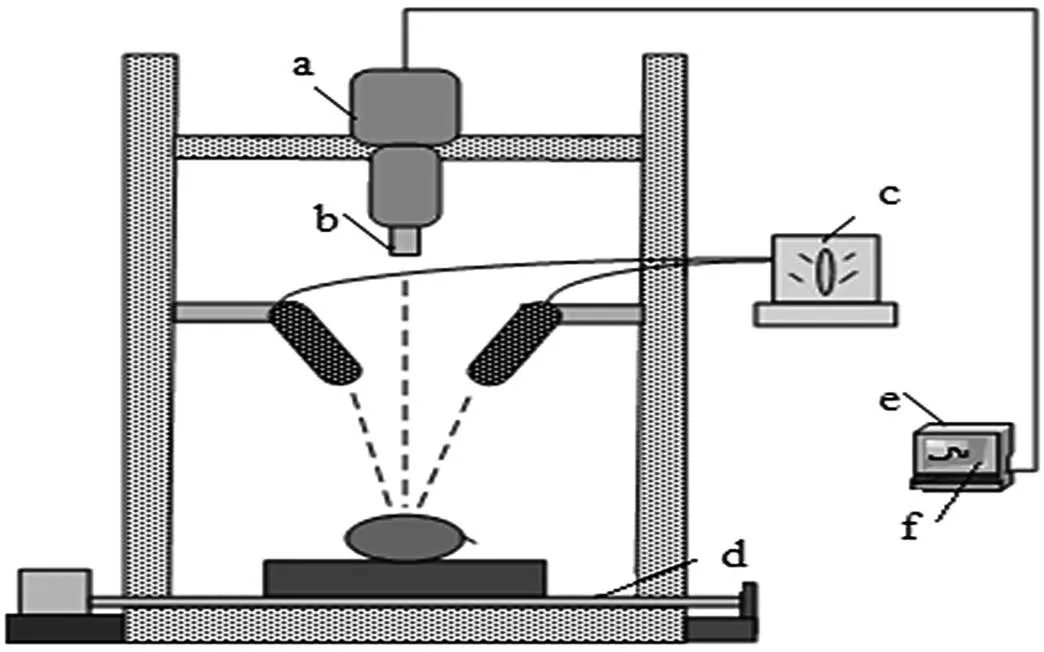
图1 VIS/NIR 高光谱成像系统
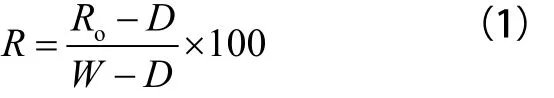
式(1)中:RO是样本原始的漫反射光谱图像,W是白板的漫反射图像(反射率约100%),D是暗图像(反射率0%),R是校正后的漫反射光谱图像。
2.4 细菌总数的测定
2.4.1 测定方法
参照食品安全国家标准GB 4789.2-2010[6],采用平板计数法对乳中细菌总数进行测定。具体方法如下。
(1)稀释:取原料乳,充分摇混,使样品呈均匀状态。以十倍稀释法,将原料乳稀释成10-1、10-2、l0-3、l0-4、10-5系列。
(2) 接种:从最大稀释度开始,各取l mL l0-3、10-4、10-5的原料乳稀释液,分别放入已标记好的培养皿内。培养基融化,待冷至45 ℃左右时,在火焰旁倒入培养皿内,细菌总数对应PCA培养基,倒完迅速盖好,放在桌面上轻轻摇转,使稀释的样品与培养基均匀混合,待平板冷却固化后,倒置。
(3)培养:于合适的培养温度下培养一定的时间,对应细菌总数选择PCA合成培养基,37 ℃培养48小时。
(4) 计数:所有平板在培养到足够的时间后,选择每皿出现30~300个菌落的稀释度为准,计算出每毫升牛奶中含细菌总数。计算公式如下。① 若只有一个稀释度平板上的菌落数在适宜计数范围内,N每毫升样品中总活菌数=同一稀释度两次重复的菌落平均数×稀释倍数×10;②若有两个连续稀释度的平板菌落数在适宜计数范围内时,按公式(2)计算:N= ∑ C/(n1+0.1n2)d 。
式(2)中:N——样品中菌落总数;∑C——(含适宜范围菌落数的平板);n1——第一稀释度(低稀释倍数),n2——第二稀释度(高稀释倍数),d——稀释因子(第一稀释度)。
2.4.2 平板计数琼脂(PCA)
胰蛋白胨5.0 g/L,酵母浸粉2.5 g/L,葡萄糖琼脂1.0 g/L,琼脂15.0 g/L,加热溶解于1 000 mL蒸馏水中,调pH值7.0,分装三角瓶,121 ℃高压灭菌15分钟,备用。取适宜稀释得样品液1 mL,加入无菌平皿中心,倾入冷至44~46 ℃的平板计数琼脂,混匀,待琼脂凝固后,放置36 ℃培养48小时进行计数菌落。
2.5 图像处理
利用高光谱成像系统采集样品图像后,使用ENVI4.6( Research System Inc,USA)软件选取感兴趣区域(Region of Interest,ROI),计算出的平均光谱值作为反射光谱。
2.5.1 模型建立
样本在采集光谱时,由于仪器噪音、暗电流等因素的影响,导致光谱曲线产生不重复和基线漂移等现象[7-8],本文采用 MSC[9]、SG[10]、OSC[11]三种预处理方法对原始光谱进行处理,多元散射校正(MSC)去除高光谱漫反射光谱中样品的镜面反射及不均匀性造成的噪声,消除漫反射光谱的基线漂移及光谱不重复性,在光谱与浓度线性关系较好和组分性质相似的情况下,MSC校正的效果较好;卷积平滑(SG)尽可能减小平滑对有用信息的影响;SG+MSC两种预处理方法相结合,可以更好地减少干扰,因此,获得更多有用信息。光谱预处理后采用偏最小二乘回归(PLSR)法建立模型。其PLSR方法是一种全新的回归模型方法,在模型预测研究中应用较为广泛。PLSR方法结合了多元线性回归(MLR)与主成分分析(PCR)两种方法的优势。在光谱矩阵中,对自变量X相互正交方向上最大方差主成分提取,同时对应变量实测值剪切力Y中光谱矩阵进行冗余信息降维处理,并将Y光谱矩阵引入X矩阵中进行快速分解,同时计算每一个新的主成分数前,将X得分与Y得分进行交换,使得X主成分数与剪切力Y最大程度的直接相关联,其作用是消除变量多重共线性,也充分利用了光谱数值、吸收值与剪切力之间的线性关系,使得模型预测能力最大程度提高。
2.5.2 模型评判
采用PLSR建立乳中细菌总数预测模型。通过校正集相关系数(Rc)与校正集均方根误差(rootmean-square error of calibration set,RMSEC)、预测集相关系数(Rp)与预测集均方根误差(rootmean-square error of prediction set,RMSEP)以及交互验证均方根误差(RMSECV)指标进行模型的性能评价。Rc、Rp值越高,其模型相关性越好;RMSEC、RMSECV、RMSEP值 越低,其模型预测能力越好。因此,利用PLSR方法所建立效果较好的模型应具有较高的Rc、Rcv、Rp与较低的RMSEC、RMSECV、RMSEP 值[12]。除此之外,也可以利用Rc+Rp说明模型总体的乳中细菌总数检测精度[13]。
2.6 数据分析
The Unscrambler X 10.4软件对光谱预处理,图像分析软件为ENVI 4.6,Matlab R2014a软件进行建模、划分样本集,绘图软件为origin 8。
3 结果与分析
3.1 筛选最佳预处理方法
本文中采用Galvao等[14]提出的 SPXY(sample set partitioning based on joint x-y distances)法,将剔除异常值的原料乳样本按3∶1划分为校正集样本和验证集样本。
表1为原始光谱以及预处理光谱的PLSR建模结果,由相关系数、均方根误差评价模型稳定性,图2为原始光谱图。可以看出使用OSC预处理方法,建模效果降低,因此,经过预处理的光谱建模效果不一定高于原始光谱模型效果;使用其他几种预处理方法,模型相关系数均高于原始光谱,均方根误差低于原始光谱,说明这几种预处理方法可以消除噪音干扰,提高建模效果;经过数据和图像对比分析,确定SG为最佳预处理方法。
3.2 光谱数据降维
3.2.1 SPA算法选取特征波长
SPA[15]算法可以在很大程度上精简模型,应用SPA选取特征波长时,设置变量范围为5-25,归一化处理后得到前五个波长变量下的RMSECV值,分别为 413、469、525、671、703 nm,特征波长占总波长的4%。
3.3.2 UVE提取特征变量
使用UVE[16]提取原料乳光谱特征波长,运行程序得到输入变量的稳定性结果,如图4所示。
图4竖线左右两侧各为125个变量(左边为波长变量,右边为随机变量)。用此方法共选取了13个特征波长,分别为 416、479、501、503、517、598、634、658、667、692、711、852、981 nm,特征波长占总波长的10.4%。

表1 不同预处理方法的PLSR模型
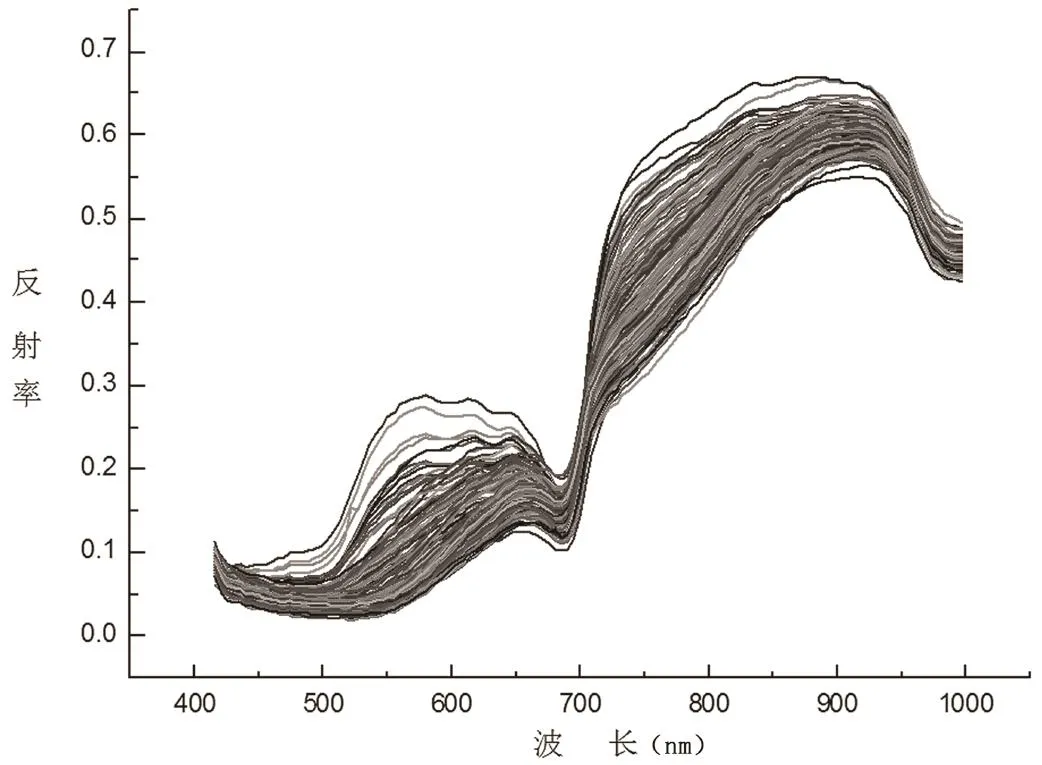
图2 原始光谱图

图3 SPA提取的特征波长数
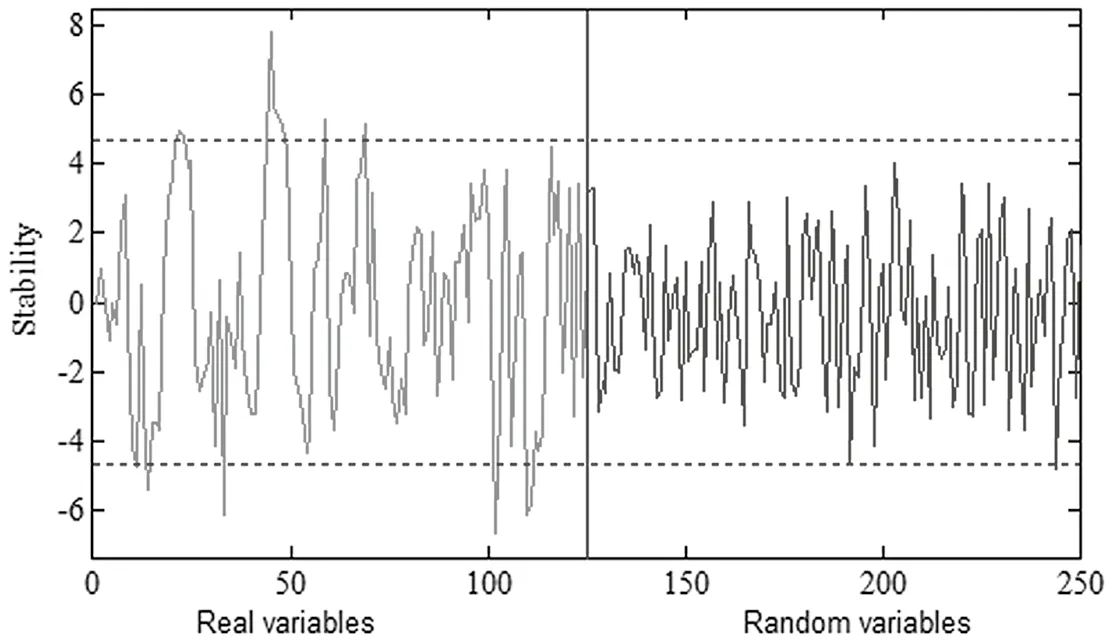
图4 UVE-PLSR稳定性分布曲线
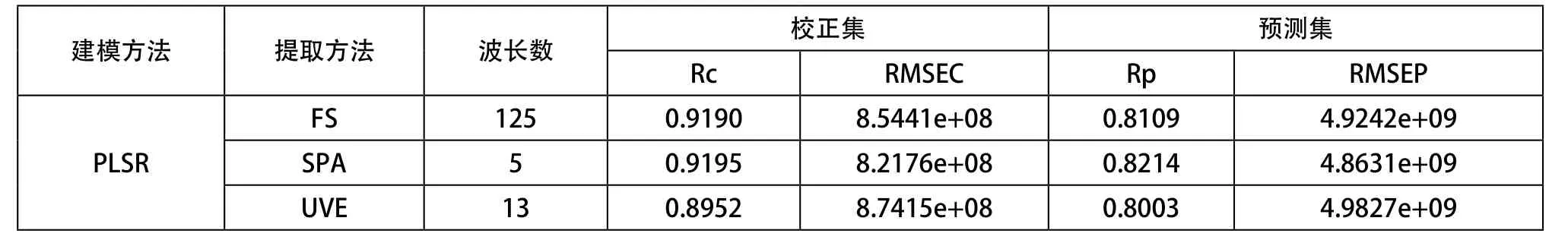
表2 不同波长提取方法建立的PLS模型的结果
3.4 建立模型
对全波段和特征波段分别建立PLSR模型,见表2。整体来看,使用SPA算法提取特征变量数后的建模效果稍高于全波段模型,而UVE建模效果不如全波段建模效果;对比分析3种模型,建模效果最优的是SPA+PLAR,Rc、Rp分别为0.91 95、0.821 4。
4 结论
本文以原料乳为研究对象,采集原料乳400~1 000 nm的光谱图像,提取反射光谱,同时采用平板计数法对乳中细菌总数进行测定,对光谱值和化学值建立PLSR模型,高光谱成像技术可以实现原料乳中细菌总数预测。主要结论如下。
(1)对原始光谱采用3种方法进行预处理,通过对比分析数据和图像信息,确定SG为最佳预处理方法,该预处理方法降低了噪音,去掉了无用信息,提高了建模效果,结果:Rc、Rp为0.9190、0.8109。
(2)基于最优预处理方法,使用了2种数据降维方法,提取特征波长数分别为5、13,占到全波段的4%、10.4%。
(3)对以上2种降维处理数据分别建立PLSR模型,SPA+PLAR为最优模型,Rc、Rp分别为0.919 5、0.821 4,提取特征波长可减少冗长数据,降低维数,实现快速检测。

