基于Oncomine数据库荟萃分析COL1A2在胃癌患者中的表达及其临床意义
肖毓,张洪攀,谭榜宪
637000 四川 南充,川北医学院 临床医学系(肖毓、张洪攀),医学影像学院(谭榜宪);637000 四川 南充,川北医学院附属医院 肿瘤科(谭榜宪)
胃癌是起源于胃黏膜上皮的恶性肿瘤,是世界上最常见的恶性肿瘤之一。根据GLOBOCAN 2018数据,2018年胃癌新发病例超过1 000 000例,占全球肿瘤发病率的第5位,其中死亡病例达到783 000例,占全球恶性肿瘤导致死亡的第3位[1]。早期胃癌检出率低,约40%的胃癌患者在确诊时已为晚期[2]。目前,化疗是晚期胃癌患者的首选治疗方法,但化疗药物不良反应重,耐药率高。作用于特定靶点的靶向药物因其药物不良反应较轻、耐药率低、患者服药方便等优点成为治疗晚期胃癌的新治疗手段之一[3]。因此从分子水平研究胃癌发生发展的机制有利于发现关键靶点,开发新的靶向药物,有利于延长患者生存时间、改善患者生活质量。
纤维胶原家族中有多种成员,其中Ⅰ型胶原α2链(collagen type1 alpha2,COL1A2)基因编码的I型胶原是最广泛表达于人体的胶原,它起到支撑基质结构的作用,形成大多数实体肿瘤的间质部分。研究发现,COL1A2基因主要通过细胞外基质受体互作通路和局部黏附通路,影响细胞的增殖、分化、黏附和转移,主要与肿瘤的侵袭和转移相关[4- 5]。多项研究显示COL1A2的表达可能与胃癌的发生、发展有关,但由于研究方法、样本量及人群的差异,其结论可靠性较低,且其表达水平与患者预后之间的关系目前尚不清楚。因此我们在Oncomine数据库中深度挖掘胃癌的基因芯片中与COL1A2表达有关的数据,二次荟萃分析COL1A2在胃癌中的表达及预后。
1 材料与方法
1.1 基于Oncomine数据库筛选目标基因
筛选条件为:①“Analysis Type:Cancer VS Normal Analysis”;②“Cancer type:Gastric cancer”;③“Data Type:mRNA”;④设定条件:over- expression。P<0.05为差异有统计学意义。
1.2 数据提取
设置的数据提取条件为:①“Gene:COL1A2”;②“Analysis Type:Cancer VS Normal Analysis”;③“Cancer Type:Gastric cancer”;④“Data Type:mRNA”;⑤临界值设定条件(Pvalue<0.05,fold change>2,gene rank=top 10%)。P<0.05为差异有统计学意义。
1.3 Kaplan Meier-Plotter(http://kmplot.com/analysis/)在线生存分析
本研究中筛选条件为:①“Cancer:Gastric Cancer”;②“Gene:COL1A2”;③“Split patients by: Auto select best cutoff ”; ④“Survival:OS”; ⑤亚组分析根据Laurane Classification,分别为:“instestinal、diffuse、mixed”。P<0.05为差异有统计学意义。
1.4 基于TCGA数据对COL1A2在胃癌中的表达及预后进行验证
UALCAN(ualcan.path.uab.edu/analysis.html)是一个基于TCGA数据集,肿瘤基因表达与生存分析的在线数据库[6]。在UALCAN数据库中验证COL1A2在胃癌中的表达及预后。筛选条件:①“gene:COLIA2”;②“cancer type: stomach adenocarcinoma”;③“Split patients by: Auto select best cutoff”。P<0.05为差异有有统计学意义。
2 结 果
2.1 筛选出目标基因COL1A2
目标基因的选择:根据我们设置的筛选条件从Oncomine数据库中得到相关众多基因中,按照中位秩次从前向后选择,排除已有相关详细报道的基因,最终选择COL1A2基因,COL1A2基因在所有差异表达基因里中位秩次为76.0,P<0.001(图1)。
2.2 COL1A2在常见肿瘤类型中的表达结果
图2显示Oncomine数据库中共纳入了348项关于COL1A2在不同肿瘤组织及对应正常组织中表达水平的研究结果,其中关于COL1A2表达有统计学意义的研究结果有102项,91项研究结果显示COL1A2在肿瘤组织中高表达。
2.3 COL1A2在胃癌中的表达结果
在Oncomine数据库中有9项研究显示COL1A2在胃癌组织和正常组织中的表达差异有统计学意义,有478个样本,包括肠型、弥漫型、混合型胃癌组织与正常组织的比较。文章分别发表于“Med Oncol”“Mol Biol Cell”“Clin Cancer”“Nucleic Acids”“Eur J Cancer”中。荟萃9个研究分析发现,与胃正常组织相比,COL1A2在胃癌组织中高表达,P<0.001(图3)。

图1 在Oncomine数据库中筛选出基因COL1A2,中位秩次76.0
Figure1.COL1A2Screened from Oncomine (Median Rank76.0)
*Inthefigure,grayindicatesdatanotdetected;whiteisequivalenttonormaltissue;redthatup.

图 2COL1A2基因在 Oncomine 数据库中所有肿瘤研究中的表达情况
Figure2.Expression ofCOL1A2in All Cancer Studies from Oncomine

图3 9项研究显示COL1A2在胃癌组织与正常胃组织中的表达差异
Figure3.Nine Studies Showing the Expression ofCOL1A2in Gastric Cancer Tissue and Normal Gastric Tissue
2.4 COL1A2在各胃癌基因芯片中的差异
图4所示为Oncomine数据库中COL1A2在不同胃癌基因芯片中的表达结果。在Wang等[7]、Chen等[8]、Cho等[9]、Cui等[10]、D′Errico等[11]5项研究中,COL1A2在胃癌中的表达量均高于正常组织,差异有统计学意义(P值均<0.001)。









图4COL1A2基因在Oncomine数据库中各项胃癌研究中的差异表达
Figure4.Differential Expression ofCOL1A2in Various Gastric Cancer Studies from Oncomine
a.WangGastric(cancervsnormal,P=2.81E-7);b.ChenGastric(intestinaltypecancervsnormal,P=6.07E-25);c.ChenGastric(diffusetypecancervsnormal,P=2.23E-10);d.ChenGastric(mixedtypecancervsnormal,P=4.51E-5);e.ChoGastric(diffusetypecancervsnormal,P=1.89E-12);f.ChoGastric(intestinaltypecancervsnormal,P=9.69E-6);g.CuiGastric(cancervsnormal,P=9.49E-12);h.D′ErricoGastric(intestinaltypecancervsnormal,P=5.42E-14);i.D′ErricoGastric(mixedtypecancervsnormal,P=2.37E-8).
2.5 COL1A2基因在胃癌中表达水平与预后关系
为了明确COL1A2表达与胃癌预后之间的关系,我们利用The Kaplan Meier- Plotter (http://kmplot.com/analysis/)在线数据库进行生存分析。结果显示[12]:COL1A2表达水平与胃癌患者总体生存时间存在相关性。与低表达组相比,COL1A2高表达组胃癌患者总生存时间缩短(P<0.001)。进一步亚组分析发现,COL1A2表达水平对肠型和弥漫型胃癌患者OS有影响(P值分别为P<0.001和P=0.001),而在混合型胃癌患者中,其表达水平对OS无影响(P>0.05)(图5)。
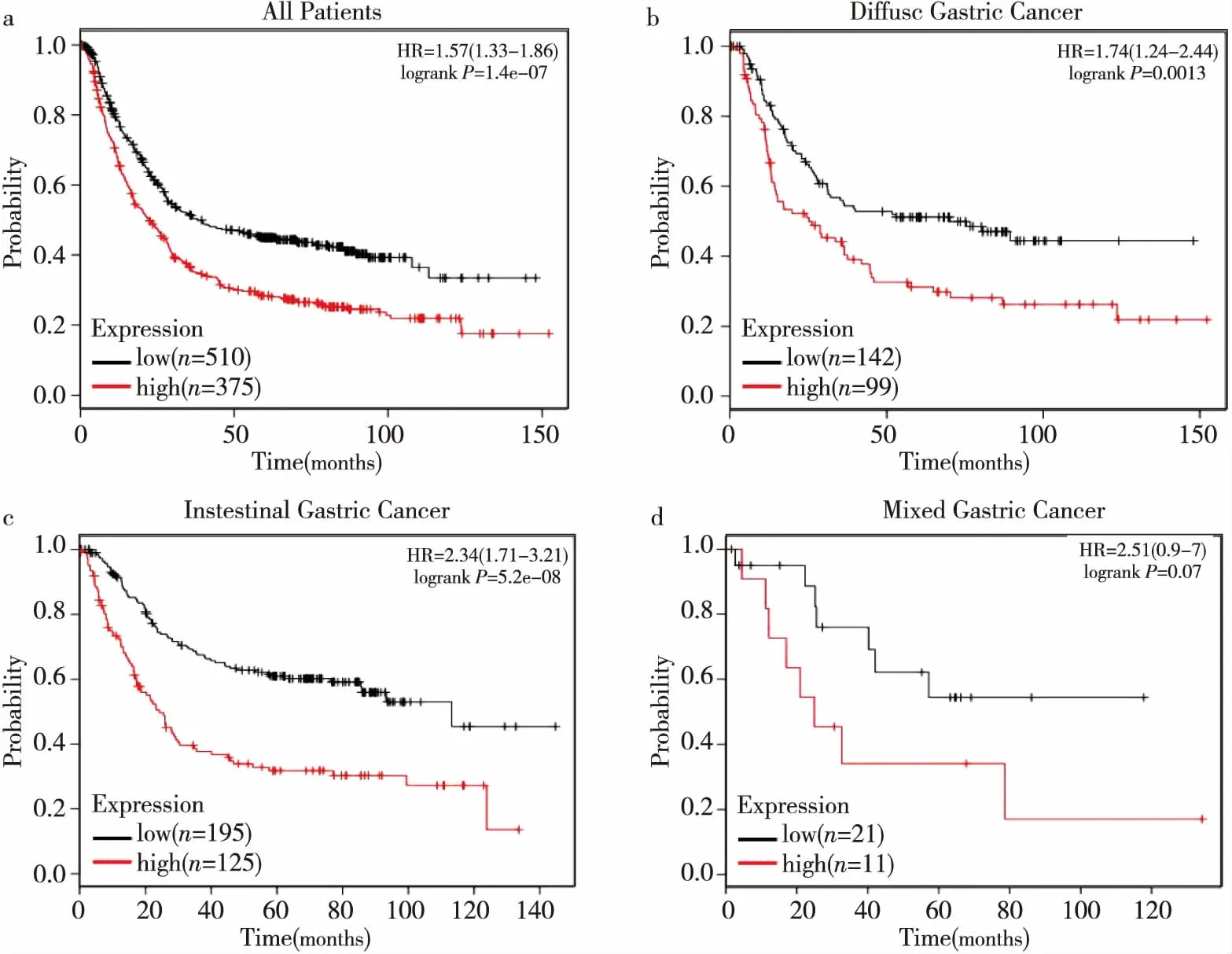
图5 Kaplan Meier-Plotter数据库对COL1A2基因在胃癌患者中的表达水平进行在线生存分析
Figure5.Online Survival Analysis of the Expression Level ofCOL1A2in Gastric Cancer Patients by Using Kaplan Meier-Plotter
a.EffectofCOL1A2expressioningastriccancerpatientsonoverallsurvival;b.EffectofCOL1A2expressionindiffusegastriccancerpatientsonoverallsurvival;c.EffectofCOL1A2expressioninintestinalgastriccancerpatientsonoverallsurvival;d.EffectofCOL1A2expressioninmixedgastriccancerpatientsonoverallsurvival.
2.6 基于TCGA数据对COL1A2在胃癌中的表达及预后进行验证
UALCAN(ualcan.path.uab.edu/analysis.html)数据库在线分析发现,与正常组织相比,COL1A2在胃癌组织中高表达(图6),与表达较低的患者相比,高表达COL1A2的胃癌患者总生存期缩短(P<0.05)。(图7)

图6COL1A2在TCGA数据集胃癌患者中的表达情况
Figure6.Expression ofCOL1A2in Gastric Cancer Patients from TCGA

图7 TCGA数据集中COL1A2表达水平与胃癌患者生存时间的相关性分析
Figure7.Analysis of Correlation betweenCOL1A2Expression Level and Gastric Cancer Patient Survival from TCGA
3 讨 论
胃癌是中国常见的恶性肿瘤之一,幽门螺杆菌是胃癌的主要危险因素,近90%的非贲门胃癌的发病归因于这种细菌[13- 14]。腌制食品的摄入、饮酒、吸烟也会增加胃癌的发病风险[14- 15]。随着手术技术的改进和传统放疗、化疗和新辅助治疗的实施,早期胃癌的5年生存率大大提高。近年来,随着分子生物学技术的发展,逐渐面市的靶向药物不仅用于晚期胃癌姑息治疗,而且在局部进展期胃癌围手术期治疗中也发挥着重要作用。因此寻找胃癌发生发展过程中的关键靶点,对于研发治疗胃癌的靶向药物具有重要的意义。
在哺乳动物细胞中,Ⅰ型胶原蛋白含量丰富,它是由2 条COL1A1基因编码的α1链和1条COL1A2基因编码的α2链组成的三螺旋结构,螺旋之间通过氢键紧密地结合在一起[16]。I 型胶原纤维参与形成细胞外基质,对于细胞的粘附及分化有重要作用,和正常细胞相比,肿瘤细胞间的粘附作用普遍降低,促进了肿瘤细胞的迁移[17]。研究表明COL1A2基因参与头颈部癌[18]、食管癌[19]、肝癌[20]、结肠癌[21- 22]、膀胱癌[23]、肾癌[24]、恶性黑色素瘤[25]等多种恶性肿瘤的增殖、转移等过程中。COL1A2基因参与编码Ⅰ型胶原,该基因的差异表达在人类恶性肿瘤发生发展中产生明显的胶原介导的作用,影响肿瘤细胞增殖及远处转移[26],但其在各种肿瘤中具体的作用机制目前仍尚未明确。
Misawa等[18]、Brooks等[23]分别发现COL1A2在头颈部鳞癌、膀胱癌中下调的机制是启动子区域的CpG高甲基化作用。CpG高甲基化的COL1A2基因失活可能有助于头颈部鳞癌、膀胱癌细胞的增殖和迁移。Fang等[19]发现COL1A2基因的高表达与食管鳞状细胞癌的发生、发展密切相关,COL1A2mRNA的表达水平可用作监测食管鳞癌早期发生及干预治疗效果的有用指标。Zhu等[20]发现由内源性非编码miRNAlet- 7g介导的COL1A2基因位点的多态性可使肝细胞肝癌风险增加1.73倍,COL1A2与肝细胞肝癌的发生发展密切相关。Yu等[21]发现COL1A2基因表达水平在结直肠癌中明显下调,其表达量与结直肠癌的分期、分化程度及淋巴结转移呈负相关(P<0.05)。在结直肠癌SW480和SW620细胞系中过表达COL1A2可以减弱肿瘤细胞增殖、迁移及侵袭的能力。并且该研究发现COL1A2基因可能是通过参与NF- κB途径调控结直肠癌细胞的功能。Li等[27]通过定量PCR检测发现,胃癌组织中COL1A2mRNA表达量显著高于癌前和正常胃组织,并且发现COL1A2表达水平与肿瘤大小及侵袭深度呈正相关。Ao等[28]研究发现COL1A2基因沉默抑制胃癌细胞增殖,迁移和侵袭,同时通过PI3K-Akt信号通路促进肿瘤细胞凋亡。Rong等[29]对75例人胃癌组织进行检测发现,与正常胃组织相比,COL1A2在人胃癌中高度表达。统计分析显示COL1A2表达水平与组织学类型和淋巴结状态显著相关。
尽管目前有研究发现COL1A2在胃癌患者中高表达,但由于各独立研究存在样本量大小及研究方法不一致等原因,其结论的真实性和可靠性有待考究。而对于COL1A2表达水平对胃癌患者预后的影响,目前为止一直缺乏强有力的证据。因此,基于目前研究背景,我们荟萃分析了Oncomine数据库中胃癌组织的基因芯片。Oncomine 数据库涵盖了TCGA数据、GEO数据和目前已发表文献的RNA和DNA-seq数据,截至目前,该数据库共收集了715个基因表达数据集,86 733个正常组织和癌症组织的数据,旨在挖掘新的生物标记物或新的治疗靶点[30]。在此数据库中可根据自己的需求设定筛选和挖掘数据的条件。我们通过Oncomine数据库挖掘COL1A2基因在众多常见肿瘤中的表达信息,结果发现,102项研究有统计学意义,91项研究发现COL1A2在常见肿瘤中高表达。利用Oncomine数据库进一步荟萃分析证实,与胃正常组织相比,COL1A2在胃癌组织中高表达,差异有统计学意义。The Kaplan-Meier Plotter数据库(http://kmplot.com/analysis/)是目前可信度较强的预后相关分析的在线分析数据库,包含了1 065例胃癌样本,可对54 675个基因进行相关预后分析。本文通过 The Kaplan-Meier Plotter数据库发现了COL1A2在胃癌患者中的预后价值,结果显示,COL1A2的表达水平与胃癌患者的总体生存时间相关,COL1A2高表达胃癌患者的总生存时间降低。亚组分析发现,COL1A2表达水平与肠型、弥漫型胃癌患者OS呈负相关,而在混合型胃癌患者中,其表达水平对OS无影响。我们进一步通过基于TCGA数据集的UALCAN数据库验证了这一结论。本研究中,我们利用基因芯片数据库荟萃分析了各独立研究,样本量大,结论的可信度较强。
4 结 论
本研究基于Oncomine数据库分析发现,在mRNA转录水平上,与正常胃组织相比,COL1A2基因在胃癌组织中高表达,进行在线生存分析发现,与COL1A2基因低表达组相比,COL1A2基因高表达的胃癌患者总体生存期降低,进一步亚组分析发现,COL1A2基因高表达的肠型、弥漫型胃癌患者总体生存期降低。本研究结果为胃癌的COL1A2分子机制的研究和靶向药物治疗提供了理论支持。
作者声明:本文第一作者对于研究和撰写的论文出现的不端行为承担相应责任;
利益冲突:本文全部作者均认同文章无相关利益冲突;
学术不端:本文在初审、返修及出版前均通过中国知网(CNKI)科技期刊学术不端文献检测系统学术不端检测;
同行评议:经同行专家双盲外审,达到刊发要求。

