宏基因组学技术在水产动物病毒鉴定中的应用
彭司华 江守文 孙丹 吴智超 陈洁

摘要:近年来水产养殖生产中病毒性疾病的发生越来越严重,所以对水产动物病毒的深入研究成为水产研究者的一个重要课题。随着宏基因组学技术的不断发展,以及测序成本的快速下降,基于宏基因组学技术的水产动物病毒鉴定技术逐渐成为水产动物病毒鉴定的主要技术之一。本文总结了基于宏基因组学的水产动物病毒鉴定技术的主要方法,详细综述了鉴定技术的实现流程,评述了几种基于宏基因组学的水产动物病毒鉴定技术的优缺点,并对基于宏基因组学的水产动物病毒鉴定技术的未来发展方向进行了展望。
关键词:水产动物病毒;宏基因组学;病毒鉴定;二代测序
中图分类号:s941.41 文献标识码:A 文章编号:1000-4440(2019)01-0229-09
中国水产养殖产业飞速发展,最近29年产量一直居世界第一。据《2016年全国渔业经济统计公报》显示,2016年中国水产品总产量达到6.9x10(7)t,其中养殖水产品产量5.1×10(7)t,,占水产品总产量的74.5%。据联合国粮食及农业组织《世界渔业和水产养殖2016》报道,中国的水产养殖产量占全球水产养殖产量的60%以上。然而,近些年来世界各国都遭遇到一个制约水产品产业发展的严重问题,即水产养殖生产中病毒性疾病的发生越来越严重。针对这一挑战,世界各国病毒学家都对水产动物病毒的研究给予了高度重视,并获得了大量的研究成果。目前已从养殖和野生水生动物中鉴定出包括虹彩病毒、呼肠孤病毒、弹状病毒、疱疹病毒和双RNA病毒等不同病毒。尤其是近年来,随着二代和三代测序组测序技术的发展,已经获得了许多水产动物病毒的全基因组序列,这给水产动物病毒的深入研究打下了良好的基础。
宏基因组学(Metagenomics)是通过直接从环境样品中提取全部微生物的DNA,构建宏基因组文库,利用基因组学的研究策略研究样品所包含的全部微生物的基因组,并可刻画其中包含的微生物的群落功能。随着宏基因组学技术的深入发展,给水产动物病毒的鉴定与发现带来了很多便利。基于宏基因组学发现病毒,过去10年来发表了很多十分有见地的综述,但随着宏基因组学技术的进步,需要介绍最近几年来新的技术进展。2018年Greninger发表了关于基于宏基因组学发现RNA病毒的很深刻的综述,但对于水产病毒领域的学者,更希望看到关于针对水产动物病毒更详尽的综述。尽管在水产动物病毒鉴定方面,Munang'andu最近发表了一篇较为详尽的综述,但文章着重点在病毒发现的结果描述方面,而在介绍病毒鉴定的实现流程方面,则较少涉及。为弥补以上综述文献的不足,我们以水产动物中的鱼类为重点,着重介绍基于宏基因组的水产动物病毒鉴定实现流程的最新进展,并展望未来技术的发展方向。
本文按测序平台推出的先后顺序介绍6种测序平台,包括二代和三代测序技术。简要介绍各种测序平台在病毒鉴定中的应用。并介绍了利用新一代测序技术(Next generation sequencing,NGS。含二代和三代測序技术)用于水产动物病毒的鉴定案例,其中以养殖和野生的鱼类病毒鉴定为重点。详细总结基于新一代测序技术发现的鱼类病毒的种类,所属病毒家族,宿主来源等信息。对基于新一代测序技术和宏基因组学技术进行水产动物病毒鉴定的详细操作流程进行综述。
1基于宏基因组学水产养殖病毒鉴定的产生背景
传统的病毒鉴定方法主要有病毒培养、电镜观察、血清学反应和PCR方法等,操作繁琐,费时费力,且鉴定的效率较低。传统的方法在鉴定新病毒方面主要缺陷有:(1)很多病毒缺乏特异的细胞系,使得至少60%以上的病毒无法获得;(2)用电镜来观察病毒其灵敏性较低,许多情况下根本无法观察到;(3)由于高效的特异性抗体很难获得,用血清学反应来鉴定病毒也不理想;(4)而基于PCR法的病毒鉴定,必须首先得到病毒的DNA序列,这对于未知序列的病毒,以及变异性大的病毒的鉴定事实上很难实现。
而基于二代测序的病毒宏基因组学方法直接利用鱼类样本中的病毒DNA序列作为研究对象。能够快速,有效地发现各种未知鱼类病毒,而且能直观地显示各种鱼类样本中病毒的种类与丰度。这样就可以方便地了解病毒的分布情况,对某些病原和潜在致病病毒的变化进行实时监测。同时有望诊断由新的或未知病原体引起的鱼类疾病。
2二代与三代测序技术简介
2.1 454测序或焦磷酸测序技术
这是基于宏基因组学病毒发现的第一种高通量测序技术。焦磷酸测序技术的原理是:引物与模板DNA退火后,在ATP硫酸化酶,DNA聚合酶,双磷酸酶和荧光素酶4种酶的共同作用下,将引物上每一个dNTP的聚合与一次荧光信号的释放偶联起来,实现实时测定DNA序列。454测序被广泛用于鉴定来自人和动物的新病毒,例如虫媒病毒(Or-biviruses),冠状病毒,y乳头瘤病毒(Gamma-papillomavirus)和蝙蝠肝炎病毒和Pegivillls等。虽然这种技术比Sanger测序有更高的通量,但是由于其成本高,错误率较高,且在测序通量方面比不上Illumina等技术,因此,该技术已被其他NGS技术所取代。
2.2 Ion torrent测序技术
这是Life Technologies/Thermo Fisher Scientific开发的一种基于半导体测序技术的测序平台,这种技术最早在2012发布,这个测序平台在基本原理上与454焦磷酸测序平台基本相似。这些快速的测序技术很快被应用到病毒的临床检测中,例如乙型肝炎病毒_24],HIV病毒,HCV。采用Ion tor-rent测序技术,对许多病毒进行了病毒基因组测序,例如正交病毒、猪繁殖与呼吸综合征病毒、多瘤病毒、托斯卡纳病毒、蓝舌病病毒、轮状病毒和流感病毒等。
2.3 ABI SOLID测序技术
SOLID使用了连接法测序的策略,该测序平台通过检测“双碱基编码原理”的颜色编码序列的原理,实现了DNA序列的检测。在得到原始数据后的的数据分析流程中,把原始颜色序列和转换成颜色编码的参考序列进行对比,从而实现把SOLID颜色序列准确定位到参考序列上,从而实现测序的目的。连接测序所采用的底物是8个碱基荧光探针的混合物,只根据序列的位置,所测DNA就可以被探针准确标记。Solid的缺点是读长只有50~75 bp,但测序的精确度较高,可达99.99%[34-35]。由于读长太短,现在在病毒鉴定中已经不采用这种测序技术。
2.4 mumina测序技术
这是Solexa/Illumina开发出的一个高通量测序平台,Illumina测序技术采用边合成边测序的策略来实现测序。首先,在DNA片段两端加上通用接头来构建文库,然后把文库加载到Flowcell芯片上,经过桥式PCR反应后,对每一条文库片段都进行扩增,结果形成一个簇,通过边合成边测序反应,在经过多次循环后,最后获得完整的核酸序列。采用这种测序技术发现的病毒有血小板减少症病毒,猿猴腺病毒,原病毒属病毒等。
2.5PacBio测序技术
PacBio测序技术是第三代基因测序技术,又被称为单分子实时DNA测序技术Single mole-cule real time(SMRTT-TM)DNA sequencing]。該方法基于纳米孔的单分子读取技术,不需要扩增即可快速读取序列。Pacific Biosciences的SMRT测序平台,被称为RS II,该技术可产生长序列PacBio测序技术非常适合病毒宏基因组学研究,因为测序读长可以达到10 kb以上。但是PacBio测序成本较高,且测序的错误率较高,测序精确度只能达到86%左右。但是由于测序读长较长,这种技术将成为未来最主要的病毒鉴定测序技术之一。
2.6 Nanopore测序技术
Nanopore是一种单分子实时测序的新一代测序方法,其以单分子DNA(RNA)通过生物纳米孔的电流变化推测碱基组成来进行测序。Nanopore测序准确率和PacBio持平,为86%左右。目前Nanopore的长序列主要应用于高质量基因组的组装,尤其是对于高杂合,高重复,大基因组等复杂基因组。2016年前,主要用于一些小基因组如大肠杆菌,酵母,线虫等,目前已经应用在人、野生番茄、香蕉、欧洲鳗等多个复杂基因组组装上。由于测序读长较长,这种技术也会成为未来病毒鉴定最重要的测序技术之一。
3新一代测序技术和宏基因组学在水产新病毒发现上的应用
病毒宏基因组学已经成功用于新的水生动物病毒发现研究,包括鱼类、甲壳类动物、软体动物、海龟和海洋哺乳动物,本文以鱼类新病毒发现研究成果为重点进行评述。
2011年以来,应用二代测序和宏基因组学技术,鉴定了许多养殖和野生鱼类病毒,例如虹彩病毒科病毒。在大西洋三文鱼、淡水鲤鱼和金体美洲鳊鱼体内鉴定了呼肠孤病毒,在欧洲鲶鱼体内鉴定了圆环病毒,在鲤鱼和金体美洲鳊鱼体内发现了小RNA病毒科病毒,在鲤鱼体内鉴定了微小核糖核酸病毒属病毒,在白亚口鱼和大太阳鱼体内鉴定了嗜肝DNA病毒,在金体美洲鳊鱼体内鉴定了整体病毒科病毒,在钓饵鱼体内鉴定了黑头豆科杯状病毒1。这些基于二代测序和宏基因组学鉴定的鱼类病毒案例的详细信息见表1。
此外,二代测序技术和宏基因组学在甲壳动物虾、贝类、海星、佛罗里达绿海龟以及加利福尼亚海狮、太平洋海豹、白喙海豚、海狗等水产动物为宿主的病毒发现中也得到广泛应用。
4病毒鉴定流程
仅仅采用二代测序技术鉴定病毒是一个费时费力的工作,且要耗费较多的资金,现在已经较少采用这种方法来鉴定病毒了。随着宏基因组学技术的成熟,二代测序加上宏基因组学技术成为鉴定病毒的主流技术。由于绝大多数微生物无法人工培养,造成仅基于二代测序的方法鉴定病毒有很大的局限性。而基于宏基因组学技术可以克服大多数微生物不能培养的缺点,使得基于宏基因组学的病毒鉴定得到广泛采用。
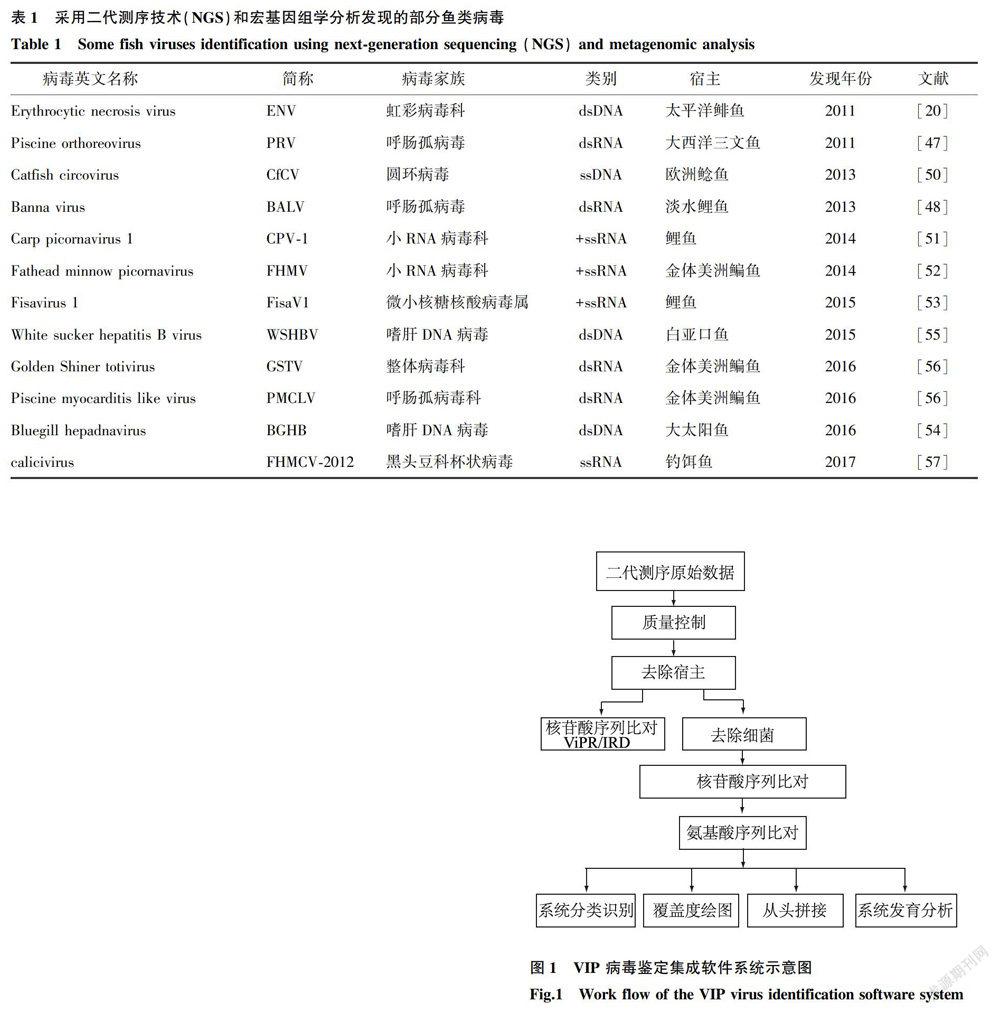
一些研究者开发了一些基于宏基因组学的病毒鉴定流程软件(Pipeline for virus identification anddiscovery),例如中国疾控中心IJi等开发的VIP病毒鉴定集成软件系统,如图1所示。
Lin等则开发了另一种集成软件系统drVM。但这些集成软件系统虽然使得系统容易使用,但它们往往只集成固定的几种软件。而实际上宏基因组学分析软件更新换代非常快,这就使得现有的集成软件系统的性能不一定是最好的。因此选择当时最好的软件来构建自己的分析流程是最好的选择。当然这需要较多的生物信息学软件使用经验。Tengs等在大西洋三文鱼的病毒鉴定中提出了一个鱼类病毒的鉴定流程,根据文中的描述,我们绘制出该病毒鉴定流程示意图(图2)。在基本分析框架不变的情况下,我们可以更换分析流程中的任何软件。例如该文作者采用了Velvet软件来做宏基因组拼装,实际上我们可以把Velvet用其他宏基因组拼接软件来代替,例如MEGAHIT、Spades、SOAPdenov02等。质量控制工具PRINSEQ也可以用TRIMOMMATIC、NGS 9c Toolkit等软件来代替。在对数据进行微生物分类学鉴定时采用的METAXA2软件也可以换成普遍采用的同类型软件QIIME。
我们课题组在研究青藏高原土壤与水体宏基因组研究中,采用Torstein Tengs等使用的Pipeline流程,成功地鉴定了一种病毒。此外,我们还开始了鲤鱼肠道宏基因组学研究,目前做的项目有鲤鱼16S RNA测序,鲤鱼肠道宏基因组测序。虽然目前我们测序的鲤鱼样本是健康的鲤鱼,但我们仍然开始了鲤鱼肠道病毒筛选工作。后续还打算对发病的鲤鱼样本进行深度测序,并初步打算增加三代测序方法,这样既可以拼接到更多的鲤鱼肠道微生物全长基因组,又可能筛选到鲤鱼肠道中的病毒。
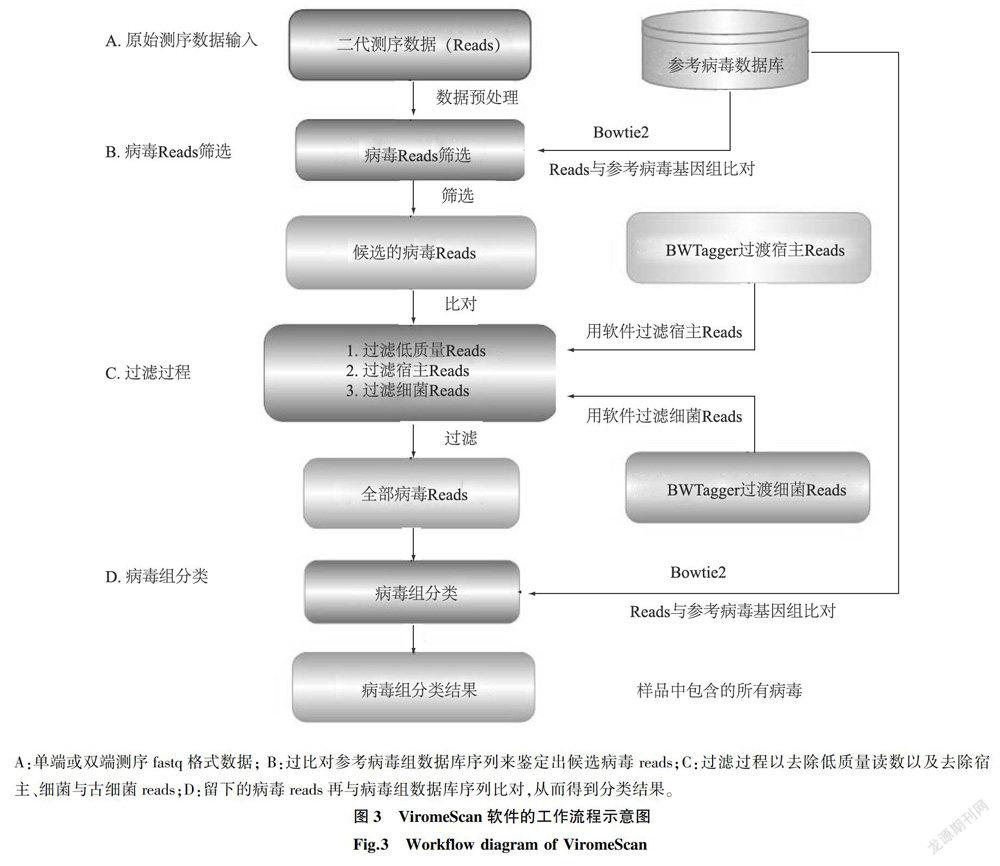
Rampelli等开发的ViromeScan,其实现流程如3所示。这个软件只要输入二代测序fastq格式的原始数据(Raw data),不需要拼接宏基因组,经过多步运算后就可以得到病毒组分类结果。其中采用的病毒组数据库可以在NCBI病毒基因组数据库下载。在过滤宿主基因组和细菌Reads时,均采用了BMTagger。
最新报道的几种Pipeline病毒鉴定方法值得关注,Ren等推出了VirFinder。Lin等开发了基于web的鉴定流程Vipie,另外一种新方法是Tithi等提出的基于单机版的病毒鉴定流程FastVirome-Explorer。这几种方法和上面介绍的3种方法实现流程虽有差别,但也基本相似,读者可以参考原文了解详细实现流程。
5展望
本文综述了基于新一代测序和宏基因组学进行水产动物病毒鉴定和发现的基本原理和主要技术。尤其是随着近年来宏基因组学技术的完善以及测序成本的持续大幅度下降,使得宏基因组学技术已经成为水产动物疾病诊断和水产养殖新病原发现的一种重要工具。就新型病毒的发现而言,从装配方法(De novo assemblies)与生物信息学注释工具相结合,简化了鉴定与获得完整病毒基因组的步骤。因此,病毒宏基因组学可以用作新型病毒鉴定,病毒系统发育分析,病毒感染诊断,以及疾病监测的工具。在病毒宏基因组学分析中面临的一个重要挑战是,使用宏基因组学技术得到的大部分病毒序列仍然未被注释,这主要是因为它们与普通数据库中的任何已知序列没有相似性_。Mokili等认为,目前注释未知病毒序列不完善的主要原因还是缺乏合适的生物信息学分析工具。IJi等指出,病毒序列在装配中的问题之一是它们有时会形成嵌合重叠群,这些嵌合重叠群在拼接时很难识别。
病毒宏基因组学分析面临的另一个重要挑战是,装配的病毒基因组的准确性还有待提高。使用宏基因组学分析鉴定新型病毒的注释方法应力求避免出现假阳性。鉴于大多数感染水生动物的病毒都没有被鉴定,而且在数据库中也没有发现这种病毒,所以新感染水生动物病毒的这种不准确性可能更高。尽管如此,我们认为基于基因组学的水产动物病毒鉴定将随着宏基因组学技术的不断成熟,测序成本的不断下降,以及已知病毒数据库的不断完善而得到更广泛的采用。
新型纳米孔测序技术Nanopore测序是一种非常有前途的测序技术。由于该测序仪的小型化以及样品准备极其简单,且不需要借助碱基、多聚酶、连接酶等就能进行测序,尤其适合野外携带。所以,Nanopore测序技术有望在水产动物病毒鉴定方面得到更广泛的采用。
我们在表1中可以发现,一种病毒可能感染多种鱼类,例如呼肠孤病毒可以感染大西洋三文鱼、淡水鲤鱼、金体美洲鳊鱼等,但感染的分子机制仍未揭示。随着宏基因组学的发展和完善,更多的病毒序列全长将会得到。这将给揭示病毒感染的机制带来很多方便,并将有力促进鱼病药物的研发,为更好地控制鱼病疫情打下基础。

