茶树光敏色素基因家族成员的生物信息学及其表达量与黄酮含量的相关性分析
莫晓丽 周子维 把熠晨 武清扬 赖钟雄 孙云

摘要:【目的】分析茶树光敏色素(PHY)基因(CsPHY)的生物学信息及不同采摘时间点茶叶中光敏色素基因表达量与黄酮含量的相关性,为茶叶适时采摘及品质调控提供理论参考。【方法】从茶树基因组数据库(CSA)中筛选出CsPHY基因家族成员,对其进行生物信息学分析,并通过邻接法(NJ)构建系统发育进化树。以一天中不同时间点0:00(R0)、6:00(R6)、12:00(R12)和18:00(R18)采摘的乌龙茶品种肉桂鲜叶为试材,通过实时荧光定量PCR(qRT-PCR)检测CsPHY基因家族成员在不同时间点的表达量,并采用三氯化铝比色法检测茶叶中黄酮含量的日变化,以皮尔逊(Pearson)相关系数评估CsPHY基因相对表达量与黄酮含量的相关性。【结果】共筛选获得6个CsPHY基因家族成员,编码蛋白的氨基酸数量674~1142个,分子量为76126.75~126714.53 Da,等电点(pI)为5.74~5.96,脂溶性系数为91.11~95.27,其中,CsPHY1基因属于PHYC亚族,CsPHY2和CsPHY4基因属于PHYA亚族,CsPHY3和CsPHY5基因属于PHYB亚族,CsPHY6基因属于PHYE亚族,说明CsPHY2和CsPHY4蛋白属于Phy I型光敏色素,CsPHY1、CsPHY3、CsPHY5和CsPHY6蛋白属于Phy II型光敏色素。6个CsPHY蛋白均与双子叶植物PHY蛋白的亲缘关系较近,其中,CsPHY2蛋白与水晶兰PHYA亚族蛋白的亲缘关系较近,CsPHY4蛋白与美味猕猴桃PHYA亚族蛋白的亲缘关系较近;CsPHY3和CsPHY5蛋白均与美味猕猴桃PHYB亚族蛋白的亲缘关系较近,且CsPHY5蛋白与葡萄PHYB亚族蛋白的亲缘关系相近;CsPHY1蛋白与河岸葡萄PHYC亚族蛋白的亲缘关系较近,CsPHY6蛋白与中华猕猴桃和河岸葡萄PHYE亚族蛋白的亲缘关系较近。CsPHY1、CsPHY3、CsPHY4和CsPHY5基因在R6時的表达量最高,其中,CsPHY4和CsPHY5基因在R6时的表达量显著高于其他时间点的表达量(P<0.05,下同),而CsPHY2和CsPHY6基因分别在R12和R18时的表达量达最高,其中CsPHY6基因在R18时的表达量显著高于R12时的表达量。在R0~R18时段内茶树鲜叶中的黄酮含量无显著变化(P>0.05),整体呈先下降后上升趋势,在R18时达最高值,在R12时最低值。6个CsPHY基因家族成员中,仅CsPHY6基因与黄酮间呈极显著正相关(P<0.01)。【结论】CsPHY基因家族中仅缺少PHYD亚族成员,尽管各成员间功能结构较相似,但表达模式存在明显差异。光照影响茶树叶片中黄酮含量,CsPHY6基因编码的光受体蛋白CsPHY6参与调控茶树叶片中的黄酮含量。
关键词: 茶鲜叶;采摘时间;光敏色素基因;qRT-PCR;黄酮
中图分类号: S571.1 文献标志码: A 文章编号:2095-1191(2019)06-1173-10
Abstract:【Objective】By analyzing the bioinformatics of tea phytochrome(PHY) gene(CsPHY) and correlation between its expression and the content of flavonoids in tea plucked at different times,the objective of this study was to provide theoretical references for timely plucking and quality control of tea. 【Method】Tea tree CsPHY gene family members were selected from Tea Tree Genome Database(CSA),their bioinformatics were analyzed and phylogenetic tree was constructed through Neighbor-Joining method (NJ). Oolong tea Rougui plucked at different times 0:00(R0), 6:00(R6), 12:00(R12), 18:00(R18) in one day were used as materials, the expression patterns of PHY gene family members at di-fferent times were analyzed by real-time fluorescence quantitative(qRT-qPCR). The daily variation of flavonoids in tea was detected by AlCl3 colorimetry, the correlation between relative express levels of CsPHY genes and the content of flavonoids were evaluated by Pearson correlation coefficient. 【Result】Six CsPHY gene family members were obtained. The amino acids number of coded protein was 674-1142, the molecular weight was 76126.75-126714.53 Da, the isoelectric point(pI) was 5.74-5.96, and the aliphatic index was 91.11-95.27. CsPHY1 gene belonged to PHYC subfamily, CsPHY2 and CsPHY4 genes belonged to PHYA subfamily, CsPHY3 and CsPHY5 genes belonged to PHYB subfamily, CsPHY6 gene belonged to PHYE subfamily, indicating that CsPHY2 and CsPHY4 proteins belonged to Phy I type phytochrome, CsPHY1, CsPHY3, CsPHY5 and CsPHY6 proteins belonged to Phy II type phytochrome. The six tea tree CsPHY proteins were closely related to the PHY protein of dicotyledonous plants. Among them,the CsPHY2 protein was closely related to the PHYA subfamily protein of Monotropastrum globosum, and the CsPHY4 protein was closely related to the PHYA subfamily protein of Actinidia deliciosa. CsPHY3 and CsPHY5 proteins were closely related to the PHYB subfamily protein of the A. deliciosa, and CsPHY5 protein was closely related to the PHYB subfamily protein of Vitis vinifera. CsPHY1 protein was closely related to the PHYC subfamily of V. riparia. CsPHY6 protein was closely related to the PHYE subfamily proteins of A. chinensis and V. riparia. The expression levels of CsPHY1,CsPHY3,CsPHY4 and CsPHY5 genes were the highest at R6. The expression levels of CsPHY4 and CsPHY5 genes at R6 were significantly higher than those at other time points(P<0.05,the same below). The highest expression levels of CsPHY2 and CsPHY6 genes were at R12 and R18 respectively, and the expression level of CsPHY6 gene at R18 was significantly higher than that at R12. There was no significant change in the content of flavonoids in fresh tea leaves during the period of R0-R18(P>0.05). The trend of flavonoids content declined first and then upward, reaching the peak at R18 and the lowest at R12. Among the six CsPHY gene family members, only CsPHY6 gene was extremely positively correlated with flavonoids(P<0.01). 【Conclusion】Only the PHYD subfamily member is missing from the tea tree CsPHY gene family. Although the functional structures are similar, there are great difference in expression patterns. Light affects the content of flavonoids in tea leaves, and CsPHY6 gene-encoded photoreceptor protein CsPHY6 is involved in the regulation of flavonoids content in tea leaves.
Key words: fresh tea leaves; plucking time; phytochrome gene; qRT-PCR; flavonoid
0 引言
【研究意义】在自然光条件下,植物通过光受体感知光周期和光质,从而影响植物次生代谢。光敏色素(Phytochrome,PHY)是一类吸收红光—远红光可逆转换的光受体蛋白(色素蛋白质),广泛存在于蓝细菌、低等植物和高等植物体中(Rockwell and Lagarias,2010),属可溶性色素蛋白。茶树次生代谢产物是茶叶重要的功能成分,对茶叶品质起决定性作用,如黄酮类化合物是乌龙茶中重要的品质和功效成分,同时影响乌龙茶滋味和汤色品质(罗丽和郭雅玲,2004;夏涛和高丽萍,2009)。研究发现,光处理对黄酮类化合物合成有明显促进效果(徐茂军等,2003;张晓燕等,2014),说明PHY间接调控茶叶的品质。茶树鲜叶采摘是茶叶生产环节的第一步,采摘时间的差异通常对成品茶品质造成影响。恰当的茶葉采摘时间不仅有利于后续加工工艺的开展,还可影响自然光条件下茶树PHY基因(CsPHY)的表达模式,从而间接影响茶叶内含物的合成,因此,研究茶树PHY相关基因的表达模式及与黄酮日变化的相关性对解析早、午、晚青付制成品茶造成的品质差异并制定适宜的茶叶采摘时间提供理论参考。【前人研究进展】植物中的PHY基因家族至少含3个成员(Mathews et al.,1995)。模式植物拟南芥PHY基因分别属于PHYA、PHYB、PHYC、PHYD和PHYE亚族,其中PHYA、PHYB和PHYC亚族基因的序列相似性低于50%,但PHYD和PHYB亚族基因的相似度较高,其原因是PHYD亚族是在拟南芥最后一次全基因组复制事件中从PHYB亚族派生出来(Sharrock and Quail,1989)。PHYA亚族基因编码的蛋白属PhyⅠ型光敏色素,接收波长700~750 nm连续远红光,对光反应不稳定,在光照下其mRNA活性受到抑制;PHYB、PHYC、PHYD和PHYE亚族基因编码的蛋白属光敏Phy Ⅱ型光敏色素,具有高度的光稳定性,不受光影响,接收600~700 nm红光,属组成型表达(Clark et al.,1994;马晓净,2018)。后续研究发现,番茄中也存在上述5个亚族的PHY基因(Alba et al.,2000);水稻中仅有PHYA,PHYB和PHYC亚族成员(王静和王艇,2007),但至今尚未在单子叶植物中发现PHYE亚族基因。目前,PHY的生理功能研究主要集中在参与植物形态建成,包括种子萌发(Franklin and Quail,2010)、开花诱导(顾建伟等,2011)、去黄化作用(刘栓桃等,2014)等,以及对不同光信号的应激反应和化学成分调节(Rockwell et al.,2006)等方面。Alba等(2000)研究发现,番茄果实中番茄红素的积累与光敏色素有关。陈碧薇(2014)研究表明,沉默番茄PHY基因下游转录因子可提高番茄果实的营养品质、抗氧化能力及类黄酮含量。李娜等(2017)研究发现,蓝光照射能提高大豆芽苗菜中类黄酮合成关键酶的活性,并上调相关基因的表达水平。刘转霞等(2017)研究发现,香蕉含PHYA、PHYB、PHYC1和PHYC2等4种PHY基因,其中PHYC1基因能积极响应红光刺激。牛骧等(2017)研究发现,玉米在不同光质和光周期处理下2个PHYC基因在玉米形态建成中发挥关键作用。【本研究切入点】至今,鲜见有关CsPHY基因家族成员的表达模式及黄酮日变化规律的研究报道。【拟解决的关键问题】从茶树基因组数据库中筛选出CsPHY基因家族成员,并对其进行生物信息学分析;以一天中不同时间点0:00(R0)、6:00(R6)、12:00(R12)和18:00(R18)采摘的乌龙茶品种肉桂鲜叶为试材,通过实时荧光定量PCR(qRT-PCR)检测CsPHY基因家族成员在不同时间点的表达量,并采用三氯化铝比色法检测茶叶中黄酮含量的日变化,以皮尔逊相关系数评估CsPHY基因相对表达量与黄酮含量的相关性,为解析早、午、晚青付制成品茶的品质差异及制定适宜的茶叶采摘时间提供理论参考。
1 材料与方法
1. 1 试验材料
供试乌龙茶品种肉桂(Camellia sinensis cv. Rougui)是福建武夷岩茶的当家品种,付制乌龙茶香气辛锐持久、带桂皮香、滋味醇厚回甘,深受消费者青睐,由福建农林大学茶学教学科研实践基地茶树资源圃提供。主要试剂:RNAprep Pure Plant Kit(DP432)试剂盒购自天根生化科技(北京)有限公司,PrimeScriptTM RT Reagent Kit with gDNA Eraser(Perfect Real Time)试剂盒(RR047A)和SYBR® Premix Ex Taq II试剂盒均购自宝生物工程(大连)有限公司,其他生化试剂均购自国药集团化学试剂有限公司。1% AlCl3溶液的配制:称取三氯化铝(AlCl·6H2O)7567 g,加水溶解并稀释至100 mL。qRT-PCR引物委托铂尚生物技术(上海)有限公司合成。主要仪器设备:Allegra 64R台式高速冷冻离心机(Beckman Coulter,美国)、超微量核酸分析仪(Themo,美国)、实时荧光定量PCR仪(Roche,美国)、UV-5900(PC)型紫外可见分光光度计(上海元析仪器有限公司)和电泳凝胶成像系统(BIO-RAD,美国)。
1. 2 试验方法
1. 2. 1 样品采集 2018年5月分别于0:00(R0)、06:00(R6)、12:00(R12)和18:00(R18)4个时间点采集乌龙茶品种肉桂的鲜叶,采摘标准为年生长无病虫害的中开面三四叶约20.0 g。其中,5.0 g鲜叶用锡箔纸包好,立即放入液氮中3 min,然后取出样品快速放入-70 ℃冰箱中保存备用,用于总RNA的抽提;剩余15.0 g鲜叶微波(中高火,3 min)烘烤,再充分研磨成粉末,用于黄酮含量检测。每次取样设3个重复。
1. 2. 2 CsPHY基因序列筛选 以茶树全基因组数据库(Tea Tree Genome Database,CSA)为基础(Xia et al.,2017),在Pfam功能注释库(Functional annotations,TSV)中以PHY基因典型蛋白结构域的登录号(PF00989)为条件进行检索,并将搜索获得的CsPHY基因序列片段依次命名为CsPHY1、CsPHY2、CsPHY3、CsPHY4、CsPHY5和CsPHY6。
1. 2. 3 生物信息学分析 采用ExPASy对CsPHY基因进行理化性质预测,并在NCBI中进行同源序列比对,利用MEGA 7.0的邻接法(NJ)构建系统发育进化树。根据CsPHY基因编码蛋白的氨基酸序列,在motif research网站上进行基序预测分析,并利用SMART预测其结构域。
1. 2. 4 总RNA提取及cDNA合成 参照RNAprep Pure Plant Kit说明以离心柱法抽提茶树叶片中的总RNA,利用超微量核酸分析仪检测其浓度和纯度,1.2%琼脂糖凝胶电泳检测其完整性。采用PrimeScriptTM RT Reagent Kit逆转录合成cDNA,将获得的cDNA文库置于-20 ℃条件下贮藏备用。
1. 2. 5 qRT-PCR检测 采用SYBR® Premix Ex Taq II试剂盒对不同采摘时间叶片的CsPHY基因表达情况进行检测。利用DNAman设计目的基因的特异性引物如表1所示。以CsGADPH、18S rRNA和CsACT为内参基因(Zhou et al.,2019)。反应体系20.0 μL:SYBR Green Master Mix 10.0 μL,正、反向引物各0.8 μL,灭菌水7.4 μL,cDNA模板1.0 μL。扩增程序:95 ℃ 30 s,95 ℃ 5 s,60 ℃ 20 s,进行40个循环。
1. 2. 6 黄酮含量测定 采用三氯化铝比色法测定茶叶中黄酮含量:取研磨成粉末的茶样1.00 g,加入40.0 mL水,于沸水浴中提取30 min后过滤,加水定容至50.0 mL后摇匀,吸取0.5 mL试液,加入1%三氯化铝定容至10.0 mL,摇匀,静置10 min后比色,以空白试剂作参比,于波长420 nm处测吸光度,计算公式如下:
其中,E为试液测得的吸光度;320为当E=1.00时相当于320 µg黄酮;L1为试液总量;L2为测定时的用液量;M为试样干重。
1. 3 统计分析
利用多内参法2-△Ct校正qRT-PCR定量试验结果(Zhou et al.,2019)。采用Graphpad Prism 6.0和PASW Statistics 18.0对试验数据进行分析及制图,利用皮尔逊相关系数(Ahlgren et al.,2003)评估PHY基因的表达量与黄酮含量的相关性。
2 结果与分析
2. 1 CsPHY基因筛选结果及其编码蛋白的理化性质分析结果
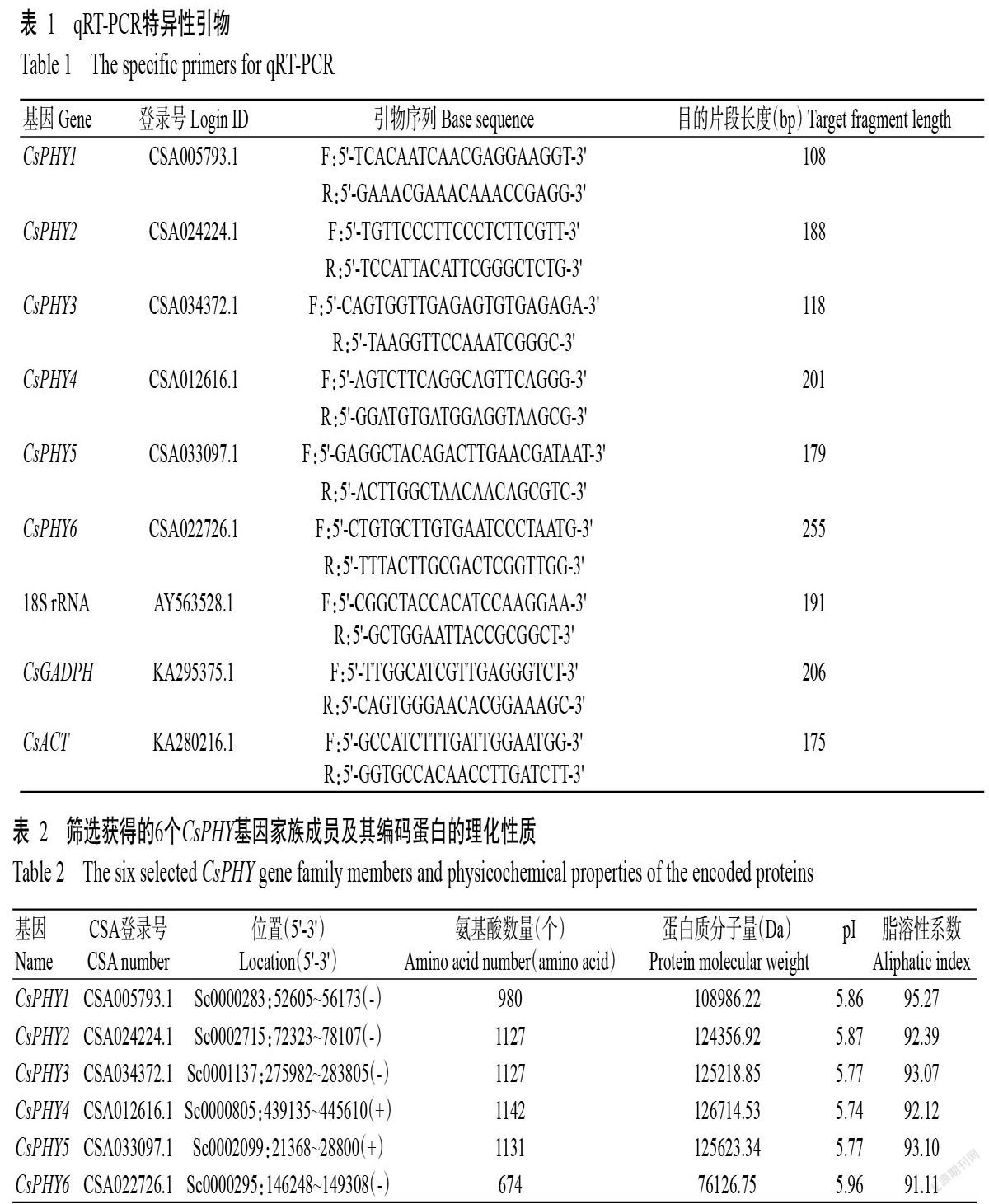
从茶树全基因组数据库中筛选获得6个CsPHY基因家族成员,依次命名为CsPHY1、CsPHY2、CsPHY3、CsPHY4、CsPHY5和CsPHY6,其相关理化性质如表2所示。6个CsPHY基因家族成员编码蛋白的氨基酸数量674~1142个,分子量为76126.75~126714.53 Da,其中以CsPHY4蛋白的氨基酸数量和分子量最多,CsPHY6蛋白的最少;等电点(pI)为5.74~5.96,以CsPHY6蛋白最大;脂溶性系数为91.11~95.27,以CsPHY1蛋白最大。
2. 2 CsPHY基因进化分析结果
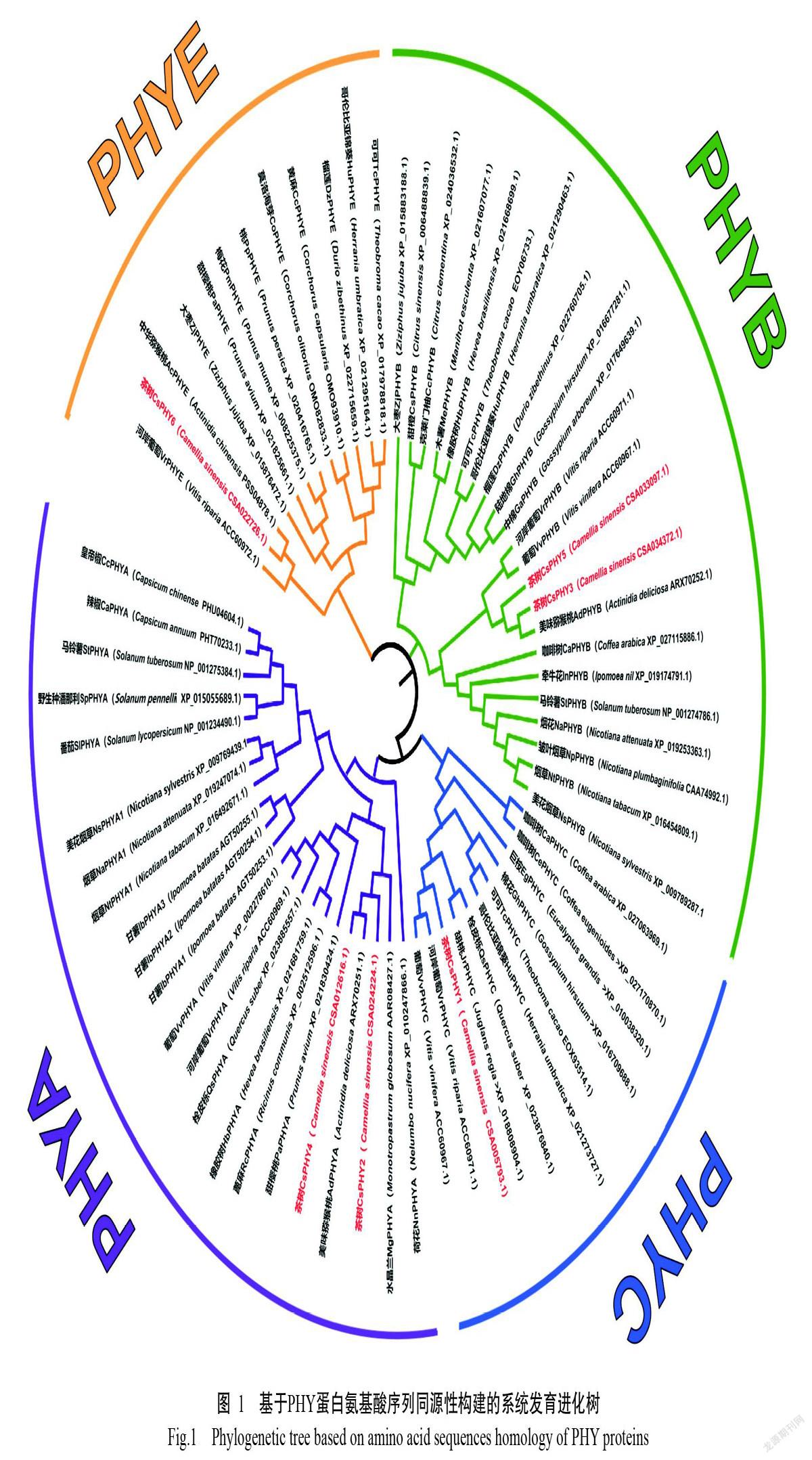
为系统了解CsPHY基因家族成员与其他物种PHY基因的亲缘关系和进化距离,对筛选获得的6个茶树PHY基因编码蛋白的氨基酸序列在NCBI上进行BLASTp同源比对分析,共获得猕猴桃、葡萄等28个其他物种的61个PHY蛋白氨基酸序列,构建的系统发育进化树如图1所示。6个CsPHY基因分别属于PHYA、PHYB、PHYC和PHYE四个亚族,其中,CsPHY1属于PHYC亚族,CsPHY2和CsPHY4基因属于PHYA亚族,CsPHY3和CsPHY5基因属于PHYB亚族,CsPHY6基因属于PHYE亚族,说明CsPHY2和CsPHY4蛋白属于Phy I型光敏色素,CsPHY1、CsPHY3、CsPHY5和CsPHY6蛋白属于Phy II型光敏色素。且6个CsPHY蛋白均与双子叶植物PHY蛋白的亲缘关系较近,其中,CsPHY2蛋白与水晶兰PHYA亚族蛋白的亲缘关系较近,CsPHY4蛋白与美味猕猴桃PHYA亚族蛋白的亲缘关系较近;CsPHY3和CsPHY5蛋白均与美味猕猴桃PHYB亚族蛋白亲缘关系较近,且CsPHY5蛋白与葡萄的PHYB亚族蛋白亲缘关系相近;CsPHY1蛋白与河岸葡萄PHYC亚族蛋白的亲缘关系较近,CsPHY6蛋白与中华猕猴桃和河岸葡萄PHYE亚族蛋白的亲缘关系较近。
2. 3 CsPHY基因结构域和Motif分析結果
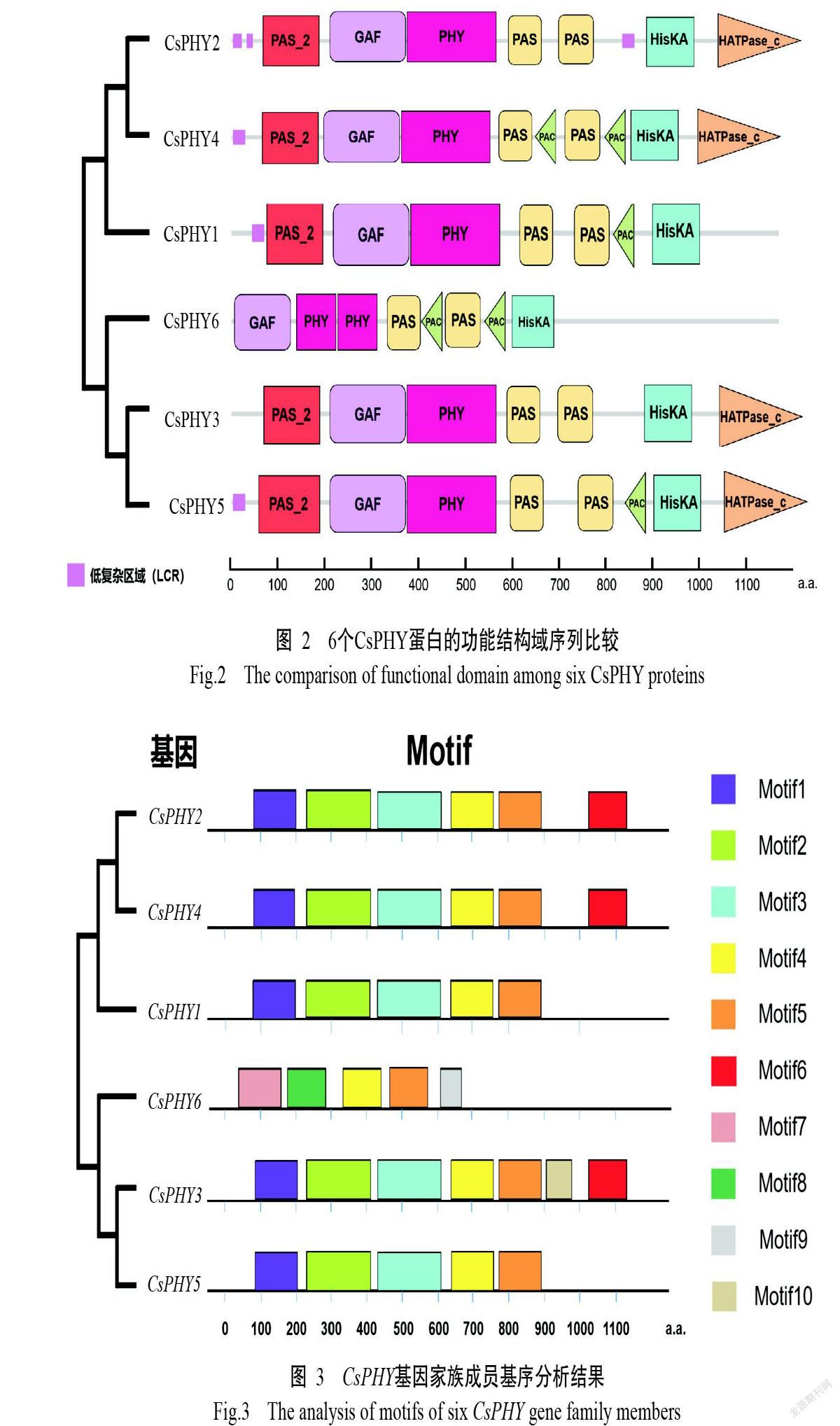
利用SMART在线分析CsPHY蛋白的氨基酸序列,结果如图2所示。6个CsPHY蛋白中共存在7种结构域,分别为PAS_2(PF08446)、GAF、PHY(PF00360)、PAS、PAC、HisKA和HATPase_c。其中,CsPHY1、CsPHY2、CsPHY3、CsPHY4和CsPHY5的N段均具有光敏色素典型的PAS-GAF-PHY结构域,而C端(除CsPHY1外)均具有PAC、HisKA和HATPase_c结构域。虽然CsPHY2和CsPHY4均属于PHYA亚族,但CsPHY4的C端多了2个PAC结构域。CsPHY1属于PHYC亚族,其结构与CsPHY4相似,但C端缺少1个PAC和HATPase_c结构域。CsPHY3和CsPHY5均属PHYB亚族,二者差异主要体现在CsPHY3的C端不具备PAC结构域。CsPHY6属PHYE亚族,与其他成员的差异主要体现在CsPHY6的N端缺少PAS结构域,且其PHY结构域的220~226 aa区间出现7个氨基酸重叠现象,而C端结构域与CsPHY4类似,但均缺乏HATPase_c结构域。
此外,CsPHY1、CsPHY2、CsPHY4和CsPHY5均存在低复杂结构域(图2)。运用Motif Search在线预测6个CsPHY蛋白的基序,结果显示,6个CsPHY蛋白共含10种Motif(Motif1~Motif10),其中,CsPHY1、CsPHY2、CsPHY3、CsPHY4和CsPHY5均存在Motif1~Motif5,而CsPHY2、CsPHY3和CsPHY4的C端还存在长约1000 aa的Motif6,而Motif7~Motif9仅在CsPHY6中存在,同时Motif10仅存在于CsPHY3中的900~950 aa区间内(图3)。
2. 4 茶树PHY基因在不同采摘时间点的表达模式检测结果
不同采摘时间点茶树鲜叶中CsPHY基因的表达量如图4所示。CsPHY1、CsPHY3、CsPHY4和CsPHY5基因在R6时的表达量最高,其中,CsPHY4和CsPHY5基因在R6时的表达量显著高于其他时间点的表达量(P<0.05,下同),而CsPHY2和CsPHY6基因分别在R12和R18时的表达量达最高,其中CsPHY6基因在R18时的表达量显著高于R12时的表达量。可见,CsPHY1、CsPHY2、CsPHY3、CsPHY4和CsPHY5基因的表达量在清晨或中午达峰值,而凌晨和傍晚的表达量较低,但CsPHY6基因的表达量日变化有所不同,在傍晚达峰值。
2. 5 黄酮含量测定结果
不同采摘时间点茶树鲜叶的黄酮含量检测结果如图5所示。在R0~R18时段内茶树鲜叶中的黄酮含量无显著性变化(P>0.05,下同),整体呈先下降后上升趋势,在R18时达最高值,为5.58 mg/g,在R12时最低,为4.61 mg/g。由于R0~R12时段的自然光由弱变强,R12~R18时段的自然光由强到弱,黄酮含量随光强的增强而下降,表明自然光的交替变化能引起茶树叶片中黄酮含量变化。
2. 6 CsPHY基因的表达量与黄酮含量的相关性分析结果
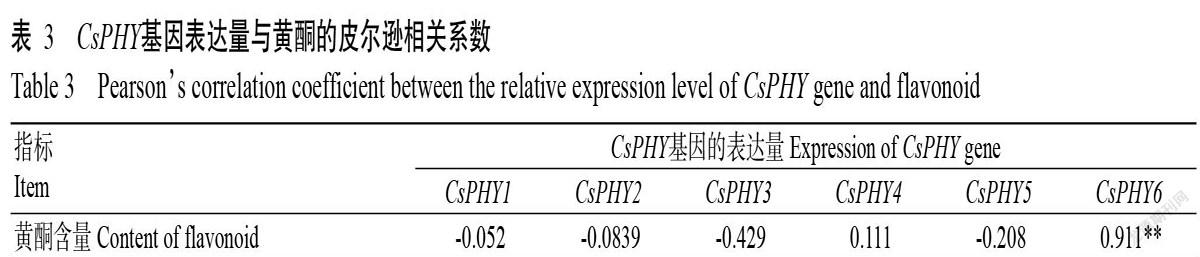
利用皮尔逊相关系数评估茶树CsPHY基因的表达量与黄酮含量的相关性,结果如表4所示。仅CsPHY6基因表达量与黄酮含量呈极显著相关(P<0.01,下同),CsPHY1、CsPHY2、CsPHY3、CsPHY4和CsPHY5基因的表达量与黄酮含量均无显著相关性。由此推测,CsPHY6基因编码的光受体蛋白CsPHY6参与调控茶树叶片中的黄酮含量。
3 讨论
3. 1 CsPHY基因家族成员的筛选及进化分析
前人研究发现,拟南芥中存在PHYA、PHYB、PHYC、PHYD和PHYE 5种不同亚族的光敏色素基因,其过表达会影响植株的株形和光合效率(Bradley et al.,1996)。本研究筛选获得6个典型CsPHY基因,将其编码蛋白与其他物种PHY蛋白进行氨基酸序列同源比对分析,结果发现属于PHYA和PHYB亚族的茶树基因各有2个,属于PHYC和PHYE亚族各有1个,其中CsPHY3(PHYB亚族)和CsPHY4(PHYA亚族)分别与美味猕猴桃PHYB和PHYA亚族蛋白的亲缘关系较近,而CsPHY6(PHYE亚族)与中华猕猴桃PHYE亚族蛋白的遗传距离较近,而CsPHY1(PHYC亚族)与河岸葡萄PHYC亚族蛋白的亲缘关系较近,CsPHY5(PHYB亚族)与美味猕猴桃和葡萄PHYB亚族蛋白的亲缘关系均较近。在漫长的进化过程中茶树基因组经历过两次全基因组复制事件,通过分析旁系同源基因对的同义替换率(Ks)分布情况,发现茶树与葡萄和中华猕猴共同经历了第一次的全基因组复制事件(Xia et al.,2017),为茶树、中华猕猴桃和葡萄基因家族间存在同源性较高提供一定佐证,但有待进一步探究。
本研究未发现茶树PHYD亚族基因,可能是茶树进化历程出现的基因功能冗余所造成。Aukerman等(1997)研究发现,拟南芥PHYD亚族蛋白的结构与PHYB亚族蛋白相近,但二者均可响应避光调节,表明植物中天然存在PHYD-1突变现象。李建平等(2014)则研究表明,在不同光下PHYD亚族基因转录和表达较稳定,但其对拟南芥幼苗的光形态建成的调控作用较微弱。上述研究结果表明,在自然环境条件下,许多植物的PHYD亚族基因并非必需。
3. 2 CsPHY基因编码蛋白的结构域及其表达模式差异分析
基因中不同外显子编码的结构域是蛋白质的结构和功能单元(Bork,1991)广泛参与植物基因转录、翻译调控和信号转导等生命过程(廉德君和许根俊,1997),而光对植物的生长发育及化学成分调节是通过特异基因的调控来实现(Quail,2002)。本研究发现,6个CsPHY蛋白中,除CsPHY6蛋白外,其他蛋白的N端均存在PAS-GAF-PHY 3个结构域组成的光敏核心区,该区段可结合吸收红光/远红光生色团的前体藻蓝胆素生色团,负责光感受(Kawai et al.,2003),但所有成员在C端均存在2个PAS和1个HisKA结构域,这些结构域可构成光信号转导体系负责光调节(Li et al.,2005)。但所有成员的C端PAS/PAC结构域在位置分布上存在差异,可能是PHY基因家族成员表达模式存在差异的原因。本研究发现,CsPHY6基因在R0~R6时段相对表达量无明显变化,而其他基因在该时段内均上调表达,其中,CsPHY4和CsPHY5基因均显著上调表达。此外,由于R6~R12时段的自然光从无到有,由弱变强,但CsPHY6基因表达显著下调,其原因可能是CsPHY6蛋白N端缺失PAS结构域,从而造成光感受障碍或延迟,致使CsPHY6基因的转录受到抑制,但需后续试验进一步证实。
3. 3 黄酮含量与CsPHY基因表达量的相关性分析
植物PHY基因作為重要的光信号受体蛋白编码基因,对于植物的生长发育、形态建成、次生代谢及抗逆性起重要作用(Baldwin and Ohnmeiss,1994;Aphalo and Lehto,1997;Deng et al.,2002;王静,2009)。朱新贵等(1999)研究发现,光照能明显促进玫瑰茄细胞黄酮含量的增加。本研究发现,茶树鲜叶的黄酮含量在R18时最高,说明自然光的减弱影响茶树叶片中黄酮类化合物的合成,而傍晚茶青黄酮含量达峰值,可能是生物合成代谢流累积的结果,在一定程度上或许可印证乌龙茶产区“早青不如午青,午青不如晚青”的说法。本研究相关性分析结果表明,黄酮含量与CsPHY6基因表达量呈显著正相关,其他5个基因的相对表达量与黄酮含量均无显著相关性,推测CsPHY6基因参与调控黄酮的合成,其原因是该基因编码蛋白CsPHY6属于PHYE亚族,在氨基酸数量、结构域及Motif数量和种类方面均与其他成员存在明显差异,说明其生物学功能也存在明显差异。何静雯等(2018a,2018b)研究发现,在近源物种葡萄中PHYE亚族基因表达量随胁迫程度的升高而降低,其与葡萄的抗逆性及生理机能存在密切联系,但该研究未提及与黄酮合成和代谢的关系。在今后的研究中,应深入研究茶树CsPHY6基因在光信号转导及黄酮代谢方面的调控机理。
4 结论
CsPHY基因家族中仅缺少PHYD亚族成员,尽管各成员间功能结构较相似,但表达模式存在明显差异。光照影响茶树叶片中黄酮含量,CsPHY6基因编码的光受体蛋白参与调控茶树叶片中的黄酮含量。
参考文献:
陈碧薇. 2014. 光敏色素下游转录因子基因沉默对番茄生长和营养品质及丁香假单胞菌Pst DC3000胁迫抗性的影响[D]. 杭州:浙江大学. [Chen B W. 2014. Effects of phytochrome downstream transcription factor gene silen-cing on the growth,nutritional quality and stress tolerance to Pst DC3000 in tomato[D]. Hangzhou:Zhejiang University.]
顾建伟,刘婧,薛彦久,臧新,谢先芝. 2011. 光敏色素在水稻生长发育中的作用[J]. 中国水稻科学,25(2):130-135. [Gu J W,Liu J,Xue Y J,Zang X,Xie X Z. 2011. Phytochrome functions in rice development[J]. Chinese Journal of Rice Science,25(2):130-135.]
何静雯,李晨,邱甜,吴燕燕,沈梓力,吴月燕. 2018a. 弱光胁迫下鄞红葡萄生理生化特性及相关基因的研究[J]. 华北农学报,33(6):123-129. [He J W,Li C,Qiu T,Wu Y Y,Shen Z L,Wu Y Y. 2018a. Effects of weak light stress on physiological and biochemical characteristics and relative gene expression of Yinhong grape[J]. Acta Agriculturae Boreali-Sinica,33(6):123-129.]
何静雯,赵晟,岳庆春,吴月燕. 2018b. 弱光胁迫下‘鄞红’葡萄光合特性及相关基因的表达[J]. 西南农业学报,31(12):2520-2526. [He J W,Zhao S,Yue Q C,Wu Y Y. 2018b. Effects of weak light stress on photosynthetic characteristics and relative fene expression of ‘Yinhong’ grape[J]. Southwest China Journal of Agricultural Scien-ces,31(12):2520-2526.]
李建平,杨建平,宋梅芳,苏亮,侯佩,郝晓燕,邵琳,陈果,足木热木,黄全生. 2014. 拟南芥光敏色素D(AtphyD)在不同光质下的表达特性分析[J]. 新疆农业科学,51(2):305-310. [Li J P,Yang J P,Song M F,Su L,Hou P,Hao X Y,Shao L,Chen G,Zumuremu,Huang Q S. 2014. Di-fferential patterns of expression of the Arabidopsis phytochrome D under different light conditions[J]. Xinjiang Agricultural Sciences,51(2):305-310.]
李娜,张晓燕,田纪元,贾礼,龚春燕,居鑫,刘自力,崔瑾. 2017. 蓝光连续光照对大豆芽苗菜类黄酮合成的影响[J]. 大豆科学,36(1):51-59. [Li N,Zhao X Y,Tian J Y,Jia L,Gong C Y,Ju X,Liu Z L,Cui J. 2017. Effect of blue light continuous illumination on flavonoid synthesis in soybean sprouts[J]. Soybean Science,36(1):51-59.]
廉德君,许根俊. 1997. 蛋白质结构与功能中的结构域[J]. 生物化学与生物物理进展,24(6):482-486. [Lian D J,Xu G J. 1997. Structure domains in protein structure and function[J]. Progress in Biochemistry and Biophysics,24(6):482-486.]
刘栓桃,张志刚,李巧云,王淑芬,赵智中,卢金东,张晓燕,徐文玲,刘贤娴,付卫民. 2014. 光敏色素B基因(PHYB)启动子突变及与大白菜开花时间的关联分析[J]. 农业生物技术学报,22(7):853-861. [Liu S T,Zhang Z G,Li Q Y,Wang S F,Zhao Z Z,Lu J D,Zhang X Y,Xu W L,Liu X X,Fu W M. 2014. Association analysis between phytochrome B gene(PHYB) promoter mutantions and flowering time in Chinese cabbage(Brassica rapa L. ssp. pekinensis)[J]. Journal of Agricultural Biotechnology,22(7):853-861.]
劉转霞,余运康,陈裕坤,冯新,刘炜婳,张梓浩,程春振,林玉玲,赖钟雄. 2017. 福州野生蕉隐花色素和光敏色素基因家族的克隆及其表达分析[J]. 热带作物学报,38(11):2089-2099. [Liu Z X,Yu Y K,Chen Y K,Feng X,Liu W H,Zhang Z H,Cheng C Z,Lin Y L,Lai Z X. 2017. Cloning and expression analysis of Cryptochrome and Phytochrome family genes in the wild banana(Musa spp.) from Fuzhou City[J]. Chinese Journal of Tropical Crops,38(11):2089-2099.]
羅丽,郭雅玲. 2004. 福建乌龙茶黄酮类化合物含量分析[J]. 中国茶叶加工,(3):32-34. [Lou L,Guo Y L. 2004. Analysis of flavonoids in Fujian Oolong tea[J]. China Tea Processing,(3):32-34.]
马晓净. 2018. 玉米PHYB1基因在拟南芥中的功能分析[D]. 北京:中国农业科学院. [Ma X J. 2018. Functional ana-lyses of maize PHYB1 in Arabidopsis thaliana[D]. Beijing:Chinese Aca-demy of Agricultural Sciences.]
牛骧,郭林,杨宗举,孙蕾,李红丹,游光霞,徐宏,孟凡华,佘跃辉,杨建平. 2017. 2个玉米光敏色素C基因的转录丰度对多种光质处理的响应[J]. 中国农业科学,50(12):2209-2219. [Niu X,Guo L,Yang Z J,Sun L,Li H D,You G X,Xu H,Meng F H,She Y H,Yang J P. 2017. Transcription abundances of two phytochrome C in response to different light treatment in Zea mays[J]. Scientia Agricultura Sinica,50(12):2209-2219.]
王静,王艇. 2007. 高等植物光敏色素的分子结构、生理功能和进化特征[J]. 植物学通报,24(5):649-658. [Wang J,Wang T. 2007. Molecular structure,physiological function and evolution of phytochrome in higher plant[J]. Chinese Bulletin of Botany,24(5):649-658.]
王静. 2009. 裸子植物PHY基因GAF和PHY-PAS1结构域的适应性进化研究[D]. 武汉:中国科学院武汉植物园. [Wang J. 2009. Adaptive evolution in the GAF and PHY-PAS1 domains of PHY gene in gymnosperms[D]. Wuhan:Wuhan Botanical Garden,Chinese Academy of Scien-ces.]
夏涛,高丽萍. 2009. 类黄酮及茶儿茶素生物合成途径及其调控研究进展[J]. 中国农业科学,42(8):2899-2908. [Xia T,Gao L P. 2009. Advances in biosynthesis pathways and regulation of flavonoids catechins[J]. Scientia Agricultura Sinica,42(8):2899-2908.]
徐茂军,朱睦元,顾青. 2003. 发芽大豆中异黄酮积累的光诱导作用研究[J]. 中国粮油学报,(1):44-47. [Xu M J,Zhu M Y,Gu Q. 2003. Light-induced effects of isoflavone accumulation in germinated soybean[J]. Journal of the Chinese Cereals and Oils Association,(1):44-47.]
张晓燕,鲁燕舞,魏圣军,崔瑾. 2014. 光质对大豆芽苗菜生长和大豆异黄酮含量及PAL活性的影响[J]. 大豆科学,33(1):46-52. [Zhang X Y,Lu Y W,Wei S J,Cui J. 2014. Effects of light quality on growth,soy isoflavone content and PAL activity of soybean sprouts[J]. Soybean Scien-ce,33(1):46-52.]
朱新贵,郭勇,林捷. 1999. 光质对玫瑰茄悬浮细胞合成花青素的影响[J]. 华南理工大学学报(自然科学版),(3):57-60. [Zhu X G,Guo Y,Lin J. 1999. Effects of monochromic light on synthesis of anthocyanin in roselle(Hibscus sabdariffa L.) cells suspension[J]. Journal of South China University of Technology(Natural Science),(3):57-60.]
Ahlgren P,Jarneving B,Rousseau R. 2003. Requirements for a cocitation similarity measure,with special reference to Pearson’s correlation coefficient[J]. Journal of the Association for Information Science & Technology,54(6):550-560.
Alba R,Kelmenson P M,Cordonnier-Pratt M M,Pratt L H. 2000. The phytochrome gene family in tomato and the rapid differential evolution of this family in angiosperms[J]. Molecular Biology and Evolution,17(3):362-373.
Aphalo P J,Lehto T. 1997. Effects of light quality on growth and N accumulation in birch seedlings[J]. Tree Physiology,17(2):125-132.
Aukerman M J,Hirschfeld M,Wester L,Weaver M,Clack T,Amasino R M,Sharrock R A. 1997. A deletion in the PHYD gene of the Arabidopsis Wassilewskija ecotype defines a role for phytochrome D in red/far-red light sen-sing[J]. The Plant Cell,9(8):1317-1326.
Baldwin I T,Ohnmeiss T E. 1994. Coordination of photosynthetic and alkaloidal responses to damage in uninducible and inducible Nicotiana sylvestris[J]. Ecology,75(4):1003-1014.
Bork P. 1991. Shuffled domains in extracellular proteins[J]. Febs Letters,286(1-2):47-54.
Bradley J M,Murphy G P,Whitelam G C,Harberd N P. 1996. Identification of phytochrome B amino acid residues mutated in three new phyB mutants of Arabidopsis thaliana[J]. Journal of Experimental Botany,47(302):1449-1455.
Clack T,Mathews S,Sharrock R A. 1994. The phytochrome apoprotein family in Arabidopsis is encoded by five genes:The sequences and expression of PHYD and PHYE[J]. Plant Molecular Biology,25(3):413-427.
Deng J M,Bin J H,Pan R C. 2002. Effect of light quality on the primary nitrogen assimination of rice(Oryza sativa L.) seedlings[J]. Acta Botanica Sinica,42(3):234-238.
Franklin K A,Quail P H. 2010. Phytochrome functions in Arabidopsis development[J]. Journal of Experimental Bo-tany,61(1):11-24.
Kawai H,Kanegae T,Christensen S,Kiyosue T,Sato Y,Imaizumi T,Kadota A,Wada M. 2003. Responses of ferns to red light are mediated by an unconventional photoreceptor[J]. Nature,421(6920):287-290.
Li F W,Melkonian M,Rothfels C J,Villarreal J C,Stevenson D W,Graham S W,Wong G K S,Pryer K M,Mathews S. 2005. Phytochrome diversity in green plants and the origin of canonical plant phytochromes[J]. Nature Communications,6:7852.
Mathews S,Lavin M,Sharrock R A. 1995. Evolution of the phytochrome gene family and its utility for phylogenetic analyses of angiosperms[J]. Annals of the Missouri Botanical Garden,82(2):296-321.
Quail P H. 2002. Phytochrome photosensory signalling networks[J]. Nature Reviews Molecular Cell Biology,3(2):85-93.
Rockwell N C,Lagarias J C. 2010. A brief history of phytochromes[J]. Chemphyschem:A European Journal of Che-mical Physics & Physical Chemistry,11(6):1172-1180.
Rockwell N C,Su Y S,Lagarias J C. 2006. Phytochrome structure and signaling mechanisms[J]. Annual Review of Plant Biology,57(1):837-858.
Sharrock R A,Quail P H. 1989. Novel phytochrome sequen-ces in Arabidopsis thaliana:Structure,evolution,and differential expression of a plant regulatory photoreceptor family[J]. Genes & Development,3(11):1745-1757.
Xia E H,Zhang H B,Sheng J,Li K,Zhang Q J,Changhoon Kim,Zhang Y,Liu Y,Zhu T,Li W,Huang H,Tong Y,Nan H,Shi C,Shi C,Jiang J J,Mao S Y,Jiao J Y,Zhang L P,Liu Y L,Liu B Y,Yu Y,Shao S F,Ni D J,Eichler E E,Gao L Z. 2017. The tea tree genome provides insights into tea flavor and independent evolution of ca-ffeine biosynthesis[J]. Molecular Plant,10(6):866-877.
Zhou Z W,Deng H L,Wu Q Y,Liu B B,Yue C,Deng T T,Lai Z X,Sun Y. 2019. Validation of reference genes for gene expression studies in post-harvest leaves of tea plant(Camellia sinensis)[J]. PeerJ:7:6385-4401.
(責任编辑 陈 燕)

