玉米根际土壤中大环内酯类抗性基因的分布特征
陈运杰,郭欣妍,杨 艺,王 娜①,杨 烨
(1.南京中医药大学医学与生命科学学院,江苏 南京 210023;2.生态环境部南京环境科学研究所,江苏 南京 210042)
抗生素作为畜禽感染性疾病治疗药物及动物生长促进添加剂,在养殖业中发挥着非常重要的作用[1]。然而,长期大量依赖抗生素的养殖方式,可诱导动物肠道菌群产生大量携带抗生素抗性基因(antibiotic resistance genes,ARGs)的耐药菌(antibiotics resistance bacteria,ARBs),且耐药菌会随排泄物最终进入环境[2-3];同时,抗生素药物母体和代谢产物随着粪便等排泄物直接进入土壤、水等环境介质中,又增加了环境微生物的进化选择压力,将诱导微生物产生耐药性[4]。
最近一些研究认为,植物根际是耐药基因转移的“热区”[5]。植物根际指受植物根系的影响,在物理、化学和生物学特性上不同于周围土体的根表面微域土区,是土壤水分、养分和有益或有害微生物进入植物根系的门户。在根际范围内,微生物种类丰富,能量和物质代谢活跃,根际生长和营养成分渗出会影响细胞密度、分布以及土壤细菌的代谢能力,一些抗生素的抗性效应在此区域内会进一步得到加强。REICHEL等[6]研究了施用含磺胺嘧啶(SDZ)和二氟沙星(DIF)的粪肥后玉米根际土壤与非根际土壤的微生物结构多样性,对63 d中宇宙试验的土壤样品磷脂脂肪酸(PLFA)进行分析发现,施用含SDZ和DIF的粪肥增加了土壤中革兰阴性菌和真菌数量,但是根际土壤微生物菌群的变化与非根际土壤相比并不十分明显;通过分析PCR-DGGE条带发现,条件致病菌假单胞菌属(Pseudomonas)在根际土壤中的增加更为显著。这表明植物根际环境可能促进土壤微生物群落产生耐药性,也可能促使粪肥中抗性基因在微生物间水平转移。因此,植物根际可能是影响ARGs增殖与传播行为的重要环境。但迄今为止,相关文献报道较少,研究角度比较单一,且所研究的抗生素品种主要集中于磺胺类抗生素,仅局限于施用含抗生素的粪肥后植物根际微生物多样性变化[6],以及植物根际部位磺胺类抗生素及其抗性基因(sul1、sul2和sul3)的表观分布规律[7-8]。
世界卫生组织(World Health Organization,WHO)报告指出,人畜共用抗生素的使用会大大增加动物与人体内抗性细菌交叉感染的风险,对人体健康造成极大危害[9]。大环内酯类抗生素(包括红霉素、螺旋霉素、乙酰螺旋霉素、交沙霉素和罗红霉素等)不仅在临床上作为青霉素过敏患者的替代药物而发挥着重要作用,对于养殖业也是广泛使用的一类兽药品种。目前,关于养殖业抗性基因的报道以四环素和磺胺类为主,但有关养殖行业使用频率同样较高的大环内酯类抗生素所导致的ARGs(主要为erm基因)传播扩散问题对人体健康的隐患更为严重。这是由于大环内酯类ARGs是一类较为稳定的抗性基因,在土壤中残留丰度相对更高。JOY等[10]在猪粪中检出金霉素、泰乐菌素及其对应的抗性基因tet和erm,在40 d的堆肥过程中,虽然泰乐菌素降解90%,但其对应的ermB基因却只降解40%。KNAPP等[11]对荷兰和苏格兰不同地区农田土壤中抗性基因进行定量分析,结果显示大环内酯类ARGs(ermB、ermC、ermE和ermF)丰度随着时间的推移而呈递增趋势。SU等[12]研究发现,广东东江流域水底沉积物中ermB、ermC和ermF这3种大环内酯类ARGs也存在较高丰度(105~1011copies·g-1)。可见大环内酯类抗性基因的环境潜在风险不容忽视。
笔者以污染隐患较大的大环内酯类抗性基因(erms)为研究对象,采用陆生微宇宙模拟试验方法,在人工控制条件下模拟目标污染物进入土壤中的分布行为。利用实时荧光定量PCR(real-time fluorescence quantitative PCR,qPCR)技术探明植物根际对erms在土壤中演变与归趋的影响规律,以及其在土壤剖面的垂直分布规律,为阐明erms在农田环境中传播扩散机制提供科学依据,也为建立抗性基因的生态环境安全评价和预警体系提供理论基础。
1 材料与方法
1.1 微宇宙试验方法的建立
1.1.1粪肥的收集
供试动物粪肥采集于江苏省农业科学院六合动物科学基地。选取1头育肥猪,首先在不饲喂任何抗生素药物情况下培育2个月,之后添加抗生素药物饲喂,方法为每天将1 g硫氰酸红霉素溶于8 L水中,令其自由饮用,连续饮用4 d,采集7 d内产生的猪粪便。粪便经短期堆肥腐化后收集并于-20 ℃条件下冷冻保存备用。
1.1.2土柱单元设置
陆生微宇宙系统的土芯取自生态环境部南京环境科学研究所实验用地,土壤为水稻土,其基本理化性质:pH为6.23,w(有机碳)为49.8 g·kg-1,离子交换量为18.0 cmol·kg-1,w(黏粒)为19.4%,w(粉粒)为75.8%,w(砂粒)为4.8%。土柱桶材质为聚乙烯,内径为17 cm,高度为60 cm(包括0.15 m厚种植层土和0.45 m厚深层土)。填装土柱前,用电子混匀机将粪肥和水稻土(以干重计)按照1∶25质量比充分混合,用水稻土填装深土层(0.45~0.6 m)后,再用粪肥-土壤混合物填装种植层(0~0.15 m)。土柱填装后先对土柱进行预淋溶,使土柱持水,之后按正常种植方式种植玉米并正常管理,每日适量补水保持土柱质量含水率为8.5%~15.2%。微宇宙系统的温度恒定设为21 ℃,采用卤钨灯(400 W)控制光照周期为t(光)∶t(暗)=16 h∶8 h[13]。以种植玉米的土柱作为试验组,以不种植玉米的土柱作为对照组(CK),其余处理方式与种植玉米的试验组完全相同。
1.2 样本采集
玉米种子购于南京润祥种业有限公司(品名:江南花糯,由江苏省农业科学院粮食作物研究所研制),每柱播种3粒种子,待预生长1周后,每柱仅保留1株生长良好的幼苗作为试验用玉米株,并以此作为玉米生长的第0天(D0)。之后分别于生长7、14、28、42、56和63 d(D7、D14、D28、D42、D56和D63)时采集土样,主要分为2个部分:(1)对试验组土柱中根际(rhizosphere,RH)和非根际(bulk soil,BK)土样进行采集。参照文献[7],采用抖动植物根际方式采集贴近根系0~4 mm的根际土壤约5 g,而非根际土壤则使用圆形取样器(高为9.2 cm,内径为1.2 cm)采集同一土柱中表层(0~0.1 m)土壤获得。(2)在每个采样时段利用特制的推土装置将试验组和对照组(CK)土芯整体推出后,同时取0~0.2、>0.2~0.4和>0.4~0.6 m这3段各自混为1个样品。所有样品均采集3个平行样,混合后置于无菌保鲜袋中,于4 ℃条件下无菌保存。
1.3 土壤总DNA(gDNA)的提取
土壤gDNA提取参照PowerSoil®DNA Isolation Kit试剂盒说明书操作,DNA洗脱液于-80 ℃条件下保存待用。采用w=1.0%的琼脂糖凝胶电泳和Nandrop1000对提取的gDNA质量和浓度进行检测。
1.4 实时荧光定量PCR(qPCR)
1.4.1PCR扩增抗性基因特异性片段
所涉及的目标基因主要包括4种大环内酯类抗性基因(ermB、ermC、ermF和ermX)和16S rDNA(作为分析时的内参基因),引物设计参照文献[14],具体信息见表1。以土壤gDNA为模板,PCR反应过程:94 ℃预变性300 s,然后进行30个循环,每个循环包括94 ℃变性30 s,适当退火温度(Tm)下退火30 s,72 ℃延伸45 s,最后72 ℃终延伸360 s结束反应,反应体系为12.5 μL 的2×ES Taq MasterMix,1 μL 上游引物(10 μmol),1 μL下游引物(10 μmol),1 μL模板DNA(<500 ng),9.5 μL的RNase-free水,反应体系总体积为25 μL。
表1 qPCR引物序列
Table 1 Primer sequence for qPCR

基因名称引物名称退火温度/℃产物大小/bpermBFW: ACCTTGGATATTCACCGAAC58119RV: CTTTGGCGTGTTTCATTGCTermCFW: ACTGCCATTGAAATAGACCA56292RV: TAGCAAACCCGTATTCCACermFFW: AAGAAATTGCCCGTTCGTT55274RV: ACTTGAACATTTCGGGCATCermXFW: AAATTTTCTCACCGACCACA62219RV: AGTACAGCGTCAGTCCA16SrDNAFW: CAATGGACGAAAGTCTGACG60146RV: ACGTAGTTAGCCGTGGCTTT
1.4.2目的基因的纯化与扩增
将含目的基因片段的PCR产物进行w=1.5%的琼脂糖凝胶电泳后,利用PCR纯化试剂盒(EasyPure Qucik Gel Extraction Kit)对目的基因条带进行纯化回收。将目的基因转染至Trans-T3感受态细胞进行克隆培养。最后,利用Axy Prep Plasmid Miniprep Kit提取质粒DNA,再由苏州金唯智生物科技有限公司测序以检测目的基因片段是否插入。
1.4.3基因拷贝数计算
利用Nanorop1000对所提取质粒纯度和DNA含量进行检测,若A260/A280介于1.8~2.0之间且A260/A230>2.0,则认为是纯DNA,否则需进一步纯化。参照文献[15],将质粒浓度换算为1 μL质粒溶液所携带的绝对模板拷贝数(y,copies·μL-1)公式为
y=[x/(a+b)×660]× 10-9× 6.02 × 1023。
(1)
式(1)中,x为质粒浓度,ng·μL-1;a为载体长度,bp;b为目的基因长度,bp。
1.4.4标准曲线的建立
将已知初始拷贝数的标准品质粒依次进行10倍梯度稀释,稀释浓度分别为10-2、10-3、10-4、10-5、10-6、10-7、10-8和10-9,再进行qPCR检测,反应结果经荧光定量软件分析得到对应循环数(Ct,Y),以其作为纵坐标,以拷贝数的lg值(X)作为横坐标,绘制标准曲线。4种大环内酯类抗性基因及16S rDNA标准曲线见表2。
在qPCR中,随着循环数的增加,荧光强度逐渐增强,在经过一段时间的指数扩增后,扩增曲线趋于稳定,出现“平台效应”,即随着模板扩增循环数的增加,由于其荧光强度和循环数不同,从而得到不同循环数与荧光强度之间的关系图,即扩增曲线。以ermB为例,其扩增曲线、标准曲线、溶解曲线和熔融峰见图1(a)~(d)。
表2 大环内酯类抗性基因及16S rDNA标准曲线
Table 2 Standard curve equation of macrolides antibiotic resistance genes and 16S rDNA
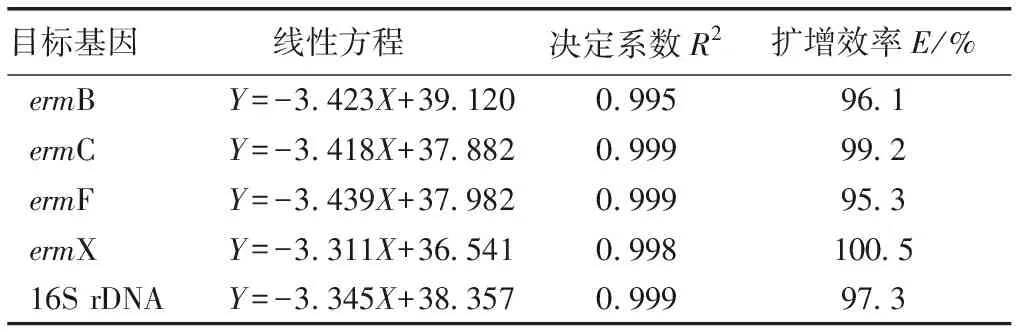
目标基因 线性方程 决定系数R2扩增效率E/%ermBY=-3.423X+39.1200.99596.1ermCY=-3.418X+37.8820.99999.2ermFY=-3.439X+37.9820.99995.3ermXY=-3.311X+36.5410.998100.516S rDNAY=-3.345X+38.3570.99997.3
X为目标基因拷贝数的lg值,Y为循环数。
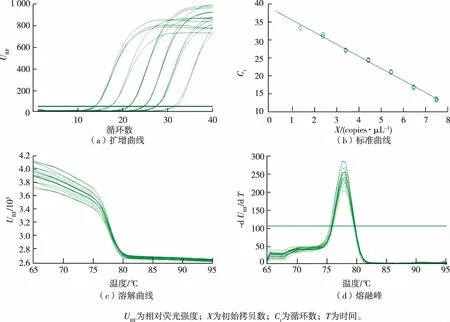
图1 ermB基因的扩增曲线、标准曲线、溶解曲线和熔融峰
1.5 抗性基因(ARGs)的定量分析
以16S rDNA为内参基因,使用qPCR技术对ermB、ermC、ermF和ermX 4种大环内酯类抗性基因进行定量分析,反应体系为5 μL 的2×UltraSYBR Mixture,0.2 μL上游引物(10 μmol),0.2 μL下游引物(10 μmol),1 μL模板DNA(10~100 ng),3.6 μL 的RNase-free 水,反应体系总体积为10 μL。
qPCR反应于CFX96 TouchTMReal-Time PCR Detextion System(Bio-Rad,USA)仪器上进行,采用三步法进行反应:95 ℃预变性10 min,然后进行40个循环,每个循环包括95 ℃变性15 s,适当退火温度(表1)下退火30 s,72 ℃延伸30 s。溶解曲线程序按照仪器默认值设置,从60 ℃上升到95 ℃,其间每隔0.5 ℃采集1次荧光以生成溶解曲线,根据溶解曲线变化检测扩增结果的特异性。
1.6 数据统计
所有试验数据均重复测定3次,以平均值±标准差形式表示,采用SPSS 22.0软件进行数据处理与分析,以P<0.05表示组间差异显著。大环内酯类抗性基因(erms)相对丰度为erms浓度与16S rDNA浓度的比值,即erms∶16S rDNA的计算值。采用OriginLab 8.0软件制图。
2 结果与分析
2.1 根际(RH)与非根际(BK)土壤中大环内酯类抗性基因的分布
根据标准曲线计算得到RH和BK土壤中大环内酯类抗性基因(erms)相对丰度。如图2所示,4种大环内酯类抗性基因均可检出,相对丰度范围大致为10-4~10-1。在检测的2种土壤中,ermF和ermX相对丰度较高,为10-3~10-1;其次为ermB,为10-3~10-2;而ermC相对丰度最低,为10-4~10-3。如表3所示,BK土壤中ermC、ermF和ermX在7 d时的相对丰度均显著高于RH土壤(P<0.05),而63 d时,除ermF基因外,RH和BK土壤中抗性基因相对丰度无显著差异(P>0.05)。
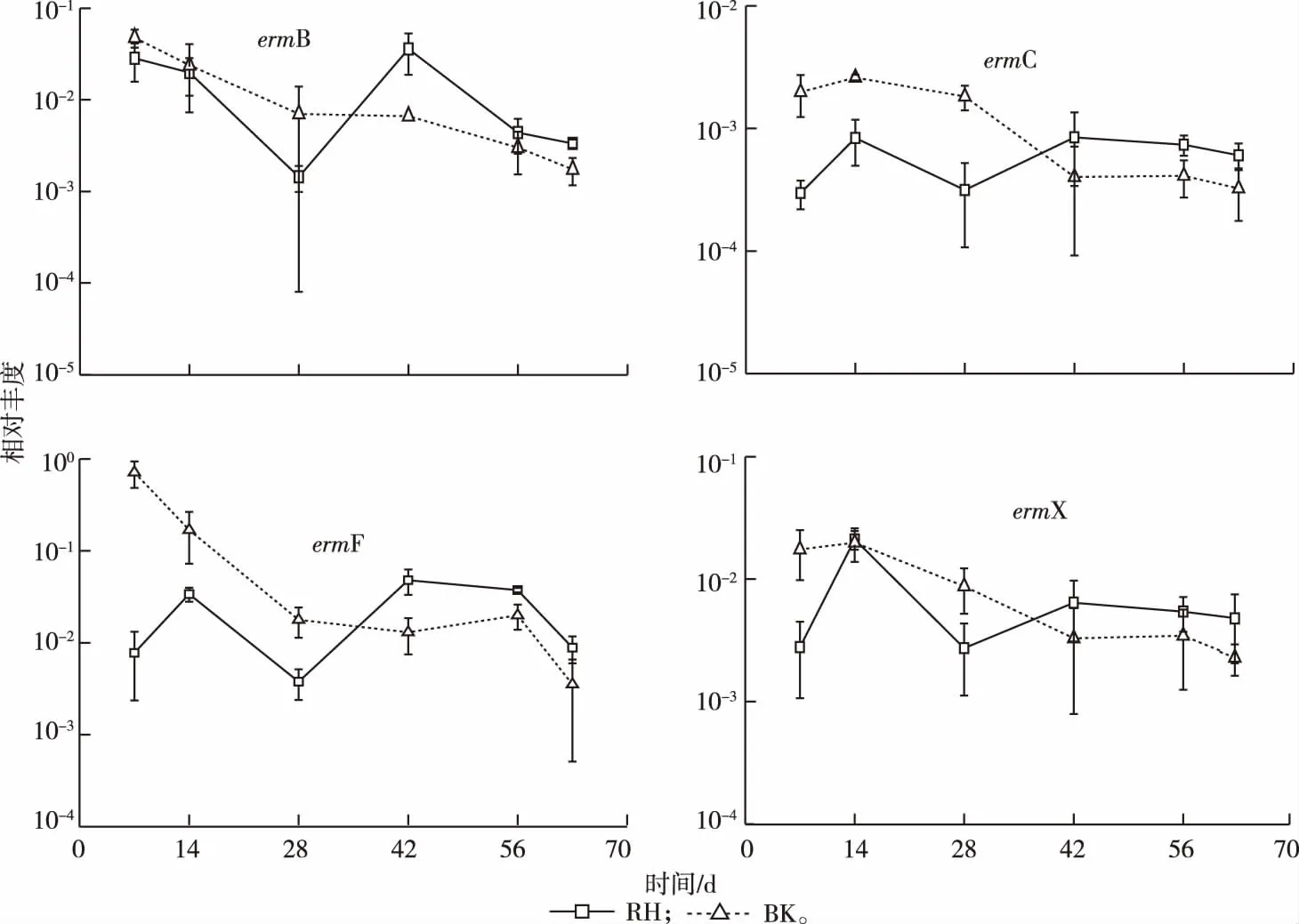
图2 根际(RH)和非根际(BK)土壤中大环内酯类抗性基因相对丰度
图2显示,在63 d的试验周期内,随着玉米生长时间的推移,BK土壤中erms相对丰度总体呈下降趋势,而RH土壤中4种抗性基因相对丰度变化均呈现波动状态,部分抗性基因在63 d时出现增殖。具体表现为7 d时,BK土壤中ermB、ermC、ermF和ermX相对丰度分别为4.72×10-2、1.98×10-3、7.13×10-1和1.75×10-2,而63 d时则分别为1.74×10-3、3.24×10-4、3.53×10-3和2.28×10-3,其增幅分别为-96.3%、-83.6%、-99.5%和-87.0%。类似地,RH土壤中4种抗性基因相对丰度增幅则分别为-88.3%、103.0%、-88.6%和71.5%。
28 d之前,土壤中抗性基因相对丰度表现为BK > RH,但28 d时,RH土壤中erms相对丰度开始呈现上升趋势,并在42 d后表现为RH > BK,且除ermB基因(42 d时)和ermF基因(42、63 d)外,其余差异性并不显著(表3)。
2.2 剖面土壤中大环内酯类抗性基因垂直分布
种植玉米(MA)和未种植玉米(CK)剖面土壤中抗性基因相对丰度见图3。在MA和CK土壤中,4种erms均能被检测到,且相对丰度随着玉米生长周期的增加呈下降趋势。与RH和BK土壤中抗性基因相对丰度的检测结果相似,MA和CK剖面土壤中ermF和ermX相对丰度较高,ermB和ermC相对丰度较低。MA土壤中ermB、ermC、ermF和ermX相对丰度分别为10-6~10-2、10-6~10-3、10-4~10-2和10-4~10-3,较CK土壤的10-7~10-3、10-6~10-4、10-5~10-2和10-5~10-3高1~2个数量级。总体而言,MA各土壤层中erms相对丰度高于CK土壤。
表3 根际和非根际土壤中抗性基因显著性差异P值分析
Table 3 Variance analysis of ARGs relative abundance in rhizosphere and bulk soil

抗性基因不同时间(d)P值71428425663ermB0.0670.9830.1300.0190.1280.368ermC0.0350.0730.0300.1580.0780.137ermF0.0280.0220.2860.0220.1920.027ermX0.0470.4610.3950.0820.0620.051
就0~0.2 m土层而言,无论是MA还是CK土壤,4种erms均可检出,其中ermF相对丰度最高,在10-1~10-2之间,ermC相对丰度最低,在10-4~10-3之间。就>0.2~0.4 m土层而言,MA土壤中除了ermB直到42 d时才被检测到以外,其余3种抗性基因在不同采样时间均可被检测到,且相对丰度呈现先增长后下降趋势;而就CK土壤而言,除了ermB在56 d之后才被检测到以外,其余3种抗性基因在不同采样时间均可被检测到;在同一采样时间,MA土壤中erms相对丰度高于CK土壤。就>0.4~0.6 m土层而言,28 d之前,MA土壤中4种erms均未检出,其中ermC和ermF在28 d之后才检出,ermB 在42 d之后才检出,ermX则在56 d之后检出;而对于CK土壤,除了ermC较早被检测到(28 d时)以外,ermB和ermF直到56和42 d之后才检出,ermX始终未检出。
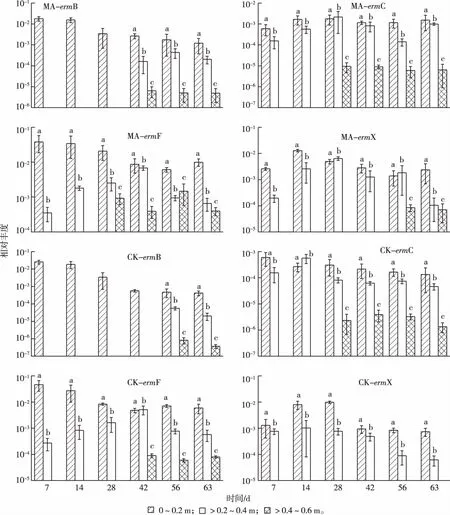
MA和CK分别指种植玉米和未种植玉米土壤不同深度土层中抗性基因相对丰度。就同一幅图而言,同一组直方柱上方
通过对剖面土壤中抗性基因垂直分布的研究发现,不同抗性基因在土层中的分布也有显著差异(图3),例如,MA土壤0~0.2 m土层中ermB基因(42 d时)相对丰度比>0.4~0.6 m土层高1 000倍,而ermC相对丰度则高100倍。随着剖面土壤深度的增加,抗性基因相对丰度逐渐下降,各土层抗性基因相对丰度由大到小依次为0~0.2、>0.2~0.4和>0.4~0.6 m。
3 讨论
笔者研究中,在63 d的采样周期内,非根际(BK)土壤中ermC、ermF和ermX相对丰度在7 d时均显著高于RH土壤(P< 0.05),这可能与非根际土壤主要取自含有粪肥的耕作层(0~0.1 m)有关。POPOWSKA等[16]从动物粪便堆肥样本和施用粪肥的农田及有机果园土壤样本中分离得到红霉素耐药菌,采用定性PCR技术检测到耐药菌携带有ermC、ermV、ermX、msrA、oleB和vg基因,可见,大环内酯类抗性基因通过粪便施肥会广泛进入环境并进一步传播扩散。随着时间的推移,BK土壤中erms相对丰度总体呈下降趋势,63 d时抗性基因相对丰度比7 d时增加-99.5%~-83.6%,这可能是因为在一段较长时间后,由于土壤中抗生素逐渐降解,在缺少外源压力的情况下,相应的抗性基因表现为相对稳定或持续性缓慢下降[17]。而在RH土壤中抗性基因相对丰度增幅则为-88.6%~103.0%,erms相对丰度总体变化趋势较BK更稳定,63 d时4种抗性基因相对丰度均超过BK土壤,暗示根际对erms丰度的维持具有一定作用,使得根际周围的土壤更有可能成为抗性基因的“蓄积池”。
在28 d之前,BK土壤中erms相对丰度均高于RH土壤,但28 d之后RH开始呈现上升趋势,在42 d之后出现逆转,表现为RH土壤中erms相对丰度高于BK土壤。RH土壤中erms的这种逆转情况可能与玉米生长过程中根际分泌物不同,进而引起根际周围携带有抗性基因的微生物群落变化有关[18]。根据玉米的生长周期,28 d之后,玉米正处于生长的关键期——穗期,此时根系继续扩展,受其分泌物的持续性影响,根际周围土壤养分更为丰富,微生物多样性和丰富度显著高于非根际[19]。研究显示,erm基因容易被质粒或转座子等水平基因移动原件(HGT)捕获,可以非常容易地在不同宿主细菌之间转移[12]。BRANDT等[20]研究发现,在添加人工根际渗出液作为碳源后,土壤中磺胺嘧啶(SDZ)的细菌群落抗性和相应的磺胺类抗性基因大大增加,这可能是因为 SDZ 耐药菌得到增殖。JECHALKE等[21]研究了植物根际对SDZ抗性转移的影响作用,发现玉米和青草的根际区域抗性质粒转移率明显增加,同时结果显示植物的不同品种及不同发育周期会对土壤微生物活性的促进作用以及抗生素抗性基因的转移作用产生影响。
通过对剖面土壤中抗性基因垂直分布的研究发现,4种erms在不同深度土层中的分布不一,且种植玉米(MA)土壤中抗性基因相对丰度显著高于未种植玉米(CK)土壤。随着纵向距离的加深,抗性基因检出频率和丰度也相应呈递减趋势。JOY等[10]研究发现,粪便耐药菌携带的sul1、sul2和ermF 基因容易随着雨水而渗透土层,径流至周边的地表水或逐渐渗透至地下水,说明抗生素抗性基因具有能够沿着土壤界面向下迁移的能力,继而可能会对地下水造成污染,具有潜在的环境风险。
随着土壤深度的增加,土壤养分和含氧量逐渐下降,从而使得土壤中微生物多样性呈现差异性分层,进而导致携带抗性基因微生物丰度随着土壤深度增加而减少[22]。RYSZ等[23]研究发现,大肠埃希菌(Escherichiacoli)在厌氧环境下易丢失抗性基因,含氧量高低将会影响微生物对抗性基因的保持能力。因此,土壤含氧量变化会对抗性基因丰度产生影响。此外,抗性基因在土壤中的传播还与可移动基因元件存在密切关系[12],因而呈现不同抗性基因在土壤垂直分层中的分布存在显著差异(P< 0.05)。MA土壤中4种erms相对丰度范围为 10-6~10-2,较CK土壤(10-7~10-2)高1~2个数量级,而在同一采样时间,CK深层(>0.2~0.4和>0.4~0.6 m)土壤中抗性基因的检出频率和丰度显著低于MA。研究结果提示植物根际对土壤中抗性基因的增殖及向下迁移具有一定作用。
4 结论
在玉米根际和非根际土壤中,4种大环内酯类抗性基因(erms)均被检出,且ermF相对丰度最高,ermC相对丰度最低。随着玉米生长时间的推移(7~63 d),BK土壤中erms相对丰度呈逐渐下降趋势,而RH土壤中erms相对丰度虽呈现一定波动,但最终趋于稳定,并高于BK土壤,提示根际对于土壤中抗性基因丰度的促进和维持具有明显作用。
通过对剖面土壤中抗性基因垂直分布的研究发现,不同抗性基因在土壤中的纵向分布差异显著,不同土层中4种erms相对丰度由大到小依次为0~0.2、>0.2~0.4和>0.4~0.6 m。种植玉米(MA)土壤中抗性基因相对丰度显著高于未种植玉米(CK)土壤,随着纵向距离的加深,抗性基因的检出频率和丰度也相应呈递减趋势。这表明植物根际的存在促进了土壤中抗性基因丰度的增加以及在剖面土壤中的纵向迁移。
总之,植物根际的存在对抗性基因在土壤中的增殖、扩散及纵向迁移具有一定推动作用,这种作用可能主要与根际分泌物对土壤中细菌群落的影响有关,因此进一步分析玉米生长过程中根际与非根际土壤中细菌群落的组成变化,将为揭示植物根际对土壤中抗性基因的影响提供一定理论基础。

