杜鹃花属(Rhododendron L.)植物 DNA 条形码鉴定
白霄霞 沈风娇 李志斌 赵建成 蒋淑磊 石硕

摘 要:杜鹃花属(Rhododendron L.)植物作为重要观赏和中药植物,市场前景广阔,发展潜力巨大。本文通过归纳杜鹃花属植物的分类、鉴定相关问题,发现利用DNA条形码可有效解决杜鹃花属植物(品种)的鉴定问题。基于杜鹃花属植物的具体情况,本文对序列参考数据库建设、分子标记开发以及杜鹃花品种的野生亲本溯源三方面内容进行综述,并结合GenBank中1 311条杜鹃花属DNA序列及作者测序获得的463条序列,初步建立杜鹃花属DNA条形码数据库,并通过非加权组平均法利用matK与psbA-trnH序列联合建立UPGMA树,部分杜鹃花属栽培品种可与野生种聚为一支。综合说明DNA条形码技术可用于对杜鹃花属栽培品种开展初步的野生亲本溯源。
关键词:杜鹃花; DNA条形码;中国珍稀濒危植物DNA条形码鉴定平台
中图分类号:S685.21; Q941+1 文献标识码:A DOI 编码:10.3969/j.issn.1006-6500.2019.10.001
Abstract: Genus Rhododendron is a group of important ornamental plants and traditional Chinese medicine resources with broad market prospects. In this paper, a DNA barcoding method for effectively solve species (cultivars) identification problems was proposed based on the review of taxonomy and identification history of genus Rhododendron. Three key topics related to DNA barcoding research on genus Rhododendron, which included reference library construction, molecular markers screening and optimization, and cultivars wild relatives tracing, were discussed. The DNA barcodes database of genus Rhododendron was preliminarily constructed with 1 311 DNA sequences in GenBank and 463 sequences generated by the authors. The UPGMA tree was constructed by matK and psbA-trnH sequences, and some cultivated varieties could be grouped with the wild species. The results suggested that DNA barcoding could be used to carry out preliminary wild relatives tracing of cultivated Rhododendron varieties.
Key words: Rhododendron L.; DNA barcoding; BREP
杜鹃花多指杜鹃花属(Rhododendron L.)植物——《中国植物志》[1]和《中国植物志》(英文版)[2]中记录本属中文名为“杜鹃属”,但由于杜鹃花的中文名来自杜鹃鸟[3]即动物界中的杜鹃属(Cuculus L., Cuculidae),为避免与动物学之“杜鹃”相混淆,此处参考耿玉英[3]的处理,将表示植物的“杜鹃”后面加一“花”字,用中文名“杜鹃花属”将植物杜鹃与动物杜鹃相区别。
杜鹃花属最初由林奈在1753年建立[4],其学名Rhododendron来源于希腊语,由rhodon(玫瑰)和dendron(树)组合而来,字面上的意思是“玫瑰树”[5]。杜鹃花属包含约1 000个野生种,虽然不同学者对于具体数字观点不一[1-3,6-8],但都承认它是种子植物中少见的大属。权威的植物学名数据库The Plant List统计了杜鹃花属下物种水平名称共计2 506个(包含杂交种),其中741个为接受名称,850个为异名,另有915个名称的地位待定(有待进一步分类研究)[7],可见野生杜鹃花属植物在物种水平的多样性非常丰富。杜鹃花的品种数目则更多,其国际品种登陆权威(the International Cultivar Registration Authority for the genus Rhododendron)收录了上万个杜鹃花品种[9]。相对如此丰富的物种和栽培品种,最初西方对杜鹃花的分类学研究和物种描述使用的材料却仅来自于部分模式标本和少量被引种到西方栽培的植株,因此当时对可利用的分类关键性状掌握并不完善;且由于杜鹃花在野外极易产生各种变异并伴有杂交,不同学者对于杜鹃花的分类存在较多争议[1-3,6,10]。面对物种数量如此之多,而且栽培品种的性状变异极其复杂的类群,仅依据形态性状进行物种和品种的分类尚存较多问题;而且形态学鉴定需要长期的研究基础和经验,面对植物分类鉴定专业人才缺失的现状,杜鹃花及其品種的鉴定问题将成为制约相关研究和产业发展的瓶颈。如今,在分类研究中注重居群内变异的观察和体现居群概念的标本馆标本研究[11]等方面工作,以进一步加深对杜鹃花属植物形态和变异的理解,这将有利于杜鹃花分类问题的更好解决。但更值得注意的是,引入分子生物学数据可以为杜鹃花分类研究提供分子层面数据,基于这些数据进行的分子系统学研究为杜鹃花分类研究开拓了新的局面[12-19]。在对杜鹃花属分类和分子系统学研究的基础上,杜鹃花分子鉴定应运而生[20-28],特别是利用分子标记鉴定物种的DNA条形码技术。DNA条形码技术也许可以为杜鹃花物种和品种鉴定难题提供一个可行的、稳定的、高效的解决方案。
DNA条形码(DNA barcoding)技术,或称条形码分子鉴定,是利用基因组中一段“短的”、“标准”的DNA片段来区分与鉴定生物物种的技术方法[29]。其名称来源于类似超市商品上的条形码——每一种商品的包装上都有代表这一商品信息的条形码,而这样的条形码在同种商品间是一致的,不同商品间却绝不相同,通过扫描条码并与数据库中对应的信息比对,即可以确定这个条形码对应的是哪种商品。所以DNA条形码鉴定的基本原理是:每个物种都有不同于其他物种的特有DNA序列;测定样本的这段特定DNA序列,并与相应的参考序列库(Library)进行比对,根据被测样品与参考序列库序列的相似性便可以将被测样本鉴定至不同物种。Ratnasingham等[30]研究表明,上述鉴定过程基本上是可行的。
作为21世纪新兴的技术之一,DNA条形码技术不仅对传统物种分类鉴定给予强有力的补充,而且它采用数字化形式,可以实现样本鉴定过程自动化和标准化,突破了对操作者经验的过度依赖,具有快速、高效、可靠、操作性强等特点,能够在较短时间内建立易于应用的物种鉴定系统。但是,DNA条形码技术尚存一些问题有待解决。第一,在DNA条形码鉴定时,需要在数据库中进行比较、搜索,而这个序列参考数据库必须在鉴定之前建设完备。只有参考序列库相对完善,其中囊括了相关的所有物种,并对于每一物种有很好的代表(且物种间的分子序列差异明显大于物种内差异),才有可能进行分子鉴定,所以建立完善的DNA形码参考序列库是影响DNA条形码应用的一个关键问题。第二,用以鉴定的分子标记(Marker),即基因组上特定位置DNA序列,是否具有变异性且易于扩增,直接决定着DNA条形码是否能够获取,影响着DNA条形码鉴定是否可以进行,所以分子标记的开发与优化是影响DNA条形码应用的另一个关键问题。第三,具体到众多栽培杜鹃花的鉴定研究,不可避免地涉及到栽培品种来源问题,特别是不同野生种通过杂交参与栽培品种形成。所以利用DNA条形码追溯杜鹃花品种的野生亲本与鉴定杜鹃花品种是不分割的一个问题的两个方面。综上所述,结合杜鹃花属植物具体情况和作者团队的工作,本文对杜鹃花属植物DNA条形码研究需要解决的序列参考数据库建设、分子标记开发以及对杜鹃花品种的野生亲本溯源三方面内容进行综述,旨在为杜鹃花资源的利用、育种与开发提供参考。
1 参考序列库(Reference library)
1.1 参考序列库的定义
参考序列库是对有代表性的、已被准确鉴定的不同物种,以及同一物种的不同个体的DNA条形码获取和汇总,序列库中某一物种的数据是代表这一物种的标准DNA条形码合集。这样的参考序列库保存了某一类生物具有代表性的条形码序列集,可以用于鉴定这一类生物的不同物种。
1.2 建设参考序列库要解决的关键问题
要保证DNA条形码鉴定的可操作性,必须有完善的参考序列库,而参考序列库的标本都应当来自于被准确鉴定的物种(品种),只有来自准确鉴定标本的参考序列库条形码才是可以用于分子鉴定的“合格”条形码,所以对序列库收录杜鹃花物种和品种的准确鉴定是建设参考序列库的一个关键问题。此外,还要考虑用于建设DNA条形码库的样品代表性,即某杜鹃花品种的参考序列来源植物应具代表性,需要采集这一品种一定数量的不同(分布区)个体,这是建设参考序列库的另一关键问题。
1.3 已有的研究基礎
对于杜鹃花的研究来说,如今可以直接使用的参考序列库数据主要来自以下网上平台:GenBank是由美国国家生物技术信息中心(National Center for Biotechnology Information,NCBI)组织建立的DNA(及蛋白质)序列数据库,其开放数据可供各国科学家免费下载使用。截止到2017年11月15日,GenBank数据库[31]共有杜鹃花属植物9 298条核酸序列,已经覆盖了567个物种。BOLD是专业的DNA条形码数据网站(Barcoding of Life Data Systems),其中整理和保存了杜鹃花属植物247个种的条形码数据,可供DNA条形码鉴定参考。我国的相关研究起步较晚,以科技部基础性工作专项“重要生物DNA条形码的技术规范体系与信息系统构建”为基础构建的我国珍稀濒危植物DNA条形码鉴定平台BREP (Barcode of Rare and Endangered Plant,http://www.brep.ac.cn),提供了准确鉴定我国珍稀濒危植物及其相关类群的DNA条形码参考序列[32]。虽然上述数据库可初步作为杜鹃花植物DNA条形码的参考序列库,但其包含物种数距离杜鹃花属上千个物种的数目尚有一定距离。同时,GenBank数据库的序列提交几乎是完全开放的,数据的可靠性不能够完全保证,如果缺乏分类学者对所研究标本的鉴定和把关,上传的序列可能是形态性状相似的近缘种的错误鉴定,如果错误鉴定的物种收录进参考序列库,将对后期的应用鉴定造成致命影响;BREP包含的数据相对较少,且多针对珍稀濒危植物进行研究;BOLD的杜鹃花数据库中多数种只有一两条参考序列,这距离同一种要有多数代表样品的要求来说还差得远[33]。因此,为了达到较好的分子鉴定效果,还需要加大力度建设杜鹃花的参考序列库:一方面,在生产新的DNA条形码数据时,要尽量吸收专业分类鉴定学者参与,确保所使用的标本被尽量准确鉴定,同时,对开放平台上的序列数据谨慎使用,以宁可不用、不可错用的原则,利用分子数据分析手段排除可能的错误鉴定样品;另一方面,新建设的序列库需要覆盖到尽量多的野生种和栽培品种,并力求同一种或品种都有不同遗传背景代表的一定数目的个体。
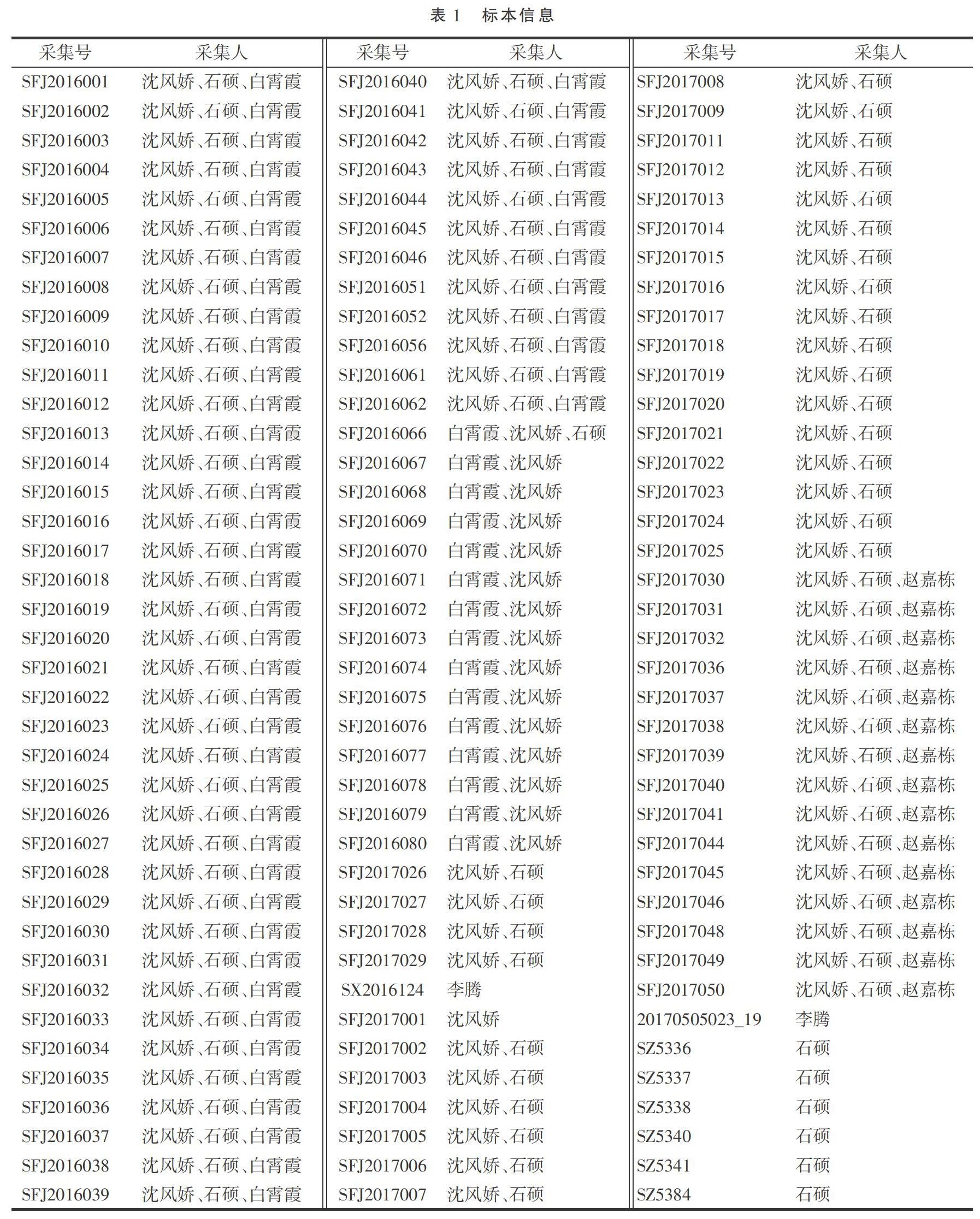
本研究对GenBank等数据库上相关序列进行了下载、分析、优化与整合,初步建立可用于鉴定杜鹃花的分子参考序列库。同时,依托现有的“高山杜鹃”及其他杜鹃花品种收集,对62个品种(个体)116号标本进行DNA条形码研究和品种序列基础库建设,对不同样品的rbcL、matK、trnH-psbA和ITS共4个序列完成了扩增和测序,已经初步建立本地的杜鹃花品种分子鉴定参考序列库(表1)。今后我们计划与各标本馆合作,利用标本馆已经相对准确鉴定过的植物标本的分子材料,进行数据生产。在收集较丰富的标本样品后,可利用二代测序技术批量获取可用的参考序列,为建设杜鹃花植物DNA条形码鉴定参考序列库提供第一手数据。
2 分子標记
2.1 DNA条形码研究中的分子标记
在DNA条形码研究中,分子标记特指基因组特定区域的用以区分物种的DNA序列。DNA条形码概念提出之初,希望能用一个短的分子标记来区分来自不同类群的生物,特别是区别不同物种[29],但实践证明,这样的条形码并不容易找到。如今在动物中一般使用COI基因,在植物中通常使用rbcL和matK(以及ITS)作为通用的分子标记[34-35],BOLD选择的分子标记就是这样。但对于不同具体类群,如杜鹃花科,或杜鹃花属植物,由于不同类群的特异性,适于其他植物的通用分子标记并不一定好用,这体现在分子标记引物的扩增效果和分子标记的分辨率等方面。
2.2 分子标记对于DNA条形码研究的影响
分子标记需要有一定的变异性,从而可以区别不同的物种;还需要有相对的稳定性,稳定性一方面体现在物种内部的遗传距离要小于物种之间的遗传距离,一方面体现在其片段两端需要有保守的区域以便于设计引物。此外,引物扩增的效率、测序的成功率,都是决定一个分子标记是否可用于DNA条形码鉴定的关键因素。当然,对于当前的应用而言,某一分子标记的参考序列库是否完善也将直接影响到这一条码在当下的可用性。
3 栽培品种野生亲本溯源
栽培品种由于来源复杂,变异性强,老品种亲本来源背景可能并不清晰,而新品种可通过杂交、选育的方式快速形成,加之栽培上对使用的品种来源、栽培历史往往未能准确记录(甚至许多品种的名称都未能规范、统一,国内有不少栽培杜鹃花培育的单位,但很少有在品种登陆权威申请登记品种名称的),故品种间遗传背景关系较难明确。而品种间自然分类关系不明确将影响对品种的进一步选育、繁殖等开发、研究。DNA条形码技术可以有效地运用于当下数以万计的杜鹃花品种遗传背景的分析,或者更准确地说,对其野生亲本溯源。通过遗传信息推测其母父本可能是哪一个野生物种,或者可能是哪几个野生物种之一,这将是分子生物学运用于杜鹃花品种分类研究的利器。本文以分辨率较高的matK与psbA-trnH序列为例,建立UPGMA树,试图对栽培品种野生亲本进行初步溯源,并将其结果进行展示(图1)。结果显示,利用matK与psbA-trnH二基因序列联合建立UPGMA树时,部分杜鹃花属栽培品种可与野生种聚为一支,这说明可以利用DNA条形码技术对栽培品种展开初步的野生亲本溯源工作。
当然,具体到DNA条形码鉴定品种问题,尤其是新品种的分子鉴定,从技术上看,最为重要的还是参考序列库的完善,所以这一问题的解决同样需要进一步加强参考序列库的建设工作。出于对上述问题的考虑,或许可以在合适时候建议新品种的审定(甚至是品种登陆权威的登记)必须提交DNA条形码序列作为特征参考指标,类似的规范和实践已经在菌物分类学研究[36-37]和中药DNA条形码研究[38-39]中得到了应用并日益完善。
4 展 望
本文通过对杜鹃花属植物DNA条形码鉴定相关的原理、技术和应用的简要介绍,以及对存在问题和需要克服困难的分析,建议杜鹃花研究者将野生种的系统分类研究与整合二代测序技术进行合理结合,建设完善的条形码参考序列库,并结合基因组学研究更深入地筛选分子标记,优化实验条件,以更好地将DNA条形码研究和鉴定技术运用于杜鹃花品种鉴定和相关研究中。
参考文献:
[1]FANG M Y, FANG R Z, HE M Y, et al. Flora of China[M]. Vol 14. Beijing: Science press; St. Louis: Missouri botanical garden press,2005.
[2]方瑞征,杨汉碧.中国植物志(第五十七卷,第一分册,杜鹃花科)[M].北京:科学出版社,1999.
[3]耿玉英.中国杜鹃花属植物[M].上海:上海科学技术出版社,2014.
[4]LINNAEUS C. Species plantarum[M]. Sweden: Stockholm,1753.
[5]HARPER, DOUGLA S. Rhododendron [DB/OL].[ 2017-01-23].http://www.etymonline.com/index.php?allowed_in_frame=0&search=rhododendron.
[6]CHAMBERLAIN D, HYAM R, ARGENT G, et al. The genus Rhododendron: its classification and synonymy[M]. Edinburgh: Royal botanic garden edinburgh, 1996.
[7]ANON. THE PLANT LIST[DB/OL].[2017-11-21].http://www.theplantlist.org.
[8]ANON. Rhododendron[DB/OL].[2017-11-21].https://en.wikipedia.org/wiki/Rhododendron.
[9]THE ROYAL HORTICULTURAL SOCIETY. RHS the international cultivar registration authority for the genus Rhododendron[DB/OL].[2017-11-21].https://www.rhs.org.uk/plants/plantsmanship/plant-registration/Rhododendron-cultivar-registration/Rhododendron.
[10]SLEUMER H. Einsystem der gattung Rhododendron L.[J]. Botanische jahrbuch systematic, 1949, 74:511-553.
[11]洪德元.关于提高物种划分合理性的意见[J].生物多样性,2016,24(3):360-361.
[12]KRON K. Phylogenetic relationships of Rhododendroideae (Ericaceae)[J]. American journal of botany, 1997, 84(7):973-980.
[13]SCHEIBER S M, JARRET R L, ROBACKER C D, et al. Genetic relationships within Rhododendron L. section Pentanthera G. Don based on sequences of the internal transcribed spacer (ITS) region[J]. Scientia horticulturae, 2000, 85(1):123-135.
[14]KURASHIGE Y, ETOH J I, HANDA T, et al. Sectional relationships in the genus Rhododendron (Ericaceae): evidence from matK and trnK intron sequences[J]. Plant systematics and evolution, 2001, 228(1):1-14.
[15]高连明,杨俊波,张长芹,等.ITS序列分析探讨杜鹃属映山红亚属的组间关系[J].云南植物研究,2002,24(3):313-320.
[16]GAO L M, LI D Z, ZHANG C Q, et al. Infrageneric and sectional relationships in the genus Rhododendron(Ericaceae) inferred from ITS sequence data[J]. Acta botanica sinica, 2002, 44(11):1351-1356.
[17]MILNE R. Phylogeny and biogeography of Rhododendron subsection Pontica, a group with a tertiary relict distribution[J]. Molecular phylogenetics and evolution, 2004, 33(2):389-401.
[18]GOETSCH L, ECKERT A J, HALL B D. The molecular systematics of Rhododendron (Ericaceae): a phylogeny based upon RPB2 gene sequences[J]. Systematic botany, 2005, 30(3):616-626.
[19]YAN LJ, LIU J, MOLLER M, et al. DNA barcoding of Rhododendron (Ericaceae), the largest Chinese plant genus in biodiversity hotspots of the Himalaya-Hengduan Mountains[J]. Molecular ecology resources, 2015, 15(4):932-944.
[20]KOBAYASHI N, TAKEUCHI R, HANDA T, et al. Cultivar identification of evergreen azalea with RAPD method[J]. Journal of the Japanese society for horticultural science, 1995, 64:611-616.
[21]劉义梅.杜鹃花属药用植物DNA条形码的鉴定研究[D].武汉:湖北中医药大学,2011.
[22]周泓.杜鹃花品种资源多样性研究及品种分类体系构建[D].杭州:浙江大学,2012.
[23]GAO L M, LIU J, CAI J, et al. A synopsis of technical notes on the standards for plant DNA barcoding[J]. Plant diversity and resources, 2012, 34(6):592-606.
[24]LIU Y F, XING M, ZHAO W, et al. Genetic diversity analysis of Rhododendron aureum Georgi (Ericaceae) located on Changbai Mountain using ISSR and RAPD markers[J]. Plant systematics and evolution, 2012, 298(5):921-930.
[25]陈建梅,王钊宇,张立娜,等.基于ITS条形码技术分析北京云蒙山杜鹃花属遗传多样性[J].中国农学通报,2014,30(25):43-48.
[26]肖政,苏家乐,刘晓青,等.32个杜鹃花品种遗传关系的SRAP分析[J].西北林学院学报,2016,31(4):123-128.
[27]沈德周,纵丹,周安佩,等.AFLP引物组合数对准确分析马缨杜鹃遗传多样性的影响[J].西南林业大学学报,2016,36(6):22-28.
[28]廖菊阳,吴林世,刘艳,等.28种杜鹃亲缘关系ISSR分析[J].经济林研究,2018,36(2):133-138.
[29]HEBERT P D N, CYWINSKA A, BALL S L, et al. Biological identifications through DNA barcodes[J]. Proceedings of the royal society b: biological sciences, 2003, 270(1512):313-321.
[30]RATNASINGHAM S, HEBERT P D N. Bold: the barcode of life data system (www.barcodinglife.org)[J]. Molecular ecology notes, 2007, 7(3):355-364.
[31]GEER L Y, MARCHLER-BAUER A, GEER R C, et al. The NCBI biosystems database[J]. Nucleic axids research, 2010, 38:492-496.
[32]李長昊,程涛,董文攀,等. “中国珍稀濒危植物DNA条形码鉴定平台”简介[J].生物多样性,2014,22(1):110.
[33]CUENOUD P, SAVOLAINEN V, CHATROU L W, et al. Molecular phylogenetics of Caryophyllales based on nuclear 18S rDNA and plastid rbcL, atpB, and matK DNA sequences[J]. American journal of botany, 2002, 89: 132-144.
[34]YU J, XUE J H, ZHOU S L. New universal matK primers for DNA barcoding angiosperms[J]. Journal of systematics evolution, 2011, 49(3): 176-181.
[35]DONG W P, CHENG T, LI C H, et al. Discriminating plants using the DNA barcode rbcL b: an appraisal based on a large data set[J]. Molecular ecology resources, 2014, 14(2): 336-343.
[36]VINCENT R, DUONG V, AMOR A B H, et al. MycoBank gearing up for new horizons[J]. Ima fungus, 2013, 4(2):371-379.
[37]HAWKSWORTH D L, CROUS P W, Redhead S A, et al. (308-310) Proposals to permit DNA sequence data to serve as types of names of fungi[J]. Taxon, 2016, 65(4):899-900.
[38]CHEN S L, YAO H, HAN J P, et al. Validation of the ITS2 region as a novel DNA barcode for identifying medicinal plant species[J]. PLoS one, DOI: 10.1371/journal.Pone.0008613,2010.
[39]陈士林.中药DNA条形码:分子鉴定[M].北京:人民卫生出版社,2012.

