鸡G蛋白α亚基基因可变剪接体的克隆和组织表达
章学东,王欢欢,葛莹,宋丹丹,张雷,李庆海,楼立峰
(杭州市农业科学研究院,杭州310024)
G 蛋白α 亚基(G-protein alpha subunit,GNAS)处于细胞膜内侧,它是跨膜G蛋白偶联受体信号转导通路的关键元件,参与很多神经递质、自分泌/旁分泌因子、激素相关的生命活动,例如5-羟色胺和多巴胺传递、胰岛素分泌、黑色素合成等[1-3]。在人和小鼠的研究中发现,GNAS基因通常具有1~12或13 个外显子,但呈现比较复杂的父源、母源或双性印记表达模式,这与该基因存在多个可变启动子和甲基化区域有关[4]。因此,GNAS基因在转录过程中会出现不同的剪接形式,可翻译为多种蛋白,如Gαs、XLαs、Nesp55(或Nespas)、A/B(ex 1A)和AS(Alex)等[5-6]。其中:Gαs 在绝大多数组织中呈双性表达,是最典型的GNAS 蛋白;而XLαs 则表现为父源等位基因转录,它与Gαs的区别在于第1外显子,XLαs的第1外显子较大且远在Gαs第1外显子的前方[7-8]。最新NCBI 数据库检索表明,鸡GNAS 基因位于第20 号染色体(NCBI 参考序列号:NC_006107.5,https://www.ncbi.nlm.nih.gov/nuccore/NC_006107.5)上,全长为130 648 bp(前一版NCBI 参考序列号NC_006107.4 中为48 456 bp,https://www.ncbi.nlm.nih.gov/nuccore/NC_006107.4),预测其存在X1~X4等4 种转录剪接体,但尚无这方面的实例报道。我们前期通过聚合酶链式反应-限制性片段长度多态性(polymerase chain reaction of restriction fragment length polymorphism, PCR-RFLP)和DNA测序发现,鸡GNAS 基因的突变与鸡肤色性状显著相关[9],但该基因的转录状态并不明确;因此,本研究旨在进一步通过5´和3´cDNA 末端快速扩增(rapid-amplification cDNA ends, RACE)技术对鸡GNAS 基因进行克隆测序,探寻该基因的不同剪接形式及其转录剪接体的组织表达情况。
1 材料与方法
1.1 试验材料
1.1.1 试验鸡
试验鸡为来自杭州市农业科学研究院培育的90日龄HW1系乌骨鸡。该品系鸡的每个世代采用群体制种方法进行保存,羽毛均为黑色片羽,皮肤以黑色为主,少部分肤色为白色。育雏育成期采用地面垫料平养;120 日龄左右上笼饲养。按种鸡免疫程序进行免疫。
1.1.2 主要试剂
MiniBEST Universal RNA 提取试剂盒(TaKaRa公司,美国);SMARTer RACE 5´/3´试剂盒(Clontech公司,美国);琼脂糖、无水乙醇、乙二胺四乙酸二钠盐(EDTA-2Na·2H2O)、NaCl、Tris-HCl、硼酸、冰乙酸等试剂均为进口或国产分析纯;异丙基-β-D-硫代半乳糖苷(IPTG)、5-溴-4-氯-3-吲哚半乳糖苷(X-gal)、氨苄青霉素[天根生化科技(北京)有限公司];质粒小提试剂盒(Qiagen公司,美国)。
1.1.3 仪器设备
Opticon®2 实时荧光定量PCR 仪、电泳仪、电泳槽(BIO-RAD公司,美国),5417R高速冷冻离心机、微量移液器、PCR仪(Eppendorf公司,德国),液氮罐、CO2恒温培养箱(Thermo公司,美国),凝胶成像系统(Alpha Innotech 公司,美国),超纯水仪(PALL 公司,美国),电子天平(Mettler Toledo公司,瑞士),超净工作台(江苏省苏州净化设备有限公司),微型台式离心机(江苏省海门市其林贝尔仪器制造有限公司),YKKY雪花制冰机(北京长流科学仪器有限公司)。
1.2 方法步骤
1.2.1 组织样本采集
屠宰采集8只90日龄试验鸡的多种组织样本,包括胸侧皮肤、胸肌、腹脂、脑、肺、心和肝等,于液氮中保存。
1.2.2 总RNA 提取
根据TaKaRa 公司总RNA 提取试剂盒(产品编号:9767)的使用说明,分别提取各组织样本的总RNA,于-20 ℃保存,备用。
1.2.3 5´和3´RACE 实验
1.2.3.1 引物设计
根据Ensemble 数据库(http://asia.ensembl.org/Gallus_gallus/Gene/Sequence?db=core;g=ENSGALG 00000030859;r=20:10918207-11045636)中的鸡GNAS基因序列,设计5´和3´RACE引物(表1)。

表1 GNAS基因的RACE引物信息Table 1 RACE primer information of GNAS gene
1.2.3.2 cDNA合成
取1 μL RNA 与1 μL 5´-CDS 引物A 和9 μL ddH2O(5´ RACE)或1 μL RNA 与10 μL ddH2O(3´RACE)混合离心后,放入PCR仪中,于72 ℃条件下孵育3 min,42 ℃条件下冷却2 min。然后加入1 μL SMARTer ⅡA 寡核苷酸、4 μL 5×第1链缓冲液、0.5 μL 二硫苏糖醇(DTT)(100 μmol/L)、1 μL dNTPs(20 mmol/L)、0.5 μL RNase 抑制剂(40 U/µL)、2 μL SMARTScribeTM反转录酶(100 U),混匀,离心,在42 ℃条件下孵育90 min,70 ℃条件下加热10 min,终止反应。
1.2.3.3 巢式PCR
将上述cDNA 溶液加入到90 μL 乙二胺四乙酸(EDTA)中进行稀释后,开始第1 轮PCR 反应;将第1 轮产物稀释50 倍后,进行第2 轮PCR 反应。反应体系(50 μL)如下。第1 轮:cDNA 稀释液2.5 μL,2×SeqAmpTM缓冲液25 μL,SeqAmpTMDNA 聚合酶1 μL,10×通用长引物(UPM long)5 μL,10 μmol/L基因特异性引物(5´GSP-1 或3´GSP-1)1 μL,ddH2O 15.5 μL。第2 轮:产物稀释液5 μL,2×SeqAmpTM缓冲液25 μL,SeqAmpTMDNA聚合酶1 μL,10×通用短引物(UPM short)1 μL,10 μmol/L 基因特异性引物(5´GSP-2 或3´GSP-2)1 μL,ddH2O 17 μL。反应程序如下。第1 轮:94 ℃变性30 s,72 ℃退火、延伸3 min,5 个循环;接着94 ℃变性30 s,70 ℃退火30 s,72 ℃延伸3 min,5 个循环;然后94 ℃变性30 s,68 ℃退火30 s,72 ℃延伸3 min,25 个循环。第2 轮:94 ℃变性30 s,68 ℃退火30 s,72 ℃延伸3 min,20个循环。
1.2.4 克隆测序
将巢式PCR 产物割胶、回收、纯化,连接到pRACE 载 体 上,取2.5 μL 反 应 液 加 入 到50 μL Stellar感受态细胞中,轻弹管壁混匀;取3 μL转化液加入到降解物阻遏的超级优化肉汤(super optimal broth with catabolite repression, SOC)培养基上,补齐至100 μL;然后将其涂布于预热的溶菌肉汤(lysogeny broth, LB)培养板(含适量氨苄青霉素)上,37 ℃培养过夜。随机挑取10个阳性菌落,摇菌扩繁,参照Qiagen 质粒小提试剂盒使用说明,抽提质粒后送杭州擎科生物技术有限公司测序;拼接得到转录剪接体全长序列。
1.2.5 生物信息学分析
运用DNAMAN 6.0 软件(Lynnon Biosoft 公司,美国)搜索克隆得到的鸡GNAS 基因转录剪接体全长序列的开放阅读框,定义编码序列(coding sequence,CDS)位置,然后进行氨基酸的编码翻译。
选择人(Homo sapiens)、小鼠(Mus musculus)2个物种,运用NCBI 数据库中的BLASTP 程序(https://blast.ncbi.nlm.nih.gov/Blast.cgi?PROGRAM=blastp&PAGE_TYPE=BlastSearch&LINK_LOC=blasthome),进行转录剪接体编码蛋白序列的同源性比对,建树方法为快速最小进化(fast minimum evolution)法,最大序列差比(max seq difference)为0.85。
运用NCBI 数据库中的COBALT 程序(https://www.ncbi.nlm.nih.gov/tools/cobalt/),进行2 种转录剪接体的编码蛋白与4条预测的鸡GNAS基因蛋白序列的同源性比对。
1.2.6 表达量检测
根据测序拼接得到的转录剪接体全长序列,设计目的基因的mRNA引物(表2),内参基因为RPL4(NCBI参考序列号:NM_001007479.1)。
定 量PCR 反 应 体 系:cDNA 1 μL,SYBR®Premix Ex Taq Mix(2×)10 μL,上、下游引物各0.5 μL,加水补至20 μL。反应程序:95 ℃预变性1 min;95 ℃变性15 s,60 ℃退火、延伸40 s,40个循环。绘制55~95 ℃范围内的熔解曲线,每0.2 ℃读板一次。每个样本进行3次重复,记录CT值,用2-△△CT法计算目的基因的相对表达量。
1.2.7 统计分析
运用GraphPad Prism 6.0 软件对鸡GNAS 基因转录剪接体的各组织表达量进行单因素方差分析并制图,P<0.05 表示差异有统计学意义,P<0.01表示差异有高度统计学意义。
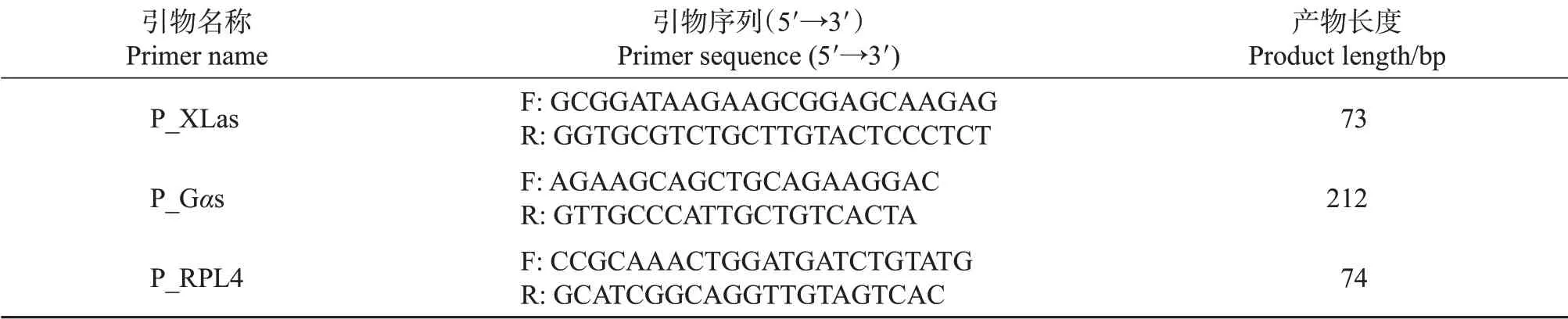
表2 表达量检测所用引物信息Table 2 Primer information of gene expression detection
2 结果与分析
2.1 RACE 和克隆结果
运用5´和3´RACE法对鸡GNAS基因进行巢式PCR 扩增,得到的琼脂糖凝胶电泳条带见图1。从中可见,5´和3´RACE均出现特异性条带,大小约为1 000 bp。测序发现存在2种克隆子,拼接得到的这2 种克隆子序列长度分别为1 554 bp 和1 796 bp。经比对,克隆子中各个外显子片段长度及所在基因组位置见表3。克隆子1与克隆子2均含有12个外显子,二者仅第1外显子的长度和位置不一样,前者在后者上游,相距46 897 bp;其余11个外显子相同。
2.2 转录剪接体全长序列分析
克隆得到的鸡GNAS 基因的2 种转录剪接体的全长序列及编码情况见图2。从中可知:克隆子1全长1 554 bp,CDS 区从5´端的第55 个碱基开始,至1 308 bp 处结束,共计编码417 个氨基酸;克隆子2全长1 796 bp,CDS 区从5´端的第411 个碱基开始,至1 550 bp 处结束,共计编码379 个氨基酸。克隆子1的第1外显子编码84个氨基酸,克隆子2的第1外显子编码46个氨基酸。2种转录剪接体的第2-12 外显子完全相同,均编码333 个氨基酸;第12 外显子中的3´非翻译区(untranslated region, UTR)长度为246 bp。
2.3 序列同源性分析
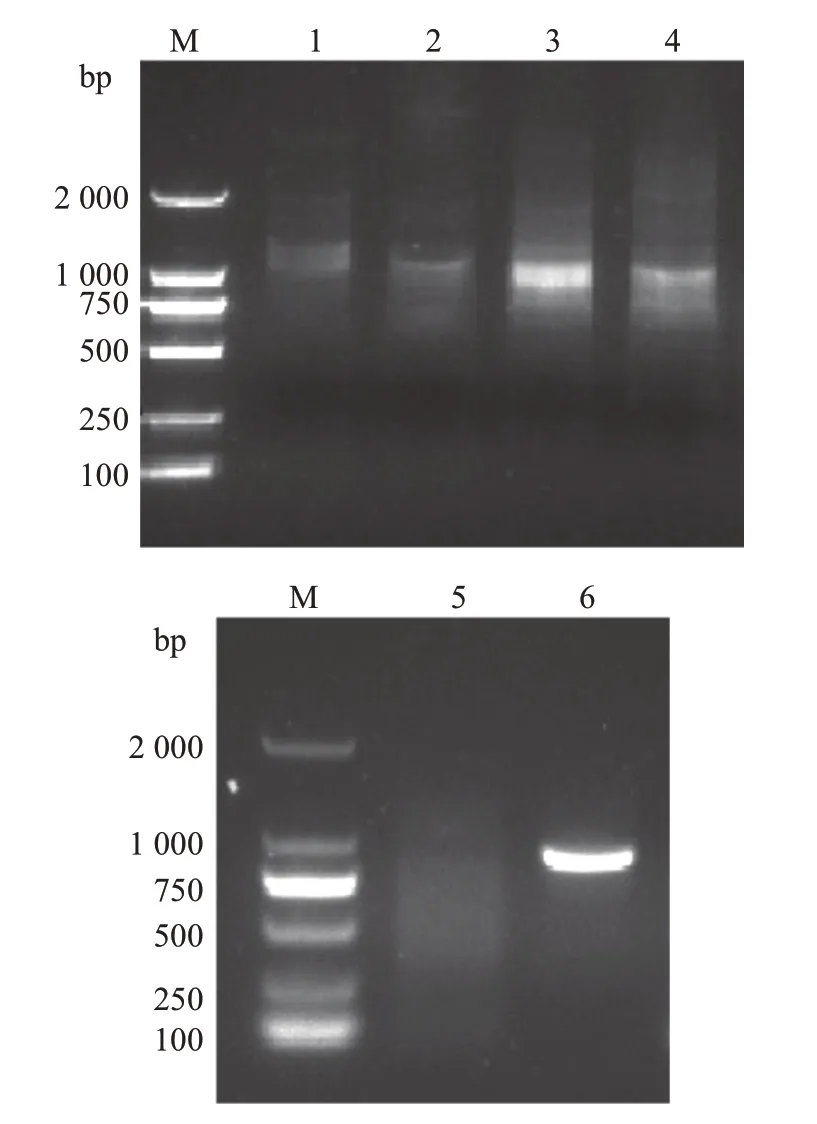
图1 鸡GNAS基因的5´和3´RACE扩增结果Fig.1 PCR products of 5´and 3´RACE of chicken GNAS gene
克隆得到的鸡GNAS 基因的2 种转录剪接体与人、小鼠、鸡等物种的蛋白同源性比对结果见图3和表4。从中可见,克隆子2编码的379个氨基酸(aa)序列与已知的人和小鼠的GNAS基因Gαs亚基同源相似度达93%,明显高于与XLαs 亚基的同源性(相似度84%~87%)。克隆子1编码的417 aa序列与已知的人和小鼠的GNAS基因XLαs亚基同源性较高,相似度最高达87%(330/379 aa),与其他2 条XLαs亚基的相似度为84%,这主要是因为这2条XLαs亚基中间多了一段15 aa序列,导致相似度降低;此外,虽然417 aa 序列与2 条Gαs 亚基的相似度也达到87%,但二者相似氨基酸的数目要比XLαs 亚基少7~8 aa(323/370 aa,322/370 aa)。经与NCBI 数据库中4 种预测的鸡GNAS 基因蛋白亚基比较发现,417 aa 序列与预测的X1亚基、379 aa 序列与预测的X2亚基的同源相似度均达到100%,但这2种预测蛋白亚基对应的mRNA 序列长度却在5 000 bp 以上,远远大于克隆得到的剪接体长度。
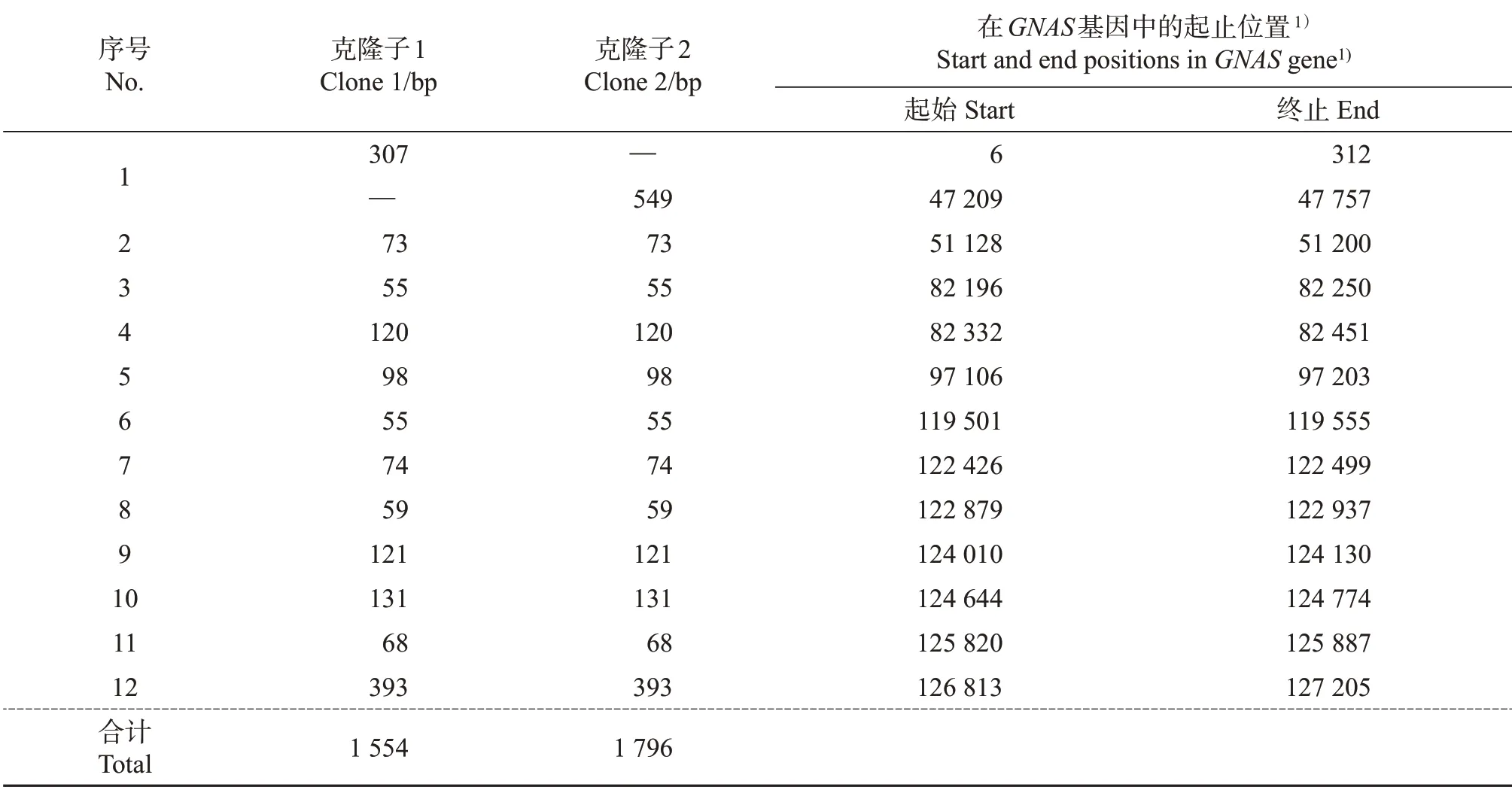
表3 鸡GNAS基因经RACE得到的克隆子信息Table 3 Information of the clones obtained from RACE of chicken GNAS gene
2.4 组织表达量分析
心、脑、皮肤等不同组织中鸡GNAS基因的2种转录剪接体的表达量检测结果见图4。从中可见,鸡GNAS 基因克隆子1 和克隆子2 在7 种组织中均有不同程度的表达。其中:在脑组织中的表达量最高,与其他6 种组织的差异均极显著(P<0.01);在皮肤组织中的表达量次之,基本极显著高于在其他5种组织中的表达量(P<0.01,除克隆子2在皮肤与肺组织中的表达量在P<0.05水平上差异有统计学意义外);在肺、胸肌、肝、心、腹脂等组织中的表达量较低,且组间差异无统计学意义(P>0.01);相对而言,克隆子1在腹脂组织中的表达量最低,克隆子2在肝组织中的表达量最低。
3 讨论与结论
本研究找到了鸡GNAS 基因的2 种转录剪接状态,而据NCBI 数据库资料,人和小鼠的GNAS 基因分别存在多达26种和16种已知的或预测的转录剪接 形 式,可 翻 译 形 成 不 同 的Gαs、Alex、XLαs、Nesp55等蛋白亚基。GNAS基因存在许多转录剪接体,这应该与mRNA 前体的选择性剪接有关,即基因在转录过程中,其mRNA前体通过不同的剪接方式(选择不同的剪接位点)产生不同的mRNA 剪接异构体。学界普遍认为,mRNA 前体的可变剪接,是调节基因表达和产生蛋白质组多样性的重要机制,也是生物体基因和蛋白质数量差异较大的主要原因[10-11]。对于作用于神经系统和免疫系统的基因,其可变剪接现象更为常见,这与2大系统的功能多样性和反应敏感性密切相关[12-13]。GNAS 基因参与了众多神经递质、免疫因子等相关的生命活动,是关键的信号通路元件,其转录剪接形式多样也在情理之中。因此,鸡GNAS基因除了本研究找到的2种转录剪接体,很可能还存在更多的剪接体。

图2 鸡GNAS基因2种转录剪接体的全长序列Fig.2 Full-length sequences of two transcriptional spliceosomes of chicken GNAS gene
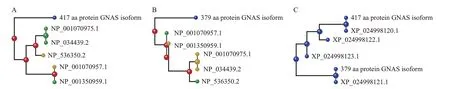
图3 鸡GNAS基因2种转录剪接体的蛋白同源性比对结果Fig.3 Protein homology alignment results of two transcriptional spliceosomes of chicken GNAS gene
通过考察人和小鼠的GNAS 基因剪接体发现,Gαs 和XLαs 在第2 外显子之后的mRNA 组成序列比较一致,二者区别主要在第1 外显子:人XLαs 的第1和第2外显子相距约40 kb(小鼠约为33 kb),而Gαs 的第1 和第2 外显子相距约6.1 kb(小鼠约为3.7 kb);且XLαs的第1外显子编码氨基酸数目大于Gαs。本研究得到的鸡GNAS基因克隆子1的第1和第2 外显子相距约50 kb,而克隆子2 的相应间距只有约3.3 kb;同时,蛋白同源性比对结果也发现,鸡GNAS 基因克隆子1 与人和小鼠XLαs 的同源性较高,而与Gαs的同源性稍低。因此,我们有理由认为找到的克隆子1 就是鸡GNAS 基因的XLαs 剪接形式。

表4 同源比对涉及的蛋白序列及相关信息Table 4 Protein identification(ID)and related information involved in homologous alignment

图4 鸡GNAS基因2种转录剪接体的组织表达量Fig.4 Tissue expression level of two transcriptional spliceosomes of chicken GNAS gene
当然,与人和小鼠XLαs 相比,鸡XLαs 克隆子的5´UTR 区长度较短,编码氨基酸数较少。同时,与鸡GNAS 基因的4 种预测剪接体相比,本研究找到的2 种转录剪接体的3´UTR 区长度要短约3 000 bp。5´UTR 和3´UTR 区虽然不是翻译蛋白的组成序列,但它们包含的特殊结构和一些顺式作用元件,如5´UTR 区uORFs 和uAUGs、3´UTR 区APA等,会参与翻译调节,影响转录后的各个方面,包括mRNA 的转运、稳定性及翻译效率等[14-15]。本研究得到的鸡GNAS 基因5´UTR 和3´UTR 区长度虽短,但可能与研究涉及的种属特异性有关,这在mRNA的表达检测结果中也有所反映。
从本研究测定的组织表达情况来看,鸡GNAS基因的2 种转录剪接体在脑、皮肤组织中的表达量与其他组织相比均要高得多,其中XLαs 剪接体在脑组织中的表达差异更为显著。前面提到,GNAS基因参与神经递质活动,它在中枢神经系统(脑)中的表达量理应较为丰富,因此鸡与人、小鼠脑中的GNAS 基因表达量检测结果[16-18]是相符的。但与人和小鼠相比,鸡GNAS 基因在皮肤中的表达水平也较高,推测这与物种差异有关。有研究发现,鸡皮肤具有无疤痕愈合现象[19],表明其抗炎再生性强、免疫因子活跃,这可能是鸡皮肤中GNAS 基因表达量高的原因之一。此外,我们发现,鸡GNAS 基因的2种转录剪接体在不同组织间的表达水平存在差异,表明二者有不同的表达方式或功能。据报道,基因的可变剪接一般具有时空(发育阶段、组织、疾病状态)表达的特异性[20-21],本研究只涉及90日龄鸡的7种组织,所以今后还需要不断完善鸡GNAS 基因的表达谱。

