人RPS6KA3基因及蛋白质的生物信息学分析
唐 乖, 杨 越, 朱家佳, 龙鼎新
(南华大学 公共卫生学院, 衡阳 421001)
核糖体蛋白S6激酶A3基因(ribosomal protein S6 kinase A3,RPS6KA3)又称为RSK2、p90RSK2、S6K-α-3或MAPKAPK-1B,位于X染色体p22.12,编码丝氨酸/苏氨酸激酶RSK2[1-2]。核糖体S6激酶2(ribosomal S6 kinase2, RSK2)属于Ras-MAPK信号通路下游的90 ku 核糖体S6激酶家族(ribosomal S6 kinase,RSK)[3-4]。RSKs家族包含 4 个亚型(RSK1-4)及 2 个结构同系物(MSK1/2)[5],该家族成员具有高度的序列同源性[6](约75% ~ 80%的氨基酸序列是相同的),只有两个功能不同的激酶结构域[7-8]。RPS6KA3通过磷酸化细胞死亡相关蛋白激酶(Death associated protein kinase,DAPK)[9]、活化T细胞核因子3(Nuclear factor of activated T-cells,NFAT3)[10]、热休克蛋白27(Heat shock protein,Hsp27)[11]等一系列细胞因子[3, 12],在细胞的增殖、迁移、侵袭、凋亡、自噬及细胞周期的调控等多项细胞生命活动中起关键作用[8]。研究发现RPS6KA3在细胞增殖、转化等过程中发挥重要作用,并与癌症等多种疾病的发生发展过程密切相关[13]。已证实,RPS6KA3基因的功能缺失突变会引起科-勒二氏综合征(Coffin-Lowry syndrome,CLS)——一种罕见的神经退行性疾病[12, 14-15]。
为了深入研究人RPS6KA3基因的性质和功能,本研究拟采用生物信息学方法,对人RPS6KA3基因及其编码蛋白的结构特点以及与其潜在相互作用的基因和蛋白进行预测分析,揭示其理化性质及功能信息,有利于了解其在疾病发生发展中的生物学作用,可为该基因的生物学功能及作用机制的深入研究奠定了实验基础。
1 材料与方法
1.1 数据的来源
从在线数据库NCBI中(http://www.ncbi.nlm.nih.gov/protein/)获取人RPS6KA3基因(Gene ID: 6197)相关序列和RPS6KA3蛋白质的氨基酸序列信息(NP_004577)。
1.2 方法
利用Neural Network Promoter Prediction(http://www.fruitfly.org/seq_tools/promoter.html)在线软件预测潜在核心启动子;利用Ensembl[16]数据库(http://asia.ensembl.org/Homo_sapiens/Gene/)获得RPS6KA3的染色体图谱。在NCBI数据库中,使用BLAST(https://blast.ncbi.nlm.nih. gov/Blast.cgi)进行同源搜索,获得其它物种的RPS6KA3序列,采用最大似然法(maximum likelihood, ML)对不同物种RPS6KA3蛋白序列进行分析,随后利用MEGA6.06软件构建系统进化树。应用NCBI的UniGene数据库中EST(Expressed Sequence Tag)结果对RPS6KA3在正常组织及癌变组织中的表达情况进行预测分析。应用PSORT II Prediction[17](https://psort.hgc.jp/form2.html)在线数据库对RPS6KA3进行亚细胞定位。采用在线数据库GeneMANIA[18-19](http://genemania.org)构建RPS6KA3与其他基因之间的相互作用网络图。将人RPS6KA3蛋白质序列(NP_004577)提交到Expert Protein Analysis System(ExPASy)[20]数据分析系统中,使用在线分析软件ProParam[21](http://web.expasy.org/protparam/),对RPS6KA3蛋白质的分子式、分子质量、理论等电点、酸碱性及稳定性等物理化学特性进行预测分析。使用ProtScale(http://web.expasy.org/protscale/)在线预测软件分析RPS6KA3蛋白质的亲水性/疏水性;利用在线软件SignalP4.1 Server[22](http://www.cbs.dtu.dk/services/SignalP/)、核定位信号(nuclear localizationsequence, NLS)Mapper[23](http://nls-mapper.iab.keio.ac.jp/cgi-bin/NLS_Mapper_form.cgi)和TMHMM Server version 2.0[24](http://www.cbs.dtu.dk/services/TMHMM/)分析RPS6KA3有无切割位点和信号肽、核定位信号及跨膜区域。用在线分析软件NetPhos 3.1 Server[25](http://www.cbs.dtu.dk/services/NetPhos/ )对人RPS6KA3蛋白磷酸化位点进行分析。应用NCBI的Conserved domain数据库(https://www.ncbi.nlm.nih.gov/Struct-ure/cdd/wrpsb.cgi)分析结构域。使用SOPMA[26](http://npsa-pbil.ibcp.fr/)、Swiss-Model[27](http://swissmodel.expasy.org)和The Structure Analysis and Verification Server[28](http://services.mbi.ucla.edu/SAVES)对蛋白质的二级结构、三级结构及拉曼图进行预测分析;采用String[29](http://string-db.org/)对相互作用蛋白质进行预测分析。各数据库软件参数如无特别说明均取默认值。
2 结果与分析
2.1 人RPS6KA3基因启动子预测
利用Neural Network Promoter Prediction对人RPS6KA3基因潜在核心启动子的位置进行在线预测(表1),结果发现其在 2820~2870 bp 位置处的得分值最大(0.99),推测此处为潜在核心启动子位置的可能性最大。
2.2 人RPS6KA3基因结构分析
人RPS6KA3基因位于X染色体上(Xp22.12),见图1,大小为7723 bp,共有28个外显子区域及27个内含子,编码740个氨基酸的蛋白质[12]。其编码蛋白核糖体S6激酶2(ribosomal S 6 Kinase 2,RSK2)是一种生长因子调节的丝氨酸-苏氨酸蛋白激酶,在Ras-MAPK信号通路中发挥重要作用[30-31]。
利用NCBI数据库中的BLAST对RPS6KA3蛋白序列进行BLASTp 搜索,获得其他物种的RPS6KA3氨基酸序列,选取Identity较高的序列,随后运用MEGA6.06软件构建系统进化树(图2)。利用NCBI 中的BLASTp对人RPS6KA3蛋白序列进行分析发现,食蟹猴 (Macaca fascicularis)的蛋白序列与人RPS6KA3的蛋白序列的相似性最好;从系统进化树中可以看到,物种被分为两个大分支,其中人、食蟹猴及白颊长臂猿聚在一个分支内,同源性最高。

表1 人RPS6KA3基因启动子预测结果

图1人RPS6KA3基因位于染色体Xp22.12上
Figure 1 Human RPS6KA3 gene was located on chromosome Xp22.12

图2 人RPS6KA3基因的系统进化树Figure 2 The phylogenefic tree of human RPS6KA3 gene
2.3 人RPS6KA3的组织表达特异性和亚细胞定位预测
根据NCBI的UniGene数据库中的EST预测结果,可知RPS6KA3在如下正常组织中均有表达,拷贝数分别为:肌肉112、甲状腺128、肝脏82、心脏22、肺47、乳腺59、胃62、食管49、膀胱33,其在甲状腺中的拷贝数最高。由此推测,RPS6KA3在多种组织中均有表达,特异性较低。使用PSORT II 亚细胞定位分析发现,RPS6KA3蛋白定位于细胞质的可能性最大(65.2%),其次分别为细胞核(30.4%)和液泡(4.3%)。据此推测,RPS6KA3主要存在于细胞质中,也可能动态存在其他亚细胞结构中并发挥作用。
2.4 基因相互作用分析
应用GeneMANIA在线搜索与RPS6KA3基因存在相互作用的基因信息,构建基因间相互作用网络图。基因互作预测分析显示,与RPS6KA3相互作用的基因主要有MAPK1、RPS6KA1、MAPK7、HMGN1、SRF等(图3)。
2.5 人RPS6KA3蛋白特性分析
使用ProtParam分析发现:RPS6KA3蛋白含有740个氨基酸残基,总分子式为 C3763H5900N1012O1097S27,相对分子量为 83 735.98 u,总原子数为11 799,理论等电点(pI, isoelectric point)预测为6.41,表明该蛋白为酸性蛋白质。在740个氨基酸残基中带正电荷的氨基酸残基(Arg + Lys)和带负电荷的氨基酸残基(Asp + Glu)的总数分别是90和97,RPS6KA3基因CDS区氨基酸组成如图4所示。消光系数(M-1cm-1γ=280 nm)为80 510,其不稳定系数为42.80(>40考虑为不稳定),属于不稳定类蛋白质,且其在哺乳动物网织红细胞内的半衰期为30 h。脂肪系数为 85.65,总平均亲水系数(Grand average of hydropathicity, GRAVY)为 -0.338,表明该蛋白是亲水性蛋白。
图4 人RPS6KA3基因CDS序列氨基酸
进一步应用ProtScale在线亲/疏水性软件,按其默认的算法预测,获得 RPS6KA3 蛋白的亲/疏水性序列分析图谱(图5)。正值越大表明该氨基酸的疏水性越强,负值越大表明该氨基酸的亲水性越强,数值介于-0.5~0.5之间的主要为两性氨基酸。由图5可知,该蛋白质存在两个亲水性最强的位点,分别是位于第458位的精氨酸(Arg)和第459位的天冬氨酸(Asp),分值为-2.878;疏水性最强的位点是位于第608位的亮氨酸(Leu),分值为2.100。ProtScale分析的732个氨基酸(5~736)中有63.80%(467个)分布在低分值区,总得分为-418.046;35.38%(259个)分布在score>0区,总得分为160.194。这表明RPS6KA3存在大量亲水域,属于亲水性蛋白质。预测结果与理化性质中的亲/疏水性结果一致。
图5 人RPS6KA3亲水性/疏水性分析
利用信号肽预测软件SignalP 4.1 Server在线分析RPS6KA3蛋白质的氨基酸序列(图6),Cut-off值设为0.450。其中C为剪切位点打分,最大值为0.110;Y为信号肽打分,最大值为0.110;S为综合剪切点打分,最大值为0.119;S-mean和D-mean值分别为0.105、0.108,由以上数据可知,人RPS6KA3蛋白不存在切割位点及信号肽序列,提示该蛋白不是分泌蛋白。进一步应用cNLS Mapper分析其核定位序列发现,在RPS6KA3蛋白质中存在一段序列为“FGKVFLVKKISGSDARQLYAMK VLKKATLK(79-108)”的NLS,其得分值为5.1分,高于设定的阈值(5分)。
图6 人RPS6KA3蛋白的信号肽分析
使用TMHMM Server. 2.0对人RPS6KA3蛋白质的跨膜区进行预测与分析,结果发现RPS6KA3不存在跨膜区域(图7),属于非跨膜蛋白。其中RPS6KA3蛋白定位于膜外(outside)的概率几乎为100%,定位于膜内(inside)及跨膜区域(transmembrane)的概率几乎为0。多肽链中跨膜区域所在的位置用加粗的紫色线来标识,由于RPS6KA3蛋白无跨膜区域存在,所以在紫色粗线上并未显示相应标记。
图7 人RPS6KA3蛋白跨膜结构分析
用NetPhos 3.1 Server对RPS6KA3翻译后修饰情况进行在线分析,发现RPS6KA3蛋白有多个潜在的磷酸化调节位点存在(图8),这些磷酸化调节位点主要位于丝氨酸(Ser)、酪氨酸(Tyr)上,表明RPS6KA3通过被丝氨酸、酪氨酸及苏氨酸激酶磷酸化来实现其功能调控作用。

图8 人RPS6KA3蛋白磷酸化位点
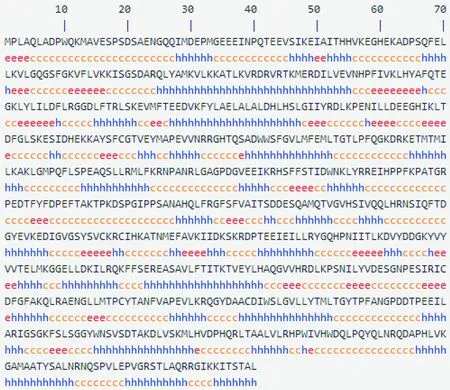
h:α-螺旋;e:延伸链; t:β-转角;c:无规卷曲
图9人RPS6KA3二级结构预测
Figure 9 Secondary structure prediction of human RPS6KA3
通过SOPMA对人RPS6KA3蛋白质所形成的二级结构进行预测分析(图9),构象状态选择3(Helix,Sheet,Coil),相似性阈值选择8。在RPS6KA3蛋白质的二级结构中,α-螺旋(h所示区域)占41.62%(308/740),延伸链(e所示区域)占13.38%(99/740),β-转角(t所示区域)占8.11%(60/740),无规则卷曲(c所示区域)占36.89%(273/740)。由此可知,RPS6KA3蛋白二级结构最大量元件是α-螺旋,无规则卷曲、延伸链、β-转角则散在分布于整个蛋白质结构中。
应用NCBI的Conserved domain数据库预测分析RPA6KA3蛋白,发现其属于PKc_like超家族,含有2个功能不同的催化结构域(STKc_RSK_N和STKc_RSK_C),它们可将ATP上的γ-磷酸基团催化转移到蛋白质底物上的丝氨酸/苏氨酸残基上,从而使蛋白底物磷酸化(图10)。

图10人RPS6KA3蛋白保守结构域
Figure 10 Conserved domain of human RPS6KA3
采用结构预测软件SWISS-MODEL预测RPS6KA3蛋白质的三级结构。首先将RPS6KA3的氨基酸序列提交至SWISS-MODEL,得到4个三级结构的预测信息(表2);然后再利用The Structure Analysis and Verification Server 对预测获得的模型进行拉曼图分析,进一步验证获得的预测模型的可靠性(图11)。表2展示了4种预测结果所选用的模板(Model)、序列相似度(Sequence Similarity)、预测范围(Range)、覆盖度(Coverage)及拉曼图分析中处于不合理区域氨基酸的比例(Residues in disallowed regions)等。图9展示了预测获得的4种蛋白质模型的三级结构、同源蛋白质相似性波形图及拉曼图。在拉曼图中区域由白到红,颜色越深则表明该区域二面角越合理。
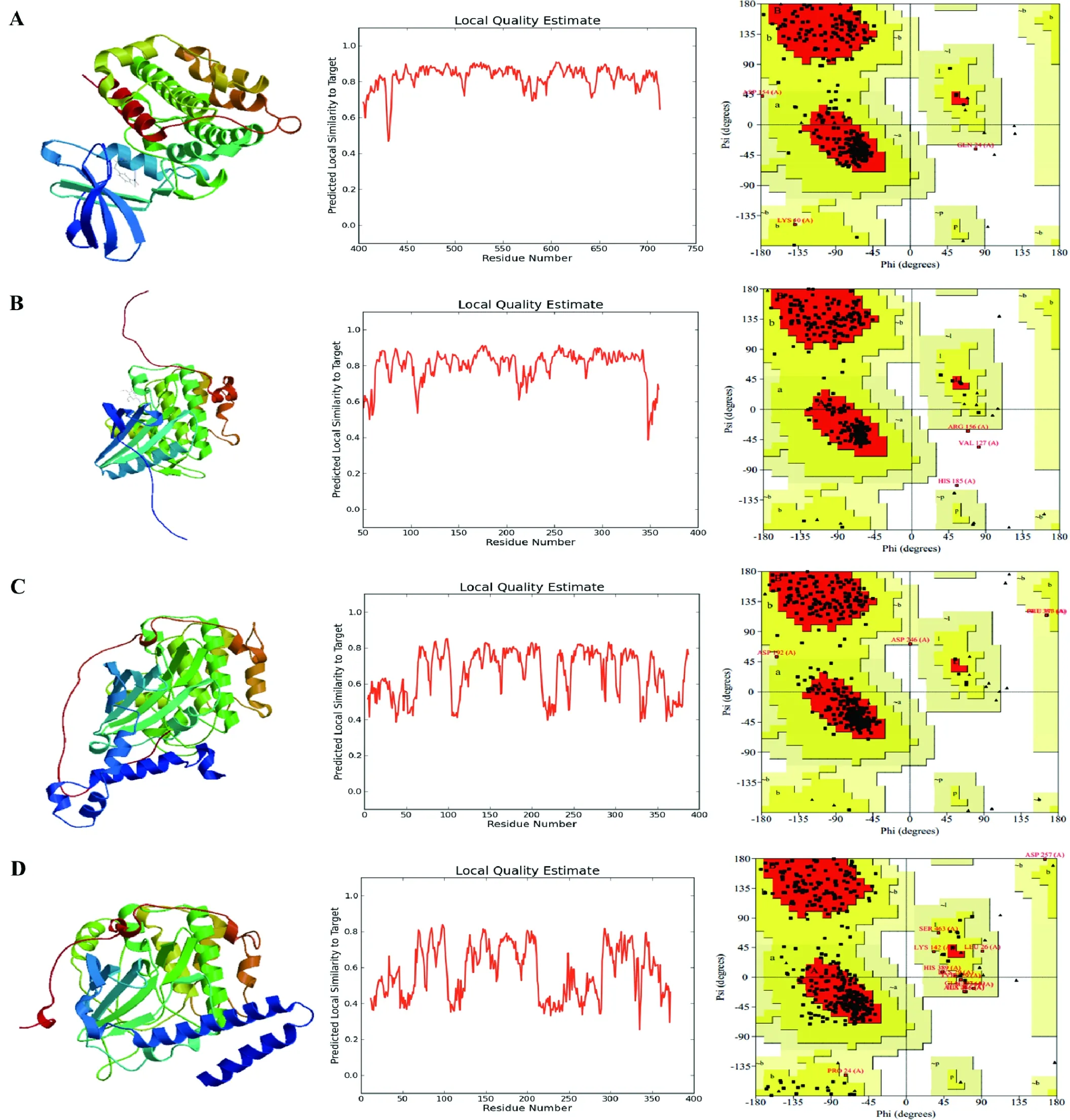
▲:甘氨酸;■:除甘氨酸外的其他氨基酸
图11人RPS6KA3三级结构预测结果及拉曼图分析
Figure 11 Tertiary structure prediction and Ramachandran
plot analysis of RPS6KA3 in human
2.6 人RPS6KA3相互作用蛋白质的预测分析
应用蛋白质-蛋白质相互作用(protein-protein, PPI)在线分析数据库STRING,对人RPS6KA3蛋白质间的相互作用进行预测分析,设置为高置信度0.7,构建RPS6KA3蛋白相互作用网络(图12)。得到10个与RPS6KA3相互作用紧密的蛋白信息及预测得分值(表3),这些蛋白质彼此之间存在紧密的相互作用关系。
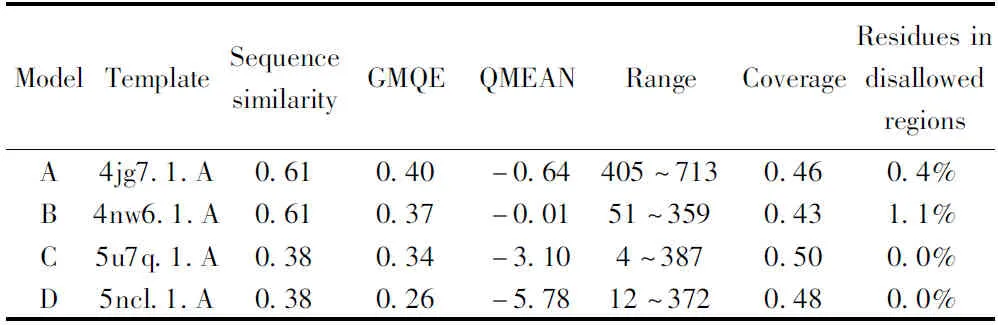
表2 RPS6KA3预测三级结构信息
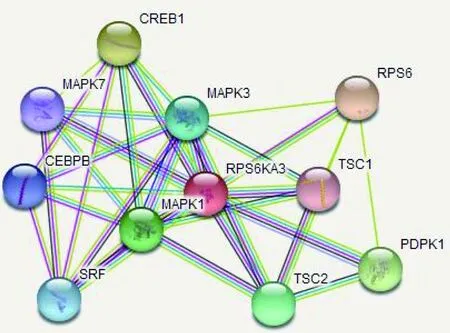
图12 与人RPS6KA3蛋白质相互作用的蛋白质预测
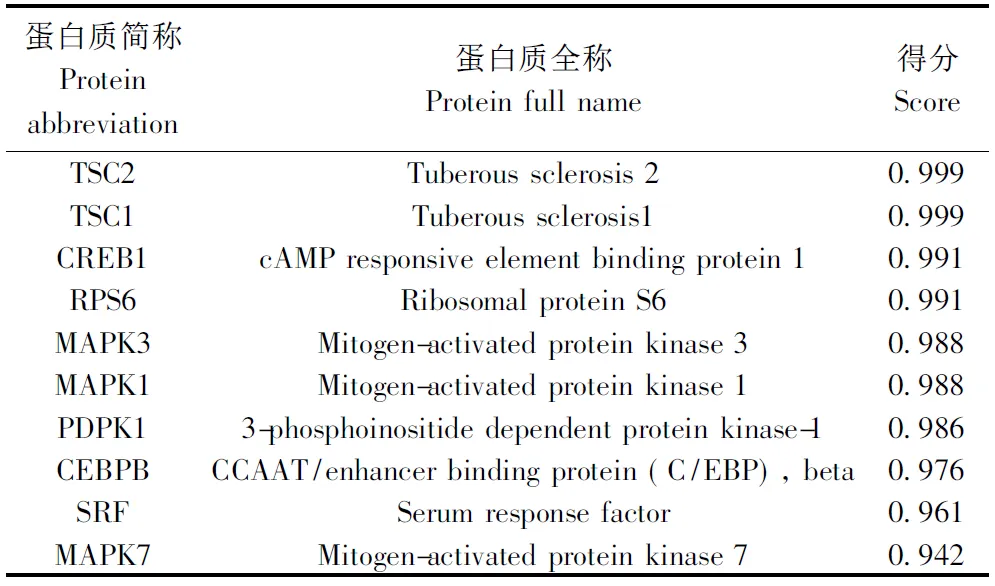
表3 与人RPS6KA3相互作用可能性较大的10种蛋白质
3 讨论与结论
生物信息学(Bioinformatics)是一门由生物学、数学、信息科学以及计算机科学等多学科相结合,以计算机为工具对生物信息进行加工、存储、分配、分析解释等,并综合运用计算机科学、数学和生物学等学科的各种工具来理解和阐明大量数据所包含的生物学意义的一门交叉性学科[32-33]。
本研究对人RPS6KA3基因及其蛋白进行生物学分析,结果显示其基因存在15个潜在启动子,其中在 2820~2870 bp 位置处得分值最大0.99,表明其为潜在核心启动子位置的可能性最大。人RPS6KA3基因定位于X染色体长臂上,共编码740个氨基酸组成的多肽;通过对不同物种间的序列进行同源比对分析可知,不同物种间的序列相似度较高,提示其在进化过程中相对保守,说明其由于具有重要的功能作用而在进化过程中得以保留。基因互作预测分析表明,与RPS6KA3相互作用的基因主要有MAPK1、RPS6KA1、MAPK7、HMGN1和SRF等;人RPS6KA3基因存在多个磷酸化位点,其编码的蛋白是一个不稳定的水溶性蛋白,其分子量为83 735.98 u,等电点为6.41,蛋白质结构主要由α-螺旋和无规则卷曲组成,不含切割位点,无明显信号肽及跨膜结构,说明RPS6KA3不参与物质的跨膜运输。蛋白质相互作用预测表明,与RPS6KA3相互作用的蛋白主要是MAPK1、MAPK3、MAPK7等MAPK信号通路相关蛋白,TSC1、TSC2等mTOR信号通路相关蛋白及蛋白合成相关蛋白等。
本研究针对人RPS6KA3基因及其编码蛋白的生物学特性进行系统分析,构建了RPS6KA3的系统进化树,分析了RPS6KA3蛋白的理化性质、空间结构特点、组织学特异性、亚细胞定位以及与其存在相互作用蛋白等,为全面认识RPS6KA3和深入研究其生物学功能提供了一定的基础。

